文章信息
- 乌云塔娜, 李洪果, 李振国, 包梅荣, 谭晓风
- Wuyun Tana, Li Hongguo, Li Zhenguo, Bao Meirong, Tan Xiaofeng
- 梨4个SFBB-γ基因的分离及遗传多态性分析
- Isolation and Genetic Polymorphism Analysis of Pear SFBB-γ Genes
- 林业科学, 2010, 46(1): 147-151.
- Scientia Silvae Sinicae, 2010, 46(1): 147-151.
-
文章历史
- 收稿日期:2008-10-06
-
作者相关文章
2. 中南林业科技大学林学院 长沙 410004;
3. 内蒙古克什克腾旗林业局黄冈梁林场 克什克腾旗 025350
2. College of Foresty, Central South University of Forestry and Technology Changsha 410004;
3. Huanggangliang Forest Farm of Keshiketeng Banner Forestry Bureau, Inner Mongolia Keshiketeng 025350
茄科(Solanaceae)、芸苔属(Brassica)、蔷薇科(Rosaceae)植物多数属于配子体自交不亲和型,即雌蕊和花粉自交不亲和基因的产物相互作用而控制自交亲和性的发生。20世纪90年代分离鉴定了雌蕊自交不亲和基因及编码产物(Tao et al., 1997; Ushijima et al., 1998),而近几年才分离鉴定花粉自交不亲和基因,配子体自交不亲和植物的花粉自交不亲和基因具有F-box基因的特点。如茄科植物中SLF (S locus F-box gene)基因控制花粉自交不亲和特异性状(McClure et al., 2006); 蔷薇科核果类自交不亲和基因和自交亲和基因的研究中提出了核果类花粉自交不亲和基因为SFB(S haplotypespecific F-box gene)基因(Sonneveld et al., 2005; Sutherland et al., 2008); 仁果类果树苹果(Malus × domestica)和日本沙梨(Pyrus pyrifolia)中也已分离鉴定了控制花粉的自交不亲和基因——S locus F-box brothers(SFBB)(Sassa et al., 2007;Hiroyuki et al., 2007;Surbanovski et al., 2007)。日本沙梨SFBB基因的克隆过程中发现,在S位点上F-box基因存在很高的遗传多态性,在1个S位点上存在3个基因a, b, γ,且连锁在S-RNase的两侧(Sassa et al., 2007;Hiroyuki et al., 2007)。但目前尚未清楚哪些是假基因,哪些真正决定梨自交不亲和性的发生。这为梨SFBB基因的功能研究和梨自交不亲和机制研究带来了很大的困难。我国梨资源丰富,但中国梨属(Pyrus)植物的花粉自交不亲和基因分离鉴定少见报道。本文通过特异性PCR扩增、目的片段的克隆测序及生物信息学分析方法分离克隆中国沙梨(P. pyrifolia)和新疆梨(P. sinkiangensis)的花粉自交不亲和基因,并进行遗传多态性分析,为梨花粉SFBB基因的功能研究、中国梨自交不亲和机制研究以及遗传改良提供基础。
1 材料与方法 1.1 试验材料1) 植物材料 中国沙梨品种‘黄冠’、‘壁山二号’、‘新雅’、‘雅青’、‘早美酥’、‘中梨一号’和新疆梨品种‘早熟句句’、‘魁克句句’采集于中国农业科学院郑州果树研究所。每个品种采集叶片2 g左右,保存于-80 ℃备用。
2) PCR引物 利用梨SFBB基因的保守区DNA序列,设计合成较特异的SFBB基因扩增引物(引物的位置见图 1),引物由上海生工生物技术有限公司合成。P1:5′-GTGTGTAATTCATGTGCAT GG-3′; P2: 5′-TGGAACGTTTCCCTCAACTC-3′。

|
图 1 梨的SFBB基因一级结构及引物位置 Figure 1 The structure and primer position of pear SFBB gene V1-V4:可变区Variable region; P1, P2:引物Primer. |
3) 药品试剂 总DNA提取试剂盒购自北京三博远志生物技术有限责任公司;β-巯基乙醇、Tris购自上海生工生物技术公司;100 bp DNA Ladder,pMD18-T载体购自大连宝生物工程公司;PCR Master Mix Kit和Gene Clean II Kit购自北京天根生物技术有限责任公司。DHα大肠杆菌(Escherichia coli)由中南林业科技大学国家林业局经济林育种与栽培重点实验室保存。其他生化试剂和常规试剂均为超纯或分析纯。
1.2 试验方法1) 梨叶片DNA的提取、纯度浓度测定 叶片DNA的提取参照北京三博远志生物技术有限责任公司总DNA提取试剂盒的说明书进行;纯度浓度测定采用DU~640核酸蛋白分析仪。
2) PCR扩增反应体系和循环条件 参考PCR Master Mix Kit的说明书,稍做改动(反应体系中Mix为说明书推荐的1/2)。
3) 回收克隆 目的片段的回收和克隆分别依Gene CleanⅡKit和pMD18-T载体的说明书进行。
4) DNA序列测定 由于PCR产物中存在2个等位基因的扩增片段,故回收克隆后随机挑选10个单菌落进行DNA双向测序,委托上海生工生物技术有限责任公司完成。
5) DNA序列分析 用DNAStar软件将双向测序DNA序列拼接后, 与GenBank的梨SFBB基因序列进行比较分析,用Clustal X软件对相应的氨基酸序列进行分析, 用Mega2软件的UPGMA法构建系统进化树。
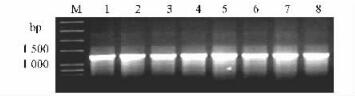
2 结果与分析 2.1 FBB-γ基因特异性引物PCR分离用SFBB-γ基因的特异性引物对‘黄冠’等8个梨品种的基因组DNA进行PCR扩增,均扩增出1条1 250 bp左右的条带(图 2)。
|
图 2 SFBB-γ基因的PCR扩增 Figure 2 PCR of the SFBB-γ 1-6.沙梨P. pyrifolia. 1.‘黄冠’‘Huangguan’;2.‘壁山二号’‘Bishanerhao’;3.‘雅青’‘Yaqing’;4.‘新雅’‘Xinya’;5.‘早美酥’‘Zaomeisu’;6.‘中梨一号’‘Zhongliyihao’.7, 8.新疆梨P. sinkiangensis. 7.‘早熟句句’‘Zaoshujuju’;8.‘魁克句句’‘Kuikejuju’. |
‘黄冠’和‘壁山二号’的S基因型均为S4S16(乌云塔娜等,2007)。对‘黄冠’和‘壁山二号’的SFBB基因进行克隆后分别挑选了10个单克隆进行测序。结果表明,‘黄冠’的1个SFBB基因序列和‘壁山二号’的1个SFBB基因序列相似性达100%,且与SFBB4-γ基因(AB270799)在DNA序列上具有12 bp的差异,在推导氨基酸序列上具有3个氨基酸的差异。GenBank中的其他SFBB-γ基因比对结果表明,该基因与SFBB12a-γ基因(EU081890)的相似性最高,但在DNA序列上具有7个碱基的差异,推导氨基酸序列上具有4个氨基酸的差异。因此,将该基因命名为SFBB16-γ基因(EU422956)。
‘新雅’和‘雅青’的S基因型均为S4S17(乌云塔娜等,2007)。克隆片段的测序结果表明,‘新雅’的5个单克隆和‘雅青’的3个单克隆的DNA序列完全一致,属于同一个基因片段,而且这些基因的DNA片段与SFBB4-γ基因(AB270799)有12个碱基和3个氨基酸的差异。进一步比对分析表明,该基因与GenBank中的SFBB12a -γ基因(EU081890)的相似性最高,但在DNA序列上具有8个碱基的差异,推导氨基酸序列上具有2个氨基酸的差异。因此,将该基因命名为SFBB17-γ基因(EU422957)。
新疆梨品种‘早熟句句’和‘魁克句句’的S基因型均为S22S28(待发表)。分别挑选10个单克隆进行测序, 结果表明,‘早熟句句’的1个单克隆和‘魁克句句’的2个单克隆的DNA序列完全一致,属于同一个基因片段,而且该基因的DNA序列与SFBB22-γ基因(EU081887)在DNA序列和推导氨基酸序列上分别有30 bp和11个氨基酸的差异。与其他SFBB基因的比对分析表明,该基因与GenBank中的SFBB13-γ基因(EU081895)的相似性最高,但在DNA序列上具有17碱基的差异,推导氨基酸序列上具有8个氨基酸的差异。故将该基因命名为SFBB28-γ基因(EU422960)。
‘早美酥’和‘中梨一号’的S基因型分别为S3S35和S4S35(乌云塔娜,2007)。‘早美酥’的10个单克隆的测序结果表明,其中4个克隆的DNA序列与SFBB3-γ基因有差异,在DNA和推导氨基酸上分别相差17 bp和9个氨基酸;‘中梨一号’的10个克隆的测序结果表明,其中6个克隆与SFBB4-γ基因有差异,在DNA和氨基酸序列上分别有25 bp和8个氨基酸的差异;而‘早美酥’和‘中梨一号’中与SFBB3-γ基因和SFBB4-γ基因有差异的片段的序列相似性达100%,属于同一个基因。进一步分析表明,该基因与GenBank中的SFBB13a-γ基因(EU081895)的相似性最高,但在DNA序列上具有6个碱基的差异,推导氨基酸序列上具有2个氨基酸的差异。因此,将它命名为SFBB35-γ基因(EU422958)。
2.3 梨SFBB-γ基因的推导氨基酸序列分析梨SFBB-γ基因推导氨基酸序列比对见图 3。SFBB-γ基因由1个F-Box区域、4个可变区(V1, V2, V3, V4)组成(Sassa et al., 2007)。F-Box区域在+1~+150 bp间,氨基酸序列是从“MSQV/MR/CE”到“FVAKHL”。在这个区域中,不同SFBB基因的变异较多,具有7个氨基酸序列产生突变。其中新分离的SFBB16-γ基因和SFBB17-γ基因中‘S’(第7个)被‘G’替换;第10个氨基酸, SFBB16-γ基因为‘P’而SFBB17-γ基因为‘L’;SFBB28-γ基因中‘V’(第4个)被‘M’替换、‘M’(第14个)被‘K’替换;SFBB35-γ基因中‘S’(第7个)被‘I’替换。

|
图 3 梨的SFBB基因的氨基酸序列的比对 Figure 3 Alignment of putative amimo acids of Pyrus SFBB gene 直线Beeline:F-Box; 双线Crevel:V1.沙梨P. pyrifolia SFBB1: AB297933;SFBB2:AB297934;SFBB3:AB297935;SFBB4:AB270799;SFBB5:AB270802;SFBB6:AB297936;SFBB7:AB297937;SFBB8:AB297938;SFBB9:AB297939.白梨P. bretschneideri SFBB12a:EU081890;SFBB13:EU081895;SFBB15:EU081893;SFBB16:EU422956;SFBB17:EU422957;SFBB18:EU081896;SFBB35: EU422958.新疆梨P. sinkiangensis SFBB22: EU081887;SFBB28: EU422958.下同。The same below. |
可变区V1(图 3的双线部分)中SFBB16-γ基因和SFBB17-γ基因未发生突变,而SFBB28-γ基因和SFBB35-γ基因发生氨基酸的替换,即‘I’(第106)被‘N’替换,‘G’(第113)被‘D’或‘N’替换。可变区V2、可变区V3、可变区V4中SFBB16-γ、SFBB17-γ、SFBB28-γ和SFBB35-γ基因均未发生氨基酸的替换(图省略)。
2.4 梨花粉SFBB基因的聚类分析梨SFBB-γ基因在推导氨基酸序列上的差异见表 1。可看出,不同的SFBB-γ基因间差异不大,只有1~12个氨基酸的区别,其中SFBB22-γ基因和SFBB35-γ基因间的差异最大,具有12个氨基酸的差异。新分离的SFBB16-γ基因和SFBB17-γ基因与其他SFBB基因之间具有1~8个氨基酸的差异,差异相对较小,而SFBB28-γ基因与其他SFBB基因之间具有5~11个氨基酸的差异,SFBB35-γ基因与其他SFBB基因之间具有7~12个氨基酸的差异。日本沙梨中分离鉴定的SFBB基因(SFBB1-SFBB8)间差异较少,只有1~6个氨基酸的差异。而日本沙梨SFBB基因(如SFBB1-SFBB8)(Sassa et al., 2007; Vieira et al., 2008)和新疆梨(如SFBB28)或白梨(P. bretschneideri)(如SFBB13-SFBB28)基因SFBB差异更大,具有8~11个氨基酸的差异。
|
|
通过Clustalx和Mega2软件对梨SFBB-γ基因聚类分析结果见图 4。从图中可以看出,根据SFBB基因的序列可以划分出3类:新疆梨(如‘早熟句句’、‘魁克句句’)基因SFBB28-γ基因为一类;第2类为中国白梨(如‘早美酥’、‘鹅梨’)基因,SFBB35-γ和SFBB13-γ;其他基因聚到一起,包括日本沙梨(如‘丰水’、‘幸水’、‘二十世纪’)和中国沙梨(‘苍溪雪梨’、‘桂冠’、‘黄花’、‘宝珠’)SFBB基因。这和物种分类规律基本相符。

|
图 4 梨的SFBB基因的聚类图 Figure 4 Clustering chart of Pyrus SFBB genes |
配子体型自交不亲和植物的花粉自交不亲和基因如茄科植物的SLF、蔷薇科核果类果树的SFB基因已经被确定(Sonneveld et al., 2005;Surbanovski et al., 2007;Sutherland et al., 2008;Vieira et al., 2008)。仁果类果树梨和苹果中也已经确定SFBB基因是控制花粉自交不亲和基因,但分离克隆过程中发现SFBB基因以高度的遗传多态性的形式存在,如梨的SFBB基因包括a、b、γ等。对于这些基因哪个是真正的梨花粉自交不亲和基因目前尚不清楚,目前日本千叶大学和神户大学的学者正在开展梨SFBB基因的功能鉴定研究。因此,梨SFBB基因的分离克隆及遗传多态性分析为梨花粉SFBB基因的准确确定提供了理论依据。梨SFBB-γ基因一级结构包括1个F-Box区域和4个可变区(V1, V2, V3, V4)(Sassa et al., 2007)。本试验克隆的SFBB基因的F-Box区域氨基酸的变异较多,可达7处,4个可变区中V1, V3, V4中具有氨基酸的变异,而V2中均未发现氨基酸变异。这和日本梨SFBB1-SFBB9基因的序列规律基本一致。
SFBB基因序列变异因梨种不同有明显的差异。如日本沙梨中分离鉴定的SFBB基因(SFBB1-SFBB8)间差异较少(Sassa et al., 2007; Vieira et al., 2008),只有1~6个氨基酸的差异。而日本沙梨SFBB基因(如SFBB1-SFBB8)和新疆梨(如SFBB28)或白梨SFBB(如SFBB13-SFBB28)基因差异更大,具有8~11个氨基酸的差异。这可能与日本沙梨与新疆梨或白梨之间的遗传差异较大,亲缘关系相对远有关。梨SFBB基因是花粉自交不亲和基因,控制花粉的自交不亲和性。该基因存在很大的多态性,随着不同梨品种不同SFBB基因的遗传多态性的更深入的研究,为梨自交不亲和机理及遗传改良提供良好的理论基础。
乌云塔娜, 谭晓风, 李秀根, 等. 2007. 13个'新世纪'梨后代品种S基因型的鉴定[J]. 林业科学, 43(9): 116-122. |
Hiroyuki K, Takeshi T, Takato K. 2007. Polymorphism of SFBB-γ and its use for S genotyping in Japanese pear (Pyrus pyrifolia)[J]. Plant Cell Reports, 26: 1619-1625. DOI:10.1007/s00299-007-0386-8 |
McClure B A, Franklin T. 2006. Gametophytic self-incompatibility: understanding the cellular mechanisms involved in 'self' pollen tube inhibition[J]. Planta, 224: 233-245. DOI:10.1007/s00425-006-0284-2 |
Sassa H, Kakui H, Miyamoto M. 2007. S locus F-box brothers: multiple and pollen-specific F-box genes with S haplotype-specific polymorphisms in apple and Japanese pear[J]. Genetics, 175: 1869-1881. DOI:10.1534/genetics.106.068858 |
Sonneveld T, Tobutt K R, Vaughan S P, et al. 2005. Loss of pollen-S function in two self-compatible selections of Prunus avium is associated with deletion/mutation of an S haplotype-specific F-box gene[J]. Plant Cell, 17: 37-51. DOI:10.1105/tpc.104.026963 |
Sutherland B G, Tobutt K R, Robbins T P. 2008. Trans-specific S-RNase and SFB alleles in Prunus self-incompatibility haplotypes[J]. Mol Genet Genomics, 279(1): 95-106. DOI:10.1007/s00438-007-0300-7 |
Surbanovski N, Tobutt K R, Konstantinovi Ac' M. 2007. Self-incompatibility of Prunus tenella and evidence that reproductively isolated species of Prunus have different SFB alleles coupled with an identical S-RNase allele[J]. Plant J, 50(4): 723-734. DOI:10.1111/j.1365-313X.2007.03085.x |
Tao R, Yamane H, Sassa H. 1997. Identification of stylar RNases associated with gametophytic self-incompatibility in almond (Prunus dulcis)[J]. Plant Cell Physiol, 38(3): 304-311. DOI:10.1093/oxfordjournals.pcp.a029167 |
Ushijima K, Sassa H, Tao R. 1998. Cloning and characterization of cDNAs encoding S-RNases from almond (Prunus dulcis): primary structural features and sequence diversity of the S-RNases in Rosaceae[J]. Mol Gen Genet, 260(2/3): 261-268. |
Vieira J, Santos R A, Ferreira S M. 2008. Inferences on the number and frequency of S-pollen gene (SFB) specificities in the polyploid Prunus spinosa[J]. Heredity, 101(4): 351-358. DOI:10.1038/hdy.2008.60 |
 2010, Vol. 46
2010, Vol. 46

