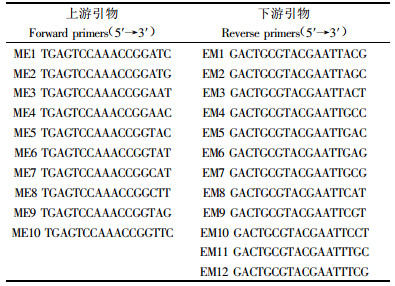
文章信息
- 刘志远, 范卫红, 沈世华
- Liu Zhiyuan, Fan Weihong, Shen Shihua
- 构树SRAP分子标记
- SRAP Marker in Broussonetia papyrifera
- 林业科学, 2009, 45(12): 54-58.
- Scientia Silvae Sinicae, 2009, 45(12): 54-58.
-
文章历史
- 收稿日期:2008-11-12
-
作者相关文章
2. 中国科学院研究生院 北京 100039
2. Graduate University of Chinese Academy of Sciences Beijing 100039
构树(Broussonetia papyrifera),主要分布于中国、马来半岛、日本、太平洋群岛(郑汉臣等,2004),属桑科(Moraceae)构树属(Broussonetia),是一种重要的速生绿化树种。构树具有重要的经济价值,构树皮是一种十分优良的造纸原料,其叶片也可用作饲料。此外,构树还具有重要的药用价值(杨小建等,2007)。目前对构树的研究主要集中于栽培、生理以及药理方面,在分子生物学方面的研究较少,仅见报道过从光叶楮(Broussonetia papyrifera)中克隆肌动蛋白基因片段(李岩等,2007)。构树集诸多优良性状于一身,具有广阔的发展前景,但由于其遗传背景不清楚,因而给遗传育种及推广带来了很大困难。
遗传标记作为基因型的易于识别的表现形式,在植物种质资源研究和育种工作中有着重要的地位。随着分子生物学的发展,遗传标记已经从形态学标记、细胞标记、生化标记发展到第4代——DNA分子标记。序列相关扩增多态性(SRAP)分子标记技术是Li和Quiros(2001)发明的可用来研究植物遗传育种的一种新颖有效的分子标记技术,又称基于序列扩增多态性(sequence-based amplified polymorphism,SBAP),其最大特点是针对基因的阅读框区域设计引物进行扩增而产生多态性,同时也具有简便、中等产量、高共显性、重复性好、易于分离条带及测序等优点(李巧燕等,2006)。本研究建立了构树基因组DNA的提取方法及构树的SRAP反应体系,通过SRAP分子标记对我国以及日本典型分布区的构树资源进行遗传多样性检测,分析不同地区构树的遗传资源多样性,同时找到一些特异性条带可转化为SCAR(sequence characterized amplified regions)标记,为研究构树的遗传多样性及育种奠定基础。
1 材料与方法 1.1 主要试剂和材料CTAB提取缓冲液:2% CTAB,100 mmol·L-1 Tris-HCl(pH 8.0),20 mmol·L -1 EDTA(pH 8.0),1% PVP(分子质量:40 ku),1.4 mmol·L-1 NaCl。
|
|
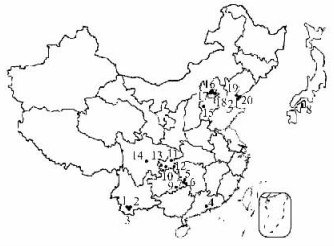
|
图 1 21份构树样品分布图 Figure 1 Distribution of 21 samples of B. papyrifera |
大量DNA提取采用改进的CTAB法。称取0.5 g新鲜构树叶片,用液氮研磨成粉末,倒入盛有5 mL CTAB溶液的离心管中,先加入0.1 g PVPP,再加入20 μL β-巯基乙醇,轻轻摇匀,65 ℃水浴45 min,冷却至室温。加入等体积的氯仿/异戊醇(体积比为24:1),颠倒数次,静置10 min。10 000 r/min-1离心10 min,取上清加入等体积氯仿抽提。10 000 r·min-1离心10 min,取上清加入1/2体积的氯化钠溶液(5 mmol·L-1),再加入等体积的预冷异丙醇-20 ℃沉淀30 min。75%乙醇洗沉淀2次,于超净工作台上晾干。加入500 μL双蒸水溶解,取20 μL DNA溶液进行琼脂糖凝胶电泳检测后保存于-20 ℃。
1.2.2 DNA扩增SRAP的PCR程序参考Li和Quiros(2001)的方法,所用SRAP引物见表 2。PCR反应程序:94 ℃预变性5 min,反应前5个循环:94 ℃ 1 min,35 ℃ 1 min,72 ℃ 1 min;后35个循环:94 ℃ 1 min,50 ℃ 1 min,72 ℃ 1 min;最后72 ℃延伸7 min。最佳反应体系建立参照正交试验设计原则,先确定反应体系中的最佳模板浓度,在此基础上再分别确定dNTP浓度和Mg2+最佳浓度比、引物和Mg2+最佳浓度比,进而确定最佳反应条件。
|
|
扩增产物在浓度为6%的聚丙烯酰胺凝胶(丙烯酰胺、甲叉双丙烯酰胺质量比为26:1)中电泳,电极缓冲液为1×TBE buffer,电流50 mA,恒电流跑约2 h,直至指示剂二甲苯青距玻璃板下部边缘1/3处停止。
1.2.4 银染参照Sanguinetti等(1994)的方法。
1.2.5 图像扫描与分析凝胶用UMAX Power Look 2100扫描仪(MaxiumTech,Taipei,China)扫描(分辨率300 dpi)。
1.3 数据处理与分析结果记录的标准是在同一迁移率上,有带记为“1”,无带记为“0”,转换成0 1矩阵。数据统计利用NTSYSpc2.10e(Rohlf,1998)软件分析,用SHAN程序中的UPGMA(unweighted pair group method using arithmetic average)方法进行聚类分析,并通过Tree plot模块生成聚类图。同时运用Popgene1.32软件进行遗传多样性分析、Shannon信息指数及物种水平的Nei基因多样性等分析。
1.4 大连构树特异性片段扩增引物上游引物M1:5′-TTCTTGCTAGTCACGGTTTTAATTT-3′,下游引物M2:5′-GTAGGAGGAGGGAGCCCATAGGTTT-3′。PCR扩增反应:预变性94 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,共40个循环;最后72 ℃延伸10 min。1%琼脂糖凝胶电泳检测PCR扩增结果,目的片段回收并测序。
2 结果与分析 2.1 构树叶片DNA的提取DNA的提取质量是决定SRAP标记成功与否的关键。由于构树叶片中含有大量的糖类及酚类等杂质,而一般CTAB方法(王关林等,1998)去除多糖效果不明显,多糖等杂质残留在点样孔周围(图 2A),所以与多糖理化性质很相近的RNA等杂质很难除去,所提的构树DNA质量较差,严重影响了后续试验操作。而本试验中加入的高浓度的盐有效地减少了多糖,改进后的方法所提的DNA质量明显提高(图 2B),可以用作进一步试验。
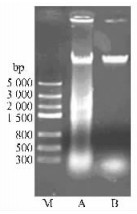
|
图 2 2种方法所获得的构树基因组总DNA Figure 2 Genomic DNA of B. papyrifera by two methods M:标记物下同。 Marker. The same below. |
通过正交试验设计,确定最佳SRAP-PCR反应体系各成分浓度为:模板DNA 4 ng·μL-1 ,Mg2+浓度2.0 mmol·L-1,dNTP浓度0.6 mmol·L-1,引物浓度0.8 mmol·L-1,Taq DNA聚合酶0.075 U·μL-1,可以扩增出清晰、多态性高及重复性好的谱带(图 3)。
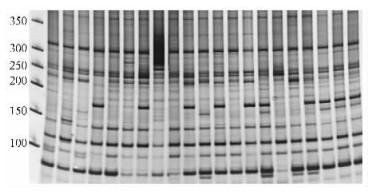
|
图 3 引物组合ME4/EM8扩增21份构树样品的结果 Figure 3 Amplification of DNA from 21 samples of B. papyrifera using primer combination ME4/EM8 |
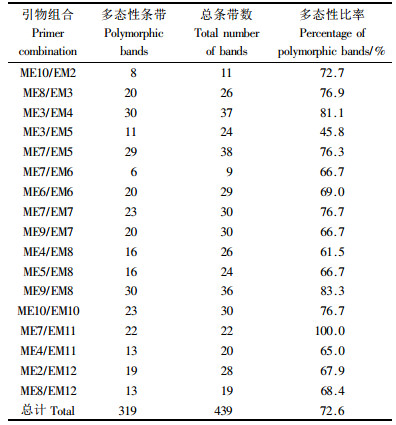
从120对引物组合中筛选出多态性信息较高的17对引物(表 3)。这17对引物对21份样品扩增共产生439条扩增带,平均每对引物产生25.5条带,其中多态性条带有319条,占72.6%以上;1对引物最多能产生30条多态性片段,多态性比率最高的有100%,可见SRAP多态性还是较高的。经聚类分析,从图 4可以看出,遗传距离差距最大的是云南西双版纳和日本的构树;在相似系数0.57附近,可以将21份构树样品分成2大类:一类为中国大陆构树,另一类为日本构树。这可能是因为日本很久以前就和大陆分离,其构树和其他地区(大陆)构树之间基因交流间断,因而进化缓慢或者很少发生变化,从而导致2个地方构树之间遗传距离差距巨大。在0.85附近,大陆构树又可以分成2大类:云南西双版纳和其他地区2个派系;在0.91附近,广东广州和湖南以及贵州构树聚为一类,重庆、四川、河北、北京以及大连构树聚为一类,基本上和地理气候分布一致(图 1)。
|
|
用Popgene1.32软件分析,Nei常数均值为0.133 6,Shannon信息指数均值为0.227 5,总体上构树的遗传多样性不高,与其近亲桑树(Morus)相差不大(赵卫国等,2008)。
2.4 大连构树特征性条带用引物ME8/EM12进行扩增发现大连构树的1条约400 bp的特征性条带,回收、测序,序列见图 5。设计引物M1/M2再对21份样品进行扩增,发现得到2条稳定的特征性条带(图 6),经测序证实条带1为以引物M2为上游引物、M1为下游引物扩增所得到的,条带2为目的条带,与图 4中特征性条带序列一致,可能成功转化为大连构树SCAR标记。

|
图 5 大连构树特异性片段全序列 Figure 5 Whole specific sequence of Dalian B. papyrifera 横线处为引物序列。 The primers are underlined. |

|
图 6 大连构树SCAR标记 Figure 6 SCAR marker of Dalian B. papyrifera |
构树作为一种优良的速生造纸原料,同时也是一种具有重要经济价值和环保价值的树种,由于其遗传背景知之甚少,严重制约了其推广应用。因此如何拓宽构树种质资源,挖掘有价值的种质资源材料已成为当今构树产业化一个急需解决的问题。随着现代科学技术的快速发展,应用现代生物技术与传统常规技术结合,可通过种质创新与分子辅助育种为构树的推广应用奠定良好的基础,而SRAP分子标记技术为实现这一目标提供了一条良好途径。
虽然SRAP标记技术具有很多优点,但它同样是一种基于PCR的分子标记技术,依然会受到诸多因素的影响,包括模板DNA质量、Mg2+、dNTP、引物浓度等(邹喻萍等,2001)。本试验中在DNA提取过程中加入了少许PVPP来吸附色素,巯基乙醇防止酚类氧化,去除酚类等杂质效果较明显,尤其加入高浓度的盐可以有效地除去多糖类物质,因而可以获得质量较高的基因组DNA(图 2B)。在SRAP反应体系中,通过设计正交试验,充分考虑各种因素,建立最佳体系,为后续试验奠定了良好基础。
目前,SRAP分子标记在遗传多样性检测(Ferriol et al., 2003;Espósito et al., 2007)、基因定位(潘俊松等,2005)、特异表型性状定位(郭庆华等,2007)、遗传图谱构建(林忠旭等,2003;Gao et al., 2008)、比较基因组学(Li et al., 2003)、遗传多样性分析(Han et al., 2008)、植物分子育种(Nagl et al., 2007)、DNA指纹图谱分析(Ahmad et al., 2008)等方面都得到了广泛应用。本试验中,17对SRAP引物扩增出439条带,其中多态性条带319条,比例超过70%。对21份材料进行聚类分析表明,10个不同地方的构树可以分为日本和大陆2大类,可能是由于日本作为一个岛国和大陆分离久远,和大陆构树之间基因交流中断,加上面积狭小,品种之间基因交流缓慢,因此二者遗传距离差距较大。大陆构树从聚类分析结果来看,其分布基本上都和特定地理条件相关,因此挖掘一些具有能够适应极端气候条件基因资源的构树,对于构树应用、品种选育具有重要实质意义。本试验找到1个大连构树特有片段并转换成稳定性更好的SCAR标记,但尚需扩大样本进一步验证,可能对实践具有一定的指导意义。
郭庆华, 郭美丽. 2007. 与红花苞叶刺性状紧密连锁的SRAP分子标记研究[J]. 药学学报, 42(7): 794-797. DOI:10.3321/j.issn:0513-4870.2007.07.019 |
李巧燕, 林瑞庆, 朱兴全. 2006. SRAP分子标记及其应用概述[J]. 热带医学杂志, 6(4): 467-469. DOI:10.3969/j.issn.1672-3619.2006.04.041 |
李岩, 李冠. 2007. 光叶楮肌动蛋白基因片段的克隆及其序列分析[J]. 生物技术, 17(4): 1-3. |
林忠旭, 张献龙, 聂以春, 等. 2003. 棉花SRAP遗传连锁图构建[J]. 中国科学通报, 48(15): 1676-1679. |
潘俊松, 王刚, 何欢乐, 等. 2005. 黄瓜SRAP遗传连锁图的构建及始花节位的基因定位[J]. 自然科学进展, 15(2): 167-172. DOI:10.3321/j.issn:1002-008X.2005.02.007 |
王关林, 方宏筠. 1998. 植物基因工程原理与技术[M]. 北京: 科学出版社, 598-600.
|
杨小建, 王金锡, 胡庭兴. 2007. 中国构树资源的综合利用[J]. 四川林业科技, 28(1): 39-43. DOI:10.3969/j.issn.1003-5508.2007.01.008 |
赵卫国, 汪伟, 杨永华, 等. 2008. 我国不同生态类型桑树地方品种遗传多样性的ISSR分析[J]. 蚕业科学, 34(1): 1-5. DOI:10.3969/j.issn.0257-4799.2008.01.001 |
郑汉臣, 黄宝康, 秦路平, 等. 2004. 构树属植物的分布及其生物学特性[J]. 中国野生植物资源, 21(6): 11-13. |
邹喻萍, 葛颂, 王晓东. 2001. 系统进化植物学中的分子标记[M]. 北京: 科学出版社, 36-97.
|
Ahmad R, Liow P S, Spencer D F, et al. 2008. Molecular evidence for a single genetic clone of invasive Arundo donax in the United States[J]. Aquat Bot, 88(2): 113-120. DOI:10.1016/j.aquabot.2007.08.015 |
Espósito M A, Martin E A, Cravero V P, et al. 2007. Characterization of pea accessions by SRAP's markers[J]. Sci Hortic, 113(4): 329-335. DOI:10.1016/j.scienta.2007.04.006 |
Ferriol M, Pico B, Nuez F. 2003. Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP marker[J]. Heor Appl Genet, 107(2): 271-282. DOI:10.1007/s00122-003-1242-z |
Gao Lixia, Liu Nian, Huang Banghai, et al. 2008. Phylogenetic analysis and genetic mapping of Chinese Hedychium using SRAP ma rkers[J]. Sci Hortic, 117(4): 369-377. DOI:10.1016/j.scienta.2008.05.016 |
Han Xiaoyan, Wang Liangsheng, Liu Zheng'an, et al. 2008. Characterization of sequence-related amplified polymorphism markers analysis of tree peony bud sports[J]. Sci Hortic, 115(3): 261-267. DOI:10.1016/j.scienta.2007.09.003 |
Li G, Gao M, Quiros C F, et al. 2003. Gene for gene alignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping[J]. Theor Appl Genet, 107(1): 168-180. DOI:10.1007/s00122-003-1236-x |
Li G, Quiros C F. 2001. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet, 103(2/3): 455-461. |
Nagl N, Weiland J, Lewelle R. 2007. Detection of DNA polymorphism in sugar beet bulks by SRAP and RAPD markers[J]. J Biotechnol, 131(2): S32-S35. |
Rohlf J F. 1998. NTSYS-pc:Numerical taxonomy and multivariate analysis system. Version 2.1 Exeter Software[M]. New York: Setauke.
|
Sanguinetti C J, Dias N E, Simpson A J. 1994. Rapid silver staining and recovery of PCR products separated on polyacrylamide gels[J]. Biotechniques, 17(5): 914-921. |
 2009, Vol. 45
2009, Vol. 45