文章信息
- 洑香香, 杨立伟, 施季森, 王桂风
- Fu Xiangxiang, Yang Liwei, Shi Jisen, Wang Guifeng
- mRNA差异显示法研究杉木木材形成相关cDNA
- Isolation cDNA Related to Wood Forming in Chinese Fir (Cunninghamia lanceolata) by mRNA Differential Display
- 林业科学, 2009, 45(12): 30-35.
- Scientia Silvae Sinicae, 2009, 45(12): 30-35.
-
文章历史
- 收稿日期:2008-08-28
-
作者相关文章
杉木(Cunninghamia lanceolata)是我国特有的针叶树种之一,广泛分布于我国南方1 5个省区,它具有生长快、材质好及产量高等特点,是重要的用材树种。通过几十年的选育,已获得了大量的杉木优良种源家系和栽培无性系,同时在基因组水平的研究上也已取得了一些进展(施季森等,2004;童春发等,2004)。
为了分离与木材形成相关的目的基因,进一步研究木材形成的分子机理及改良林木的材性,大量的研究工作侧重于构建木材形成组织的cDNA文库并进行大量EST测序(Lorenz et al., 2002; Cato et al., 2006; 洑香香等, 2008;Wang et al., 2007)、从木材组织中分离相关蛋白(Du et al., 2006)和cDNA微阵列分析(Yang et al., 2004)。这些研究为木材相关基因的表达和分离提供了有用的信息。本研究以杉木的自然突变体独干杉(Cunninghamia lanceolata var. dugan)和句容0号杉(Cunni nghamia lanceolata var. jurong 0)为材料,利用mRNA差异显示法(differential display reverse transcriptase polymerase chain reaction,DDRT-PCR)获得与木材形成相关的差异片段,为木材形成相关基因的分离和克隆奠定基础。
1 材料与方法 1.1 植物材料研究材料为杉木的2个自然变异类型“句容0号杉”和“独干杉”。句容0号杉针叶扁硬、稀疏,生长较快,其茎干生长呈现出明显的一年一轮状分枝性状,每轮节间距离长,平均可达1 m左右,最长时可达2~3 m;每轮盘分枝数为4~6个(图 1B)。独干杉的特点是在自然状态下,主干没有分枝,高生长明显,径生长十分不明显,每年仅有顶芽萌发向上生长。因主干纤细,正常状态下无法直立,生长至一定高度后出现下垂弯曲现象和折断;同时独干杉还表现为灌木性状,易从基部产生萌条(图 1A)。

|
图 1 杉木的2个自然变异类型 Figure 1 Two natural mutants of Chinese Fir |
试验样品取自2个杉木突变体的生长期,取材部位为2~3年生茎段皮层,包括初生木质部、形成层的分生组织区、初生韧皮部、次生韧皮部和周皮组织。取材时用蒸馏水把取材部位冲洗干净,连同整个枝条剪下,迅速置于液氮中,带回实验室后,置于-70 ℃超低温冰箱中保存备用。
1.2 总RNA提取采用改良CTAB法提取总RNA(Chang et al., 1993)。获得的总RNA用RQ1 RNase-Free DNase消化后再过柱纯化。1%琼脂糖凝胶电泳和紫外分光光度计(GeneQuant pro)检测总R NA的质量。
1.3 DDRT-PCR分析转录所用的锚定引物(AP)和随机引物均来自Genomyx的HIEROGLYPHTM mRNA Profile。
1.3.1 反转录(第1链cDNA合成)在20 μL第1链反应体系中先加入总R NA 2.0 μL(0.1 μg·μL-1)、锚定引物2.0 μL(2.0 μmol·L- 1)和8.0 μL RNase-free水;混匀后,在-70 ℃条件下温育5 min,迅速置于冰上。在上述混合物中依次加入:4.0 μL SuperScript Ⅱ RT Buffer(5×)、2.0 μL dN TPs(250 μmol·L-1)、2.0 μL DTT(100 μmol·L-1)和0.2 μL SuperScript Ⅱ RT enzyme(200 U·μL-1),混匀后运行下列程序:25 ℃ 5 min;42 ℃ 15 min;50 ℃ 50 min;70 ℃ 10 min;4 ℃保持。
1.3.2 DDRT-PCR(第2链cDNA合成)在20 μL PCR反应体系中加入2.0 μL cDNA第1链产物、2.0 μL 10×PCR Buffer、2.0 μL MgCl2(25 mmol·L- 1)、2.0 μL dNTPs mix(25 mmol·L-1)、1.0 μL与第1链合成一致的锚定引物(5 μmol·L-1)、2.0 μL随机引物(2 μmol·L-1)、0.2 μL Taq酶(5 U·μL-1)、8.8 μL无菌水。充分混和后,按下列程序运行:95 ℃ 2 min;5个循环:94 ℃ 30 s,50 ℃ 30 s,72 ℃ 2 min;25个循环:94 ℃ 30 s,57 ℃ 30 s,72 ℃ 2 min;72 ℃ 10 min;4 ℃下保持。
1.3.3 DDRT-PCR产物的银染检测及回收扩增产物采用6 %变性聚丙烯酰胺凝胶电泳,胶长47 cm、宽17 cm、厚0. 4 cm,电泳功率70 W,银染法显示cDNA电泳条带。将凝胶上对应差异条带切下,放入400 μL DNA回收溶液中[20%乙醇、1 mol·L-1 LiCl、10 mmol·L-1 Tris-Cl(pH 7.5)]置于室温下放置24 h后,65 ℃温育2 h,无水乙醇沉淀后离心,回收的差异片段即为二次扩增的模板。用T7和M13引物进行二次扩增,以增加差异条带的量;二次扩增产物用1%琼脂糖凝胶电泳,并回收纯化。
1.3.4 反向Northern点杂交cDNA差异片段与pGEM-T Eeasy载体连接,转入Escherichia coli DH5α,通过蓝白斑筛选获得阳性克隆;碱法提取质粒。用EcoR Ⅰ酶切质粒进行电泳鉴定;另外用质粒(约600 ng·μL-1)点到Hybond N+尼龙膜上,在80 ℃烘箱中烤膜2 h以固定DNA。以地高辛(DIG)标记的独干杉和句容0号杉总RNA为探针,将膜置于适量预热的DIG标准杂交液在杂交炉中65 ℃预杂交30 min~2 h,然后加适量探针在65 ℃杂交过夜。洗膜显色后,扫描或照相。
1.3.5 差异片段的测序与分析将经检测正确并经过反向Northern点杂交证实为阳性的cDNA克隆的菌液送至上海博亚生物有限公司测定序列,利用GenBank数据库进行Blast分析。
2 结果与分析 2.1 总RNA的完整性和纯度杉木组织中富含酚类等大量次生代谢物质,用常规的方法提取总RNA并不是十分有效。本文采用改良CTAB法获得了完整的总RNA,28S RNA的量约为18S RNA的2倍,说明RNA无降解,提取效果较好。总RNA经DNase Ⅰ消化后,用紫外分光光度计测定RNA的纯度,260 nm/280 nm值在1.9~2.0之间,260 nm/230 nm的值在2.1~2.3间,说明RNA的纯度高,没有蛋白质及其他物质的污染。
2.2 DDRT-PCR 2.2.1 差异片段的获得利用12个锚定引物和12个随机引物组合,进行RT -PCR。结果表明,锚定引物T7(dT12)AP3、T7(dT12)AP5、T7(dT12)AP6、T7(dT12)AP7和随机引物M13r-ARP3、M13r-ARP4、M13r-ARP5、M13r-ARP7、M13 r-ARP8都有产物且获得差显片段(图 2)。

|
图 2 DDRT-PCR产物银染显示 Figure 2 DDRT-PCR products display by silver stain M:Marker.1-16:锚定引物AP3和随机引物ARP1-ARP8组合PCR products primed by anchor primer AP3 combined with random primers ARP1-ARP8 respectively. 17-32:锚定引物AP4和随机引物ARP1-ARP8组合PCR Products of anchor primer AP4 combined with random primers ARP1-ARP8 respectively.箭头指示不同引物组合对2个样本扩增出的差异片段Arrows indicate the differentially displayed fragments in two samples. |
根据是否表达和表达量的强弱,初步把差异条带的情况分为以下4类:Ⅰ类,在句容0号杉中表达,而独干杉未表达;Ⅱ类,在独干杉表达,句容0号杉不表达;Ⅲ类,在句容0号杉中表达强,独干杉表达量弱;Ⅳ类,句容0号杉中表达弱,独干杉表达量强。据此,统计共获得54条差异条带。其中Ⅰ类条带占40.7%,Ⅱ类占38.9%,Ⅲ类和Ⅳ类合占20.4%。Ⅲ类和Ⅳ类切取的条带少,可能主要是由于试验误差所引起,因此仅选择了亮度差异十分显著的条带。
2.2.2 差异片段的回收总体来看,回收片段长度位于300~900 bp之间,大部分集中在350~500 bp,占74.1%;大于800 bp的片段仅占3.7%;500~800 bp的片段有20.4%。
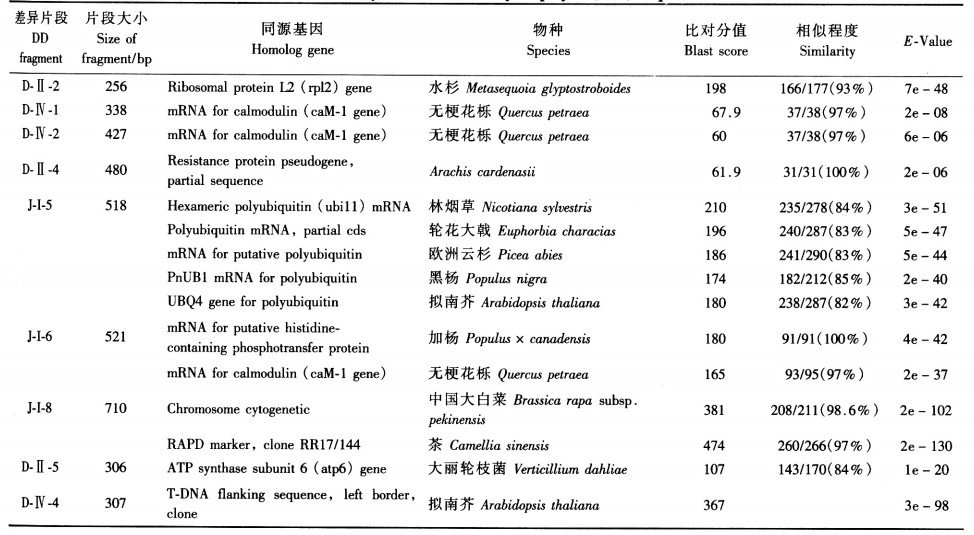
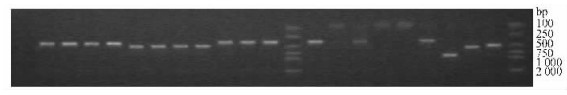
回收的差异片段DNA含量很低,不能满足后续的克隆和测序用量,需作二次扩增放大。对二次PCR扩增产物的电泳检测(图 3)发现:差异片段(共54个片段)的二次PCR有41个获得了较强的扩增产物,且都是单条带,没有出现2条带或多带的情况;有6个片段的产物量较低;还有7个片段没有得到扩增产物,回收率可达75.9%。
|
图 3 部分差异条带的二次扩增产物的电泳检测 Figure 3 The second PCR products of differentially displayed fragments separated in agrosegel |
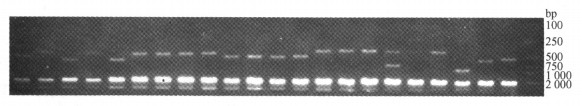
差异片段经纯化与T载体连接后,转化子用大肠杆菌DH 5α来扩大繁殖,并通过蓝白斑来筛选。质粒提取后进行酶切,电泳检测产物是否和原差异片段大小相符。结果表明有35个质粒酶切产物片段大小正确;有2个质粒的酶切获得了2个片段(图 4),推测是否其片段内部也存在着酶切位点,有待进一步研究;另外,还有2个质粒酶切后无产物,2个质粒酶切产物与原片段大小不符,这4个片段弃之不用。
|
图 4 EcoR Ⅰ酶切质粒的电泳检测 Figure 4 Examination of plasmids enzymed using EcoR Ⅰ by agrose gel electrophoresis |
DDRT-PCR存在的主要问题是假阳性偏高。为了筛除假阳性片段,本试验采用反向Northern法来检测获得的差异片段。根据杂交膜上杂交信号对比(图 5),发现在句容0号有杂交信号,而在独干杉上无杂交信号的点有17个;在独干杉有杂交信号而在句容0号无信号的点有10个;而在2张膜上杂交信号有差异的点有8个;在2张膜上杂交信号无差异的点(包括无杂交信号和有杂交信号但信号强度难以说明问题的点)共25个。综合统计,共获得35个阳性克隆,去掉4个克隆(质粒酶切出现2个片段及酶切无产物),还有31个有效的阳性片段;假阳性片段为6个(14.6%)。

|
图 5 反向Northern杂交检测阳性片段 Figure 5 Reverse Northern-blot analysis of differentially displayed fragment Ⅰ:以句容0号总RNA为探针The probe is total RNA of jurong 0;Ⅱ:以独干杉总RN A为探针The probe is total RNA of dugan. F1,F2,F3:阳性对照(28S RNA) Posit ive control (28S RNA); F4:阴性对照Negative control. |
测序以M13为引物进行序列测定,除去2个质量不高的序列,共获得29条序列。剪切掉载体序列后,和GeneBank数据库进行比对分析。
根据Blastn的比对条件(分值>60),有9个cDNA克隆在GeneBank中找到了相似的功能(表 1)。其中D-Ⅱ-2片段与水杉(Metasequoia glyptostroboides)中所获得的核糖体蛋白[L2(rpl2)]基因功能相似;D- Ⅳ-1、D-Ⅳ-2和J-Ⅰ-6具信号传导功能,与无梗花栎(Quercus petraea)的钙调蛋白基因(CaM -1)相似。另外有关抗性功能的片段有D-Ⅱ-4;J-Ⅰ-6号片段还与加杨(Populus× canadensis)含组氨酸的磷酸转移蛋白的功能相关。而与细胞遗传相关的为J-Ⅰ-8片段,D-Ⅱ-5片段是和大丽轮枝菌(Verticillium dahliae)中的能量代谢功能相似。
|
|
12个差异片段进行Blastx分析,并依此进行功能推测:其中7个片段(包括J-Ⅰ-1,J- Ⅲ-1,D-Ⅱ-1,J-Ⅲ-5,D-Ⅳ-3,J-Ⅰ-7及D-Ⅳ-5)与细菌的一些基因功能相似,可能涉及膜结构、细胞因子、代谢过程中酶的活性等;5个cDNA片段(J-Ⅰ-2,J- Ⅰ-3,D-Ⅱ-3,J-Ⅰ-4和J-Ⅰ-9)与一些植物[拟南芥、玉米(Zea mays)和水稻(Oryza sativa)]的基因功能相似,功能涉及锌指蛋白、激酶蛋白、cf2蛋白、抗性蛋白及过氧化氢酶(CAT-2)等。
还有8个cDNA克隆在数据库中没有找到任何匹配的信息。
3 结论与讨论目前在对针叶树基因组的研究中,有关木材形成和次生木质部的形成成为研究的热点,而火炬松(Pinus taeda)成为研究的代表树种。测定的序列主要来自于火炬松有关木材发育过程(次生木质部)所构建的6个cDNA文库(Quackenbush et al., 2000);Pavy等(2005)对这6个cDNA文库的共7 732个不同Contigs的EST鉴别出了260个差异表达基因序列。Ujino-Ihara等(2000)以日本柳杉(Cryptomeria japonica)树皮内层构建了cDNA文库,测定了2 231个ESTs,发现其中有3%克隆与细胞壁形成相关。
本研究第1次通过DDRT-PCR法研究杉木木材形成相关的基因。在29个阳性克隆中,有31%的cDNA克隆获得了同源基因的信息,还有41.4%cDNA克隆可推测蛋白功能,但仍有27.6%在数据库中没有相关信息,即杉木有很高比例的cDNA克隆属于未知领域。Wang等(2007)利用SS H研究杉木时发现有43%的EST无比对结果或属于未知功能的蛋白;洑香香等(2008)构建杉木皮部cDNA文库测序进行EST分析时也获得了相似的结果(近50%无比对结果)。这说明杉木中可能存在大量的新基因未被认知,但也可能是因为EST的测序数量太少或序列太短所造成。
在所获得的差异cDNA克隆中,在独干杉中表达量比较高的钙调蛋白是一种热稳定蛋白,能结合钙离子的磷酸二酯酶激活蛋白,具有4个EF-hand motifs(保守的helix-loop-helix结构和1个钙离子结合)。CaM和相关蛋白的相互作用及对细胞周期具有广泛的调节作用,受C aM调控的生长因子包括代谢酶、结构蛋白、转录因子、离子通道等(Snedden et al., 2001)。Allona等(1998)在对火炬松的木质部形成层的cDNA文库进行EST测序研究中,发现了3个cDNA片段,它们与钙平衡及细胞对钙的调节有关,这是非常有意义的,因为钙和钙调蛋白在植物中和木材密度有关(Stinemetz et al., 1987; Young et al., 1994)。
泛素基因家族(ubiquitin family)的多聚泛素(J-Ⅰ-5)只在句容0号中显示,它在进化过程中是高度保守的。泛素-蛋白酶体途径是Hershko等(2000)发现的一种高效蛋白降解途径,其主要作用是对细胞内蛋白质进行降解。它能够选择性降解蛋白质,使细胞内各种蛋白质的降解受到精确的调控,这对于维持细胞的许多重要功能(如细胞周期的调控、转录调控、凋亡等)有着极其重要的作用(Doherty et al., 2002)。Pozo等(1998)在拟南芥中发现生长素抑制基因(auxin-resistant)AXR1可降低生长素的活性;洑香香等(2008)在杉木茎段皮层的cDNA文库中也发现了编码泛素降解蛋白的ARP(auxin-repressed protein)基因片段。生长素的活性反应途径是泛素途径,通过泛素途径可降低生长素的活性,这说明泛素途径可能调节生长素的活性。因此推测在独干杉中未发现泛素,因而不能降解生长素,从而抑制了侧枝的萌发。这验证了作者的另一个试验,即去掉独干杉的顶芽,侧芽就开始萌发(未发表)。
锌指蛋白作为普遍存在于真核生物中的基因转录因子来调控基因的转录,它可以和DNA、RNA和蛋白质结合,也涉及到蛋白质-蛋白质之间的相互作用。本研究中获得的J-Ⅰ-2差异片段与水稻(日本栽培品种)的锌指(CCCH-type)类蛋白相似。在水稻的抗寒反应中发现了2种锌指蛋白基因(CCCH型),并且2种基因对水分胁迫有反应(Lee et al., 2002)。Prigge等(2001)研究了拟南芥的se突变体,发现突变体在子叶和胚萌发后器官横向初始生长和伸长有缺陷。经分离克隆后发现SE基因编码了1个在正常茎发育过程中所必需的锌指蛋白(C2H2型)。在整个生长过程中其转录体出现于茎的分生组织和器官原基,进一步分析表明SE基因产物通过修饰染色质来调节基因表达的变化。在对火炬松的木质部形成层的EST研究中,发现了与锌指蛋白形成的特定结构域相似的克隆(Allona et al., 1998)。另外,Pavy等(2005)在火炬松的6个cDNA文库中,发现1个序列和RING蛋白相似,它大量出现于拟南芥中,但功能还不十分清楚(Kosarev et al., 2002)。
综上所述,在句容0号杉中特异表达的多聚泛素和含组氨酸磷酸的转移蛋白分别和生长素及细胞分裂素的代谢途径相关,而这些激素都是影响植株高生长、径生长及分枝性状的重要调控因子。这些基因在独干杉中未见表达,独干杉的表型特征(有株高生长、无径生长且无分枝)可能和这些基因的调控密切相关,但如何调控仍有待进一步分析。
洑香香, 杨立伟, 施季森, 等. 2008. 杉木茎段皮层cDNA构建及EST分析[J]. 分子植物育种, 6(4): 683-688. DOI:10.3969/j.issn.1672-416X.2008.04.010 |
施季森, 童春发. 2004. 利用改进的复合区间作图法和F1代群体进行杉木的QTL作图[J]. 分子植物育种, 2(1): 1-6. DOI:10.3969/j.issn.1672-416X.2004.01.001 |
童春发, 施季森. 2004. 利用杉木的F1代群体构建遗传连锁图谱[J]. 遗传学报, 31(10): 1149-1156. |
Allona I, Quinn M, Shoop E, et al. 1998. Analysis of xylem formation in pine by cDNA sequencing[J]. Proc Natl Acad Sci USA, 95: 9693-9698. DOI:10.1073/pnas.95.16.9693 |
Cato S, McMillan L, Donaldson L, et al. 2006. Wood formation from the base to the crown in Pinus radiata: gradients of tracheid wall thickness, wood density, radial growth rate and gene expression[J]. Plant Mol Biol, 60: 565-581. DOI:10.1007/s11103-005-5022-9 |
Chang S, Puryear J, Cairney J. 1993. A simple and efficient method for isolation RNA from pine tree[J]. Plant Mol Biol Rep, 11(40): 114-117. |
Doherty F J, Dawson S, Mayer R J. 2002. The ubiquityin-proteasome pathway of in tracellular proteolysis[J]. Essays Biochem, 38: 51-63. DOI:10.1042/bse0380051 |
Du Juan, Xie Hongli, Zhang Deqiang, et al. 2006. Regeneration of the secondary vascular system in poplar as a novel system to investigate gene expression by a proteomic approach[J]. Proteomics, 6: 881-895. DOI:10.1002/(ISSN)1615-9861 |
Hershko A, Ciechanover A, Varshavsky A. 2000. Basic medical research award: The ubiquitin system[J]. Nat Med, 6(10): 1073-1081. DOI:10.1038/80384 |
Kosarev P, Mayer K F, Hardtke C S. 2002. Evaluation and classification of RING-finger domains encoded by the Arabidopsis genome[J]. Genome Biol, 3(4): research 0016.1-0016.12. |
Lee J M, William M E, Tingey S V, et al. 2002. DNA array profiling of gene expression changes during maize embryo development[J]. Funct Integr Genomics, 2: 13-27. DOI:10.1007/s10142-002-0046-6 |
Lorenz W W, Dean J F. 2002. SAGE profiling and demonstration of differential gene expression along the axial developmental gradient of lignifying xylem in loblolly pine (Pinus taeda)[J]. Tree Physiol, 22: 301-310. DOI:10.1093/treephys/22.5.301 |
Pavy N, Laroche J, Bousquet J, et al. 2005. Large-scale statistical analysis of secondary xylem ESTs in pine[J]. Plant Mol Biol, 57(2): 203-224. DOI:10.1007/s11103-004-6969-7 |
Pozo J C, Timpte C, Tan S, et al. 1998. The ubiquityin-related protein RUB1 and auxin response in Arabidopsis[J]. Science, 280: 1760-1763. DOI:10.1126/science.280.5370.1760 |
Prigge M J, Wagner D R. 2001. The Arabidopsis serrate gene encodes a zinc-finger protein required for normal shoot development[J]. The Plant Cell, 13: 1263-1279. DOI:10.1105/tpc.13.6.1263 |
Quackenbush J, Liang F, Holt I, et al. 2000. The TIGR gene indices: reconstruction and representation of expressed gene sequences[J]. Nucleic Acids Res, 28: 141-145. DOI:10.1093/nar/28.1.141 |
Snedden W A, Fromm H. 2001. Calmodulin as a versatile calcium signal transducer in plants[J]. New Phytologist, 151: 35-66. DOI:10.1046/j.1469-8137.2001.00154.x |
Stinemetz C L, Kuzmanoff K M, Evans M L, et al. 1987. Correlation between calmodulin activity and gravitropic sensitivity in primary roots of maize[J]. Plant Physiol, 84: 1337-1342. DOI:10.1104/pp.84.4.1337 |
Ujino-Ihara T, Yoshimura K, Ugawa Y, et al. 2000. Expression analysis of ESTs derived from the inner bark of Cryptomeria japonica[J]. Plant Mol Biol, 43(4): 451-457. DOI:10.1023/A:1006492103063 |
Wang Guifeng, Gao Yan, Yang Liwei, et al. 2007. Identification and analysis of differentially expressed genes in differentiating xylem of Chinese fir (Cunninghamia lanceolata) by suppression subtractive hybridization[J]. Genome, 50(12): 1141-1155. DOI:10.1139/G07-091 |
Yang S H, van Zyl L, No E G, et al. 2004. Microarray analysis of genes preferentially expressed in differentiation xylem of loblolly pine (Pinus taeda)[J]. Plant Sci, 166: 1185-1195. DOI:10.1016/j.plantsci.2003.12.030 |
Young L M, Evans M L. 1994. Calcium-dependent asymmetric movement of 3H-indole-3-acetic acid across gravistimulated isolated root caps of maize[J]. Plant Growth Regulation, 14: 235-240. DOI:10.1007/BF00024798 |
 2009, Vol. 45
2009, Vol. 45