文章信息
- 魏志刚, 高玉池, 刘桂丰, 刘关君, 杨传平.
- Wei Zhigang, Gao Yuchi, Liu Guifeng, Liu Guanjun, Yang Chuanping
- 白桦核心种质初步构建
- Preliminary Construction of Core Collection of Betula platyphylla Germplasm
- 林业科学, 2009, 45(10): 74-80.
- Scientia Silvae Sinicae, 2009, 45(10): 74-80.
-
文章历史
- 收稿日期:2008-07-23
-
作者相关文章
植物种质资源是品种选育的重要物质基础,因此其搜集和保护受到世界各国的高度重视。截止2001年,己有610万份植物种质资源被收集和保存(贾继增等,2001)。种质资源库数量的不断增加给保存、评价、研究和利用带来了巨大困难,Frankel等(1984)在种质资源库的基础上提出了核心种质的概念,其目的是以最少数量的种质资源最大限度地保存整个群体的遗传多样性,从而解决种质资源规模过大难以管理和利用的难题(Gepts, 2006)。至20世纪末,己有51个物种构建了63个核心种质(李自超等,2003)。林木种质资源即森林植物种质资源,是陆地生物基因的“避护所”和“主基因库”, 森林植物物种及种质丢失,将引起邻近生物种以4~13倍的速率丢失(顾万春等,2007),因此林木种质资源不仅是森林生态系统最重要的影响因子,而且是一个国家的重大战略资源(顾万春等,2007;顾万春,2005)。目前,林木核心种质构建工作严重滞后于作物,如国外进行过相关研究的树种仅有咖啡(Coffea arabica)(Milene et al., 2008)、可可(Theobroma cacao)(Lerceteau et al., 1997)、大叶千斤拔(Flemingia macrophylla)(Meike et al., 2006)和桃(Prunus persica)(Wunschl et al., 2006)等,我国仅限于中国蜡梅(Chimonanthus praecox)(赵冰等,2007)、中国茶树(Camellia sinensis)(李娟等,2004)、桃(Prunus persica)(李天红,2005)和珙桐(Davidia involucrata)(宋丛文等,2005)等树种。
白桦(Betula platyphylla)耐寒性强、山地适应性好、生长速度快,集中分布于我国东北三省及内蒙古自治区,是我国极为重要的树种之一(郑万钧,1985)。“八五”攻关以前,人们对白桦经济价值及重要性认识不足,经常将它作为营林生产中的伐除对象,导致其种质资源严重流失。为了保护白桦的优良种质资源和维持其可持续性遗传改良,急需开展该树种核心种质的构建。本文以白桦种源试验林中240个家系的胸径、树高、材积和纤维素含量数据为依据,通过比较和分析不同抽样比例、遗传距离、抽样策略和聚类方法下构建的核心种质各性状评价参数后,初步构建起较为合理的白桦核心种质库。
1 材料与方法 1.1 材料白桦240个家系材料来源于东北林业大学帽儿山试验林场1999年营造的白桦种源试验林,该试验林按完全随机区组设计,重复5次,单行排列,株行距1.5 m×1.5 m。种源分别来自宁夏的六盘山,青海的西宁和新疆的天山西部,内蒙古的绰尔和莫尔道嘎,辽宁的草河口、桓仁、清源,吉林的汪清、辉南和露水河,黑龙江的小北湖、东方红、乌伊岭、凉水和帽儿山。每个种源包括15个家系。
1.2 方法 1.2.1 性状测定对每个区组内的每个家系,随机挑选10株树测量胸径及树高,并取胸径处的木芯用来测量纤维素含量,以5个区组平均值作为家系各性状值。根据测定的单株树高和胸径值,按照张广材岭地区白桦二元材积表计算单株材积,为了分析数据时统一小数位数,材积计算结果均×100作为每个单株材积大小。按屈维均(1990)的方法测量单株纤维素含量,纤维素含量经反正弦转化后进行Z分数转换,转换公式:Z=(x-μ)/σ,随后对于分数Z进行T分数线性转换,转换公式:T=10Z+50。
1.2.2 抽样比例设定5%,10%和15%抽样比例。
1.2.3 遗传距离计算遗传距离的计算分别采用马氏距离和欧氏距离。
1.2.4 聚类方法分别采用最短距离法、最长距离法、中间距离法、重心法、类平均法、加权配对算术平均法、可变法和离差平方和法8种系统聚类方法进行家系聚类。
1.2.5 抽样策略按胡晋等(2000)和徐海明等(2000)提出的多次聚类随机抽样法、多次聚类优先抽样法和多次聚类偏离度抽样法进行组内抽样。
1.2.6 核心材料的评价通过以下4个指标评价不同方法构建的白桦核心种质代表性,其分别是:均值差异百分率(mean difference percentage, MD),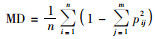
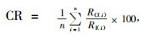
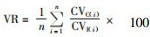
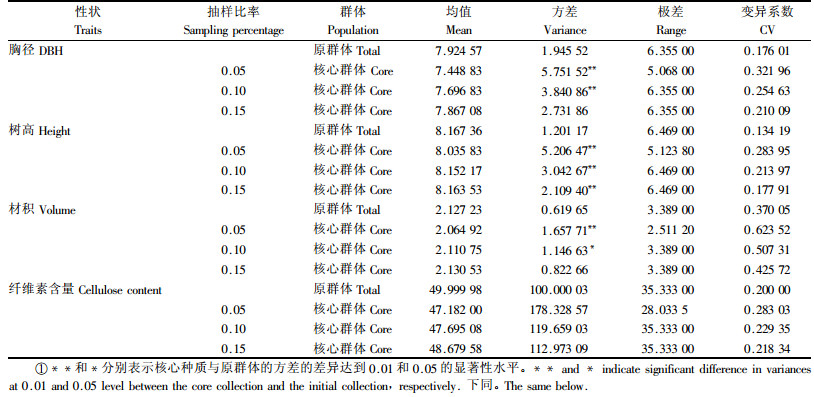
在采用欧氏距离、优先取样法和最短距离系统聚类法的前提下,抽样比例分别为5%,10%和15%的白桦核心种质遗传差异见表 1。结果表明,3种抽样比例下,所有核心种质与原有群体间的各性状均值无显著差异,MD为0。10%和15%抽样比例下,各性状CR为100%,而5%的抽样比例时,仅为75%,低于核心种质CR最低80%的标准。因此,5%的抽样比例明显不符合要求。在10%的抽样比例下,核心种质的胸径和树高与原群体的差异极显著、材积差异显著、纤维素含量差异不显著,而在15%抽样比例下,除树高性状外,核心种质其他3个性状与原群体均无显著差异。10%和15%的抽样比例下,核心种质与原群体性状的VD分别为75%和25%,VR分别为138.97%和119.04%,说明10%的抽样比例在保持了原群体的遗传变异前提下,极大地提高了各性状的方差和极差,是构建白桦核心种质的合理抽样比例。
|
|
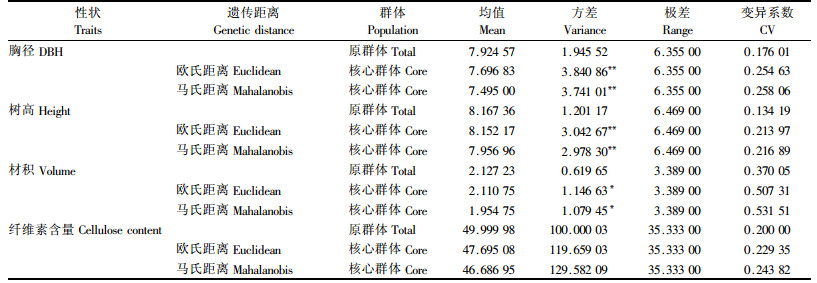
在确定抽样比例为10%,最短距离聚类和优先取样的情况下,采用马氏距离和欧氏距离构建白桦核心种质与原群体的遗传差异(表 2)。结果表明,采用2种遗传距离构建的2个白桦核心种质4个性状MD为0,CR为100%,即核心种质完全保持了原群体的均值和极差,表明2种遗传距离构建的白桦核心种质均能保持原群体的遗传变异和遗传结构。虽然构建的2个核心种质与原始群体之间各性状的VD均达到75%、胸径和树高差异均极显著、材积差异均显著、纤维素含量均不显著,但采用欧氏距离构建的白桦核心种质各性状的VR为138.97%,而采用马氏距离构建的核心种质VR为143.45%,而且采用欧氏距离的胸径、树高和材积的方差只略大于马氏距离,而纤维素含量方差则明显小于马氏距离。由此可见,在构建白桦核心种质时马氏距离稍优于欧氏距离。
|
|
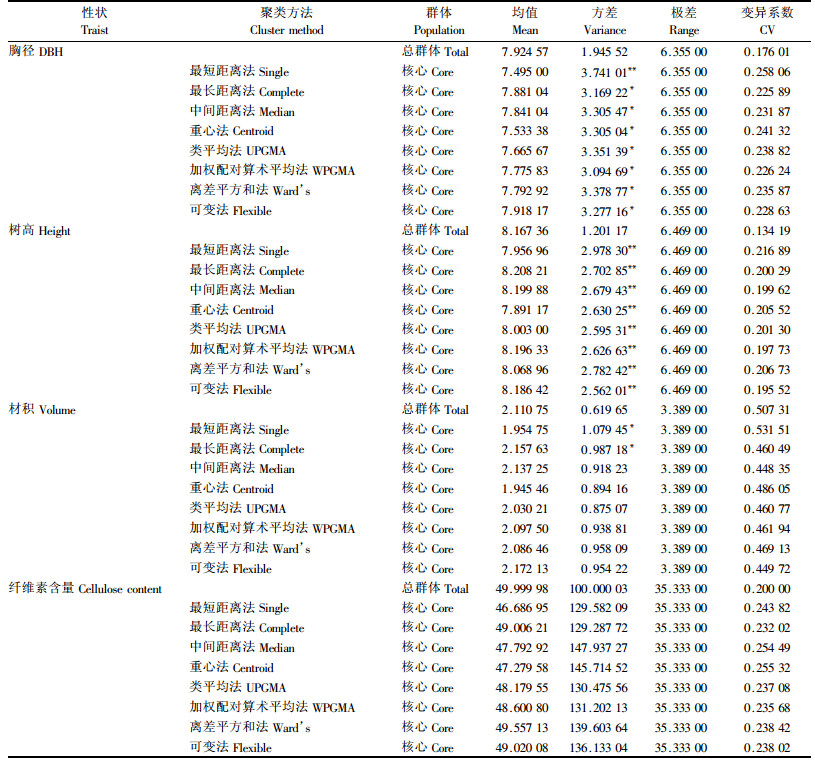
在确定马氏距离、优先取样法和10%抽样比率下,采用8种不同系统聚类方法构建的白桦核心种质遗传差异见表 3。结果表明,采用8种聚类方法分别构建的白桦核心种质与原有群体间均值无显著差异,而且极差相同,即MD为0,CR为100%,说明这8种聚类方法均能保存原群体的遗传变异与遗传结构。8种聚类方法构建的核心种质树高和胸径方差均极显著地高于原群体的方差,但纤维素含量方差与原群体差异不显著;材积性状只有最短距离与最长距离法构建的核心种质方差显著高于群体,其余方法构建的核心种质与原群体间差异均不显著;最短距离法、最长距离法、中间距离法、重心法、不加权类平均法、可变类平均法、可变法和离差平方和法构建的核心种质与原群体4个性状的VD与VR依次为75.00%和143.45%,25.00%和129.52%,25.00%和132.23%,25.00%和137.32%,50.00%和132.19%,25.00%和129.64%,25.00%和129.034%,50.0%和133.51%,其中,最短距离法构建的白桦核心种质的VD和VR最大,其次为离差平方和法和加权平均法。以上分析表明,采用最短距离多次聚类法构建的核心种质在保持了原群体遗传变异与遗传结构的同时,使各性状的方差和变异系数均得到极大提高,是构建白桦核心种质的最佳聚类方法。
|
|
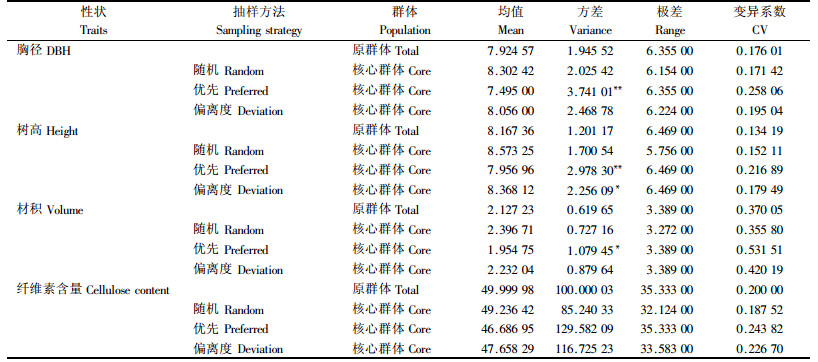
在确定采用马氏遗传距离、最短距离聚类法和10%的抽样比率下,3种抽样策略构建的白桦核心种质遗传差异见表 4。结果表明,3种抽样方法构建的白桦核心种质的均值都与原群体没有显著差异、MD为0,在极差指标上,优先取样法与原群体CR为100%,其他2种方法均低于原群体。采用多次聚类随机取样法,核心群体的4个性状的VD仅为25%,与原群体均无显著差异,CR和VR分别为93.32%和100.16%。采用多次聚类偏离度取样时,除树高外,其余3个性状与原群体均无显著差异,4个性状的VD,CR和VR分别为25%,98.25%和117.86%。采用多次聚类优先取样法时,白桦核心种质与原群体的胸径和树高存在极显著差异,材积存在显著差异,4个性状的VD,CR和VR分别为75%,100.00%和143.45%。以上分析表明,优先取样法构建的核心种质具有较大的遗传变异,偏离度取样法次之,随机取样法最差。
|
|
确定合理的抽样比例是构建核心种质的重要环节,过高的抽样比例可能会包含冗余较高的样品,反之则会导致重要核心种质材料的丧失。目前,多数植物核心种质的抽样比例为全部收集种质的5%~30%,一般为10%左右(李自超等,2000;2003;Brown, 1989a;1989b)。如Upadhyaya等(2001)在构建鹰嘴豆(Cicer arietinum)核心种质时,抽样比例约为10%。Zewdie等(2004)构建的高粱(Sorghum bicolor)核心种质也采用了10%的取样比例。王丽侠等(2004)构建的长江春大豆(Glycine max)核心种质只占原始群体的8.58%。徐海明等(2004)构建的棉花(Gossypium hirsutum)核心种质,抽样比例高达30%。尽管目前并无一个确定的抽样比例,但核心种质代表性的评价参数却能一致。如胡晋等(2000)和徐海明等(2004)提出的以VD,MD,CR和VR作为核心种质评价参数,可以用作确定核心种质抽样比例的依据。一旦选择了评价参数,在一定程度上就确定了核心种质的抽样比例。本研究在以MD小于20%、CR大于80%为最低标准的情况下发现:5%的抽样比例时,各性状CR均低于80%;而15%的抽样比例下,虽然各性状的CR为100%,但VD仅为25%。因此,确定了10%的抽样比例,这样既保证了各性状的CR为100%,也使VD提高到75%。
如何准确地评价种质资源的相似性是合理构建核心种质的前提。目前,在核心种质构建过程中,一般采用欧氏距离计算群体的相似性,然后利用1种或多种聚类方法进行分组。例如,李自超等(2000)在云南地方稻(Oryza)核心种质取样方案研究中就采用了欧氏距离结合类平均法分组取样。余萍等(2003)在研究中国普通野生稻初级核心种质的取样策略时,也采用了欧氏距离结合类平均法。然而,欧氏距离计算方法不仅受不同性状量纲的影响,而且无法排除性状间存在的相关性。而马氏距离则不受性状纲量的影响,而且能顾及各性状间的相关性,是评价材料间相似性的理想方法。如徐海明等(2004)在棉花核心种质构建研究中就发现马氏距离优于欧氏距离。本项研究中,马氏距离与欧氏距离构建的核心种质在保持原群体遗传变异的同时均能使性状方差极大提高,但马氏距离构建的初级核心种质VR达143.45%,而欧氏距离核心种质VR为138.97%。
物种的遗传多样性不是随机分布在群体中,而是有一定的组织和结构(Balakrishnan et al., 2000),因此在构建核心种质时不能随机取样,而应先对群体进行分组,然后在不同组内采用不同方法抽取核心材料组成核心子集整合成核心种质(Frankel et al., 1984)。到目前为止,将种质资源组分成亚组构建核心子集最常用的方法是聚类分析(李自超等,2000;孙传清等,2001;余萍等,2003)。然而,不同聚类方法会产生不同的分组,进而影响核心种质的代表性。如Zhang等(2000)构建芝麻(Sesamum indicum)核心种质时,比较了8种聚类方法的构建效果后认为离差平方和聚类法效果最好。Hu等(2000)在构建棉花核心种质研究中,结合马氏距离比较了3种聚类方法,结果表明不加权类平均法和离差平方和法效果较好。Li等(2004)在构建水稻核心种质的过程中发现,不加权类平均法是6种常用的聚类方法中构建效果最好的方法。但在本研究中,最短距离法构建的白桦初级核心种质能极显著地增加性状的方差和变异系数,比类平均法更能使核心种质的VD和VR值提高。
构建核心种质的方法是先对样品进行遗传分类,根据分类结果采用一定的抽样方法抽取核心材料。目前,构建核心种质的抽样方法主要有随机抽样、按常数抽样、按比率抽样、按对数抽样和按遗传变异度抽样等(Brown, 1989b)。Yonezawa等(1995)评价了上述方法后,认为按遗传变异抽样的效果最好,其次是按比率抽样法,最差为随机抽样法。Hu等(2000)先后提出采用多次聚类随机取样、多次聚类优先取样和多次聚类偏离度取样构建核心种质的策略,并比较了这3种抽样方法的优劣,结果表明用优先取样法和变异度取样法得到的核心种质比用随机取样法得到的核心种质更能代表原种质群体的遗传多样性。本项研究表明,在确定10%的抽样比例、马氏距离和最短距离聚类时,这3种方法构建的核心种质各性状均值与原群体无显著差异、CR超过80%、变异系数均大于原群体,其中多次聚类优先取样法能极大地提高核心种质VD和VR值,是白桦初级核心种质构建中的最优抽样方法。
虽然性状数目是影响核心种质构建的重要因素,但许多研究表明,构建核心种质时采用的性状数目并不是越多越好。当取样比例较小时,适当增多性状数可以增加群体的代表性,也能更准确地计算种质材料间遗传距离;反之,可适当减少。如Basigalup等(1995)在苜蓿(Medicago satiya)核心种质构建中发现,用主成分比采用全部数据构建的核心种质更有效。Malosetti等(2001)采用17个性状的数据主成分分析后,采用前11主成分构建玉米(Zea mays)核心种质。Upadhyaya等(2006)先将龙爪稷(Oryza sativa)的14个数量性状进行主成分分析,然后取前5个主成分性状结合聚类分析进行分组抽取核心样品。林木体积大、生存环境复杂,种质资源管理较为困难,实际工作中取样比例较小,构建核心种质需较多的性状。因此,对本项研究中通过4个性状初步构建的白桦核心种质有必要通过增加性状数目和采用分子标记方法对其做进一步的分析和评价。
顾万春, 李文英. 2007. 我国林木种质资源共享现状及建议[J]. 世界林业研究, 20(1): 66-69. DOI:10.3969/j.issn.1001-4241.2007.01.012 |
顾万春. 2005. 中国林木种质资源本底现状与保护对策//薛达元. 中国生物遗传资源现状与保护. 北京: 中国环境出版社, 148-194.
|
胡晋, 徐海明, 朱军. 2000. 基因型值多次聚类法构建作物种质资源核心库[J]. 生物数学学报, 15(1): 103-109. DOI:10.3969/j.issn.1001-9626.2000.01.016 |
贾继增, 张启发. 2001. 为第二次"绿色革命"发掘基因资源[J]. 中国基础科学, (7): 4-8. DOI:10.3969/j.issn.1009-2412.2001.07.001 |
李娟, 江昌俊. 2004. 中国茶树核心种质的初步构建[J]. 安徽农业大学学报, 31(3): 282-287. DOI:10.3969/j.issn.1672-352X.2004.03.007 |
李天红. 2005. 桃遗传资源核心种质的研究[J]. 园艺园林科学, 21(8): 296-298. |
李自超, 张洪亮, 曾亚文, 等. 2000. 云南地方稻种资源核心种质取样方案研究[J]. 中国农业科学, 33(5): 1-7. DOI:10.3321/j.issn:0578-1752.2000.05.001 |
李自超, 张洪亮, 曹永生, 等. 2003. 中国地方稻种资源初级核心种质取样策略研究[J]. 作物学报, 29(1): 20-24. DOI:10.3321/j.issn:0496-3490.2003.01.004 |
屈维均. 1990. 制浆造纸实验[M]. 北京: 中国轻工业出版社.
|
宋丛文, 包满珠. 2005. 珙桐种质资源保存样本策略的研究[J]. 植物生态学报, 29(3): 422-428. DOI:10.3321/j.issn:1005-264X.2005.03.012 |
孙传清, 李白超, 王象坤. 2001. 普通野生稻和亚洲栽培稻核心种质遗传多样性的检测研究[J]. 作物学报, 27(3): 313-318. DOI:10.3321/j.issn:0496-3490.2001.03.007 |
王丽侠, 李英慧, 李伟, 等. 2004. 长江春大豆核心种质构建及分析[J]. 生物多样性, 12(6): 578-585. DOI:10.3321/j.issn:1005-0094.2004.06.003 |
徐海明, 胡晋, 朱军. 2000. 构建作物种质资源核心库的一种有效抽样方法[J]. 作物学报, 26(2): 157-162. DOI:10.3321/j.issn:0496-3490.2000.02.005 |
徐海明, 邱英雄, 胡晋, 等. 2004. 不同遗传距离聚类和抽样方法构建作物核心种质的比较[J]. 作物学报, 30(9): 932-936. DOI:10.3321/j.issn:0496-3490.2004.09.016 |
余萍, 李白超, 张洪亮, 等. 2003. 中国普通野生稻初级核心种质取样策略[J]. 中国农业大学学报, 8(5): 37-41. DOI:10.3321/j.issn:1007-4333.2003.05.010 |
赵冰, 张启翔. 2007. 中国蜡梅种质资源核心种质的初步构建[J]. 北京林业大学学报, 29(增刊1): 54-60. |
郑万钧. 1985. 中国树木志[M]. 北京: 中国林业出版社.
|
Balakrishnan R, Nair N V, Screenivasan T V. 2000. A method for establishing a core collection of Saccharum offtcinarum L.germplasm based on quantitative-morphological data[J]. Genetic Resources and Crop Evolution, 47: 1-9. DOI:10.1023/A:1008780526154 |
Basigalup D H, Barnes D K, Stucker R E. 1995. Development of a core collection for perennial Medicago plant introductions[J]. Crop Science, 35: 1163-1168. DOI:10.2135/cropsci1995.0011183X003500040042x |
Brown A H D. 1989a. The case for core collections//Brown A H D, Frankel O H, Marshall R D, et al. The Use of Plant Genetic Resources. Cambridge: Cambridge University Press.
|
Brown A H D. 1989b. Core collection:A practical approach to genetic resources management[J]. Genome, 31: 818-824. DOI:10.1139/g89-144 |
Frankel O H, Brown A H D. 1984. Current plant genetic resources—a critical appraisal//Genetics: new frontiers (Vol Ⅳ). New Delhi: Oxford and IBH Publishing Co., 1-11.
|
Gepts P. 2006. Plant genetic resources conservation and utilization:the accomplishments and future of a societal insurance policy[J]. Crop Science, 46: 2278-2292. DOI:10.2135/cropsci2006.03.0169gas |
Hu J, Zhu J, Xu H M. 2000. Methods of constructing core collection by stepwise cluster with three sampling strategies based on genotypic values of crops[J]. Theoretical and Applied Genetics, 101: 264-268. DOI:10.1007/s001220051478 |
Lerceteau E, Robert T, Petiard V, et al. 1997. Evaluation of the extent of genetic variability among Theobroma cacao accessions using RAPD and RFLP markers[J]. Theoretical and Applied Genetics, 95: 10-19. DOI:10.1007/s001220050527 |
Li C T, Shi C H, Wu J Q, et al. 2004. Methods of developing core collections based on the predicted genotypic value of rice (Oryza sativa L.)[J]. Theoretical and Applied Genetics, 108: 1272-1176. |
Malosetti M, Abadie T. 2001. Sampling strategy to develop a core collection of Uruguayan maize landraces based on morphological traits[J]. Genetic Resources and Crop Evolution, 48: 381-390. DOI:10.1023/A:1012003611371 |
Meike S A, Michael P, Rainer S K, et al. 2006. Molecular characterization of a collection of the tropical multipurpose shrub legume Flemingia macrophylla[J]. Agroforest Systems, 68: 231-245. DOI:10.1007/s10457-006-9014-9 |
Milene S, Mirian P M, Maria B S, et al. 2008. Genetic diversity of a coffea germplasm collection assessed by RAPD markers[J]. Genetic Resources and Crop Evolution, 55: 901-910. DOI:10.1007/s10722-007-9295-5 |
Upadhyaya H D, Gowda C L L, Pundir R P S, et al. 2006. Development of core subset of finger millet germplasm using geographical origin and data on 14 quantitative traits[J]. Genetic Resources and Crop Evolution, 53: 679-685. DOI:10.1007/s10722-004-3228-3 |
Upadhyaya H D, Ortiz R. 2001. A mini core subset for capturing diversity and promoting utilization of chickpea genetic resources in crop improvement[J]. Theoretical and Applied Genetics, 102: 1292-1298. |
Wunschl A, Carrera1 M, Hormaza J I. 2006. Molecular characterization of local Spanish peach [Prunus persica(L.)Batsch] germplasm[J]. Genetic Resources and Crop Evolution, 53: 925-932. DOI:10.1007/s10722-004-6697-5 |
Yonezawa K, Nomura T, Morishima H. 1995. Sampling strategies for use in stratified germplasm collections//Hodgkin T, Brown A H D, Vanhintum Th J L, et al. Core collections of plant genetic resoures. Chichester, Published by John Wiley & Sons.
|
Zhang X, Zhao Y, Cheng Y, et al. 2000. Establishment of sesame germplasm core collection in China[J]. Genetic Resources and Crop Evolution, 47: 273-279. DOI:10.1023/A:1008767307675 |
Zewdie Y, Tong N K, Bosland P. 2004. Establishing a core collection of Capsicum using a cluster analysis with enlightened selection of accessions[J]. Genetic Resources and Crop Evolution, 51: 147-151. DOI:10.1023/B:GRES.0000020858.96226.38 |
 2009, Vol. 45
2009, Vol. 45