文章信息
- 齐刚, 苏智先, 李劲涛, 阮期平.
- Qi Gang, Su Zhixian, Li Jingtao, Ruan Qiping
- 休眠期珙桐种子cDNA文库构建及EST分析
- Construction of cDNA Library and Analysis of the Expressed Sequenced Tags (ESTs) Characteristics of Dormant Seeds of Davidia involucrata
- 林业科学, 2009, 45(10): 69-73.
- Scientia Silvae Sinicae, 2009, 45(10): 69-73.
-
文章历史
- 收稿日期:2008-08-06
-
作者相关文章
2. 四川省生态学与环境保护技术重点实验室 绵阳 621000;
3. 绵阳师范学院分子生物学与生物制药重点实验室 绵阳 621000
2. Key Laboratory of Ecology and Environmental Protection Technology of Sichuan Province Mianyang 621000;
3. Key Laboratory for Molecular Biology and Biopharmaceuticals of Mianyang Normal University Mianyang 621000
珙桐(Davidia involucrata)落叶乔木,起源古老,系第三纪古热带植物区系的孑遗种,有“活化石”之称,为我国单属种的特有植物,被列为国家一级重点保护植物,世界著名的珍贵稀有观赏植物,被誉为植物界的“大熊猫”(苏智先等,1999;Li et al., 2003)。珙桐天然分布在贵州、湖南、湖北、陕西、四川、云南及甘肃7省,分布范围较窄,资源量较少,仅限于一些边远山区人迹罕至之处(贺金生等,1995)。
珙桐种子的休眠期较长,一般2~3年才萌发,珙桐种子的“高休眠性”是长期适应复杂环境而形成的生理生态特性。为了研究其休眠的机制,迄今为止,已从果实形态结构(万朝琨,1988;陶金川等,1993;罗世家,2002)、果皮化学组成(戴大临等,1995)、次生代谢物质(李卓杰等,1989;陈坤荣等,1998;雷泞菲等,2003;董社琴等,2004;万斌等,2005)、组织培养(毕世荣等,1983;Sato,1996;罗世家等,2003;夏晗等,2003)等多种角度对珙桐种子的休眠及催芽问题进行了大量的试验研究;然而在分子层面上即蛋白质和基因方面的研究却是空白。本研究以休眠期的珙桐种子为材料,构建国内外第1个珙桐种子cDNA文库,进行表达序列标签(EST)分析,以期研究其适应环境的相关的功能蛋白及基因,并为从分子水平上研究其机制提供依据。
1 材料与方法 1.1 材料珙桐种子于2007年12月采自四川卧龙自然保护区三江镇鹿尔坪村的白泥岗,去果肉于室温保存。
1.2 试剂使用SMARTTM cDNA Library Construction Kit(购自Clontech公司)进行文库构建;载体为 λ TriplEx2,菌株为 Escherichia coli XL1-Blue(购自Clontech公司),包装蛋白Packagene Lambda DNA Packaging System(购自Promega公司)。其他试剂均为国产或进口分析纯。
1.3 方法 1.3.1 珙桐种子总RNA提取采用改良的LiCl法提取总RNA(Li et al., 2003)。取60 mg珙桐种子于盛有液氮研钵中速冻10 min后,加入4%PVP,研磨至粉末并转移至一盛有900 μL预冷的抽提缓冲液(8 mol·L-1 LiCl,在使用前加入已于-20 ℃下预冷的4% β-巯基乙醇,10 mmol·L-1 半胱氨酸和2%可溶性PVP,28%无水乙醇) 1.5 mL离心管中,充分混匀后4 ℃下过夜。取出过夜后的离心管瞬时离心(约5 s)。将上清液转移至另一干净的1.5 mL离心管中,离心30 min,弃上清液。用70%乙醇(-20 ℃)洗沉淀,风干约10 min,把沉淀溶于650 μL溶解液中(1.4% SDS,150 mmol·L-1 NaCl,50 mmol·L-1 EDTA,10 mmol·L-1 Tris-HCl,pH7.5,2% β-巯基乙醇)。依次用等体积的Tris饱和酚(pH7.8),苯酚:氯仿:异戊醇(25:24:1),氯仿:异戊醇(24:1)和氯仿抽提。每次抽提前,将有机相和RNA溶液充分混匀后离心15 min,取上清液。向最后1次抽提的上清液中加入1/10体积的4 mol·L-1 醋酸铵和2倍体积的冷乙醇(4 ℃),-20 ℃下沉淀30 min。混合物离心30 min,去上清液,向沉淀加入250 μL 4 mol·L-1 醋酸铵沉淀RNA。混合物离心10 min,用75%和80%冷乙醇洗沉淀,风干沉淀后溶于20 μL水中。上述离心条件均为4 ℃,13 000 r·min-1。取1 μL样品用于分光光度计纯度和浓度检测,取3 μL用于琼脂糖凝胶电泳分析。
1.3.2 cDNA文库构建根据SMARTTM cDNA Library Construction Kit实验指导说明构建cDNA文库。取3 μL RNA为模板,用CDSⅢ/3′ PCR Primer和SMART IVTM Oligonucleotide,逆转录合成单链cDNA。再用5′ PCR Primer和CDSⅢ/3′ PCR Primer通过引物延伸合成双链cDNA。然后经过蛋白酶K消化,SfiⅠ酶切,再经Chroma Spin-400柱洗脱,分离并收集大于500 bp cDNA片段。该片段与载体λTriplEx2的左右臂连接后,使用Packagene Lambda DNA Packaging System进行体外包装,扩增后所得噬菌体裂解液分装于1.5 mL离心管,于-80 ℃保存,即为构建的珙桐种子的cDNA文库。
1.3.3 文库的扩增和质量检测从cDNA文库中取出5 μL噬菌体裂解液,按照1:10稀释,然后取1 μL与200 μL用10 mmol·L-1 MgSO4 重悬的过夜培养的 E. coli XL1-Blue混合,37 ℃吸附15 min,与2 mL融化的上层琼脂混匀,倾注在37 ℃预热的(LB/MgSO4)平板上,37 ℃培养6~10 h,根据噬菌斑个数,计算文库滴度。根据cDNA文库滴度,取适量噬菌体裂解液加到含有IPTG(50 μg·mL-1)和X-gal (50 μg·mL-1)的上层琼脂中,37 ℃培养8~12 h,计数平板上形成的蓝斑和白斑数目,计算噬菌体的重组率。
1.3.4 cDNA文库插入片段检测随机挑取噬菌斑,溶于100 μL 1×λ 稀释液(MgCl2 0.01 mol·L-1, Tris-HCl pH 7.8,0.035 mol·L-1,明胶0.01%,去离子水0.01 mol·L-1)。取5 μL作为模板,使用 λTriplEx2 Kit提供的5′-和3′-端筛选引物进行PCR扩增,用1%琼脂糖凝胶电泳检测PCR片段的大小,即可检测出cDNA文库中插入DNA片段的长度大小。
1.3.5 文库转化用经PCR检测含有阳性克隆的噬菌体转化 E. coli BM25.8,在Cre重组酶的作用下,重组噬菌体DNA环化成质粒,提取质粒,转化 E. coli DH5α,保存转化子。
1.3.6 序列测定及分析以上游引物(5′-CTCCGAGATCTGGACGAGC-3′), 下游引物(5′-TAATACGACTCACTATAGGG-3′)送至上海英骏生物有限公司测序。借助生物软件DNASTAR和Bio Edit, 将测序所得到的序列进行拼接,去除载体、引物序列后,分别使用BlastN和BlastX检索National Center for Biotechnology Information (NCBI)的核苷酸和蛋白质数据库,进行序列的同源性分析。
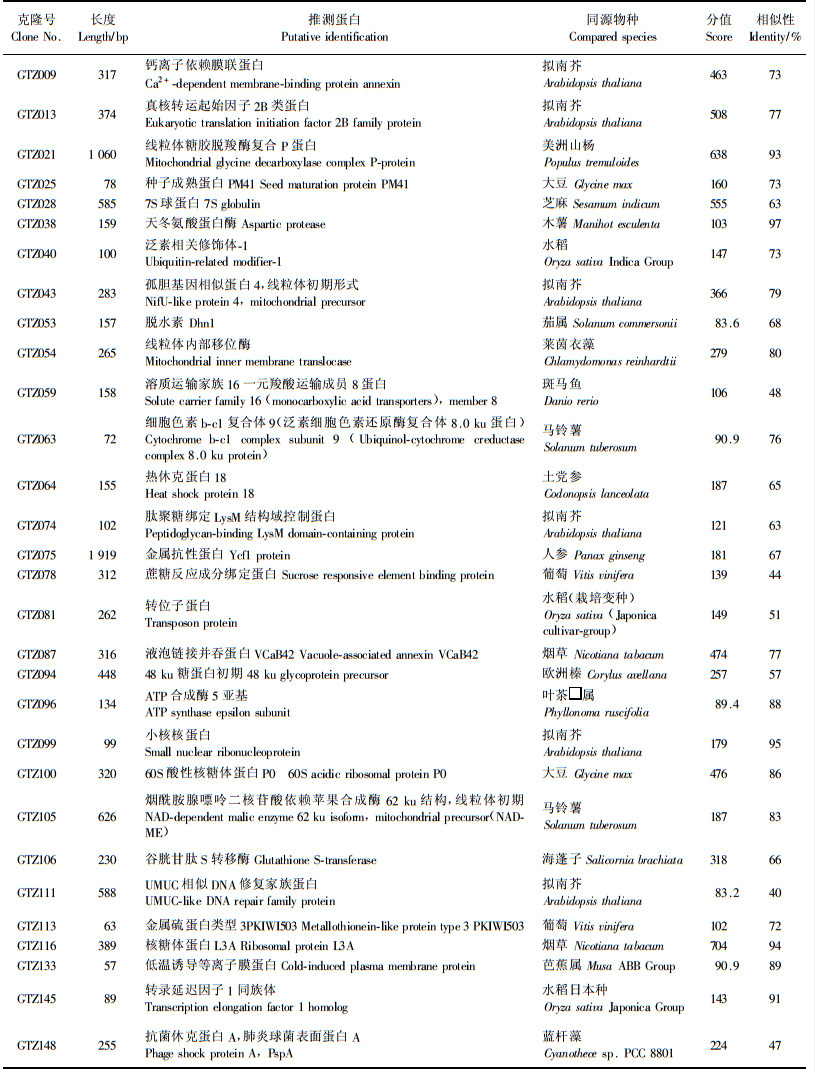
2 结果与分析 2.1 珙桐种子总RNA质量检测高质量的RNA是构建cDNA文库的关键,取3 μL总RNA进行1.1%的琼脂糖凝胶电泳检测(图 1),表明28S和18S rRNA电泳条带完整清晰,28S条带亮度约为18S的2倍。取1 μL样品用于分光光度计检测,测得OD260 nm/OD280 nm=2.0,OD260 nm/OD230 nm=2.1,说明所提取的总RNA质量较高,可适用于cDNA文库的构建。
|
图 1 珙桐种子总RNA Figure 1 Total RNA of Davidia involucrata seeds |
将具有黏性末端的cDNA与载体 λTriplEx2连接,蛋白包装后转化大肠杆菌 E. coli XL1-Blue。在含有IPTG和X-Gal的平板上统计噬菌斑数。计算得出,所建cDNA文库滴度为1.5×106 pfu·mL-1,经蓝斑和白斑计数,cDNA文库的重组率达到98%。
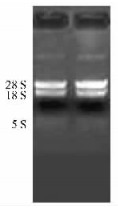
随机挑取白色噬菌斑10个,利用PCR技术检测插入DNA片段的大小。结果表明,插入DNA片段主要集中在0.5~1.0 kb之间(图 2)。

|
图 2 电泳检测经PCR扩增出的克隆子插入DNA片段大小 Figure 2 Gelelectrophoresis of PCR amplification of the DNA fragments inserted in plasmids from Davidia involucrata seeds cDNA library M:DL2000 marker; 1-10:经PCR扩增出质粒中DNA插入片段PCR amplification of the DNA fragments inserted in plasmids. |
随机挑取150个噬菌斑,测序获得148个EST序列,经过EST序列有效拼接后共获得127个EST序列,占总EST的85.8%,序列长度为932~1 891 bp,平均长度为1 462 bp。利用NCBI的Vec Screen程序识别并去除载体信息的序列,去除小于100 bp的序列7条,占全部测序量的4.7%。经过拼接和去除载体序列后,获得有效序列120条,占总EST的81.1%,序列长度为103~1 718 bp,平均长度为523 bp。
通过BlastX和BlastN软件120条有效EST序列分别在NCBI上的非冗余蛋白质数据库以及核酸序列数据库中进行搜索分析,结果表明,共有75条EST序列在蛋白质或者核苷酸水平上具有同源序列,占总数的62.5%,其中22条为功能未知蛋白,占18.3%,有32条EST序列通过比对相似性较低,占19.2%,13条EST序列无匹配,占18.3%(表 1)。
|
|
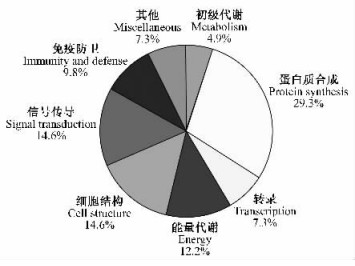
对这53条EST序列按照同源比对结果进行生物学功能分类可大致分为8类(高琪等,2005),包括初级代谢、蛋白质合成、转录、能量代谢、细胞结构、信号传导、免疫防卫和其他类。本文试验结果见图 3。其中蛋白质合成相关的序列所占比例最多(29.3%),其次是信号转导(14.6%)和细胞结构(14.6%)。大多数EST可在模式植物拟南芥(Arabidopsis thaliana)中找到同源序列,与之匹配的功能蛋白中有珙桐种子中富含的多糖蛋白、成熟蛋白,以及种子中与环境适应性相关的功能蛋白。珙桐种子部分功能蛋白对比结果见表 1。
|
图 3 53个珙桐种子EST序列的生物学功能分类 Figure 3 Functional classification of 53 ESTs of Davidia involucrata seed |
提取高质量的RNA,是构建cDNA文库的基础和关键。由于珙桐种子中含大量的多糖和多酚,不易获得高纯度高产量的RNA,本研究对LiCl法提取总RNA进行改进获得了高质量的总RNA,完全适用于cDNA文库构建的要求。文库的滴度、重组率及插入片段的大小是鉴定cDNA文库质量的重要指标。一般来说,文库的滴度能达到105 pfu·mL-1以上即为有效的文库(Li et al., 1998)。本研究构建的珙桐种子cDNA文库滴度为1.5×106 pfu·mL-1,重组率为98%,说明该文库质量较高。
EST技术被认为是一种从基因文库中快速获得基因组中的未知特征基因表达和编码序列的方法(Adams et al., 1991;周祥明等,2006)。本试验所获得的EST序列能够用于获取其全长cDNA,进而获得其全长基因序列以用于功能研究。通过对珙桐种子cDNA文库的EST测序及序列比对分析,有37.5%EST序列在数据库中的相似性较低或无匹配,可能是新基因,44.2%EST序列同源性较高,得到了一些功能基因。
通过分析得到了珙桐种子与环境相适应功能蛋白的EST序列,如热休克蛋白(heat shock protein)、脱水素(dhn1)、低温诱导等离子体膜蛋白(cold-induced plasma membrane protein)、转录延伸因子-1(transcription elongation factor 1 homolog)等。这些蛋白都在珙桐生长的高海拔、低温湿润的环境中起着重要作用,也是珙桐种子适应其环境的重要蛋白。热休克蛋白是生物的整体、组织、细胞等当其生活的温度范围急剧地从低温移向高温时,可显著地促进合成的一组蛋白质,在细胞生命活动中起重要作用,部分热休克蛋白还与细胞防脱水蛋白存在序列上的相似性,这能减轻冷胁迫对膜的损伤,减少溶质渗漏,抑制细胞脱水,从而增强了组织的抗冷性。郭九峰等(2007)在沙冬青(Ammopiptanthus mongolicus)cDNA建库和EST分析中对热休克蛋白进行了分析,表明其对沙冬青抗冻性的作用。脱水素具有高度的亲水性,它能够在植物胚胎发育后期以及处于干旱、低温、盐碱等逆境的植株中大量表达。当植物细胞氧代谢的平衡在低温等逆境中破坏时,它可以清除活性氧自由基的产生,保护细胞膜系统。近年来关于脱水素的研究发展很快,特别是逆境下脱水素对植物的保护作用引起了人们广泛的关注(郭九峰等,2007)。低温诱导等离子体膜蛋白,是在珙桐受到低温胁迫时,能直接保护细胞免受胁迫伤害的功能蛋白。转录延伸因子-1是一种在细胞内普遍存在且大量表达的多聚核糖体蛋白,在抗低温和抗紫外线等方面可能也起着关键的作用(赵嘉庆等,2005)。本研究还得到了种子成熟蛋白(seed maturation protein)、泛醇-细胞色素还原酶(cytochrome b-c1 complex subunit 9)等,这些为今后珙桐种子的进一步研究提供基础。
由于珙桐是一古老物种,且资源越来越少,种子不易萌发,已成为珍稀物种,对珙桐的保护尤为重要。本研究通过构建珙桐种子的cDNA文库,建立起了其基因资源,对今后研究珙桐的系统演化、种子休眠萌发及适应环境等提供了宝贵的资源和分子生物学依据。本研究通过ESTs测序和与已知功能基因蛋白同源性分析,对珙桐种子的特性以及与适应环境相关的功能蛋白进行了初步的探讨,为今后进一步研究其功能蛋白的特性机理以及基因的克隆表达调控奠定了基础。
毕世荣, 何立明, 孔凡伦, 等. 1983. 珙桐组织培养[J]. 植物生理学通讯, (4): 43-44. |
陈坤荣, 陈玉惠, 田广红, 等. 1998. 珙桐种子层积期间过氧化物酶同工酶的变化[J]. 西南林学院学报, 18(3): 143-147. DOI:10.11929/j.issn.2095-1914.1998.03.002 |
戴大临, 文艺, 施寒梅. 1995. 珙桐果皮中镍、钙元素的EDS分析[J]. 电子显微学报, (2): 99-103. |
董社琴, 李冰雯, 王爱荣. 2004. 植物生长调节剂对珙桐种胚离体培养的影响[J]. 湖北农学院学报, 24(4): 291-293. |
高琪, 逄越, 吴毓, 等. 2005. 日本七鳃鳗(Lampetra japonica)口腔腺表达序列标签(EST)分析[J]. 遗传学报, 32(10): 1045-1052. |
郭九峰, 孙国琴, 沈传进, 等. 2007. 沙冬青cDNA文库的构建和EST分析[J]. 华北农学报, 22(4): 37-41. |
贺金生, 林洁, 陈伟烈. 1995. 我国珍稀特有植物珙桐的现状及其保护[J]. 生物多样性, 3(4): 213-221. DOI:10.3321/j.issn:1005-0094.1995.04.005 |
雷泞菲, 苏智先, 陈劲松, 等. 2003. 珍稀濒危植物珙桐果实中的萌发抑制物质[J]. 应用与环境生物学报, 9(6): 607-610. DOI:10.3321/j.issn:1006-687X.2003.06.010 |
李卓杰, 陈润政, 傅家瑞, 等. 1989. 珙桐种子休眠和萌发中酸性磷酸酶同工酶的研究[J]. 西南林学院学报, 9(1): 8-13. DOI:10.11929/j.issn.2095-1914.1989.01.002 |
罗世家. 2002. 珙桐种子的解剖研究[J]. 湖北民族学院学报:自然科学版, 20(4): 18-19. |
罗世家, 周光来, 王建明. 2003. 珙桐芽体组织培养研究[J]. 湖北民族学院学报:自然科学版, 21(4): 11-13. |
苏智先, 张素兰. 1999. 珙桐种群生殖物候及其影响因子研究[J]. 四川师范学院学报:自然科学版, 20(4): 313-318. |
陶金川, 黄致远, 宗世贤. 1993. 珍稀植物幼苗形态初步研究(三)[J]. 江西林业科技, (2): 20-23. |
万斌, 秦帆. 2005. 促进珙桐种子发芽的技术研究[J]. 西南园艺, 33(2): 9. |
万朝琨. 1988. 珙桐种子休眠的解剖学研究[J]. 中南林学院学报, 8(1): 35-39. |
夏晗, 张健, 尚旭岚, 等. 2003. 珙桐初代培养研究[J]. 四川农业大学学报, 21(4): 356-358. DOI:10.3969/j.issn.1000-2650.2003.04.019 |
赵嘉庆, 丁淑琴, 王健, 等. 2005. 细粒棘球中国大陆株线粒体苹果酸脱氢酶基因的克隆和序列分析[J]. 宁夏医学杂志, 27(8): 510-513. DOI:10.3969/j.issn.1001-5949.2005.08.002 |
周祥明, 张冰玉, 苏晓华, 等. 2006. 美洲黑杨雄性花芽cDNA克隆测序及表达序列标签(ESTs)特性分析[J]. 林业科学, 42(11): 37-41. |
Adams M D, Kelley J M, Gocayne J D, et al. 1991. Complementary DNA sequencing: expressed sequence tags and human genome project[J]. Science, 252: 1651-1656. DOI:10.1126/science.2047873 |
Li Taiwu, Xiang Jianhai, Liu Ruiyu. 1998. Construction of cDNA library of shrimp Penaeus chinensis (Crustacea: Decapoda)[J]. Acta Zoological Sinica, 44: 237-238. |
Li Yunxiang. 2003. Cloning, sequence analysis, and prokaryotic expression of cDNA encoding a putative non-specific lipid-transfer protein from the bracts of dovetree (Davidia involucrata Baill.)[J]. Journal of Plant Biology, 46(3): 167-172. DOI:10.1007/BF03030445 |
Li Yunxiang, Chen Li. 2003. Isolation of total RNA from dry seeds of Davidia involucrata[J]. Journal of Southwest China Normal University: Natural Science(西南师范大学学报:自然科学版), 28(1): 108-111. |
Sato T. 1996. Microprogation of hankachinoki (Davidia involucrata)[J]. Journal of the Japanese Forestry Society, 78: 309-311. |
 2009, Vol. 45
2009, Vol. 45