文章信息
- 黄武刚, 程丽莉, 周志军, 刘建立.
- Huang Wugang, Cheng Lili, Zhou Zhijun, Liu Jianli
- 板栗野生居群与栽培品种间叶绿体微卫星遗传差异初探
- Diversity of Chloroplast DNA SSRs in the Wild and Cultivated Chestnuts(Castanea mollissima)
- 林业科学, 2009, 45(10): 62-68.
- Scientia Silvae Sinicae, 2009, 45(10): 62-68.
-
文章历史
- 收稿日期:2009-02-11
-
作者相关文章
2. 昌平林业局 北京 102200;
3. 北京林业大学水土保持与荒漠化教育部重点实验室 北京 100083
2. Changping County Forest Department Beijing 102200;
3. Key Laboratory of Soil and Water Conservation & Desertification Combating of Ministry of Education Beijing Forestry University Beijing 100083
栗属(Castanea)约有10个种,其中板栗(C.mollissima)、茅栗(C. seguinii)及锥栗(C.henryi)原产于中国。在中国的秦岭南麓、长江流域及大巴山地区分布着大面积的野生板栗,在其生长地主要用作栽培栗的砧木(张宇和,1989)。20世纪80年代以来,随着板栗商业化栽培在野生栗分布区域内的快速发展(宣善平,1988;邱国金,1993;沈泉,1993;李英敏等,1995;范启荣,1998;徐凌帆等,2004),板栗野生群体数量逐年下降,对该资源的保护和合理利用已成为中国栗属资源研究的主要内容之一。然而,目前对于野生板栗的了解较少,在其分布范围、遗传多样性及与板栗地方品种群关系等方面的了解仍然十分模糊。此外,有研究认为其分类学地位仍未明确(张宇和等,2005)。
叶绿体SSR是近些年发展起来的分子标记。与核SSR相比, cpSSR既有cpDNA呈单亲遗传模式、几乎不发生重组、进化速率极慢的特点,又有微卫星的高突变特性,已经成为近年发展起来的一种新型高效的分子标记技术,目前广泛用于植物群体遗传分析及系统发育分析研究(陈伯望等,2000;Parducci et al., 2001; Modliszewski et al., 2006)。在板栗群体遗传研究方面,多采用等位酶、RAPD、AFLP、SSR等分子标记方法(黄宏文等,1996;Goulão et al., 2001;Marinoni et al., 2003;肖正东等,2005),鲜见利用cpSSR标记研究板栗遗传多样性的报道。
本研究使用cpSSR对板栗栽培品种及天然野生居群进行分析,从细胞质遗传差异着手探讨板栗遗传多样性以及野生居群与栽培品种群间的关系。
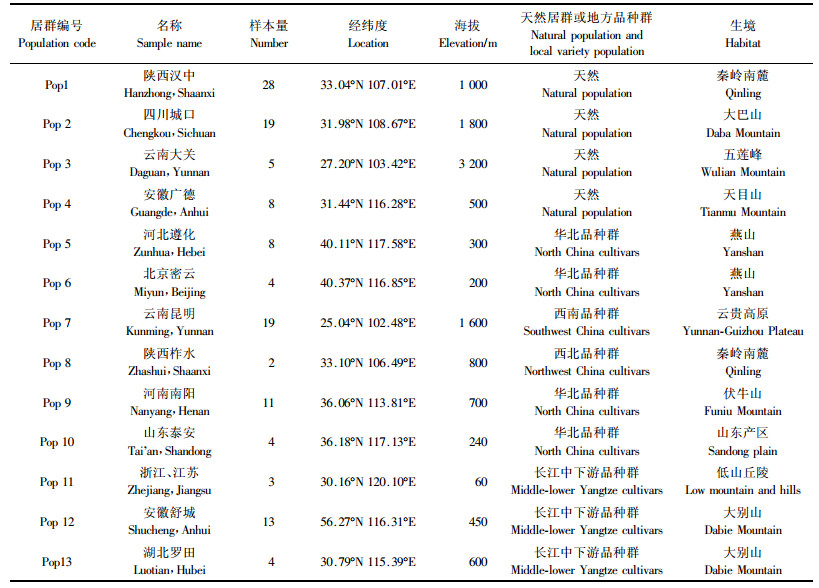
1 材料与方法 1.1 材料试验材料为板栗的128个样本,按不同地域分为13个群体,其中包括9个板栗地方品种群的68个品种和4个天然野生居群的60个个体,其中安徽省的野生板栗样本由安徽省林业科学院提供。
|
|
采用CTAB法对采集到的128个样本的新鲜嫩叶提取高质量的基因组DNA(王艳梅等,2007)。在1%琼脂糖凝胶电泳(北京六一仪器厂,DYY-Ⅲ型)检测DNA的完整性,用紫外分光光度计(Eppendorf Biophotometer 6131)测定DNA的浓度和纯度。
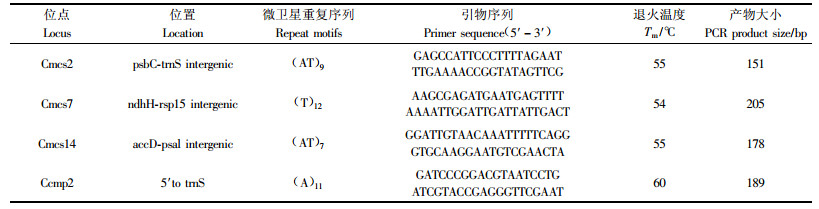
1.2.2 叶绿体微卫星的扩增从已公布的24对叶绿体SSR引物(Weising et al., 1999;Sebastiani et al., 2004)中筛选出4对,引物由上海英俊生物技术公司合成,其序列及目标片段特征见表 2。
|
|
SSR扩增在Biometra公司的T1 Theromcycler PCR仪上进行。25 μL反应体系含有:10 mmol·L-1 Tris-HCl(pH 8.3),1.5 mmol·L-1MgCl2,0.2 mmol·L-1dNTPs,0.2 μmol·L-1引物,1.0 U Taq DNA聚合酶,50~100 ng的模板DNA(上述药品均来自华美生物工程公司)。扩增程序为:94 ℃预变性3 min,94 ℃变性30 s,51~56 ℃复性40 s,72 ℃延伸40 s,共40个循环,最后在72 ℃延伸8 min。
扩增产物用聚丙烯酰胺凝胶电泳检测。在扩增产物中加入等体积的变性载样缓冲液(98%甲酰胺,10 mmol·L-1 EDTA,0.025%溴酚蓝和二甲苯兰),混匀,95 ℃变性5 min后,立即放入冰中,上样量为5 μL,6%变性聚丙烯酰胺凝胶电泳,60 W电泳2.5 h,银染染色。染色方法:乙醇固定5 min,双蒸水漂洗60 s,硝酸氧化5 min,双蒸水漂洗60 s,硝酸银中染色30 min,双蒸水漂洗60s,碳酸钠+400 μL·L-1甲醛+0.2 mg·L-1硫代硫酸钠溶液中显色,显色后用10%乙酸固定。
1.2.3 数据分析按照每个位点不同等位基因的条带大小统计单倍型种类,获得共显性标记数据矩阵,应用POPGENE 1.31软件(Francis et al., 1999)计算等位基因的频率,不同居群间的遗传关系则通过GenAlEx6.1软件(Peakall et al., 2001)获得。
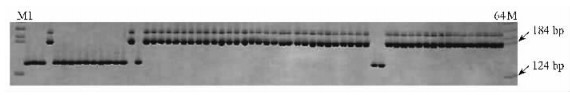
2 结果与分析 2.1 板栗野生居群和地方品种群中的cpSSR单倍型24对cpSSR引物均能从样本中扩增出PCR产物(图 1),片段大小变化在120~240bp之间,但不同引物的扩增效率有一定差异。与核基因组微卫星相比,cpSSR的变异率较低,大部分位点表现为单态,仅有4个位点(Cmcs2,Cmcs7,Cmcs14,Ccmp2)表现出多态性,位点的等位基因数量在2~4个之间,平均为3个(表 3)。
|
图 1 野生板栗样本在位点Cmcs14扩增结果 Figure 1 The allele distribution of the locus cpSSR Cmcs14 in four populations of the wild chestnut |
|
|
通过对4个cpSSR位点的分析,从4个板栗野生居群和9个地方品种群中共组合出8种单倍型,每个野生居群和品种群的单倍型组成和杂合度见表 4。从统计结果中可以看出单倍型种类在各群体中的分布不尽相同, 且在区域分布上也有差异。8个单倍型中,单倍型A出现在板栗2个野生居群(汉中和广德)及全部地方品种群中,共享率最高;单倍型B在汉中野生居群以及河北和山东地方品种群中出现;单倍型C和D存在于3个野生居群中,而其他4个单倍型(E,F,G,H)仅在板栗广德野生居群中出现,为该居群所特有。
|
|
板栗野生居群拥有全部的8个单倍型。其中,汉中居群拥有分布广泛的4种单倍型(A,B,C,D),涵盖了除广德居群外的其他板栗野生居群及地方品种群的几乎所有的单倍型,单倍型多样性为0.671;因拥有4个特异单倍型,广德野生居群的单倍型多样性也高达0.781;城口居群有C和D单倍型,而昭通居群仅有单倍型C,杂合度极低。
板栗4个地方品种群的68个品种仅拥有2个单倍型。华北地方品种群中的河北居群较为特殊,8个品种中7个拥有单倍型B,而山东居群的部分品种也拥有单倍型B;西北品种群、长江中下游品种群和西南品种群的50个品种仅有单倍型A(图 2)。
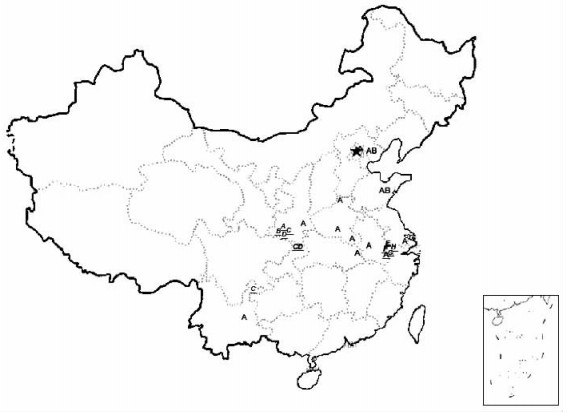
|
图 2 cpSSR单倍型在中国栗的4个野生居群和9个地方品种群中的地理分布 Figure 2 The geographical distribution of cpSSR haplotypes in chestnut populations 下划线字母代表不同野生居群拥有的单倍型,正体字母代表地方品种群中的单倍型。 The letters with underline is cpSSR haplotypes in natural populations, and others are in cultivar groups. |
利用POPGENE 1.31软件和GenAlEx6.1软件从cpSSR位点和群体2个层次上进行等位基因观察数(Na)、有效等位基因数(Ne)、基因多样度(Hs)等遗传参数分析。不同cpSSR位点的分析表明,板栗的Shannon多样性指数均值为0.617,不同位点的Nei′s多样性参数差别明显,说明不同的位点对群体基因多样度的贡献率是不一样的,其中以Cmcs2位点最高(0.509),贡献度最大。Shannon多样性指数的数值要略高于Nei′s基因多样性指数,且各参数值的趋势基本一致(表 3)。
表 5给出了13个居群的群体遗传多样性及群体分化,13个群体的Shannon指数平均为0.214,Nei基因多样度为0.149,各居群的多样性指数存在差异,其中安徽广德地区>陕西汉中地区>其他地区,可见广德居群天然野生板栗居群内蕴含更丰富的遗传变异。基因分化系数(Gst)平均值为0.6,显示板栗群体内分化较为明显。
|
|
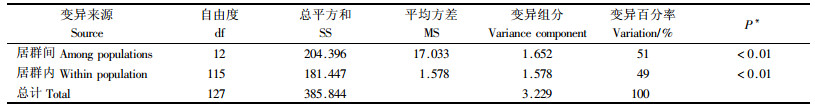
叶绿体微卫星的分子方差分析,对板栗居群间及居群内的AMOVA分析表明51%的遗传变异来源于居群间,49%来源于居群内,居群间变异略大于居群内,遗传分化达到了显著水平(P<0.01)(表 6)。而从表 3中看出,基因分化系数(Gst)在不同位点有较大差异,变化范围在0.513~0.728,平均0.600,即有60%的遗传变异存在于居群间,而40%存在于居群内。AMOVA的分析结果与 Gst 值的2个分析结果一致,显示在居群间及居群内同时存在着较大的变异来源,而居群间的分化还稍大一些。
|
|
叶绿体微卫星标记,既具有叶绿体DNA单系遗传、几乎不发生重组的特点,又具有微卫星高突变的特性,成为近年发展起来的一种新型的分子标记技术,而cpSSR具有比nSSR更高的侧翼序列保守性,其引物可以跨越种属的局限,在亲缘关系较远的科之间使用(Provan et al., 1999)。本研究使用24对cpSSR引物从板栗中成功地扩增出PCR产物,引物通用率达到100%,然而仅有4对引物扩增出的DNA片段显示出片段长度的变化,且多态位点的平均多态信息含量为0.323,Nei′s遗传多样性水平为0.377,显示其多态性水平一般。这表明cpSSR应用于板栗的遗传分析时,同样存在着引物的通用性好,而位点的多态性差的特点(Provan et al., 1998)。
3.2 板栗野生居群和地方品种中cpSSR位点的单倍型多样性板栗的高水平遗传多样性已被证实,无论是同工酶还是SSR等分子标记(郎萍等,1999;肖正东等,2005),其各多样性指标均高于本研究中cpSSR标记的结果,两者的差异反映了叶绿体基因组在群体内进化的保守特性(Weising et al., 1999)。然而从板栗的地理分布看,cpSSR能更好地揭示居群内和居群之间的遗传多样性,Nei多样性指数h和Shannon多样性指数I为群体遗传多样性的2个重要指标,从表 4可以看出,这2个指标的变化趋势是一致的,板栗汉中(秦岭南坡)及广德(天目山)天然野生居群的遗传多样性明显高于其边缘及区域性栽培群体,这与叶绿体单倍型变异分析结果相一致。
3.3 板栗野生和地方品种群中cpSSR单倍型的地理分布在野生板栗的60个样本中发现的8种单倍型呈现明显的区域性分布。其中,秦岭地区的汉中居群拥有的4种主要单倍型(A,B,C,D)出现在13个群体的95.3%样本中,涵盖本次研究涉及的全部地方品种群和绝大部分野生植株(表 4)。而以秦岭居群为中心,随着地理距离的增加,单倍型种类在居群中逐渐减少,距离中心区域最远的大关和广德居群仅拥有4个单倍型中的1个,显示了初始种群扩散过程中在cpSSR单倍型水平上形成的广泛地理差异,而4种特异单倍型仅存于天目山地区的广德野生居群中也进一步表明cpSSR单倍型的区域分布特征。对cpSSR在野生大豆(Glycine soja)群体的非随机分布(Powell et al., 1996;Tozuka et al., 1998),Abe(2000)认为可归于野生大豆种子不具备长距离扩散能力。受板栗顽拗种子特性的影响(傅家瑞,1991),cpSSR单倍型在板栗野生群体中则表现出更为明显区域性分布。
在4个板栗地方品种群的68个地方品种中仅发现2个cpSSR单倍型,其中3个地方品种群(西北、长江流域和西南)的50个品种仅有1个单倍型,而4个野生居群拥有全部8个单倍型,相对于目前板栗地方品种,板栗的野生居群具有非常高的遗传多样性;同时,也显示在板栗栽培品种形成中曾经遭遇了严重细胞质瓶颈(cytoplasmic bottleneck),一些研究表明同样的现象也在其他栽培作物的驯化过程中出现(Vaillancecourt et al., 1992;Provan et al., 1997; Ladizinsky, 1999; Friesen et al., 1999)。而8个cpSSR单倍型的地理分布及在板栗居群间的差异还暗示了板栗城口和大关野生居群似乎并未从胞质遗传方面参与当地板栗地方品种的形成,而广德野生居群在板栗地方品种形成中的作用有待进一步研究。此外,板栗地方品种群中单倍型种类远少于野生居群,且没有特异单倍型出现,显示品种群与天然野生居群关系密切,不支持目前关于野生板栗分类学地位待定的观点。
3.4 板栗地方品种的起源基于对板栗地方品种群和野生居群的cpSSR单倍型分析,认为拥有广泛存在单倍型的秦岭南麓(陕西汉中)野生板栗分布区应为我国板栗的遗传多样性中心,其他3个野生居群应是从该中心向外散布后,逐渐形成的板栗野生多样性的次级中心,但扩散期间有单倍型的丢失,以及新单倍型的产生。单倍型A存在于板栗秦岭野生居群和4个地方品种群中以及单倍型B仅存在秦岭野生居群以及河北和山东的部分品种中的事实强烈暗示现有板栗各地方品种可能全部起源于秦岭南麓野生居群中的特殊群体。考虑栽培植物的形成和扩散常与大山脉的总走向相一致(瓦维洛夫,1982)、生境对板栗扩散的限制(张宇和等,1989)以及板栗种子的生理特点(傅家瑞,1991),推测野生板栗向地方品种的演化路线可能有南北2条:北线从秦岭南麓→伏牛山→太行山→燕山→山东半岛,或经太行山后,分别向燕山和山东半岛扩散,在扩散过程中经过长期的驯化栽培,形成了现在燕山及山东的地方类型。南线从秦岭南麓沿伏牛山向南,在大别山中南部分2条,一条沿大别山南缘向东,经长江下游山地向东南沿海扩展,最终在江苏省北端与北线扩散至山东半岛的板栗汇合;另一路则经长江中游地区向南,随人口迁移,最终扩展至云贵高原。
中国现有板栗品种超过300个,几乎全部从板栗分布地的实生树中选出。基于坚果性状和栽培区域,上述品种被划分为6个地方品种群,即华北品种群、长江中下游品种群、西北品种群、东南品种群、西南品种群和东北品种群,其中东北品种群为日本栗(Castanea crenata)血统(张宇和等,2005)。本研究中涉及的品种涵盖了除东北品种群外板栗5个品种群中的4个,基本上反映了各产地的地方品种现状。国内对于板栗野生部分的关注始于20世纪60年代,然而直到最近,才开展与野生板栗的分布和遗传多样性相关的研究,未见有关于栽培板栗起源、野生板栗和栽培板栗之间关系的报道。
叶绿体SSR分析不仅可以揭示群体的遗传多态性,同时也是探讨栽培品种起源,及与野生种或近缘种间关系的有力工具(Powell et al., 1996; Provan et al., 1999; Xu et al., 2002),本文研究也显示cpSSR标记在探讨栽培板栗起源与演化方面的潜力,而增加cpSSR位点、板栗地方品种和野生居群数量将有助于进一步揭示板栗栽培品种起源、演化以及与野生板栗间的关系。
陈伯望, 洪菊生, 施行博. 2000. 杉木和秃杉群体的叶绿体微卫星分析[J]. 林业科学, 36(3): 46-51. |
范启荣. 1998. 大面积野生板栗林改造技术[J]. 中国南方果树, 27(1): 41. |
傅家瑞. 1991. 顽拗性种子[J]. 植物生理学通讯, 27(6): 402-406. |
黄宏文, FennyD, JosephD N, 等. 1996. 栗种间杂交系的同工酶基因与形态标记的遗传连锁关系研究[J]. 武汉植物学研究, 14(2): 101-109. |
郎萍, 黄宏文. 1999. 栗属中国特有种居群的遗传多样性及地域差异[J]. 植物学报, 41(6): 651-657. DOI:10.3321/j.issn:1672-9072.1999.06.017 |
李英敏, 杨道康. 1995. 从野生板栗嫁接改造到建立板栗丰产园[J]. 陕西林业, (5): 8-9. |
邱国金. 1993. 野生板栗资源的开发与利用[J]. 江苏农业科技, (1): 50-51. |
沈泉. 1993. 野生板栗嫁接利用的研究[J]. 浙江林业科技, 13(1): 36-39. |
瓦维洛夫. 1982. 董玉琛, 译. 主要栽培植物的世界起源中心. 北京: 农业出版社.
|
王艳梅, 程丽莉, 翟明普, 等. 2007. 中国榛属植物DNA提取与SSR初步分析[J]. 河南师范大学学报:自然科学版, 35(2): 129-132. |
肖正东, 何定华, 何锡山, 等. 2005. 中国栗AC/GT微卫星的提取和多态性及对安徽省板栗品种的分析[J]. 分子植物育种, 3(4): 509-514. DOI:10.3969/j.issn.1672-416X.2005.04.011 |
徐凌帆, 罗成荣, 龚固堂, 等. 2004. 四川秦巴山区野生板栗的改造[J]. 经济林研究, 22(2): 81-83. DOI:10.3969/j.issn.1003-8981.2004.02.027 |
宣善平. 1988. 大别山地区利用野生板栗就地嫁接的研究[J]. 经济林研究, 6(2): 53-56. |
张宇和, 王福堂, 高新一, 等. 1989. 板栗[M]. 北京: 中国林业出版社.
|
张宇和, 柳鎏, 梁维坚, 等. 2005. 中国果树志:板栗榛子卷[M]. 北京: 中国林业出版社.
|
Abe J. 2000. The genetic structure of natural populations of wild soybean revea led by isozymes and RFLPs of mitochondrial DNAs: possible influence of seed dis persal, cross-pollination and demography//Oono K. 7th MAFF Int Workshop Genet Resource. AFFRC and NIAR, Tsukuba, Japan, 143-138.
|
Francis C Y, Yang R C. 1999. POPGENE VERSION 1. 31.
|
Friesen N, Pollner S, Bachmann K, et al. 1999. RAPDs and noncoding chloroplast DNA reveal a single origin of the cultivated Allium fistulosum from A. altaicum(Alliaceae)[J]. American Journal of Botany, 86(4): 554-562. DOI:10.2307/2656817 |
Goulão L, Valdiviesso T, Santana C, et al. 2001. Comparison between phenetic characterization using RAPD and ISSR markers and phenotypic data of cultivated chestnut(Castanea sativa Mill.)[J]. Genetic Resources and Crop Evolution, 48: 329-338. DOI:10.1023/A:1012053731052 |
Ladizinsky G. 1999. Identification of the lentil's wild genetic stock[J]. Genet Res Crop Evol, 46: 115-118. DOI:10.1023/A:1008626128871 |
Marinoni D, Akkak A, Bounous G, et al. 2003. Development and characterization of microsatellite markers in Castanea sativa (Mill.)[J]. Molecular Breeding, 11(2): 127-136. DOI:10.1023/A:1022456013692 |
Modliszewski J L, Thomas D T, Fan C, et al. 2006. Ancestral chloroplast polymorphism and historical secondary contact in a broad hybridzone of Aesculus (Sapindaceae)[J]. American Journal of Botany, 93: 377-388. DOI:10.3732/ajb.93.3.377 |
Parducci L, Szmidt A E, Madaghiele A, et al. 2001. Genetic variation at chloroplast microsatellites(cpSSRs) in Abies nebrodensis (Lojac.) mattei and three neighboring Abies species[J]. Theoretical and Applied Genetics, 102: 733-740. DOI:10.1007/s001220051704 |
Peakall R, Smouse P E. 2001. GenAlEx 5: Genetic Analysis inExcel. Population G enetic Software for Teaching and Research. Australian National University, Canber na, Australia.
|
Powell W, Morgante M, Doyle J J. 1996. Genepool variation in genus Glycine subgenus Soja revealed by polymorphic nuclear and chloroplast microsatellites[J]. Genetics, 144(2): 793-803. |
Provan J, Corbett G, McNicol J W, et al. 1997. Chloroplast DNA variability in wild and cultivated rice(Oryza spp.) revealed by polymorphic chloroplast simple sequence repeats[J]. Genome, 40: 104-110. DOI:10.1139/g97-014 |
Provan J, Powell W, Dewar H, et al. 1999. An extrem cytoplasmic bottleneck in the modem European cultivated potato(Solanum tuberosum) is not reflected in decreased levels of nuclear diversity[J]. Proceedings of the Royal Society of London Series B, 266: 633-639. DOI:10.1098/rspb.1999.0683 |
Provan J, Soranzo N, Wilson N J, et al. 1998. Gene pool variation in Caledonian and European Scots pine(Pinus sylvestris L.) revealed by chloroplast simple sequence repeats[J]. Proceedings of the Royal Society of London Series B, 265: 1697-1750. DOI:10.1098/rspb.1998.0491 |
Sebastiani F, Carnevale S, Vendramin G G. 2004. A new set of mono-and dinucleotide chloroplast microsatellites in Fagaceae[J]. Molecular Ecology Notes, 4: 259-261. DOI:10.1111/men.2004.4.issue-2 |
Tozuka A, Fukushi H, Hirata T, et al. 1998. Composite and clinal distribution of Glycine soja in Japan revealed by RFLP analysis of mitochondrial DNA[J]. Theoretical and Applied Genetics, 96: 170-176. DOI:10.1007/s001220050724 |
Vaillancecourt R E, Weeden N F. 1992. Chloroplast DNA polymorphism suggests Nigerian center of domestication for the evolution of different chloroplast haplotypes[J]. Theoretical and Applies Genetics, 101: 724-732. |
Weising K, Gardner R C. 1999. A set of conserved PCR primers for the analysis of simple sequence repeat polymorphisms in chloroplast genomes of dicotyledonous angiosperms[J]. Genome, 42: 9-19. DOI:10.1139/g98-104 |
Xu D H, Abe J, Gai J, et al. 2002. Diversity of chloroplast DNA SSRs in wild and cultivated soybeans: evidence for multiple origins of cultivated soybean[J]. Theoretical and Applied Genetics, 105: 645-653. DOI:10.1007/s00122-002-0972-7 |
 2009, Vol. 45
2009, Vol. 45