文章信息
- 王玲, 卓丽环, 杨传平, 张捷.
- Wang Ling, Zhuo Lihuan, Yang Chuanping, Zhang Jie
- 兴安落叶松等位酶水平的遗传多样性
- Allozymic Genetic Diversity in Larix gmelinii
- 林业科学, 2009, 45(8): 170-174.
- Scientia Silvae Sinicae, 2009, 45(8): 170-174.
-
文章历史
- 收稿日期:2009-02-26
-
作者相关文章
兴安落叶松(Larix gmelinii)是我国高寒地区主要造林和用材树种,其群体内的遗传多样性可以反映种群进化的潜力,对基因资源保护、育种战略的制定有重要的参考价值。裸子植物遗传多样性研究可采用DNA分子标记(Kozyrenko et al., 2004;李因刚等,2008;Xu et al., 2008;张蕊等,2009),也可以采用等位酶分析技术,后者已成功地应用到松属(Pinus)(葛颂等,1988;祖元刚等,1999;虞泓等,1999)、落叶松属(Larix)(杨传平,1997;张学科等,2002)、云杉属(Picea)(张含国等,2002;罗建勋等,2006)等种群遗传多样性的研究中。
关于兴安落叶松酶水平遗传多样性,Larionova等(2004)采用等位酶分析了西伯利亚中部种群的遗传多样性;乔辰等(1995)采用垂直板状聚丙烯酰胺凝胶电泳技术利用3个同工酶分析了我国兴安落叶松的种群内遗传与分化,证明其种群间存在差异,杨传平(1997)利用2个同工酶分析了群体平衡状况和分化程度。等位酶是基因变化的一个标志,水平切片淀粉凝胶电泳等位酶分析为群体遗传学研究提供了有力工具,是在居群、种甚至属的水平上研究生物遗传多样性的重要方法,也是现代植物系统和进化研究所必不可少的手段(王中仁,1994a),因此有必要采用单株取样、随机多个酶系统的水平切片淀粉凝胶电泳技术,进一步研究我国兴安落叶松种群居群内、居群间不同个体之间在遗传上的变异,从基因水平上发现这些变化,从而有效地度量种群中遗传变异的大小及分布进而探讨进化问题。
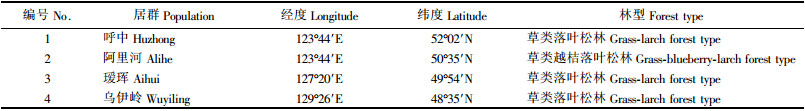
1 材料与方法 1.1 材料根据兴安落叶松的分布现状,按照种源区划的标准(杨传平等,1991),把兴安落叶松分布区分为4个小区:1)大兴安岭北部种源区,编号为Ⅰ; 2)大兴安岭中南部种源区,编号为Ⅱ; 3)小兴安岭北部种源区,编号为Ⅲ; 4)小兴安岭中南部种源区,编号为Ⅳ。在每一个种源区内选取一林区代表该区的分布式样。在Ⅰ区选取呼中,Ⅱ区选取阿里河,Ⅲ区选取瑷珲,Ⅳ区选取乌伊岭。把这4个地区作为4个居群,进行居群取样(表 1)。
|
|
秋季落叶后,在兴安落叶松4个天然居群中,每个居群选取10个以上单株,居群内样株间距至少50 m。每株取4个枝条,枝条带回实验室经处理萌发新叶,叶长至1 cm左右选作试验材料。在居群内分单株取嫩叶,每株取5 g为1个样。
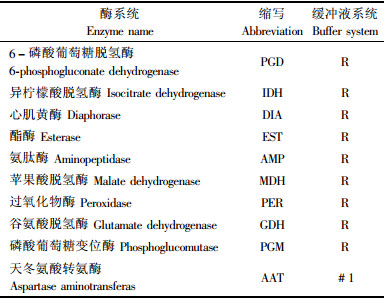
1.2 等位酶电泳采用的缓冲液系统配方见表 2。选用12个酶系统,并从12个酶系统中选出10种分离良好、谱带清晰的酶系统(表 3)做兴安落叶松种群遗传结构、遗传变异的研究。
|
|
|
|
参考云南松(Pinus yunnanensis)群体遗传学研究的等位酶分析方法(虞泓等,1999)。
1.4 数据处理和遗传分析试验数据利用BIOSYS_11.7和POPGENE1.3232软件处理和运算。以各基因位点的等位基因频率为基本数据,估算基础遗传多样性参数并进行参数分析。
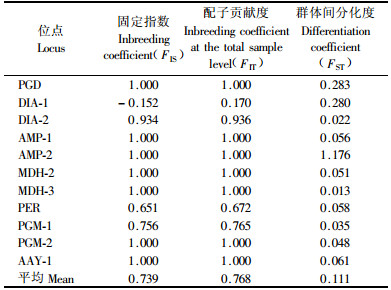
2 结果与分析 2.1 等位基因位点多样性4个居群73个样本材料的10个酶系统,确定16个等位基因位点,其中有5个单态位点:IDH,EST,GDH,AAT-2,MDH-1;11个基因位点是多态的,多态位点中有2个等位基因的位点有MDH-2,MDH-3,PGD,DIA-1,AMP-1,AMP-2,PGM-2,AAT-1;有3个等位基因的位点有DIA-2,PER,PGM-1。
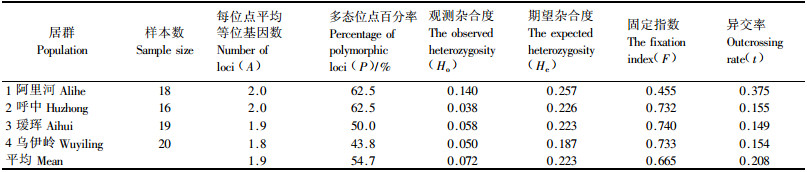
2.2 兴安落叶松居群遗传多样性等位酶位点上衡量兴安落叶松4个居群遗传变异的指标值见表 4。多态位点百分率(P)的变动范围在0.438~0.625,平均值为0.547;每个位点等位基因平均数(A)为1.8~2.0个,平均值为1.9个; 观测杂合度(Ho)为0.038~0.140,平均值为0.071 5;期望杂合度(He)变动范围在0.187~0.257,平均值为0.223 3;固定指数(F)为0.455~0.740,平均值为0.655;异交率(t)为0.149~0.375,平均值为0.208。可见兴安落叶松种群中有54.7%的位点呈现多态现象,且每个位点等位基因数达1.9个,期望杂合度高达0.223 3,即种群处于平衡状况时杂合体比例应为22.33%。
|
|
4个居群的固定指数F值都明显大于0,表明种群中杂合体不足。天然种群中杂合体不足是较常见的现象,个体间仅有20.8%的异交,异交水平相当低,因而过高的自交率或近交是造成该种群杂合体不足的主要原因。观测杂合度(Ho)低于预期杂合度(He),表明实际的等位基因频率偏离哈迪-温伯格(Hardy-Weinberg)平衡,群体存在轻微的纯合子过量现象。
2.3 兴安落叶松的种群分化采用F统计量(表 5)来推测整个分布区内兴安落叶松天然群体的遗传变异、分化在群体内和群体间的分配情况。从表 5可见:多态位点中只有DIA-1位点杂合体过量,其余位点均存在杂合体不足现象,FIT,FIS平均值都为正值,杂合体偏离期望比例的程度较严重,主要由于大多数位点上的等位基因在种群中发生了固定。由兴安落叶松4个居群11个多态位点的FST均值可知居群间分化程度为11.1%,即总体遗传变异有11.1%的变异来源于不同居群间,有88.9%的变异存在于居群内。群体存在轻微的纯合子过量现象。
|
|
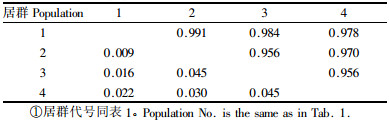
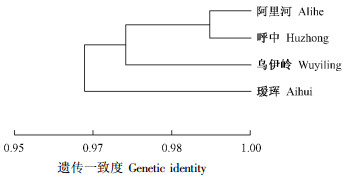
采用遗传一致度和遗传距离对居群之间相同之处和不同之处加以量化(表 6)。兴安落叶松居群间遗传一致度较高,平均值可达0.972 5;遗传距离较小,在0.009~0.045之间,平均值为0.078,各居群之间亲缘关系较近。较近的程度可采用加权平均聚类法来具体而形象地描述,以遗传一致度为基础的聚类结果见图 1。由图可见,阿里河与呼中亲缘关系最近,乌伊岭次之,瑷珲最远,遗传距离最大。从而推测种群遗传变异可能存在地理隔离关系。
|
|
|
图 1 兴安落叶松4个居群的遗传一致度聚类图 Figure 1 UPGMA dendrogram of the four Larix gmelinii populations based on Nei's genetic identities |
本研究中有几个酶系统与松科(Pinaceae)其他属或本属不同树种种群研究结果不一致,控制酶位点的数量存在差异,可能主要有以下几个原因导致电泳谱带数量有所不同:1)不同树种本身的差异;2)不同器官或组织酶系统表达不同和不同发育时期酶的活性可能不同(王中仁,1994b),在以往酶系统研究中用的一般都是种子胚乳,而本研究中用的是针叶幼叶,不同器官或组织,酶的活性存在差异,在表达上自然不同;3)凝胶电泳系统不同,以往采用的是垂直板状聚丙烯酰胺凝胶电泳,而本研究则采用水平切片淀粉凝胶电泳,不同电泳系统分辨率不同;4)不同的缓冲液系统和染色方法。
3.2 兴安落叶松居群、种群遗传分化兴安落叶松居群间的分化程度要高于裸子植物的平均分化程度(FST=6.8%),也高于长寿多年生木本植物平均分化程度(FST=7.6%)和异交风媒植物的平均分化程度(FST=9.9%)。对所有的居群,尤其是自然群体,一般都是适应环境的,复杂的环境变化需要居群内有较大的变异分布,否则该群体就不可能稳定地生存,相对来说,同一物种在它的自然分布区内,居群之间从适应方式上看应该是比较一致的;居群内这种变异一般对选种战略的参考意义不大,因为分布在居群内的变异主要反映居群的稳定性,而分布在居群间的变异才真正反映了群体在不同环境中的适应情况。换言之,分布在居群间的变异大小,在某种程度上说明了该生物对不同环境适应的广泛程度,它越大,该生物适应的环境就越广,这种变异也与地理、生殖隔离有关(庞广昌等,1995)。由此可见,兴安落叶松居群间较高的变异对其适应所处环境、维持种群稳定性起主导作用。居群间的分化是兴安落叶松种群对不同环境适应的体现方式。
在植物界中,裸子植物具有较高的变异水平,具有较高水平变异的植物种多为寿命长、地理分布广、风媒传粉、结实率高和处于演替末期的物种(葛颂等,1988),兴安落叶松就具备了这些特性。从分类群角度看,裸子植物的遗传多样性平均值P=57.7%,A=1.93,He=0.160;从生活型来看,木本植物的变异平均是P=50.0,A=1.79,He=0.149;从繁育角度来看,异交风媒植物的变异平均是P=41.6%,A=1.64,He=0.133(虞泓,1996)。兴安落叶松种群的P、A值处于中偏上水平,He则处于高水平,这与杨传平(1997)研究结果一致,也与Larionova等(2004)对西伯利亚中部兴安落叶松种群遗传多样性研究结果一致,说明兴安落叶松种群遗传多样性水平较高。并且我国的兴安落叶松种群P、A、He各项基因丰富度指标由北向南按阿里河、呼中、瑷珲、乌伊岭依次减小(阿里河、呼中A、P相同),由于散布出去的居群丰富度要比原来的居群单调得多,具有较少的等位基因或杂合性(Wang,1992),由此可以推断种群可能的历史迁移方向,可以认为兴安落叶松在我国的分布是由北向南散布的,这与乔辰等(1995)分析结果较一致。兴安落叶松种群保持相当的进化灵活度,具有较充分的进化潜力。
我国亚洲东北部的兴安落叶松是由比较古老的西伯利亚落叶松演化而来的,是一类分化较晚较年轻的种群(唐谦等,1995)。其由北向南扩展延伸,在小兴安岭由于分布不连续,这种不连续可能是人为或气候变化、物种替代等原因导致的,在自然向南延伸过程中,只有部分基因漂移,加上地理隔离的影响,减少了与其他居群的频繁的基因流动造成自交或近亲杂交频率加大,基因丰富度下降,还有对不同环境的各自适应性,使得居群间遗传相似性减小,遗传距离加大。而在大兴安岭分布区内兴安落叶松的分布是连续的,异交风媒传粉,频繁基因交流导致遗传距离较小,一致度较高。居群间遗传距离和空间距离存在一定相关性,地理隔离可能是群体分化的主要原因,当然群体间的遗传异质性也与基因型的差异选择或基因漂变有关。
等位酶分析结果支持了这样一个结论:进化中的自然选择在兴安落叶松遗传多样性和多基因位点遗传结构产生和维持中也起重要作用。
葛颂, 王明庥, 陈岳武. 1988. 用同工酶研究马尾松群体的遗传结构. 林业科学, 24(4): 399-408. |
李因刚, 周志春, 金国庆. 2008. 三尖杉种源遗传多样性. 林业科学, 44(2): 64-69. DOI:10.3321/j.issn:1001-7488.2008.02.010 |
罗建勋, 顾万春, 陈少瑜. 2006. 云杉天然群体遗传多样性的等位酶变异. 植物生态学报, 30(1): 165-173. DOI:10.3321/j.issn:1005-264X.2006.01.023 |
庞广昌, 姜冬梅. 1995. 群体遗传多样性和数据分析. 林业科学, 31(6): 543-550. DOI:10.3321/j.issn:1001-7488.1995.06.002 |
乔辰, 包玉莲, 霍德文. 1995. 十五个产地兴安落叶松种子同工酶的研究. 内蒙古林学院学报, 17(4): 5-8. |
唐谦, 顾万春, EnnosR A, 等. 1995. 几种落叶松树种叶绿体DNA的分化及其系统演化关系的含义. 林业科学, 31(4): 373-378. DOI:10.3321/j.issn:1001-1498.1995.04.008 |
王中仁. 1994a. 植物遗传多样性和系统学研究中的等位酶分析. 生物多样性, 2(1): 38-43. |
王中仁. 1994b. 植物遗传多样性和系统学研究中的等位酶分析. 生物多样性, 2(2): 91-95. |
杨传平. 1997. 兴安落叶松优良种源遗传结构. 东北林业大学学报, 25(3): 1-5. |
杨传平, 杨书文, 夏德安, 等. 1991. 兴安落叶松种源试验研究(Ⅲ)——种源区划. 东北林业大学学报, 19(育种专刊): 77-83. |
虞泓. 1996.云南松遗传多样性与进化研究.昆明: 云南大学博士学位论文. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y236263
|
虞泓, 钱韦, 黄瑞复. 1999. 云南松居群遗传学研究的等位酶分析方法. 云南植物研究, 21(1): 68-80. DOI:10.3969/j.issn.2095-0845.1999.01.012 |
张含国, 王辉, 肖彦利, 等. 2002. 红皮云杉等位酶群体遗传的多样性分析. 东北林业大学学报, 30(6): 21-25. DOI:10.3969/j.issn.1000-5382.2002.06.006 |
张蕊, 周志春, 金国庆, 等. 2009. 南方红豆杉种源遗传多样性和遗传分化. 林业科学, 45(1): 50-56. DOI:10.3321/j.issn:1001-7488.2009.01.010 |
张学科, 毛子军, 宋红. 2002. 五种落叶松遗传关系的等位酶分析. 植物研究, 22(2): 224-230. DOI:10.3969/j.issn.1673-5102.2002.02.021 |
祖元刚, 张恒庆, 颜廷芬, 等. 1999. 天然红松林等位酶研究. 植物研究, 19(1): 75-79. |
Kozyrenko M M, Artyukova E V, Reunova G D, et al. 2004. Genetic diversity and relationships among Siberian and Far Eastern Larches inferred from RAPD analysis. Russian Journal of Genetics, 40(4): 401-409. DOI:10.1023/B:RUGE.0000024978.25458.f7 |
Larionova A Y, Yakhneva N V, Abaimov A P. 2004. Genetic diversity and differentiation of Gmelin Larch Larix gmelinii populations from Evenkia (Central Siberia). Russian Journal of Genetics, 40(10): 1127-1133. DOI:10.1023/B:RUGE.0000044756.55722.d8 |
Wang Zhongren. 1992. A comparative allozyme study on one Asian and two American populations of ostrich fern (Matteuccia struthiopteris) and its biogeographic implication. Chthaya, 4: 1-8. |
Xu Shiqin, Tauer C G, Nelson C D. 2008. Genetic diversity within and among populations of shortleaf pine (Pinus echinata Mill.) and loblolly pine (Pinus taeda L.). Tree Genetics & Genomes, 4(4): 859-868. |
 2009, Vol. 45
2009, Vol. 45