文章信息
- 齐靖, 董祯, 毛永民, 申连英, 张玉星, 刘杰, 王晓玲.
- Qi Jing, Dong Zhen, Mao Yongmin, Shen Lianying, Zhang Yuxing, Liu Jie, Wang Xiaoling
- 枣高密度遗传图谱的构建与树干直径的QTL分析
- Construction of a Dense Genetic Linkage Map and QTL Analysis of Trunk Diameter in Chinese Jujube
- 林业科学, 2009, 45(8): 44-49.
- Scientia Silvae Sinicae, 2009, 45(8): 44-49.
-
文章历史
- 收稿日期:2008-09-09
-
作者相关文章
2. 河北女子职业技术学院 石家庄 050091
2. Hebei Women's Vocational College Shijiazhuang 050091
枣(Zizyphus jujuba)原产我国, 是我国重要的经济树种之一. 枣在我国栽培面积极广, 有700多个品种, 种质资源十分丰富. 如何充分利用这些种质资源, 培育综合性状优良枣树品种成为枣树育种工作的一个重要目标. 但由于枣树花器小、座果率低, 部分品种出仁率低或没有种仁, 给枣树人工杂交育种带来了极大的困难, 致使枣育种工作远落后于其他果树. 随着生物技术的高速发展, 分子标记和遗传图谱的出现为枣树育种工作开辟了新的思路. 通过构建枣遗传连锁图谱, 对控制枣农艺性状的数量性状位点(QTL)进行定位, 并在此基础上进行精细定位和图位克隆, 必将会加快我国枣树育种和种质改良进程. 相对其他植物而言, 枣遗传连锁图谱的研究尚属起步阶段, 目前仅有申连英(2005)以冬枣(Zizyphus jujuba ‘Dongzao’)×临猗梨枣(Zizyphus jujuba ‘Linyilizao’)杂交F1代为作图群体, 构建的包含333个AFLP标记、14个连锁群的枣属植物首张遗传连锁图谱, 但由于其标记种类单一且数量有限, 致使图谱还存在很多标记空缺区段, 仍需增加标记种类和数量以提高图谱饱和度.
树干直径的生长是其木质部形成、树干形成层细胞分裂和伸长的过程(王沙生等, 1991), 是检测枣树生长势的重要指标之一. 树干直径的增加不但可以为枣树生长、结果提供更加有力的骨架支撑, 还可以增强枣树对水分和养分的疏导能力, 从而有助于提高枣树抗寒、抗旱和抗盐碱能力. 目前有关枣树干直径的遗传变化规律和相关QTL定位的研究仍属空白. 本研究在申连英(2005)研究成果的基础上, 利用RAPD标记和AFLP标记对已有的枣遗传图谱进行了加密, 并将控制枣树干直径的QTL定位在了新构建的图谱上, 旨在提高原有图谱饱和度, 并为今后深入了解枣树干直径遗传机制与克隆相关基因提供依据.
1 材料与方法 1.1 试验材料应用申连英(2005)构建的冬枣×临猗梨枣杂交F1代为作图群体. 亲本果园位于河北省任县, 1999年建园, 面积0.3 hm2, 父本为临猗梨枣, 母本为自花不育品种冬枣. 2品种等比例栽植, 均为嫁接苗. 亲本园附近5 km内无其他枣园, 2 km内也无其他单株枣树. 2001年10月采摘亲本果园中冬枣的果实, 自然晾干后取出种仁, 于2002年4月播种于冬枣实生后代园中, 株行距30 cm × 50 cm, 普通果树栽培措施栽植. 共获得冬枣实生后代165株. 经AFLP分析鉴定, 其中161株为冬枣×临猗梨枣杂交子代. 从中随机选取150株作为作图群体试材.
1.2 样品DNA提取总DNA提取参照Paterson等(1993)所述的CTAB法, 取新鲜叶片进行DNA提取.
1.3 AFLP分析AFLP分析采用的2个限制性内切酶为EcoRⅠ和MseⅠ, DNA酶切片段的接头、预扩增引物和选择性扩增引物由上海生工公司合成. 反应体系参照申连英(2005)所述枣AFLP扩增体系建立. PCR反应在德国Biometra公司生产的T Gradient PCR仪中进行. AFLP选择性扩增产物在6%变性聚丙烯酰胺凝胶上电泳分离, 采用银染的方法检测DNA多态性.
1.4 RAPD分析RAPD引物参照刘平等(2005)筛选出的在枣上多态性较好的引物, 购自上海生工公司. PCR反应总体系为25 μL, 其中包含1×PCR buffer、2.0 mmol·L-1Mg2+、0.25 mmol·L-1 dNTPs、1.5 U Taq酶、1.25 μmol·L-1引物和30 ng模板DNA. PCR扩增程序为: 94 ℃预变性5 min, 94 ℃变性30 s, 36 ℃退火40 s, 72 ℃延伸60 s, 共40次循环; 72 ℃完全延伸10 min, 4 ℃保存. 扩增结束后加入5 μL 6×Loading Buffer, 在1.6%琼脂糖凝胶上电泳分离, 紫外灯下显影、照相.
1.5 数据的统计与分析分离后代中特异性条带的有无用“1”、“0”表示, 带型不清或缺失者记为“—”. 根据已知分子质量的marker计算各多态性带的分子质量, AFLP标记用“引物组合+分子质量”表示, RAPD标记用“引物名称十分子质量”表示. 运用JoinMap3.0软件的CP作图模型构建遗传图谱, 采用Kosambi函数计算图距. 利用JionmapQTL4.0分析软件, 采用区间作图法(interval mapping)进行QTL分析, 以LOD值的大小来判定是否存在QTL效应. 当LOD≥2.5时, 认为存在QTL效应.
2 结果与分析 2.1 标记在作图群体中的分离情况利用亲本与F1代进行AFLP引物组合的筛选, 最后从80对引物中筛选出8对条带清晰、多态性高的引物组合进行AFLP分析. 8对引物共产生212个条带, 其中多态性带51条, 平均每对引物扩增多态性带为6.38条. 共筛选出22个RAPD引物用于遗传图谱的构建, 扩增得到213条带, 平均每个引物扩增出9.7条, 其中多态性带有41条, 占RAPD条带总数的19.2%. 对总共92个多态性标记进行卡方分析, 检测得到偏分离标记36个(χ2 < χ0.10, 12=2.71), 占多态性标记总数的39.13%. 其中偏分离AFLP标记22个, 占AFLP多态标记总数的43.14%; RAPD偏分离标记14个, 占RAPD多态标记总数的34.15%.
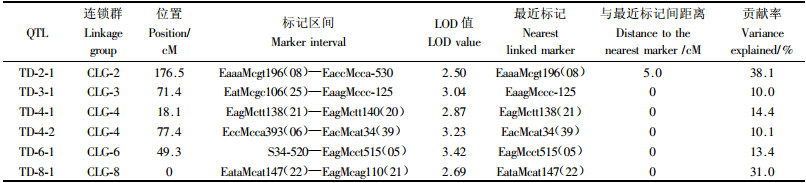
2.2 遗传图谱的构建将申连英(2005)研究获得的419个AFLP标记(包括已构图谱包含的333个标记与未定位到图谱上的86个标记)与本研究获得的92个标记整合, 利用Joinmap3.0软件对总共511个标记进行连锁分析, 构建了一张由423个分子标记组成的、包含15个连锁群的枣遗传连锁图谱(图 1), 覆盖基因组总长度1 309.4 cM, 标记间平均距离3.1 cM. 每个连锁群上的标记数4~70个, 连锁群的长度18.6~183.8 cM, 平均图距2.1~11.8 cM, 没有较明显的标记密集区. 423个分子标记中, 包含AFLP标记388个, 占91.7%, RAPD标记35个, 占8.3%. 共有137个偏分离AFLP标记和10个偏分离RAPD标记定位到了图谱中(标记“*”为偏分离位点), 偏分离标记占图谱标记总数的34.8%.

|
图 1 基于AFLP和RAPD标记的冬枣×临猗梨枣遗传连锁图谱及枣树干直径性状在图谱中的定位 Figure 1 The AFLP-RAPD genetic linkage map and QTLs for trunk diameter in Chinese jujube 连锁群上灰色区域为QTL位置. The gray regions on the linkage group are the position of QTLs for trunk diameter. |
本研究获得的47个AFLP标记和35个RAPD标记定位在图谱上. 在这些新增加的标记的作用下申连英(2005)研究获得的、但在原图谱构建过程中未利用的13个标记得到了利用, 定位在新的连锁图中, 其中3个还与2个新增标记组建了1个小的连锁群CLG-15. 但同时也发现有5个在原图谱上连锁的标记, 未被包含在新构图谱中, 分别为EacaMcat187(14)、EacaMcct139(19)、EtcMcag132(31)、EaggMat224(28)和EataMcat160(14). 新增标记填补了原图谱中部分标记空缺, 使大于10 cM的间隔由原来的21个减少到了14个, 并使图谱的总图距由原来的1 237.4 cM增加到了1 309.4 cM, 增长了5.8%, 标记间平均距离由原来的3.9 cM减少到了3.1 cM, 提高了图谱饱和度.
新标记的加入, 打破了原图谱上部分标记间的连锁关系, 致使连锁群分布发生了一些改变, LG2、LG3和LG4连锁群在8个新增标记的作用下, 组建为1个连锁群CLG-2; LG13连锁群在2个新增AFLP标记和2个新增RAPD标记的作用下, 打断为2个连锁群CLG-8和CLG-13, 其中CLG-8包含4个新增标记, CLG-13仅包含4个原LG13连锁群上的AFLP标记; 在LG14连锁群, 由于2个新增标记的存在, 原有的9个标记分为2个连锁群, CLG-11和CLG-14.
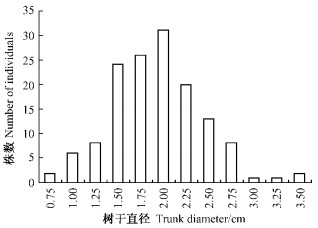
2.4 树干直径的QTL定位和分析对2005年作图群体树干直径数据进行统计分析后发现, 树干直径在作图群体中表现为连续变异, 其峰值和偏度均小于2, 在次数分布图上均表现为单峰正态分布(图 2), 具有典型数量遗传的特征, 适合进行QTL定位的研究.
|
图 2 树干直径性状表型值在作图群体中的分布 Figure 2 Segregation of trunk diameter traits in the mapping population |
利用JionmapQTL4.0分析软件, 采用区间作图法对树干直径进行QTL分析, 共获得与树干直径相关QTL 6个(图 1, 表 1), LOD值在2.50~3.42. 其中LOD值最大的为TD-6-1, 为3.42, 与标记EagMcct515(05)共分离; 其次为TD-4-2, LOD值为3.32, 与标记EacMcat34(39)共分离; TD-2-1的LOD值最小, 为2.50, 与标记EaaaMcgt196(08)距离较近, 图距为5.0 cM. 6个QTL分布在5个连锁群之上, 其中TD-4-1和TD-4-2共同分布在CLG-4连锁群上, 其余4个QTL分别分布在CLG-2、CLG-3、CLG-6和CLG-8上. 6个QTL单独存在时可解释的表型变异均在10%以上, 其中TD-2-1和TD-8-1可解释的表型变异较高, 分别为38.1%和31.0%; TD-3-1可解释的表型变异最小, 为10.0%.
|
|
本研究利用AFLP和RAPD 2种标记构建枣遗传图谱, 由结果可知AFLP多态性检出效率明显高于RAPD. 说明AFLP是一种多态性丰富、高通量的分子标记类型, 对于构建图谱和增加图谱密度是高效的. 这与其他研究者的试验结果相一致(金梦阳等, 2006; 安静等, 2007; 房守敏等, 2007). 然而在作图利用率方面, RAPD标记的利用率为85.4%, 略高于AFLP标记的利用率, 后者为82.6%. 这可能是因为本研究所利用AFLP标记均来源于EcoRⅠ/MseⅠ内切酶组合的酶切产物, EcoRⅠ受甲基化胞嘧啶的影响较小, 使EcoRⅠ/MseⅠ组合所检测的位点多聚集在着丝粒两端甲基化程度较高的重复序列区域(于拴仓等, 2003; 王美等, 2004); 而RAPD标记分布随机性强, 理论上可均匀分布于整个基因组, 更容易定位到图谱中.
分子标记发生偏分离是一种普遍的生物学现象, 被认为是生物进化的动力之一(Lyttle, 1991). 虽然偏分离可能会影响标记间遗传距离的估计值和QTL定位结果, 但在遗传连锁图构建和QTL定位中偏分离的影响常常被忽视(朱成松等, 2007), 因为如果将偏分离标记剔除将有可能造成大量信息的损失、降低基因组的覆盖率和漏掉某些重要的QTL(Luo et al., 2005). 在本研究构建图谱所包含的标记中, 共有147个标记发生了偏分离, 占图谱标记总数的34.8%, 与其他已知植物遗传图谱相比, 属正常范围. 但偏分离标记在各连锁群中分布不均匀, CLG-1, CLG-2, CLG-4和CLG-5四个连锁群上集中了半数以上的偏分离位点; 在147个偏分离位点中有81个位点(55.1%)偏向于父本, 有66个位点(44.9%)偏向于母本. 造成这种现象的原因可能是在该连锁群上某位点存在不亲和或部分不亲和的等位基因, 致使与另一等位基因相连锁的标记在子代中过度出现, 从而表现出偏分离在特定连锁群上集中分布和杂交子代群体基因型偏向父本或偏向母本的分离现象.
原枣遗传图谱包含14连锁群, 大于枣染色体数目(n=12), 申连英(2005)认为这是因为部分染色体尚存在标记空缺区段造成的, 增加标记种类和数量将使部分连锁群连接起来, 最终与染色体数目相对应. 在本研究中, 新增标记的加入虽然使原图谱的3个连锁群连接成一个大的连锁群, 但同时也发现2个原图谱连锁群被打断的现象. 其中以LG13连锁群最为明显, 在新增标记的作用下, 4个LG13上的AFLP标记从连锁群上断裂出来, 形成了一个小的连锁群CLG-13. 导致这种现象的原因可能与AFLP标记的缺点有关. 熊立仲等(1998)研究表明AFLP标记存在重要的缺点, 即相当数量的AFLP标记在统计学上严谨度较高的条件下不表现出与其他标记的连锁, 降低严谨度后则表现连锁松散而使整个连锁图的长度显著增加, 这与图谱的标记密度低有较大的关系. 原枣遗传图谱上的5个AFLP标记未被定位在新构建图谱上, 也可能是这个原因造成的.
在新构建的图谱上, 还发现部分定位在原图谱上的标记间的遗传距离和相对位置发生改变, 这种现象在各连锁群上较为普遍, 除CLG-12及CLG-15外均有发生. 吕蓓等(2003)、辛翠花等(2006)在进行图谱加密时, 也发现同样的现象. 这很可能与构图时采用的多点检测有关. 多点测验通常先对各种可能的标记排列顺序进行最大似然估计, 然后通过似然比检验确定出可能性最大的顺序. 随着标记数量的增多, 每次多点测验可能的排列数将会呈几何数目上涨, 这会使作图软件在最佳排列顺序的选择上发生变化, 也就导致了部分标记间距离和位置的改变. 这种推测正确与否还有待进一步证实, 但这种标记位置的变化应该不会影响QTL定位的准确性.
在枣性状的QTL研究领域, 仅有二次枝节间长度、枣吊节间长度、叶片形态、叶片氮磷钾含量、抗寒性和抗病性的QTL获得了定位(申连英, 2005), 而有关枣其他性状QTL定位的研究, 目前尚属空白. 本研究首次对控制枣树干直径性状的QTL进行了定位, 从而为全面进行枣性状的QTL定位增添了新的理论基础. 但是由于QTL定位是在对数量性状表型值进行分析基础上的研究, 环境和发育阶段在一定程度上影响表型值与基因型之间的对应程度. 而本研究定位的枣树干直径的QTL仅来源于2005年1年的数据, 未能对其性状进行生长及环境的追踪, 所定位的QTL难免不够全面, 有待进一步完善. 深入了解单个数量基因座位(QTL)在性状发育过程中基因表达的时空模式, 以及增加QTL与环境互作等方面的研究, 将是今后研究的方向.
安静, 胡勇胜, 张宝玺, 等. 2007. 辣椒分子连锁遗传图谱的构建及抗疫病QTL定位. 中国蔬菜, (10): 9-12. DOI:10.3969/j.issn.1000-6346.2007.10.004 |
房守敏, 余泉友, 李斌, 等. 2007. 家蚕RAPD?AFLP和SSR标记的研究及比较分析. 中国农业科学, 40(12): 2926-2930. DOI:10.3321/j.issn:0578-1752.2007.12.038 |
金梦阳, 刘列钊, 付福友, 等. 2006. 甘蓝型油菜SRAP、SSR、AFLP和TRAP标记遗传图谱构建. 分子植物育种, 4(4): 520-526. DOI:10.3969/j.issn.1672-416X.2006.04.012 |
刘平, 彭建营, 彭士琪. 2005. 应用RAPD标记技术探讨枣与酸枣的分类学关系. 林业科学, 41(2): 182-185. DOI:10.3321/j.issn:1001-7488.2005.02.032 |
吕蓓, 张丽霞, 宛煜嵩, 等. 2003. 用AFLP标记饱和大豆SSR遗传连锁图. 分子植物育种, 3(2): 163-172. |
申连英.2005.枣遗传连锁图谱构建及性状的QTL定位研究.保定: 河北农业大学博士学位论文. http://cdmd.cnki.com.cn/article/cdmd-11920-2005104347.htm
|
王美, 张凤兰, 孟祥栋, 等. 2004. 中国白菜AFLP分子遗传图谱的构建. 华北农学报, 19(1): 28-33. DOI:10.3321/j.issn:1000-7091.2004.01.009 |
王沙生, 高荣孚, 吴贯明. 1991. 植物生理学. 北京: 中国林业出版社.
|
辛翠华, 朱美云, 吴俊, 等. 2006. 利用AFLP对桃遗传连锁图的加密及加密前后连锁图的比较分析. 农业生物技术学报, 14(3): 387-390. DOI:10.3969/j.issn.1674-7968.2006.03.017 |
熊立仲, 王石平, 刘克德, 等. 1998. 微卫星DNA和AFLP标记在水稻分子标记连锁图上的分布. 植物学报, 40(7): 605-614. DOI:10.3321/j.issn:1672-9072.1998.07.004 |
于拴仓, 王永健, 郑晓鹰. 2003. 大白菜分子遗传图谱的构建与分析. 中国农业科学, 36(2): 190-195. DOI:10.3321/j.issn:0578-1752.2003.02.013 |
朱成松, 王付华, 王建飞, 等. 2007. 回交、DH和RIL偏分离群体遗传图谱的重新构建. 科学通报, 52(8): 918-922. DOI:10.3321/j.issn:0023-074X.2007.08.011 |
Luo L, Zhang Y M, Xu S. 2005. A quantitative genetics model for viability selection. Heredity, 94: 347-355. DOI:10.1038/sj.hdy.6800615 |
Lyttle T W. 1991. Segregation distorters. Annu Rev Genet, 25: 511-545. DOI:10.1146/annurev.ge.25.120191.002455 |
Paterson A, Brubaker C, Wendle J. 1993. A rapid method for extraction of (Gossypium spp.) genomic DNA suitable for RFLP or PCR analysis. Plant Molecular Bilolgy Reporter, 11(2): 122-127. DOI:10.1007/BF02670470 |
 2009, Vol. 45
2009, Vol. 45