文章信息
- 高瑞, 李向东, 王洁, 刘金亮, 竺晓平, 张广民.
- Gao Rui, Li Xiangdong, Wang Jie, Liu Jinliang, Zhu Xiaoping, Zhang Guangmin.
- 绣线菊丛枝病病原的分子鉴定
- Molecular Identification of the Phytoplasma Associated with Spiraea Witches'Broom
- 林业科学, 2007, 43(11): 72-75.
- Scientia Silvae Sinicae, 2007, 43(11): 72-75.
-
文章历史
- 收稿日期:2006-12-25
-
作者相关文章
植原体(Phytoplasma), 原称Mycoplasma-like Organism(MLO),是一类无细胞壁、不能人工培养、存在于植物筛管内的专性寄生菌。植原体主要依靠叶蝉等从韧皮部取食的半翅目昆虫传播,也可由菟丝子(Cuscuta spp.)和嫁接等传播,能够引起植物丛枝、黄化、簇生、矮化、顶枯等症状(Bai et al, 2006)。我国已报道的植原体病害约有70多种,造成了巨大的经济损失(李永等, 2005)。
传统上,主要是根据寄主的种类及其症状等生物学性状和介体昆虫的特性对植原体进行鉴定和命名。但该方法非常复杂和费力,难以鉴定多种植原体复合侵染引起的病害,经常导致片面甚至错误的结论(Lee et al, 1998a)。近年来,随着分子生物学的迅速发展,核酸杂交、RFLP、PCR等技术的广泛应用极大地推动了植原体分子鉴定等方面的研究。Lee等(1998b)根据植原体的16S rRNA基因的RFLP分析将植原体34个典型的株系划分为14个组32个亚组,其中翠菊黄化组(16SrⅠ)变异最大、分布最广。Schneider等(1997)对翠菊黄化组、stolbur组和X-d isease组的tuf基因进行RFLP分析,认为tuf基因可以作为划分植原体的依据,并且对植原体的检测可以达到单拷贝基因的水平。Marcone等(2000)将翠菊黄化组划分为6个亚组。2004年,国际比较菌原体学研究计划署(IRPCM)将植原体划分为17个组,24个种,至少40个亚组,并制定了相应的分类标准。
绣线菊(Spiraea bumalda)为蔷薇科绣线菊属落叶小灌木,因其具有适应性强、易繁殖、生长快、观赏价值高等特点,被广泛用于路边、林缘、公园等的绿化。
本研究利用巢式PCR方法,首次对表现丛枝症状的绣线菊进行植原体分子检测,并将克隆到的16S rRNA基因序列与已知植原体16Sr各组中植原体序列进行同源性比较分析,初步明确了该分离物的分类地位。
1 材料与方法 1.1 材料表现丛枝、黄化及小叶症状的绣线菊样品采自山东省青州市;克隆载体pMD18-T、PCR产物回收试剂盒、限制性内切酶及其他酶类等分子生物学试剂产品均购自TaKaRa公司。
1.2 总DNA的提取提取方法参照文献(漆艳香等, 2004)的方法进行,总DNA于-20 ℃保存备用。
1.3 16S rRNA基因PCR扩增参照文献(Lee et al, 1993; Gundersen et al, 1996)所报道的植原体16S rRNA基因通用引物R16mF2/R16mR1进行PCR扩增,然后以此扩增产物为模板用R16F2/R16R1进行巢式PCR(表 1)。
|
|
PCR产物回收后与pMD18-T连接,连接产物转化大肠杆菌DH5α感受态细胞,挑取筛选平板上的白色菌落培养,提取质粒,经PCR和酶切鉴定为阳性的重组质粒送上海英骏生物技术有限公司测序。将所得DNA序列输入GenBank进行Blast检索,采用DNASTAR和MEGA3.1软件对所得到的核苷酸序列与GenBank中收录的相应基因的核苷酸序列进行比较和分析,并构建系统进化树。
2 结果与分析 2.1 绣线菊丛枝病病株症状表现感病绣线菊植株表现为节间短缩,生长点长出大量嫩枝丛生呈鸟巢状,叶片黄化、变小畸形、顶部嫩梢枯死,植株矮化(图 1)。

|
图 1 绣线菊丛枝病症状 Fig. 1 Symptoms of spiraea witches'broom 左Left:发病枝条Diseased branch;右Right:健康枝条Healthy branch |
以表现丛枝、叶片黄化等症状的绣线菊总DNA为模板,经巢式PCR扩增,得到长度约为1.2 kb的片段(图 2),与预期大小一致,表明该绣线菊植株中存在植原体。将此植原体分离物暂命名为绣线菊丛枝植原体(spiraea witches'broom,SWB)。

|
图 2 绣线菊丛枝病基因PCR扩增产物凝胶电泳 Fig. 2 PCR amplification of 16S rDNA from phytoplasma associated with spiraea witches'broom M: DNA分子量标准V DNA Marker V; 1:泡桐丛枝植原体16S rDNA作为阳性对照1 6S rDNA o f phytoplasma associated with paulownia witches'broom as positive control; 2:绣线菊丛枝植原体16S rDNA 16S rDNA of phytoplasma associated with spiraea witche s'broom; 3:健康绣线菊DNA作为阴性对照DNA from healthy spirae plants as negativ e control; 4:双蒸水作为空白对照Double distilled water as blank control. |
通过测定2个片段的序列,确定了SWB的16S rDNA基因含有1 236个核苷酸,G+C的含量为47. 0 9%。GenBank登录号为EF176608。RFLP分析表明:SWB(图 3A)与西方翠菊黄化植原体(M86340)有相同的AluⅠ、HhaⅠ和MseⅠ限制性酶切图谱(图 3D)。在翠菊黄化组中,16SrI-A(图 3B), 16Sr I-B(图 3D)和16Sr I-E(图 3C)有相同的MseⅠ酶切图谱(Přibylová et al.,2001),16SrⅠ-A在第52位多一MseⅠ酶切位点,并且16SrⅠ-A和16SrⅠ- E在第1094位少一HhaⅠ酶切位点。因此,可以将侵染绣线菊造成丛枝症状的植原体划分到翠菊黄化组的16SrⅠ-B亚组中。

|
图 3 引起绣线菊丛枝的植原体与翠菊黄化组及其他组中相近植原体316S rRNA的RFLP分析 Fig. 3 RFLP analysis of 16S rRNA of phytoplasmas associated with spiraea witch es'broom and other closely related groups A:绣线菊丛枝病Spiraea witches'broom; B:洋葱黄化病Onion yellows (AY180931, 16SrⅠ-A); C:蓝莓矮化病Blueberry stunt (AY265213, 16SrⅠ-E); D:西方翠菊黄化病Severe aster yellows (M86340, 16SrⅠ-B); E:草莓花瓣变绿症Strawberry vi rescenc e (AY377868, 16Sr Ⅻ); F:苹果丛簇病Apple proliferation phytoplasma (AJ542542, 1 6SrⅩ). |
将该分离物与GenBank中17个植原体分离物的核苷酸序列进行比对和系统进化分析。从系统进化树中可以看出,SWB聚类到16SrⅠ组中,与16SrⅠ-B亚组的西方翠菊黄化植原体、马里兰翠菊黄化植原体及16SrⅠ-D亚组的泡桐丛枝植原体聚集为一簇,从而表明该分离物属于16 SrⅠ-B亚组。SWB分离物与植原体各个组的核苷酸同源性为89.3%~99.6%,与16S rⅠ组中各亚组的分离物核苷酸同源性均达到99%以上,其中与16SrⅠ-B亚组中的西方翠菊黄化植原体(SAY)同源性高达99.6%,说明SWB分离物属于西方翠菊黄化植原体。该分离物与植原体僵顶病组(stolbur group)(16Sr Ⅻ)的草莓花瓣变绿症(strawberry virescence,AY377868)植原体核苷酸的同源性为95.5%,与其他各组的核苷酸同源性在89.3%~92.6%。
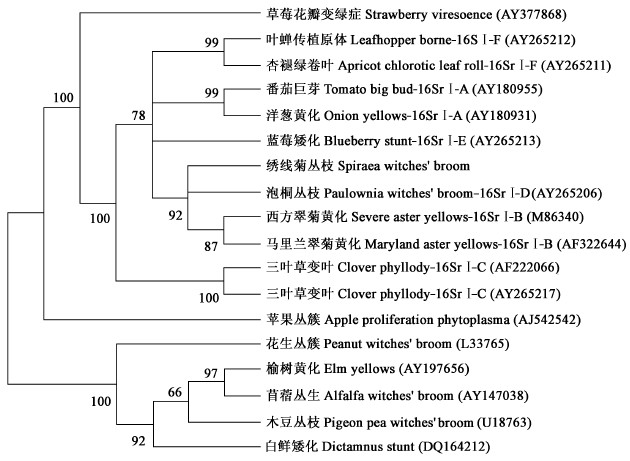
|
图 4 引起绣线菊丛枝的植原体与17个植原体16S rRNA核苷酸序列同源性比较 Fig. 4 Phylogenetic tree based on 16S rRNA nucleotide sequence of phytoplasmas associated with spiraea withces'broom and other 17 phytoplasmas |
本研究用植原体16S rRNA基因的通用引物,通过巢式PCR的方法扩增到了1 236 bp的片段,表明自然表现丛枝症状的绣线菊中存在植原体,该植原体为16Sr I-B亚组的西方翠菊黄化植原体。此前国内未见有植原体侵染绣线菊造成病害的报道。前人对植原体侵染绣线菊研究较少,国外仅报道由植原体16Sr Ⅷ-E亚组引起的绣线菊矮化病(廖晓兰等, 2002)。
The IRPCM photo plasma /spiroplasma working team(2004)认为16S rRNA基因的核苷酸同源性低于97.5%,就可确认为不同候选种。本试验通过对植原体16S rRNA基因的核苷酸同源性比较和RFLP分析,发现该分离物与16SrⅠ组的核苷酸同源性均在99%以上,与16SrⅠ-B亚组中的西方翠菊黄化植原体同源性高达99.6%,故可将绣线菊丛枝植原体初步划分于16SrⅠ-B亚组。
在进行序列比对及构建系统进化树时发现:该分离物与16SrⅠ-B亚组的西方翠菊黄化植原体、马里兰翠菊黄化植原体及16SrⅠ-D亚组的泡桐丛枝植原体的同源性分别为99.6%、99.5%和99.5%,并且在构建的系统进化树中聚集成一簇。Lee等(1998b)认为在基于16S rDNA基因进行序列比对时,16SrⅠ-D亚组的泡桐丛枝植原体会聚集到16SrⅠ-B亚组中,本研究结果与此相符。根据国际比较菌原体学研究计划署(2004)的建议,泡桐丛枝植原体、SAY植原体等都属于同一个种,即‘Candidatus phytoplasma asteris’。
李永, 田国忠, 朴春根, 等. 2005. 我国几种植物植原体的快速分子鉴别与鉴定研究. 植物病理学报, 35(4): 293-299. DOI:10.3321/j.issn:0412-0914.2005.04.002 |
廖晓兰, 朱水芳, 罗宽. 2002. 植原体的分类及分子生物学研究进展. 植物检疫, 16(3): 167-172. DOI:10.3969/j.issn.1005-2755.2002.03.018 |
漆艳香, 谢艺贤, 张辉强, 等. 2004. 植原体DNA提取方法的改良. 生物技术通报, 4: 44-46. |
Bai X, Zhang J, Ewing A, et al. 2006. Living with genome instability: the adaptation of phytoplasmas to diverse environments of their insect and plant hosts. Journal of Bacteriology, 188: 3682-3696. DOI:10.1128/JB.188.10.3682-3696.2006 |
Gundersen D E, Lee I M. 1996. Ultrasensitive detection of phytoplasmas by nested-PCR assays using two universal primer pairs. Phytopathol Mediterr, 35: 144-151. |
Lee I M, Hammond R W, Davis R E, et al. 1993. Universal amplification and analysis of pathogen 16S rRNA for classification and identification of mycoplasmalike organisms. Phytopathology, 83(8): 834-842. DOI:10.1094/Phyto-83-834 |
Lee I M, Gundersen D E, Bertaccini A. 1998a. Phytoplasma: ecology and genomic diversity. Phytopathology, 88: 1359-1366. DOI:10.1094/PHYTO.1998.88.12.1359 |
Lee I M, Gundersen D E, Davis R E, et al. 1998b. Revised classification scheme of phytoplasmas based on RFLP analyses of 16S rRNA and ribosomal protein gene sequences. Int J Syst Evol Microbiol, 48: 1153-1169. |
Marcone C, Lee I M, Davis R E. 2000. Classification of aster yellows-group phytoplasmas based on combined analyses of rRNA and tuf gene sequences. Int J Syst Evol Microbiol, 50: 1703-1713. DOI:10.1099/00207713-50-5-1703 |
Přibylová J, Spak J, Fřnová J. 2001. Association of aster yellows subgroup 16SrI-B phytoplasmas with a disease of Rehmannia glutinosa var. purpurea.. Plant Pathology, 50: 776-781. DOI:10.1046/j.1365-3059.2001.00638.x |
Schneider B, Gibb K S, Seemüller E. 1997. Sequence and RFLP analysis of the elongation factor Tu gene used in differentiation and classification of phytoplasmas. Microbiology, 143: 3381-3389. DOI:10.1099/00221287-143-10-3381 |
The IRPCM Phytoplasma/Spiroplasma Working Team. 2004. `Candidatus Phytoplasma', a taxon for the wall-less, non-helical prokaryotes that colonize plant phloem and insects. Int J Syst Evol Microbiol, 54: 1243-1255. DOI:10.1099/ijs.0.02854-0 |
 2007, Vol. 43
2007, Vol. 43


