文章信息
- 谢国文, 彭晓瑜, 郑燕玲, 张金杏.
- Xie Guowen, Peng Xiaoyu, Zheng Yanling, Zhang Jinxing.
- 濒危植物永瓣藤遗传多样性的ISSR分析
- Genetic Diversity of Endangered Plant Monimopetalum chinense in China Detected by ISSR Analysis
- 林业科学, 2007, 43(8): 48-53.
- Scientia Silvae Sinicae, 2007, 43(8): 48-53.
-
文章历史
- 收稿日期:2006-07-06
-
作者相关文章
群体遗传多样性研究是保护遗传学的基础, 种质资源保护在某种程度上讲就是保护该物种的遗传多样性(顾万春等, 1998;李斌等,2005)。遗传变异在群体间的分配及其分化能够用来阐明该树种的进化史、适应性以及群体遗传结构等(Ge et al., 1998; 邹喻苹等, 2001)。永瓣藤(Monimopetalum chinense)是雷德尔(Rehder, 1926)根据秦仁昌在安徽祁门县棕里村潘坑坞采集的标本发表的新属新种,因其花瓣宿存在果实上不凋落而得名。永瓣藤为卫矛科(Celastraceae)单种属植物,是我国长江中下游南部中亚热带向北亚热带过渡的生态环境中残存的珍稀树种。本属的现代分布范围为赣北、皖南和鄂东南,即北纬28°30′—30°10′, 东经114°30′—118°10′。数量稀少,其分布区域狭窄,且呈间断分布,是本地起源的单型特有属,被列为国家二级珍稀濒危保护植物(傅立国,1989),收入《中国植物红皮书》(傅立国等,1992)。
对永瓣藤的研究以往仅限于生态生物学特征、地理分布、形态解剖、细胞学等方面(谢国文等,1998;1999a;1999b;廖军,1995);笔者亦曾从生物学特性方面探讨过其濒危原因(谢国文,1998)。至今未见有关永瓣藤居群遗传多样性方面的研究报道。野外调查发现,其花不具备引诱昆虫传粉的特征,尚未见到昆虫参与传粉。其次是开花结果初期正逢江南降雨频繁,加之居群规模小和密度低,都严重影响了风媒传粉的效率,从而影响到永瓣藤的有性生殖。克隆植物由于其有效居群的大小难以确定、遗传变异的损失难以觉察,它们的遗传多样性可能在人们意识到这些植物需要保护并采取恰当措施之前严重丧失(Sydes et al., 1998)。因此, 加强克隆植物遗传多样性研究,并采取有效措施加以保护乃是当务之急。本文应用简单重复序列区间扩增多态性(inter-simple sequence repeat, ISSR)这种新型DNA分子标记技术(Zietkiewicz et al., 1994;沈永宝等,2005),分析永瓣藤的遗传多样性,旨在为探讨其濒危机制和制定有效保护措施提供科学依据。
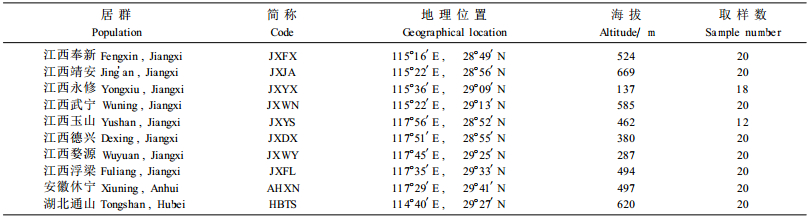
1 材料与方法 1.1 植物材料植物材料分别采自江西、安徽和湖北三省共10个居群,覆盖了永瓣藤整个分布区域,其地理位置、取样数等详见表 1。根据居群的大小,每个居群随机选取了12~20个个体,采样植株的间距30 m以上。每个居群分单株取新鲜叶片,分别置于盛有硅胶的塑胶袋中干燥保存。
|
|
采用CTAB微量法(Doyle et al., 1987)从经硅胶干燥的叶片中提取总DNA,并用电泳法检测其含量和质量。从购自于加拿大哥伦比亚大学的100个引物(UBC set No.9)中筛选出效果好的10个引物(808、810、811、835、841、857、876、880、889和890)用于PCR扩增(周延清,2005)。采用20 μL反应体系:2μL 10×Buffer(10 mmol·L-1 Tris-HCl pH9.0, 50 mmol·L-1 KCl), 2.375 mmol·L-1 MgCl2, 2% Formamide, 0.15 mmol·L-1 dNTPs, 225 mmol·L-1 Primer, 1.5 U Taq DNA聚合酶, 20 ng μL-1模板DNA。扩增程序:首先94 ℃预变性5 min;而后进行35个循环,每个循环包括94 ℃变性15 s、48~50 ℃退火(退火温度随引物而定)45 s、72 ℃复性1 min;最后72 ℃延伸10 min。将扩增产物置于4 ℃冰箱内保存。
1.3 PCR产物鉴定将PCR扩增产物在1.5%琼脂糖凝胶上电泳分离,以上海生工生物工程有限公司的100 bp DNA ladder(100~1 000 bp)作为分子质量标准,溴化乙锭(EB, 0.1 μg·mL-1)染色显带,紫外成像系统(LabWorks Software Version 3.0;UVP, Upland, CA 91786, USA)成像、观察、记录。
1.4 数据统计分析ISSR为显性标记,电泳图谱中的每一条带均视为一个分子标记,并代表一个引物的结合位点。按凝胶同一位置上DNA带的有无进行统计,有带的记为1,无带的记为0, 构成表型数据矩阵。用POPGEN软件(Yeh et al., 1999)对全部群体进行遗传参数分析。分别计算了多态位点百分率(PPB)、等位基因数(Ao)、有效等位基因数(Ae)、基因多样性(He)、Shannon多样性指数(Ho) (Ho= -∑pi log2 pi) (Lewontin,1972)、居群水平上基因多样性(Hpop)、物种水平上基因多样性(Hsp)、居群总基因多样性(HT)、居群内基因多样性(HS)、居群间遗传分化系数(GST)(GST = 1-HS / HT)、Nei's(Nei,1972)遗传距离(D)和遗传一致度(I)。根据Nei's的遗传距离,利用NTSYS PC-2.0(Rohlf, 1994)软件对永瓣藤居群进行聚类分析。同时用Mantel test(Miller, 1997)检测永瓣藤10个居群之间的遗传距离与地理距离之间的关系。
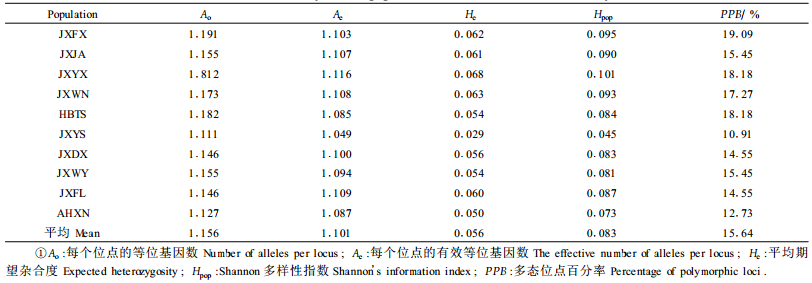
2 结果与分析 2.1 永瓣藤居群遗传多样性10个ISSR引物对永瓣藤10个居群190个单株共扩增出110条带,其中40条是多态带,多态带百分率为39.2%。永瓣藤10个居群中,单个居群的多态带百分率(PPB)为10.91%~19.09%,平均约15.64%。有效等位基因数(Ae)平均为1.049~1.116,平均为1.101。平均期望杂合度指数(He)为0.029 ~ 0.068,平均值为0.056。Shannon多样性指数(Ho)为0.045 ~ 0.101,居群水平上平均值(Hpop)为0.083,物种水平上(Hsp)为0.183。10个居群中,江西永修县居群的遗传变异最高(HPOP为0.101), 而江西玉山县居群的遗传变异最低(HPOP为0.045)(详见表 2), 因为前者居群最大,而后者居群最小。
|
|
根据总的基因多样性(HT=0.128 7)和居群内基因多样性(HS=0.055 7)计算居群间的分化水平(GST)。所分析的10个永瓣藤居群间GST =0.567 2,表明总的遗传变异中有56.72%的变异存在于居群间,居群内遗传变异为43.29%。基因多样性显示,永瓣藤居群间出现了较大的遗传分化。
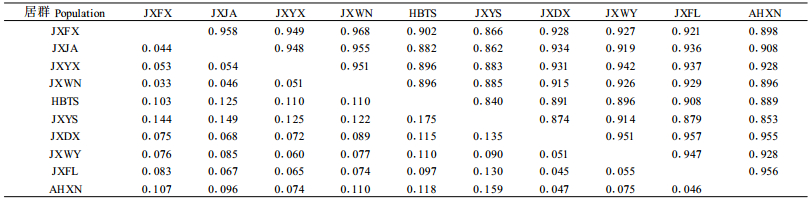
2.3 永瓣藤居群间关系永瓣藤居群间的遗传距离分析(表 3)表明,江西奉新(JXFX)居群和江西武宁(JXWN)居群间的遗传距离最小(D = 0.033),而遗传一致度最大(I = 0.968);江西玉山县(JXYS)居群和湖北通山(HBTS)居群间的遗传距离最大(D = 0.175),而遗传一致度最小(I = 0.840)。前者可能是由于地理位置近、基因交流频率较高所致;而后者可能是由于地理距离最远,使基因交流在一定程度上受阻造成。10个居群间的平均遗传距离D =0.09,Nei's的遗传距离也说明永瓣藤居群间存在着较大的遗传分化。
|
|
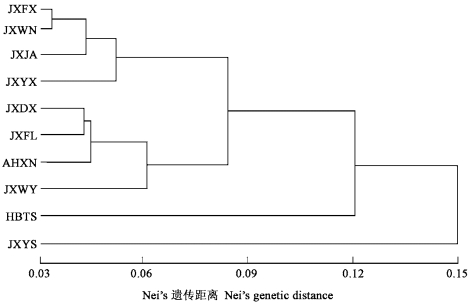
永瓣藤居群间地理距离和遗传距离的Mantel检测表明,地理距离与遗传距离间相关性极显著(r = 0.420, P = 0.001)。永瓣藤10个居群的Nei's遗传距离UPGMA聚类结果(图 1)也反映了遗传距离与地理距离间的相关性。由聚类图看出,间断分布的3块,它们中的居群各自基本聚在一起,即九岭山脉的江西奉新(JXFX)居群、江西武宁(JXWN)居群、江西靖安(JXJA)居群和江西永修(JXYX)居群聚在一起,怀玉山脉江西德兴(JXDX)居群、江西浮梁(JXFL)居群、江西婺源(JXWY)居群和安徽休宁(AHXN)居群聚在一起,湖北通山(HBTS)居群为孤立的一块,而江西玉山(JXYS)居群最小,可能由于居群退化严重、取样数偏少没有与江西德兴(JXDX)居群聚在一起。分析结果表明,永瓣藤居群间分子系统格局与其地理空间格局基本一致。
|
图 1 永瓣藤10个居群的Nei's遗传距离UPGMA聚类图 Fig. 1 UPGMA dendrogram for ten populations of M. chinense based on Nei' s genetic distance |
珍稀濒危保护植物的遗传多样性是近年研究的热点之一。虽然近来不少研究发现有些特有和濒危物种保持较高的变异水平(葛颂等, 1999), 如中国鹅掌楸(Liriodendron chinense)(刘丹等,2006);但一般认为特有种和狭窄分布种与广布种相比, 其遗传多样性较低(金则新等,2004),本研究材料永瓣藤就是如此。
物种的遗传多样性水平受许多因素的影响,包括进化历史、地理分布和物种本身的生物学特征。赣北古板块构造演化历史(舒良树等,1995; 谢国文等, 1991)表明,在地质时期(约在震旦纪),存在一个九岭—怀玉联合体,说明九岭山脉与赣东北—皖南交界的怀玉山脉曾连在一起,并统一增生到扬子板块的东南缘(舒良树等,1995)。永瓣藤在第三纪中新世起源以后沿着九岭—怀玉山脉连续扩散至皖南山区。后来由于九岭—怀玉山脉联合体的分离和鄱阳湖的形成,第四纪后永瓣藤呈间断分布(谢国文等, 1999b)。由于人类活动的干扰使植被破坏严重,永瓣藤呈零星分布。很可能在这一进化过程中,永瓣藤经历了严重的瓶颈效应,丢失了大量的遗传多样性。
永瓣藤的遗传多样性低,可能与其繁育系统有关。永瓣藤主要通过匍匐茎进行无性繁殖,而且其花不具备引诱昆虫传粉的特征,尚未见到昆虫参与传粉(谢国文等, 1999a)。其次是开花结果初期正逢江南降雨频繁,加之居群规模小和居群密度低,这些因素都严重影响了风媒传粉的效率,从而影响到永瓣藤的有性生殖,因此,靠有性生殖补充遗传变异的损失、提高居群内的遗传多样性水平并降低居群间的差异对于永瓣藤居群来说是很难的。并且小的居群规模和密度往往容易导致自交。这些因素都会导致永瓣藤遗传多样性水平的降低。对于克隆植物来说,体细胞突变是其遗传变异的一个持续来源(Esselman et al., 1999),而突变发生的概率非常小。
3.2 永瓣藤的遗传结构与有性繁殖的种相比较,克隆植物的遗传结构有一些变化,一些专性克隆植物,其居群间的遗传多样性远远高于居群内的遗传多样性(Jefferies et al., 1983)。王可青等(1999)在对沙鞭(Psammochloa villosa)的克隆多样性的研究中发现62.16%的遗传变异存在于居群之间,种群分化十分强烈。本研究中,永瓣藤的居群间分化系数GST =0.567 2,说明居群间存在较大的遗传分化。
一个物种的群体遗传结构往往受很多进化因素的影响,包括生境片断化、居群分离、繁育系统、基因流等(Ge et al., 2005; Hamrick et al., 1990; Schaal et al., 1998)。其中,繁育方式对克隆植物的遗传多样性和遗传分化影响很大。永瓣藤基本上以克隆繁殖为主,通过有性生殖和基因流的扩散降低居群间的分化,其作用是相当微弱的。当居群间基因交流存在较大障碍时,其居群间遗传变异的比例将增加,居群内的遗传变异水平将有所降低(夏立群等,2002)。
另外,居群的分布格局和居群规模也会影响物种的遗传结构,由于地质历史的变迁和人为活动的破坏导致永瓣藤呈间断、零星分布,居群规模变小。间断分布的居群由于基因流长期受阻,小居群容易产生自交和基因漂变,从而加剧居群间的遗传分化。
3.3 濒危机制及保护策略永瓣藤遗传多样性和遗传结构的分析表明,永瓣藤居群内遗传多样性很低,居群间分化较大,结合永瓣藤在生态学、生物学特征等方面的研究资料(谢国文, 1998;谢国文等, 1999a),认为永瓣藤目前的濒危状况是许多因素的叠加效应,既来源于该种本身进化中的系统压力,也有外界环境随机因素的作用。
3.3.1 生殖障碍永瓣藤本身的进化潜力低是其濒危的重要原因之一。进化潜力低最直接的表现是繁殖能力与繁殖力低,不足以使受损种群得以恢复和扩展。间接表现为遗传多样性低,不能产生足够的遗传变异,对变化的生境条件适应性差。对永瓣藤的繁殖阶段调查表明,永瓣藤的落花落果和胚珠败育现象较普遍,有的种群很难见到几个果实发育到成熟,甚至8月果枝上还剩下当年暂存的果柄。永瓣藤花小、雄蕊花丝极短,缺乏引诱昆虫传粉的结构特征,定位观察未见到昆虫参与传粉。而且在开花、结果初期正逢江南降水频繁,加之种群规模小和种群密度低,都将影响到风媒传粉的效率,必然导致传粉受精不良,胚珠败育而结实率偏低(谢国文等, 1999a)。永瓣藤种子成熟于秋高气爽的干旱季节(10月左右),因气温仍较高,种子易迅速失水而胚失活或休眠(谢国文等, 1999a)。而且永瓣藤种子小,种皮坚硬,寿命较短,种子萌发障碍难以消除,致使发芽率极低甚至不发芽,自然条件下成苗困难,以致野外调查几乎见不到实生苗。
种群调查表明,永瓣藤能通过匍匐生长习性进行无性繁殖,形成无性系种群。但是永瓣藤总的无性繁殖效率并不高,仅仅靠无性繁殖,难以大幅度提高其种群数量,尤其是不能远距离开拓新的分布区域,这也是永瓣藤分布狭窄的主要原因。
3.3.2 生态因素永瓣藤种群在生境条件、分布格局、规模大小、结构动态等方面表现出一系列问题,证明该物种受到很大的外部环境压力,因分布区和种群缩小,天然更新不良,使物种濒危速度加快(谢国文, 1998)。
3.3.3 保护策略永瓣藤作为国家级珍稀濒危保护植物,其生存环境正在受到威胁,分布区域在逐渐缩小,如果不及时加以保护,该物种将更加濒危直至灭绝。因此应积极采取科学的保护策略。1)由于永瓣藤种群的遗传多样性水平都很低,而且种群间的分化较大。为了尽可能多地保护永瓣藤的遗传资源,建议将所有种群进行保护。对稀有濒危物种来说迁地保存虽然也是必不可少的保护途径,但毕竟受到人力物力的限制。无性繁殖本质上只是无性系遗传物质的转移,因此要保护好永瓣藤的遗传多样性,首先应基于原地保护。在其种群规模较大的地方建立自然保护区,如江西武宁。并结合种群间引种的方法,更有效地保护永瓣藤的遗传多样性。2)加强生殖生理学等基础研究,消除其有性生殖障碍,提高永瓣藤的种子繁殖率。3)开展药用成分研究,在利用的基础上实现有效的保护。据初步分析,永瓣藤是一种具有药用价值的种质资源,若能在大量引种繁殖苗木的基础上开发利用,就可以更好地迁地保护与持续利用。
傅立国. 1989. 中国珍稀濒危植物. 上海:上海教育出版社, 97. |
傅立国, 金鉴明. 1992. 中国植物红皮书:第一册. 北京: 科学出版社.
|
葛颂, 洪德元. 1999. 濒危植物裂叶沙参及其广布种泡沙参遗传多样性的对比研究. 遗传学报, 26(4): 410-417. |
顾万春, 王棋, 游应天, 等. 1998. 森林遗传资源学概论. 北京: 中国科学技术出版社, 1-296.
|
金则新, 李钧敏. 2004. 七子花种群遗传多样性的RAPD分析. 林业科学, 40(4): 68-74. DOI:10.3321/j.issn:1001-7488.2004.04.012 |
廖军. 1995. 永瓣藤植物染色体数研究. 江西农业大学学报, 17(2): 189-190. |
李斌, 顾万春. 2005. 白皮松保育遗传学——天然群体遗传多样性评价与保护策略. 林业科学, 41(1): 57-64. |
刘丹, 顾万春, 杨传平. 2006. 中国鹅掌楸遗传多样性研究. 林业科学, 42(2): 116-119. |
沈永宝, 施季森, 赵洪亮. 2005. 利用ISSR DNA标记鉴定主要银杏栽培品种. 林业科学, 41(1): 202-204. DOI:10.3321/j.issn:1001-7488.2005.01.034 |
舒良树, 施央申, 郭令智, 等. 1995. 江南中段板块——地体构造与碰撞造山运动学. 南京: 南京大学出版社.
|
王可青, 葛颂, 董鸣. 1999. 根茎禾草沙鞭的等位酶变异及克隆多样性. 植物学报, 41(5): 537-540. |
夏立群, 李建强, 李伟. 2002. 论克隆植物的遗传多样性. 植物学通报, 19(4): 425-431. DOI:10.3969/j.issn.1674-3466.2002.04.006 |
谢国文. 1998. 永瓣藤濒危因素的探讨. 热带亚热带植物学报, 6(1): 52-56. |
谢国文, 丁宝章, 王遂义. 1991. 赣北云居山植物区系地理探讨. 云南植物研究, 13(4): 391-401. |
谢国文, 谭策民. 1998. 中国珍稀特有植物永瓣藤生物学特性的研究. 植物学通报, 15(3): 29-33. DOI:10.3969/j.issn.1674-3466.1998.03.006 |
谢国文, 文林. 1999a. 永瓣藤的分布现状及其保护. 生物多样性, 7(1): 15-19. |
谢国文, 孙叶根. 1999b. 中国特有的永瓣藤属植物区系地理性质与特征. 地理研究, 18(2): 130-134. |
周延清. 2005. DNA分子标记技术在植物研究中的应用. 北京: 化学工业出版社, 143-161.
|
邹喻苹, 葛颂, 王晓东. 2001. 系统与进化植物学中的分子标记. 北京: 科学出版社, 1-266.
|
Doyle J J, Doyle J L. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem Bull, 19: 11-15. |
Esselman E J, Li Jianqiang, Crawford D, et al. 1999. Clonal diversity in the rare Calamagrostis porteri ssp. insperata(Poaceae): comparative results for allozymes and random amplified polymorphic DNA(RAPD) and inter simple sequence repeat(ISSR) markers. Molecular Ecology, 8: 443-451. |
Ge S, Hong D Y, Wang H Q, et al. 1998. Population genetic structure and conversation of an endangered conifer, Cathaya argyrophylla (Pinaceae). Int J Plant Sci, 159(2): 351-357. DOI:10.1086/297556 |
Ge X J, Zhang L B, Yuan Y M, et al. 2005. Strong genetic differentiation of the East-Himalayan Megacodon stylophorus (Gentianaceae) detected by inter-simple sequence repeats (ISSR). Biodiversity and Conservation, 14: 849-861. DOI:10.1007/s10531-004-0655-6 |
Hamrick J L, Godt M J W. 1990.Allozyme diversity in plant species//Brown A H D, Clegg M T, Kahler A L, et al. Plant population genetics, breeding, and genetic resources. Sunderland, M A: Sinauer Associates, 43-63
|
Jefferies R L, Gottlieb L D. 1983. Genetic variation within and between population of the asexual plant Puccinelliax phryganodes. Canadan Journal Botany, 61: 774-779. DOI:10.1139/b83-086 |
Lewontin R C. 1972. Apportionment of human diversity. Evolutionary Biology, 6: 381-398. |
Miller M P. 1997.Tools for population genetic analysis: Version 1.3.Arizona, USA: Department of Biological Sciences, Northern Arizona University
|
Nei M. 1972. Genetic distance between populations. American Nature, 106: 283-292. DOI:10.1086/282771 |
Rehder A. 1926. Monimopetalum, a new genus of Celastraceae. J of the Arnol Arboretum, Ⅶ(4): 233-234. |
Rohlf F J. 1994.NTSYS-PC: Version 2.10. New York: Applied Biostatistics Inc
|
Schaal B A, Hayworth D A, Olsen K M, et al. 1998. Phylogeographic studies in plants: problems and prospects. Molecular Ecology, 7: 465-474. DOI:10.1046/j.1365-294x.1998.00318.x |
Sydes M A, Peakall R. 1998. Extensive clonality in the endangered shrub Haloragodendron lucasii (Haloragaceae) revealed by allozymes and RAPDs. Molecular Ecology, 7: 87-92. DOI:10.1046/j.1365-294x.1998.00314.x |
Yeh F C, Yang R, Boyle T, et al. 1999.POPGENE, Microsoft Windows-based freeware for population genetic analysis: Release 1.31.Edmonton, Canada: University of Alberta
|
Zietkiewicz E, Rafalski A, Labuda D. 1994. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification. Genomics, 20: 176-183. DOI:10.1006/geno.1994.1151 |
 2007, Vol. 43
2007, Vol. 43