文章信息
- 邓柳红, 罗明武, 曾会才, 杨卫帆, 张春发.
- Deng Liuhongx, Luo Mingwux, Zeng Huicaix, Yang Weifanx, Zhang Chunfax.
- 用抑制消减杂交法分离巴西橡胶树胶乳特异表达基因
- Isolation of Differentially Expressed Genes in Latex of Hevea brasiliensis by Suppressive Subtractive Hybridization
- 林业科学, 2006, 42(6): 32-36.
- Scientia Silvae Sinicae, 2006, 42(6): 32-36.
-
文章历史
- 收稿日期:2004-10-29
-
作者相关文章
2. 华南热带农业大学 儋州 571737
2. South China University of Tropical Agriculture Danzhou 571737
世界上有2 000多种植物可以生产天然橡胶, 而大戟科(Euphorbiaceae)的巴西橡胶树(Hevea brasiliensis)是唯一能生产高质、高量天然橡胶、具有商业价值的树种, 该植物因经济寿命长、易栽培采割, 胶质好、产胶多而得以大量种植(何康等, 1987)。随着巴西橡胶树的大规模种植, 有关巴西橡胶树形态、产胶生理及栽培学等方面的研究已取得了较大的进展。但是在分子生物学方面的研究仍很薄弱, 所报导克隆的cDNA大都用异源同类基因从巴西橡胶树构建的cDNA文库获取, 从巴西橡胶树克隆的新基因不多, 难以从整体水平上解释胶乳再生的分子机制。特别是有关橡胶转移酶(橡胶生物合成关键酶)基因的克隆及其调控的研究都还有待进一步深入。
天然橡胶是在乳管的细胞质——胶乳里合成的, 因此在胶乳里特异表达的基因可能与橡胶生物合成有关。Kush等(1990)研究表明几个与橡胶合成相关的基因在胶乳中特异表达。为了进一步了解橡胶合成的分子机制, 找出与橡胶生物合成相关的基因, 美国密歇根州立大学通过EST和cDNA-AFLP获得了一些差异片段, 并对它们进行了综合分析, 但是没有获得与cis-prenyltransferase基因(橡胶合成关键酶基因)有同源性的差异片段(Han et al., 2000; Ko et al., 2003)。为了找出与橡胶生物合成相关的基因, 本研究首次采用能有效分离低丰度差异表达基因的抑制消减杂交(SSH)技术构建了一个胶乳特异表达基因差减文库, 序列比较结果表明所分离的基因不仅包括一些已报导的, 还有一些未报导的基因, 获得了一个与cis-prenyltransferase基因有同源性的差异片段。这些基因在橡胶生物合成中可能起着直接或间接的作用, 它们的功能还有待进一步的探索与研究。
1 材料与方法 1.1 材料试验材料为巴西橡胶树优良品系“热研73397”, 胶乳及叶片采集后立即放入液氮中保存备用。
1.2 方法 1.2.1 巴西橡胶树叶片及胶乳总RNA的提取和mRNA纯化胶乳总RNA的提取参照Kush等(1990)的胶乳提取方法。叶片总RNA的提取采用SDS-LiCl法。检测总RNA浓度、纯度及完整性。采用Promega公司的PolyATtract® mRNA Isolation Systems Ⅲ进行mRNA纯化。
1.2.2 抑制消减杂交及差减cDNA文库构建抑制消减杂交具体操作参照Clontech公司的抑制消减杂交试剂盒(Clontech PCR-SelectTM cDNA Subtraction Kit)进行, 以巴西橡胶树常割树胶乳为试验方(Tester), 叶片为驱动方(Driver)。将正向差减PCR产物克隆至TVector pMD18, 保存于Escherichia coli XL1-Blue菌株中, 建立正向差减cDNA文库。
1.2.3 菌落PCR鉴定差减cDNA文库重组子将保存的菌种对应接种到96孔细胞培养板中培养, 取2 μL做模板用于菌落PCR。以差减片段接头即Nested PCR Primer 1(5′-TCGAGCGGCCGCCCGGGCAGGT-3′), Nested PCR Primer 2R(5′-AGCGTGGTCGCGGCCGAGGT-3′)为引物, 检测文库插入片段大小。
1.2.4 Reverse Northern Dot-Blot筛选鉴定阳性差异片段采用Roche的地高辛标记和检测试剂盒(DIG High Prime DNA Labeling and Detection Starter Kit11)进行杂交检测。挑取PCR阳性克隆, 在每个阳性克隆PCR产物中加入1 mol·L-1 NaOH及200 mmol·L-1 EDTA, 使其终浓度为0.4 mol·L-1 NaOH/10 mmol·L-1 EDTA, 在100 ℃变性10 min, 将各管离心5 s。将样品点于带正电荷的尼龙膜上(准备2套重复拷贝膜)。胶乳(Tester)和叶片(Driver)的总RNA用地高辛进行反转录标记后, 作为探针, 分别与点在尼龙膜上的差减cDNA文库阳性克隆的菌落PCR产物进行杂交, 然后洗膜及免疫检测。
1.2.5 Northern杂交和RT-PCR分析取胶乳和叶片总RNA各15 μg, 甲醛变性凝胶电泳, 转膜。从Reverse Northern Dot-Blot筛选的阳性克隆中随机挑选5个cDNA克隆, 用Northern Blot杂交验证, 具体操作按Roche的地高辛标记和检测试剂盒(DIG High Prime DNA Labeling and Detection Starter Kit11)进行。对在胶乳Tester和叶片Driver中均不能检出杂交信号的克隆, 用RT-PCR进一步验证。分别以等量的叶片和胶乳总RNA为模板, 进行反转录, 合成单链cDNA, R329的RT-PCR的正义引物为5′-CGGAATTCATGTCAAAGAGTTACTTCA-3′, 反义引物为5′-GTCGACTTAGACAATCCTCACGGAA-3′; R291的RT-PCR的正义引物为5′-GAAACTGGAATGTTGACAA GGCA-3′, 反义引物为5′-GACATTTGTTTTGGACCCTCAGT-3′。以18S rRNA的扩增作为内参来校正模板的用量。R329 RT-PCR程序为:94 ℃变性5 min, 然后94 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 50 s, 25个循环, 再72 ℃ 10 min; R291 RT-PCR程序为:94 ℃变性5 min, 然后94 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 50 s, 20个循环, 再72 ℃10 min。
1.2.6 cDNA片段测序及序列分析随机挑选18个通过Reverse Northern Dot-Blot筛选鉴定的阳性差异片段送上海生物工程技术有限公司测序, 所有测出的序列去除载体序列及引物序列后, 通过网络提交NCBI进行BLASTN和BLASTX分析, 推测差异片段的功能。
2 结果与分析 2.1 巴西橡胶树叶片和胶乳总RNA的提取结果取5 μg巴西橡胶树叶片和胶乳的总RNA样品进行甲醛变性凝胶电泳, 电泳结果说明所提取的巴西橡胶树叶片和胶乳总RNA完整性好, 几乎无降解, 28S rRNA与18S rRNA的比率大于1:1。紫外分光光度计检测结果表明几乎没有蛋白质污染、酚和多糖类物质等污染。本研究提取的巴西橡胶树叶片和胶乳的总RNA的纯度和完整性均符合抑制消减杂交实验的要求。
2.2 巴西橡胶树胶乳和叶片表达差异SSH结果 2.2.1 PCR扩增结果分析以2次消减杂交后获得的cDNA为模板再进行2次PCR扩增, 扩增后的cDNA群体与克隆载体连接, 转化, 构建成差减cDNA文库。在第1次PCR扩增中, 由于进行抑制PCR, 只有差异表达基因得到指数级扩增, 而随后进行的第2轮巢式PCR扩增, 可进一步降低背景, 富集差异表达基因。差减产物与未做减法的Control相比, 带型有差异, 而且经第2轮巢式PCR扩增后, 差异表达基因得到了富集(图 1)。
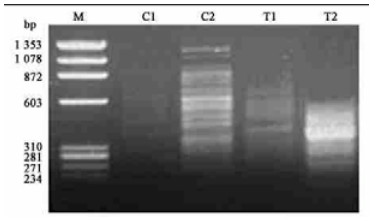
|
图 1 第1次PCR扩增和第2次PCR扩增电泳结果 Fig. 1 Results of the primary and secondary PCR products of SSHM:2 000 bp ladder marker; C1:未扣除的对照第一次PCR Primary PCR of control; C2:未扣除的对照第二次PCR Secondary PCR of control; T1:扣除的Tester第一次PCR Primary PCR of SSH; T2:扣除的Tester第二次PCR Secondary PCR of SSH. |
构建好的差减cDNA文库经蓝白斑筛选和菌落PCR鉴定后, 获得398个含插入片段的阳性克隆。菌落PCR用Nested Primer 1和Nested Primer 2R为引物进行扩增, 所扩增的片断介于200~600 bp之间(图 2)。由于用于构建cDNA差减文库的cDNA序列须经4个碱基的识别酶RsaⅠ(理论上每44=256个碱基中就有一个酶切位点)切割, 而本研究的插入片断介于200~600 bp间, 和理论预计的结果大致相符。
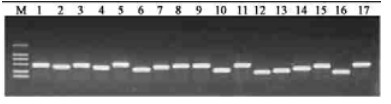
|
图 2 菌落PCR检测SSH文库插入片段大小 Fig. 2 Analysis of the insert size of SSH library by PCR M:2 000 bp ladder marker; 1~17:随机挑选的克隆Clones picked at random. |
将胶乳(Tester)和叶片(Driver)的总RNA用地高辛进行反转录标记后, 作为探针, 分别与点在尼龙膜上的差减cDNA文库阳性克隆的菌落PCR产物进行Reverse Northern Dot-Blot, 经洗膜、压片、显影和定影后, 将胶乳(Tester)X-光片上的杂交信号与叶片(Driver)X-光片上的杂交信号进行对比分析后, 找出在胶乳(Tester)中杂交信号强于叶片(Driver)的克隆, 及在胶乳(Tester)有杂交信号而在叶片(Driver)中没有杂交信号的克隆共79个, 另有一些克隆在胶乳(Tester)和叶片(Driver)中均无杂交信号。
2.2.4 Northern Blot分析和RT-PCR分析从Reverse Northern Dot-Blot筛选的阳性克隆中随机挑选5个cDNA克隆用Northern Blot杂交进一步验证(图 3), R363、R183、R30共3个克隆在胶乳Tester中有杂交信号, 而在叶片Driver中没有; 而R291、R329两个克隆在胶乳Tester和叶片Driver中均不能检出任何杂交信号。推测原因可能是丰度太低以至无法由总RNA进行的Northern杂交检出。对在胶乳Tester和叶片Driver中均不能检出任何杂交信号的R291、R329两个差异片段进一步进行RT-PCR分析后发现, 2个差异片段均只在胶乳中表达(图 4)。
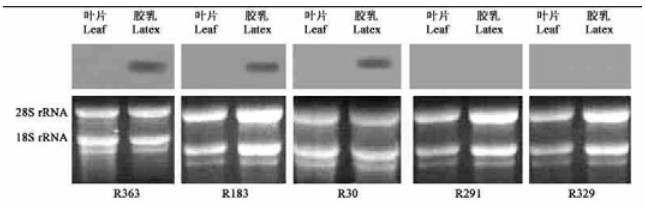
|
图 3 Northern blot验证阳性克隆结果 Fig. 3 Verification of the positive clones by Northern blot |
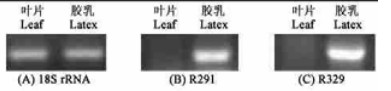
|
图 4 RT-PCR验证阳性克隆 Fig. 4 Verification of the positive clones by RT-PCR (A)对照Control(18S rRNA); (B)R291:mRNA在叶片和胶乳中不同表达mRNA expression in leaf and latex; (C)R329:mRNA在叶片和胶乳中不同表达mRNA expression in leaf and latex. |
从Reverse Northern Dot-Blot鉴定的79个阳性克隆中随机挑选18个克隆送上海生物工程技术有限公司测序, 所有测出的序列去除载体序列及引物序列后, 在GenBank中进行核酸和蛋白质同源比较, 结果表明:有5个克隆与巴西橡胶树中已知的基因相同, 这些基因在橡胶生物合成中有积极的作用(Oh et al., 1999; Light et al., 1989; Asawatreratanakul et al., 2003); 2个克隆与GenBank上公布的巴西橡胶树顺式-异戊烯基转移酶同源性高; 11个克隆在巴西橡胶树中是新发现的, 其中6个克隆通过RACE技术获得了5个全长cDNA序列(另文发表), 5个在GenBank中查询不到相关的同源序列, 它们可能是功能未知的新基因, 或是靠近3′末端的基因片段, 同源区段太短, 难以通过同源比较推测其功能(表 1)。
|
|
SSH是由Diatechenko等(1996)建立的差异表达基因分离技术, 它具有假阳性率低, 能有效分离低丰度差异表达基因的优点。因此本研究在构建胶乳特异表达基因的差减cDNA文库时, 采用了该技术, 同时选取了胶乳再生比较旺盛时期——刚结束排胶的常割树胶乳作为样品。初步结果表明所分离的基因不仅包括一些已报导的, 还有一些未报导的基因, 它们在橡胶生物合成中起着直接或间接的作用; 同时还获得了一个与cis-prenyltransferase基因高度同源的差异基因片段, 通过RACE扩增该片段的5’和3’末端, 获得了一个cDNA全长序列(登录号AY461414), 该结果另文介绍。而美国密歇根州立大学通过EST和cDNA-AFLP 2种方法从胶乳中得到的差异片段中并不含有与cis-prenyltransferase基因(橡胶合成关键酶基因)有同源性的差异片段(Han et al., 2000; Ko et al., 2003)。
橡胶的生物合成由蔗糖开始, 经过糖酵解, 三羧酸循环生成乙酰辅酶A。然后以乙酰辅酶A为原料通过一系列相关的酶促反应合成橡胶。因此橡胶的生物合成与巴西橡胶树的生理代谢是密切相关的(Cornish et al., 1993; Kekwick, 1989)。
本研究通过对部分差异片段进行序列分析及比较, 发现由SSH法获得的差异片段与橡胶生物合成、信号传导和物质代谢运输等相关。割胶是一种机械刺激, 它可诱导巴西橡胶树体内产生伤害乙烯, 而研究发现乙烯可以提高乳管细胞膜的透性, 加速胶乳流速, 影响胶乳的代谢(Amalon et al., 1992)。由此推测割胶可能通过影响巴西橡胶树的生理代谢功能进而影响到胶乳的再生。割胶后的排胶因引起乳管细胞质的流失, 需要在乳管内建立一种新的代谢平衡, 来补充每次割胶后损失的细胞质(包括蛋白), 这时可能就会启动与胶乳再生相关基因的表达, 作者推测橡胶生物合成是由细胞质再生需求所诱导的, 而割胶等伤害刺激则通过影响巴西橡胶树的生理代谢来对橡胶生物合成起到间接的促进作用。
何康, 黄宗道. 1987. 热带北缘巴西橡胶树栽培. 广州: 广东科技出版社, 398-402, 407-416.
|
Amalon Z, Bangratz J, Chrestin H. 1992. Ethrel induced increases in the adenylated pool and transtonoplast pH with latex cells. Plant Physiol, 98: 1270-1276. DOI:10.1104/pp.98.4.1270 |
Asawatreratanakul K, Zhang Y W, Wititsuwannakul D, et al. 2003. Molecular cloning, expression and characterization of cDNA encoding cis-prenyltransferases from Hevea brasiliensis. Eur J Biochem, 270: 4671-4680. DOI:10.1046/j.1432-1033.2003.03863.x |
Cornish K, Pan Z, Backhaus R A. 1993.Engineering new domestic sources of natural rubber//Janick J, Simon J E (eds).New Crops.New York: Wiley, 192-196
|
Diatechenko L, Lau Y F C, Campbell A P, et al. 1996. Suppression subtractive hybridization:A method for generating differentially regulated or tissue-specific cDNA probes and libraries. Proc Natl Acad Sci USA, 93: 6025-6030. DOI:10.1073/pnas.93.12.6025 |
Han K H, Shin D H, Yang J M, et al. 2000. Genes expressed in the latex of Hevea brasiliensis. Tree Physiology, 20: 503-510. DOI:10.1093/treephys/20.8.503 |
Kekwick R G O. 1989.The formation of isoprenoids in Hevea latex//d'Auzac J, Jacob J L, Chrestin H. Physiology of rubber tree latex. CRC Press, Inc, Boca Raton, 145-164
|
Ko J H, Chow K S, Han K H. 2003. Transcriptome analysis reveals novel features of the molecular events occurring in the laticifers of Hevea brasiliensis (the para rubber tree). Plant Molecular Biology, 53(4): 479-492. DOI:10.1023/B:PLAN.0000019119.66643.5d |
Kush A, Goyvaerts E, Chye M L, et al. 1990. Laticifer specific gene express in Hevea brasiliensis. Proc Natl Acad Sci USA, 87: 1787-1790. DOI:10.1073/pnas.87.5.1787 |
Light D R, Dennis M S. 1989. Rubber elongation factor from Hevea brasiliensis. J Biol Chem, 264(31): 18608-18617. |
Oh S K, Kang H, Shin D H, et al. 1999. Isolation, characterization and functional analysis of a novel cDNA clone encoding a small rubber particle protein from Hevea brasiliensis. J Biol Chem, 274(24): 17132-17138. DOI:10.1074/jbc.274.24.17132 |
 2006, Vol. 42
2006, Vol. 42


