文章信息
- 乌云塔娜, 谭晓风, 李秀根, 曹玉芬, 张琳.
- Wuyun Tana, Tan Xiaofeng, Li Xiugen, Cao Yufen, Zhang Lin.
- 梨自交不亲和新基因S12-RNase的分离鉴定及序列分析
- Isolation and Identification of S12-RNase Gene of Pyrus bretschneideri
- 林业科学, 2006, 42(4): 117-121.
- Scientia Silvae Sinicae, 2006, 42(4): 117-121.
-
文章历史
- 收稿日期:2005-03-16
-
作者相关文章
2. 中国农业科学研究院郑州果树研究所 郑州 450009;
3. 中国农业科学研究院果树研究所 兴城 121600
2. Zhengzhou Fruit Tree Research Institute, The Chinese Academy of Agricultural Sciences\ Zhengzhou 450009;
3. Fruit Tree Research Institute, The Chinese Academy of Agricultural Sciences Xingcheng 121600
植物自交不亲和性(self-incompatibility, 简称SI)是植物生殖过程中的一种普遍现象(Hiscock et al., 1996; Andrew et al. , 1996)。它作为防止近亲繁殖和物种退化的异花授粉机制,为物种的生存、发展以及种群的相对独立性提供了有力的保障(Stone, 2002; Yunjun, 1990)。但该性状给有些植物的生产栽培带来了不便,如蔷薇科(Rosaceae)植物苹果(Malus domestica)、沙梨(Pyrus pyrifolia)、西洋梨(Pyrus communis)、甜樱桃(Cerasus avium)、樱桃(Cerasus pseudocersus)、李(Prunus salici na)、梅(Armeniaca mume)、山杏(Armeniaca vulgaris)等物种的多数栽培品种均存在配子体自交不亲和性(gametophytic self-incompatibility, 简称GSI),生产栽培中必须配置授粉品种或通过人工授粉等措施来保证高产稳产;而配置授粉品种时必须考虑品种间的亲和性。因此确定品种的自交亲和程度和品种间的亲和性是丰产栽培中的关键环节之一。过去,国内外主要通过品种间的杂交座果率来确定其亲和性(Nettancourt, 1977; Lewis, 1979),而近来日本学者发现不同品种S-RNase基因型与杂交亲和性间存在密切的关系,即当相同S-RNase基因型的品种进行杂交时则表现为不亲和,否则表现为亲和,因此国外主要采用分子生物学方法确定品种S-RNase基因型,然后通过适当的杂交试验准确确定品种间的亲和性(Ishimizu et al., 1999; Shogo et al., 1999),因此,分离鉴定品种S-RNase基因或确定S-RNase基因型可以为授粉品种的配置提供理论依据。我国梨品种资源非常丰富,栽培品种数量多,但对这些品种的S-RNase基因以及S-RNase基因型的确定工作均未进行。生产栽培中,常常靠经验来判断品种间的亲和程度。为了减少授粉品种配置中的盲目性,为授粉品种配置提供科学依据,作者从2001年开始开展了中国沙梨( P. pyrifolia)和白梨(P. bretschneideri)S-RNase基因的分离鉴定和S-RNase基因型确定方面的研究工作,共试验了白梨92个品种和沙梨78个品种,并从中分离鉴定了18个新的S-RNase基因,结合分子生物学方法和杂交试验方法确定了35个梨品种的S基因型(谭晓风等, 2005)。本文报道白梨S12-RNase基因的分离鉴定及序列分析情况。
1 材料与方法 1.1 植物材料从中国农业科学院果树研究所采集白梨品种大凹凹和硬枝青的嫩叶300 mg左右,存储于液氮中。带回实验室后于-70 ℃冷冻保存。
1.2 总DNA提取和纯化总DNA提取主要依据改良的CTAB方法(乌云塔娜等, 2003),纯化按照《精编分子生物学实验指南》(奥斯伯等,1998)的方法。
1.3 酶、试剂、试剂盒DNA回收试剂盒、高保真酶购自日本TAKARA公司,Taq DNA聚合酶购自上海生工。
1.4 引物梨S基因(图 1)由5个保守区、一段高变区(hyper variable re gion,HV)以及一段内含子序列组成。而梨S-等位基因的多态性主要来自于HV区的变异,且S-RNase HV区正是与花粉S基因产物相互识别的部位,通常根据HV区的不同来确认不同的雌蕊S-等位基因。因此根据梨S1~S10等位基因的HV区两端的保守序列C1区的‘FTQQYQ’ (F1)和‘IIWPNV ’(R)设计合成引物(引物位置如图 1所示)。F2引物是根据起始密码子上游区32 bp附近保守序列来合成的。引物由上海基康生物工程有限公司合成。引物序列分别为F1:‘FTQQYQ’(5′-TTTACGCAGCAATATCAG-3′);R:‘IIWPNV’(5′-AC(A/G)TTCGGCCAAATAATT-3′);F2:‘5′-32bp’(5′-TGCCTCGCTCTTGAACAAA-3′)。F1和R一对引物可以扩增出梨S基因F1+C1+C2+HV+内含子+R的片段。F2和R一对引物可以扩增出梨S基因-32 bp+信号肽+ F1+C1+C2+HV+内含子+R的片段。
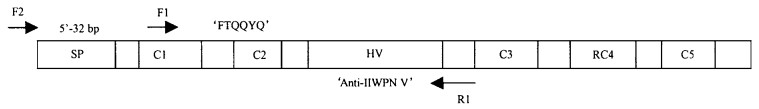
|
图 1 梨S基因结构及引物的位置 Fig. 1 The structure of pear S-gene and the position of primers SP:信号肽The signal peptide; HV:高变区(含内含子) Hyper variable region(containing intron); C1~C5:保守区Conserved region; 空格Blank:外显子Exon. |
PCR扩增按照谭晓风等(2002)的PCR扩增反应体系和循环条件。PCR产物测序由上海基康生物技术有限公司进行。
1.6 HV区域的确定S基因识别花粉的特异功能区是S-RNase的HV区,变异非常丰富,因此通常根据HV区的差异来区别不同的S基因。梨S-RNase基因HV中含有一段内含子序列。该研究根据双子叶植物内含子/外显子保守的边界序列(5′GT—AG3′)(Norilka et al.,1996)和日本沙梨(P. pyrifolia)S-RNase基因的一级结构特点(Castillo et al.,2002),查找HV区中的内含子序列,并获得外显子序列,推导出HV区+周边区的氨基酸序列,进一步比较确认该S基因。
1.7 田间杂交试验田间杂交试验选择S基因型已经确定的梨品种与含有新S-RNase基因的大凹凹和硬枝青进行杂交,并统计座果率。当座果率≤30%时,将影响产量,认为不亲和;座果率≥60%,不影响产量,认为亲和;而30%≤座果率≤60%,为难以确定(Ishimizu et al.,1999)。杂交试验方法参考刘助生(2001)的方法,所有杂交试验均选择成熟雌蕊和成熟花粉,去雄。
2 结果与分析 2.1 梨品种自交不亲和基因的特异性PCR扩增利用S-RNase基因引物‘FTQQYQ’和‘IIWPNV’,对白梨品种大凹凹和硬枝青基因组DNA进行S-RNase特异性PCR扩增结果表明,PCR产物中出现一条530 bp左右的片段(图 2),而该片段PCR产物相当大小的片段在日本沙梨70多个品种和韩国沙梨近20个品种的PCR扩增中均未出现。已鉴定的梨属(Pyrus)S-RNase基因PCR产物大小S1-RN ase基因为375 bp,S2-RNase为1 355 bp,S3- RNase为384 bp,S4-RNase为3 76 bp,S5-RNase为384 bp,S6-R Nase为355 bp,S7-RNase为360 bp,S8- RNase为442 bp,S9-RNase为1 323 bp,S10-RNase为1 718 bp,S11-RNase为363 bp。由此可见,所分离的片段显然不是S1~11-等位基因,因此对该片段进行了回收和测序。
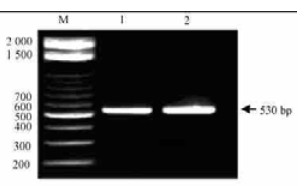
|
图 2 白梨品种S基因特异性引物PCR扩增 Fig. 2 Amplification of S-RNase fragments from Pyrus bretschneideri M. DNA marker;1.硬枝青Yingzhiqing;2.大凹凹Daaoao. |
分别回收和测序大凹凹和硬枝青的530 bp左右的片段。测序后确认了该片段大小为536 bp。与GenBank+EMBL+DDBJ+PDB数据库的基因资源进行BlastN相似性比较结果显示,至少能搜索到80个其他植物种的S-RNase序列,并与所搜索的序列具有部分序列高度同源性,相似性达70%~90%,说明该片段是S-RNase片段。
2.3 HV区的比较利用相似性比较和序列分析,获得了该片段的HV区+周边区DNA和推导氨基酸序列。该片段与梨S1~11- RNase基因间的推导氨基酸的一致性分别为67%、72%、66%、67%、68%、68%、65%、64%、64%、56%和67%。S1~11-RNase基因的HV区长度为13~18个氨基酸,新分离片段的长度为13个氨基酸。序列比较结果(图 3)表明,在HV区中,新分离片段与S1~11-RNase基因分别有11、9、12、12、11、12、15、13、8、11、13个氨基酸存在突变、缺失或插入。因此该片段是一个新的S-RNase,继沙梨11个S-RNase基因后,将该基因命名为S12-RNase基因(GenBank中的接受号为AY249427)。

|
图 3 S1~12-等位基因的HV+周边区氨基酸序列比较 Fig. 3 Alignment of amino acid sequences around the HV region of S1~12-RNase in the pear HV(hyper variable region) |
以S基因型已知的沙梨品种作为母本与硬枝青进行杂交试验,结果见表 1。由表 1可看出,沙梨品种今村秋(S1S6)、二十世纪(S2S4 )、丰水(S3S5)、晚三吉(S5S7)、明月(S1S8 )、新高(S3S9)(Ishimizu et al., 1999)与硬枝青的杂交座果率为64.00%~90.12%,说明硬枝青与含有S1~9-RNase的沙梨品种间的杂交是亲和的,同时说明了白梨品种硬枝青不含梨S1~9-RNase。分别以今村秋、二十世纪、丰水、晚三吉、明月、新高为母本与大凹凹进行杂交的试验也出现类似的情况。今村秋、二十世纪、丰水、晚三吉、明月、新高与大凹凹的杂交座果率在62.45%~88.67%(表略),大凹凹与以上品种的杂交是亲和的,证明了大凹凹中不含梨S1~9-RNase。
|
|
S12-RNase等位基因DNA和推导氨基酸序列见图 4。利用已知的梨S1~11-RNase基因一级结构和网上在线资源对该片段DNA序列进行了生物信息学分析。结果表明,该片段的内含子大小为341 bp(图 4虚下划线部分),插入在HV区(图 4双下线部分)中。分离片段中还包含了保守的半胱氨酸残基Cys-44、Cys-51、Cys-77 (图 4粗线部分),以及保守的组氨酸残基His-61(图 4斜黑体H)。S1~12-RNase基因内含子大小排序:S10(1 303 bp)>S 2(1 153 bp)>S12(341 bp)>S8(234 bp)>S3(179 bp)>S 5(169 bp)>S1、S4(168 bp)>S11(167 bp)>S7(151 bp)>S6(147 bp)。用另一对引物F2和R对大凹凹和硬枝青2个品种的基因组DN A扩增或测序结果表明,PCR产物长度为632 bp(图略)。S1~12-RNase基因信号肽+C1+C2+HV+内含子+R区域DNA大小排序为:S10(1 606 bp)>S2(1 444 bp)>S12(632 bp)>S8(534 bp)>S3、S5(473 bp)>S 4(465 bp)>S1、S11(464 bp)>S7(451 bp)>S6(44 4 bp)。S1~12-RNase基因信号肽+C1+C2+HV+内含子+R区域氨基酸长度排序为:S 10(101个)>S1、S4、S6、S7、S8、S11(99个)>S3、S5、S21、S22(98个)>S2、S12(97个)。

|
图 4 S12-等位基因的HV+周边区序列特点 Fig. 4 Alignment of amino acid sequences around the HV region of S12-RNase in the P. bretschneideri 保守区C1:下划线;内含子:虚下划线;半胱氨酸残基:粗线;组氨酸残基:粗斜体;高变区:双下划线。 C1:Underline; Intron:Dashed underline; Cys:Heavy line; His:Italic;HV region: Crewel. |
自交不亲和性是显花植物中普遍存在的防止近亲繁殖和促进远缘杂交的一种遗传机制,其分子控制是由S位点上的2个单独的基因所决定,这2个基因分别控制雌蕊自交不亲和性和花粉自交不亲和性(Sijacic et al., 2004;Hong et al., 2004)。目前对自交不亲和性的机理具有2种假设,但尚未用直接的试验结果来证明哪种模型更为准确。无论雌蕊自交不亲和因子消化花粉RNA导致自交不亲和(Steinbachs et al., 2002;Tomimoto et al.,1996),或是花粉自交不亲和因子与雌蕊自交不亲和因子相结合并导致自交不亲和性的产生(Sijacic et al., 2004), 必须先要确定雌蕊自交不亲和因子和花粉自交不亲和因子,才能进一步研究各种植物自交不亲和性及其在生产中的应用。而日本对日本沙梨的自交亲和性进行了大量的研究, 在1992—2000年间分别从蛋白质水平和DNA水平上分离鉴定了日本沙梨的7个雌蕊S-RNase (Sassa et al.,1993;Ushijima et al.,1998),2000—2001年,用分子生物学方法和杂交试验方法分离鉴定了S8-和S9-RNase基因(佐藤昭宏,2001)。2003年,韩国学者在韩国沙梨中发现S10-RNase基因(提交至GenBank),中国沙梨中发现S11-RNase基因(谭晓冈等,2005)。本研究依据PCR产物大小差异、HV区大小及氨基酸序列差异、内含子大小以及杂交试验方法鉴定了S12-RNase基因。S1~11-RNase基因的PCR产物大小分别为S1=464 bp,S2=1 444 bp,S3=473 bp,S4=465 bp,S5=473 bp,S6=444 bp,S7=451 bp,S8=534 bp, S9=1 333 bp,S10=1 606 bp,S11=4 64 bp, 而S12基因大小为632 bp,目前为止在日本沙梨、韩国沙梨中均未发现与该基因相当大小的基因,而在中国白梨和沙梨的几个地方品种中发现了S12基因的存在,如作者从大凹凹、硬枝青、海棠酥、红皮酥、天生伏等白梨品种中成功地分离到了S12基因;HV区是区别不同S基因的主要区域,生物信息学分析表明S12 基因HV区氨基酸为13个,而S1~11-RNase基因分别为S1、S4、S6、S7、S8、S9=15AA,S2=12AA,S3、S5=14AA,S10=18AA,而且与S1~11-RNase基因分别有11、9、12、12、11、12、15、13、8、11、13个氨基酸存在差异;S12基因内含子大小为41 bp,而S1~11-RNase基因的内含子大小分别为S1=168 bp、S2=1153 bp、S3=179 bp、S4=168 bp、S5=169 bp、S6=147 bp、S7=151 bp、S8=234 bp、S9=1 129 bp、S10=1 303 bp、S11=167 bp,而且在内含子中40%以上的序列发生突变、插入或缺失;含有S12基因的梨品种与含有S1~9-RNase基因的沙梨品种今村秋(S1S6)、二十世纪(S2S4)、丰水(S3S5)、晚三吉(S5S7)、明月(S1S8)、新高(S3S9)的正反交座果率均大于60%,表现为亲和,进一步证明了该基因的存在。目前正在进行S12基因的基因组全长克隆和结构与功能关系研究。
奥斯伯F M, 金斯顿R E, 赛德曼J G.1998.精编分子生物学实验指南.马学军, 舒跃龙, 译.北京: 科学出版社
|
刘助生. 2001. 日本梨授粉品种的选择及人工授粉技术. 柑桔与亚热带果树信息, 17(3): 38-39. |
谭晓风, 胡芳名, 张党权. 2002. 香榧主要栽培品种的RAPD分析. 园艺学报, (1): 67-69. |
谭晓风, 乌云塔娜, 中西 テツ, 等. 2005. 中国梨品种自交不亲和新基因的分离鉴定. 中南林学院学报, 25(1): 1-3. DOI:10.3969/j.issn.1673-923X.2005.01.009 |
乌云塔娜, 张党权, 谭晓风. 2003. 梨不同DNA提取方法的效果研究. 中国生物工程杂志, 23(7): 98-101. |
佐藤昭宏.2001.RT-PCRによるセィョナシS遗传子型の推定.修士学位论文(日本)
|
Andrew G, McCubbin, Tehhui K. 1996. Molecular mechanisms of self-incompatibility. Plant Biotechnology, 13: 150-154. |
Castillo C, Takasaki T, Saito T, et al. 2002. Cloning of the S8-RNase(S8-RNase) of Japanese pear (Pyrus pyrifolia Nakai). Plant Biotechnology, 19(1): 1-6. DOI:10.5511/plantbiotechnology.19.1 |
Hiscock S J, Kues U, Dickinson H G. 1996. Molecular mechanisms of self-incompatibility in flowering plants and fungi-different means to the same end. Trends Cell Biol, 6: 421-428. DOI:10.1016/S0962-8924(96)10037-4 |
Hong Q, Fei W, Lan Z. 2004. The F-Box protein AhSLF-S2 controls the pollen function of S-RNase-based self-incompatibility. The Plant Cell, 16: 2307-2322. DOI:10.1105/tpc.104.024919 |
Ishimizu T, Inoue K, Shimonaka M, et al. 1999. PCR-based method for identifying the S-genotypes of Japanese pear cultivars. Theor Appl Genel, 98: 961-967. DOI:10.1007/s001220051156 |
Lewis D.1979.Incompatibility in plants.Limitde: Edward Arnold
|
Nettancourt D.1977.Incompatibility in angiosperms.New York: Spriner-Verlag
|
Norilka N, Norioka S, Ohnishi Y. 1996. Molecular cloning and nucleotide sequences of cDNAs encoding S-allele specific stylar RNases in a self-incompatible mutant of Japanese pear, Pyrus serotina Nakai. Biochem, 120: 335-345. DOI:10.1093/oxfordjournals.jbchem.a021418 |
Sassa H, Hirano H, Ikehashi H. 1993. Identification and characterization of stylar glycoproteins associated with self-incompatibility genes of Japanese pear. Mol Gen Gent, 241: 17-25. |
Shogo M, Sadao K, Kentaro K. 1999. S-genotypes of 15 apple cultivars and self-compatibility of `Megumi'. Japan Soc Hort Sci, 68(2): 563-566. |
Sijacic P, Wang X, Skirpan A L. 2004. Identification of the pollen determinant of S-RNase-mediated incompatibility. Nature, 429: 302-305. DOI:10.1038/nature02523 |
Steinbachs J E, Holsinger K E. 2002. RNase-mediated gametophytic self-incompatibility is ancestral in eudicots. Mol Biol Evo, 19(6): 825-829. DOI:10.1093/oxfordjournals.molbev.a004139 |
Stone J L. 2002. Molecular mechanisms underlying the breakdown of gametophytic self-incompatibility. Rev Biol, 77(1): 17-32. |
Tomimoto Y, Nakazaki T, Ikehashi H. 1996. Analysis of self-incompatibility related ribonucleases (S-RNase)in two entic. Horticulture, 66: 159-167. |
Ushijima K, Sassa H, Tao R. 1998. Cloning and characterization and cDNAs encoding S-RNases from almond:Primary structural features and sequence diversity of the S-RNase in Rosaceae. Mol Gene Genet, 260: 261-268. DOI:10.1007/s004380050894 |
Yunjun A. 1990. Self-incompatibility in Petunia inflata:isolation and characterization of cDNAs encoding three S-allele-associated. Plant Reprod, 3: 130-138. |
 2006, Vol. 42
2006, Vol. 42


