文章信息
- 李斌, 顾万春.
- Li Bin, Gu Wanchun.
- 白皮松保育遗传学——天然群体遗传多样性评价与保护策略
- Conservation genetics of Pinus bungeana——evaluation and conservation of natural populations' genetic diversity
- 林业科学, 2005, 41(1): 57-64.
- Scientia Silvae Sinicae, 2005, 41(1): 57-64.
-
文章历史
- 收稿日期:2003-02-09
-
作者相关文章
树种的生活史特征不同,其遗传多样性的分布式样也不同(Hamrick et al., 1989), 遗传变异在群体间的分配及其分化能够用来阐明该树种的进化史、适应性以及群体遗传结构等(Ge et al., 1998; 邹喻苹等,2001)。群体遗传多样性研究是保护遗传学的基础,种质资源保护在某种程度上讲就是保护该物种的遗传多样性(顾万春等,1998)。
白皮松(Pinus bungeana)是我国北方与西部地区的特有树种,分布于陕、晋、甘、豫、川、鄂、湘7省的部分地区(吴中伦,1956;赵焱等,1995;王小平,2002;李斌等,2003a)。白皮松早期生长十分缓慢,且大都生长在干旱、瘠薄、微碱性石灰质的山地上,自然条件比较恶劣,因此在一定程度上阻碍了其快速自然更新(赵焱等, 1995;李斌等,2003a)。该树种能够在恶劣的自然条件下顽强生长,因此成为当前以生态环境林建设为主的我国林业生态工程建设的优良树种之一;同时由于其抗旱节水、树姿优美等优点,白皮松在城乡绿化中也越来越受到重视。最近对白皮松天然群体种实形态多样性的研究(李斌等,2002),对揭示白皮松的群体遗传学提供了大量基础信息,在等位酶水平上研究白皮松群体保育遗传也有报道(李斌等,2003b)。本文研究目的是在李斌等(2003b)研究的基础上重点阐述白皮松天然群体的遗传参数和群体遗传多样性分布特征;揭示白皮松群体遗传结构,进一步探索其遗传资源或种质资源保护策略等。
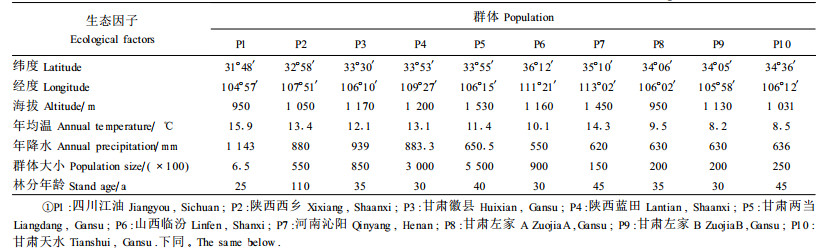
1 材料与方法 1.1 试验样本参与同工酶试验的共有10个白皮松群体(P1、P2、P3、P4、P5、P6、P7、P8、P9、P10),这10个群体的地理位置详见表 1。10个群体中7个群体(P3、P4、P5、P6、P8、P9、P10)分单株采集球果和种子,每群体随机确定30棵母株, 母株间距约5倍树高;另外3个群体(P1、P2、P7),单株采种存在困难,所以采取混合样本(李斌等, 2002), 混合采集母树样本数分别为20、50、35株,群体的大小与平均年龄见表 1。
|
|
等位酶分析采用水平切片淀粉凝胶电泳技术(王中仁, 1996),共分析了16种分离良好、谱带清晰的酶系统:Aat、Aco、Adh、Amp、Dia、Fba、Gdh、G6pd、Idh、Mdh、Me、Mnr、Pgd、P gi、Pgm、Skd(李斌等,2003b)。每单株随机分析6~8粒种子的胚乳。混合群体每群体分别随机测定120粒种子的胚乳。切胶、染色与酶谱判读依据Wendel等(1989)、Soltis等(1983)与王中仁(1996)的原则和方法。
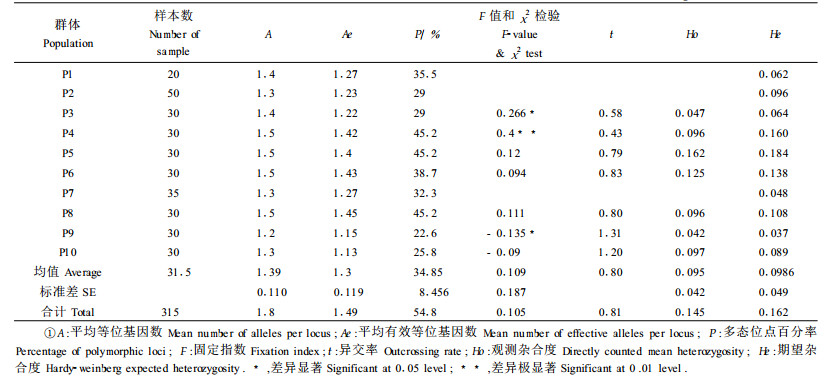
1.3 数据分析首先将7个群体各30株的电泳酶谱数据按基因型格式(Type Ⅰ)进行统计运算,然后将参试的10个群体(包括3个混合群体),按群体基因频率格式(Type Ⅱ,可分析混合样本)进行统计运算。分析程序参见BIOSYS-1 1.7(Swofford et al., 1989)。遗传多样性统计中,所有10个群体都分别统计了多态位点百分率(P)、平均等位基因数(A)、期望杂合度(He)3项指标,其中观测杂合度(Ho)只有7个分单株采种群体才能计算。F-统计量参数根据Wright (1965)进行分析。用Nei(1972)的遗传一致度I(genetic identity)和遗传距离D(genetic distance)来衡量各群体之间的遗传分化关系,并基于遗传一致度I,采用UPGMA法对各群体进行聚类分析。上述各项指标的计算均用BIOSYS-1程序完成。此外,根据F=1-Ho/He计算7个群体的固定指数F(Wright, 1965)(表 2),并利用此F值对群体的异交率t进行近似估计(王崇云等, 1999)。基因流(gene flow)采用群体每代迁移数Nm来间接估计,Nm=(1-FST)/4FST(Wright, 1951)。
|
|
共得到31个等位酶位点,其中多态位点17个,总的多态位点百分率为PS=54.8%,31个位点总共拥有等位基因53个,总的平均等位基因数AS=1.742,总的平均有效等位基因数AeS=1.49, 总的期望杂合度HeS=0.162(表 2)。
2.1.2 群体水平的遗传多样性白皮松10个群体水平的遗传多样性平均统计如下:AP=1.39±0.11, AeP=1.30±0.12, PP=34.85±8.46, HeP=0.0986±0.049。7个群体的平均观测杂合度HoP=0.095±0.042(表 2)。白皮松遗传多样性在10个群体间存在一定差异,综合各群体的多样性参数值,可以看出,甘肃两当(P5)、陕西蓝田(P4)和甘肃左家A(P8)3群体的多样性程度最高(A=1.5, P= 45.2%), 山西临汾(P6)次之,四川江油(P1)与甘肃徽县(P3)的多样性中等,而陕西西乡(P2)、河南沁阳(P7)、甘肃麦积山(P10)等群体的多样性程度较低, 多样性程度最低的为甘肃左家B(P9)群体(表 2),这与种实性状表型多样性分析结果有吻合之处(李斌等,2002)。
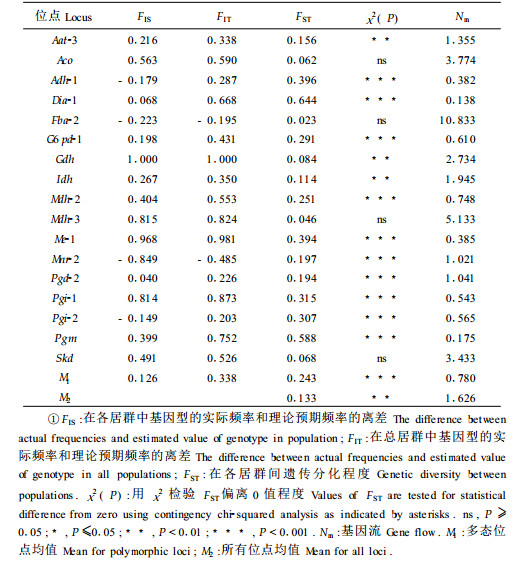
2.2 群体间基因分化与基因流群体间的基因分化水平在不同多态位点间差异较大,其中Fba-2位点的基因分化很小,FST=0.023, Dia-1位点的基因分化最大FST=0.644。根据17个多态位点计算的总基因分化FST=0.243, 根据所有位点计算的基因分化系数为0.133(表 3), 即白皮松遗传多样性在群体间仅占13.3%, 而群体内占绝大部分(86.7%),这与大多数针叶树种分析结果相一致。
|
|
根据基因分化系数计算得到白皮松群体间的基因流变化范围从Dia-1位点的0.138到Fba-2位点的10.833,位点间差异很大。根据多态位点估计的群体间基因流值为N m=0.78, 所有位点的基因流估计均值为1.626(表 3),介于1.0~4.0,表明白皮松天然群体间的基因流动基本能保证群体维持平衡,群体间没有明显的异化或同化趋势。
2.3 固定指数与遗传距离为了检验群体的基因型频率是否符合Hardy-Weinberg平衡,计算了7个分单株测定群体的固定指数(表 2)。从表 2看出,固定指数(F)在群体间差异明显,其中P3、P4群体的固定指数分别达到0.266和0.400,经χ2检验,2群体观测杂合度显著低于期望杂合度,群体内的纯合子较多,杂合子缺乏。相反,P9、P10群体的固定指数为负数,但经χ 2检验表明,只有P9群体观测杂合度显著高于期望杂合度,群体内的杂合子过剩,纯合子缺乏。经检验,白皮松总群体符合Hardy-Weinberg平衡(α=0.05),纯合子略偏高(F=0.105)。近似估计的异交率在群体间差异明显,异交率最低的P4群体t=0.43, 而P9、P10两群体的异交率均接近1.0, 总群体的异交率近似估计值为0.81,低于松属的平均值0.9(赖焕林等,1997),说明白皮松天然群体内存在自交现象。关于白皮松的交配系统还有待于进一步试验分析。
10个群体的平均遗传一致度I=0.901, 平均遗传距离D=0.128。其中P8与P9是遗传一致度最高而遗传距离最近的2个群体(I=0.969,D=0.031),P1和P9则是遗传一致度最低而遗传距离最远的2群体(I=0.772, D=0.259)(表 4)。将每个群体与其他9个群体的遗传距离进行平均分析,结果P1距离其他9个群体的平均遗传距离最大为D=0.18 3, 其余依次为P3(D=0.146)、P9(D=0.135)、P8(D =0.131)、P2(D=0.129)、P4(D=0.118)、P6(D=0.112)、P7(D=0.111)、P5(D=0.109)、P10(D=0.103)。
|
|
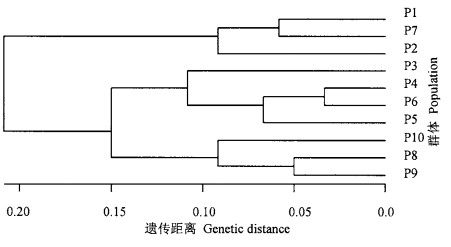
根据Nei(1972)遗传距离聚类图, 可将10个群体分为3组(图 1)。其中A组包括3个群体(P1, P2, P7), B组包括4个群体(P3, P4, P5, P6),C组包括3个群体(P10, P8, P9)。其中A组3个群体均为混合采样,这与采样方式是否有关,本文不予讨论,其分析方法建立在统一标准上。遗传距离与地理距离相关分析显示,白皮松10个群体的遗传距离与地理距离相关性不明显(r=0.311),尚未达到显著水平〔r0.05(19)=0.433,r 0.10(19)=0.368〕。
|
图 1 白皮松10个群体的遗传聚类图 Fig. 1 UPGMA dendrogram based on the Nei's genetic distances between populations showing the clustering pattern of ten populations in P. bungeana |
松属树种是典型的以风媒传粉为主的裸子植物,具有较高的遗传多样性(Hamrick et al., 1989; 1992)。平均而言,裸子植物的遗传多样性为:A=2.25,P=0.677, He =0.207(Hamrick et al., 1989;王中仁, 1996)。在松属植物中,遗传多样性最低的台地松(P. torreyona),He=0.00, P=0.00;遗传多样性水平较高的为欧洲赤松(P. sylvestris),He=0.357, P=100%。白皮松种水平的遗传多样性为:AS=1.742, PS=54.8%, HeS=0.162;群体内的平均遗传多样性为:A=1.39, P=0.348, He=0.099,略低于裸子植物平均值,因此白皮松天然群体具有中等略偏低的遗传多样性。
各群体在分布区的分布格局表明,天然资源分布较集中的甘肃东南部、山西西南部与陕西中西部等地的群体规模较大、群体遗传多样性参数值(平均等位基因数、多态位点百分率、期望杂合度)较高,如P4、P5及P8等(表 1、图 2、表 2);天然资源分布较零散、位于白皮松分布区北部、南部与东南部边缘的群体遗传多样性程度较低,如P1、P2、P7(表 1、图 2、表 2)。若完全随机状态下群体规模将影响白皮松的群体遗传多样性程度,然而群体规模在一定程度上受分布区位置影响, 除了群体规模、分布区位置效应外,还包括很多其他因素,如起源、人为干扰、自然灾害等。结合白皮松种实性状多样性分析和该物种资源分布等(李斌,2002),总的来说,白皮松遗传多样性呈现中心分布的总特征,即遗传多样性高的区域主要集中在白皮松分布区的中段(图 2),该区域也是白皮松表型多样性富集区和资源分布中心区,这证实了白皮松属于生态趋中适应策略树种(王兰州,1996;李海山等,2000)。
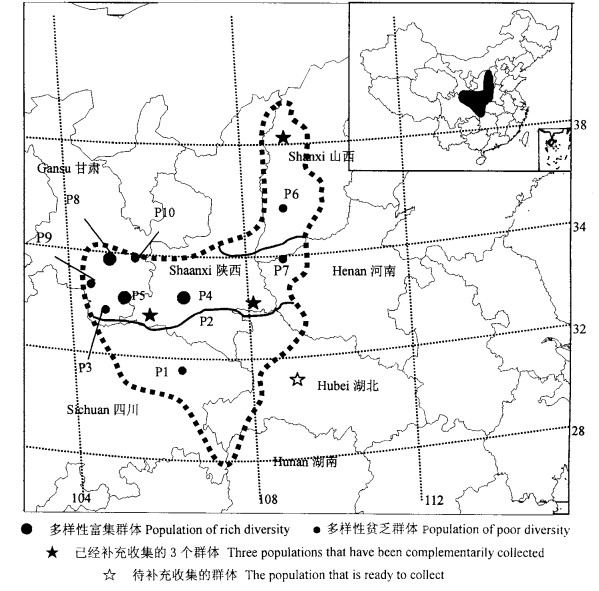
|
图 2 白皮松天然群体多样性分布与区划 Fig. 2 Regionalization and distribution regulation of natural population diversity in P. bungeana |
根据白皮松遗传多样性分布总特征,将其分布区按多样性进行区划,可分为北部(Ⅰ区)、中部(Ⅱ区)和南部(Ⅲ区)共3个区(图 2)。中部区主要包括甘肃东南部、陕西中部及晋陕豫三省交界处等,该区为多样性富集区,也是白皮松最适生区域,可能也是该物种的起源区和潜在的优良种源区。北部区主要位于山西吕梁山、太岳山、关帝山等地,该区多样性较富集,生境条件相对干燥、寒冷和贫瘠,该区的种源适应旱寒的能力较强、是潜在的北方生态造林种质资源的供应源。南部区位于陕西汉中以南,包括四川江油、陕西西乡、湖北西部及湖南西北部,该区多样性较贫乏,可能为该物种扩张的区域,其生境条件相对湿热,水热条件较好,该区的种源适应于在长江流域无积水的丘陵、坡地等微酸至微碱土壤上引种。
根据相关分析结果,白皮松群体遗传距离与地理距离相关不紧密,因此其遗传聚类图(图 1)和多样性分布区划图(图 2)在理论上可以不一致。
3.2 白皮松群体间遗传分化与松属其他树种相比,白皮松群体间遗传分化程度较大(FST=0.133)。松属树种群体间分化系数虽然在不同的研究材料间存在一定的差异,但基本介于0.03~0.078 (李斌等,2003c)。据Hamrick等(1989)报道,裸子植物的平均分化系数为FST=0.068,松属植物的平均分化系数为0.08左右。与白皮松类似的等位酶变异模式,在其他针叶树中也有报道(Ge et al., 1998; Szmidt et al., 1996)。对于高度异交的风媒树种——松树,地理隔离是限制其基因流动的有效因素(Hamrick et al., 1989)。很多研究显示,那些生境地片段化的树种其群体间的分化大于那些连续分布的树种。如,刚松(P. rigida)(Moran et al., 1988)的天然分布区被减少并分割为5块群体,等位酶分析结果,刚松的群体分化是松属树种平均分化水平的1倍(FST= 0.162)。在环地中海地区呈不连续分布的阿勒颇松(P. halepensis)(Schiller et al., 1986), 其群体间的分化达0.300。据Ge等(1998)研究报道,我国特有的呈明显间断分布的濒危裸子植物银杉(Cathaya argyrophylla)的群体间分化达到0.441,是目前报道的针叶树种中分化最剧烈的种。白皮松群体间遗传分化较大与该树种的生活史等因素有关。通过花粉与种子等发生的较高的群体间的基因流动,将导致群体在遗传上趋同化,反之,较低的基因流动则导致群体分化和遗传漂变。据报道(赵焱等,1995;李斌等,2003a),古时候白皮松曾遍布于我国西北诸省,但自唐朝以来由于战乱和无计划的乱砍乱伐,使得原本在我国西北地区广泛分布的白皮松,变成如今的间断分布,不利于群体间基因流动;而且有些块状或岛状分布群体,数量很少,易发生自交现象,产生遗传漂变;特别是解放后“大炼钢铁时期”,由于白皮松松脂含量高,是很受欢迎的薪柴,因此被大量砍伐,甚至皆伐,一方面造成该树种自然资源的大量减少,另一方面,某些砍伐迹地由留下的少量母树天然下种更新,出现了人为的瓶颈效应,子代个体间容易发生自交或近交。总之,白皮松天然群体的间断分布,群体内数量少,个体间亲缘关系近,存在自交或近交现象,这是导致白皮松天然群体内遗传多样性减少,群体间遗传分化大的主要因素。
3.3 白皮松群体遗传多样性保护的必要性及策略白皮松与处于同一分布区的其他松树在系统发育上存在明显分化,很多学者认为白皮松属于一个原始的单调世系(Wang et al., 1993;李湘萍,1993;宋湛谦等,1998; 陆素娟等,1999)。同时,白皮松的扩张策略比较保守,属于生态趋中策略(王兰州,1996;李海山等,2000),加上白皮松早期生长缓慢,天然更新周期长等原因,导致白皮松的分布区没有太大变化,而资源锐减。因此,人为的保护、栽培以及引种试验对于协助白皮松的生物扩张策略是很有帮助的。为了发展和利用好白皮松遗传资源,今后有必要对白皮松现有遗传资源或种质采取科学合理的保护。
由于白皮松的生态属性强、人工造林具有一定难度,所以首先应考虑原地保护,如在甘肃两当(P5:目前群体规模最大的白皮松天然林)等地设立白皮松原地保存林,进行连续观测和管理。除了原地保存外,还应有选择地进行异地保存。白皮松群体内遗传多样性偏低,而群体间分化剧烈、具有较大的遗传距离(D=0.128),这表明白皮松天然遗传资源若采取异地保存,保存效率较高。异地保存时应考虑抽取较多的群体,群体内可以保存相对较少的个体。这与呈连续分布的松属树种有所不同,对于那些连续分布且遗传多样性主要存在于群体内的树种,则可以保存较少的代表性群体,群体内则需要较多的个体(Dvorak et al., 1999)。
根据聚类分析,白皮松10个天然群体分为3组。其中处于分布区西北域的P10、P8、P9在地理上相隔很近,在遗传上也聚在了一起(C组),说明这3个群体的亲缘关系较近,该组P8群体的遗传多样性最丰富,适合被保护。群体P3、P4、P5、P6聚在了一起(B组),从分布位置(图 1)看,4个群体虽然东西距离较远,但大都处于分布区的水平中心位置,其中P3群体多样性程度低,且与P5群体地理距离和遗传距离很近,因此该组适合保护除P3群体的3个群体。事实上,经过子代苗期测定分析(何燕等,2001),中心分布区的群体如P4、P5不但多样性丰富,而且生长快,生物量大,是潜在的优良种源群体,而P6群体抗旱性能突出。最后,从东南的P7到西南的P2、P1三个群体聚为一类(A组),这3个群体的多样性程度均较低(表 2),而且位于白皮松分布区的南部和东南部边缘区(图 1),但是却拥有较多的局域基因(sporadic genes)和特有基因(unique genes) (李斌等,2003b),具有较强的抗逆性和特有的性状组合,应予以保护。从上述分析看出,一共需要保护7个群体:P1、P2、P4、P 5、P6、P7、P8。对上述7个群体进行基因捕获率和遗传多样性捕获率分析,结果捕获全部53个测定基因,基因捕获率100%,遗传多样性(7群体/10群体)捕获率达99.6%(李斌等,20 0 3b)。考虑到未参与试验的群体中仍含有某些有价值的基因, 如山西北部的孝义群体(李斌等,2002)和待采的湖北西部群体。因此,总共需要异地保存的群体为“8+1”模式,即8个已采集的天然群体和1个待收集的聚合群体。
这一结果不仅给白皮松群体保存所需要的样本容量提供了指导,也给松属树种或其他植物种类天然群体保护提供了一定的参考。通常,在天然群体保护的抽样问题上存在2种观点:一种是保存大量的群体,如Ellstrand等(1993)认为一个树种需要保存10个甚至几十个群体;也有的观点认为群体数并不重要,1~3个群体足以保护该物种,只要具有一定数量的个体就可以(Frankel, 1984;Dvorak et al., 1999)。本研究认为遗传多样性取样保护策略的制定应基于群体的遗传结构(Ge et al., 1998; 顾万春等,1998),对于群体分化大、群体间遗传距离大的树种,需要抽取较多的群体,而且根据遗传多样性分布格局进行选择性抽取比随机取样效果更佳; 反之,对于群体分化甚小,群体内遗传多样性占绝对优势的物种,则可保存较少的群体。
另外,本研究中有3个混合取样群体,虽然在测定和分析时,采取了相应的方法,如在遗传距离分析中采用了群体基因频率(TYPE-Ⅲ)而不是群体基因型频率(TYPE-Ⅰ)以达到所有群体统一格式进行运算的目的等,但关于抽样方法本身所造成的影响本文未予考虑,今后可做这方面的分析研究。
顾万春, 王棋, 游应天, 等. 1998. 森林遗传资源学概论. 北京: 中国科学技术出版社, 1-296.
|
何燕, 李斌, 卢宝明, 等. 2001. 白皮松种源苗期变异与选择. 林业科技通讯, (6): 5-8. DOI:10.3969/j.issn.1671-4938.2001.06.002 |
赖焕林, 王章荣, 陈天华. 1997. 林木群体交配系统研究进展. 世界林业研究, (5): 10-15. |
李斌, 顾万春, 卢宝铭. 2002. 白皮松天然群体种实性状表型多样性研究. 生物多样性, 10(2): 181-188. DOI:10.3321/j.issn:1005-0094.2002.02.008 |
李斌, 顾万春. 2003a. 白皮松天然分布特点与研究进展. 林业科学研究, 16(2): 225-232. |
李斌, 顾万春, 周世良. 2003b. 白皮松的保育遗传学(Ⅰ)——基因保护分析. 生物多样性, 11(1): 28-36. |
李斌, 顾万春. 2003c. 松属植物遗传多样性研究进展. 遗传, 25(6): 740-748. |
李海山, 曹文侠, 王兰州, 等. 2000. 油松与白皮松微弱电波信号振荡的研究. 西北师范大学学报(自然科学版), 36(4): 77-81. DOI:10.3969/j.issn.1001-988X.2000.04.019 |
李湘萍, 朱政德. 1993. 白皮松种子油中脂肪酸成分分析及其分类学问题. 南京林业大学学报, 17(1): 27-34. |
陆素娟, 李乡旺. 1999. 松属的起源、演化与扩散. 西北林学院学报, 14(3): 1-5. DOI:10.3969/j.issn.1001-7461.1999.03.001 |
宋湛谦, 梁志勤, 刘星. 1998. 白皮松松脂的化学特征及其分类学意义. 植物分类学报, 36(6): 511-517. |
王崇云, 党承林. 1999. 植物的交配系统及其进化机制与种群适应. 武汉植物学研究, 17(2): 163-172. DOI:10.3969/j.issn.2095-0837.1999.02.013 |
王兰州. 1996. 植物群落趋中生态适应对策的机制. 西北师范大学学报(自然科学版), 32(2): 46-51. |
王小平. 2002. 白皮松生物学及种子生理生态. 北京: 中国环境科学出版社, 1-226.
|
王中仁. 1996. 植物等位酶分析. 北京: 科学出版社, 1-196.
|
吴中伦. 1956. 中国松属的分类与分布. 植物分类学报, 5(3): 131-163. |
赵焱, 张学忠, 王孝安. 1995. 白皮松天然林地理分布规律. 西北植物学报, 15(2): 161-166. DOI:10.3321/j.issn:1000-4025.1995.02.015 |
中国树木志编委会. 1976. 中国主要树种造林技术. 北京: 农业出版社.
|
邹喻苹, 葛颂, 王晓东. 2001. 系统与进化植物学中的分子标记. 北京: 科学出版社, 1-266.
|
Dvorak W S, Hamrick J L, Hodge G R. 1999. Assessing the sampling efficiency of ex situ gene conservation efforts in natural pine populations in central America. Forest Genetics, 6(1): 21-28. |
Ellstrand N C, Elam D R. 1993. Population genetic consequences of small population size: implications for plant conservation. Ann Rev Ecol Syst, 24: 217-242. DOI:10.1146/annurev.es.24.110193.001245 |
Frankel O H.1984. Genetic perspectives of germplasm conservation. In: Arber W, Limensee K, Peacock W J, et al(eds). Genetic manipulation: Impact on man and society. Cambridge, UK: Cambridge Univ Press, 1-324
|
Ge S, Hong D Y, Wang H Q, et al. 1998. Population genetic structure and conversation of an endangered conifer, Cathaya argyrophylla(Pinaceae). Int J Plant Sci, 159(2): 351-357. DOI:10.1086/297556 |
Hamrick J L, Godt M J.1989. Allozyme diversity in plant species. In: Brown A D H, Clegg M T, Kahler A L, et al(eds). Plant population, breeding and genetic resources. Sinauer Associates Inc., Sunder land, MA, 43-63
|
Hamrick J L, Loveless M D. 1992. Factors influencing levels of genetic diversity in woody plant species. New Forests, 6: 95-124. DOI:10.1007/BF00120641 |
Moran G F, Bell J C, Griffin A R. 1988. The genetic structure and levels of inbreeding in Pinus rigida. Silvae Genetica, 28(5-6): 190-193. |
Nei M. 1972. Genetic distance between populations. American Naturalist, 6: 283-293. |
Schiller G, Conkle M T, Grunwald C. 1986. Local differentiation among Mediterranean populations of Aleppo Pine in their isozymes. Sil Genet, 35: 11-19. |
Soltis D E, Haufler C H, Darrow D C, et al. 1983. Starch gel electrophoresis of ferns: a complication of grinding buffers, gel and eletrode buffers, and staining schedules. American Fern Journal, 73: 9-27. DOI:10.2307/1546611 |
Swofford D L, Selander R B.1989.BIOSYS-1: a computer program for the analysis of allelic variation population genetics and biochemical systematics, release 1.7. Swofford, Illinois Natural History Survey, IL, USA
|
Szmidt A E, Wand X R, Changtragoon S. 1996. Contrasting patterns of genetic diversity in two tropical pines: Pinus kesiya and P. merkusii. Theor Appl Genet, 92: 436-441. DOI:10.1007/BF00223690 |
Wang X R, Szmidt A E. 1993. Chloroplast DNA-based phylogeny of Asian Pinus species. Plant systematics and Evolution, 188(3-4): 197-211. |
Wendel J F, Weeden N F.1989. Visualization and interpretation of plant isozymes. In: Soltis D E, Solis P S(eds). Isozymes in plant biology. Dioscorides Press, Portland, 5-45
|
Wright S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution, 19: 395-420. DOI:10.1111/j.1558-5646.1965.tb01731.x |
Wright S. 1951. The genetical structure of populations. Annals of Eugenics, 15: 323-354. |
 2005, Vol. 41
2005, Vol. 41