文章信息
- 孟丽, 郑国生.
- Meng Li, Zheng Guosheng.
- 部分野生与栽培牡丹种质资源亲缘关系的RAPD研究
- Phylogenetic Relationship Analysis among Chinese Wild Species and Cultivars of Paeonia sect. Moutan Using RAPD Markers
- 林业科学, 2004, 40(5): 110-115.
- Scientia Silvae Sinicae, 2004, 40(5): 110-115.
-
文章历史
- 收稿日期:2003-01-13
-
作者相关文章
2. 北京林业大学园林学院;莱阳农学院生命科学学院, 青岛 266109
2. School of Life Science, Laiyang Agricultural College Qingdao 266109
芍药属(Paeonia)牡丹组(sect. Moutan)植物为我国特有的资源,世界各地广为种植的牡丹(P.suffruticosa)都起源于中国。我国在2000多年前就发现了牡丹的药用价值,用作庭院观赏栽培也已经有1 500多年的历史,它作为我国的传统名花在我国传统花文化中占有极其重要的地位。研究该组植物种质资源对于维持我国生物多样性具有重要意义,探讨牡丹种与品种间的亲缘关系则是牡丹研究的热点问题之一。刘淑敏等(1987)、张益民等(1988)通过对花瓣基部彩斑和叶片解剖结构的分析,对栽培牡丹单元起源的观点提出质疑。于玲等(1997)通过对甘肃紫斑牡丹(P. rockii)和中原牡丹类群进行染色体核型、C带带型的系统研究发现两者之间既有区别又有联系,从细胞学的角度说明中原牡丹变异幅度大,起源较复杂,应属多元起源。王莲英(1997)、李嘉钰(1998)等根据形态特征的分析比较,认为中原牡丹受矮牡丹(P. jishanensis)、紫斑牡丹和杨山牡丹(P. ostii)的影响较大,是否受四川牡丹(P. decomposita)和卵叶牡丹(P. qiui)的影响还不清楚。因此,对于野生种与中原牡丹间的亲缘关系应进一步从携带更多遗传信息的核酸大分子物质进行研究。
RAPD-PCR技术自1990年建立起(Welsh et al., 1990;Williams et al., 1990),就被证明是一种研究植物种间和品种间遗传分化有效的分子标记技术,可以对没有任何分子生物学研究基础的物种直接从基因组DNA上分析其遗传多态性,对于牡丹组植物,裴颜龙等(1995)证明了RAPD技术用于检测野生牡丹居群内与居群间的遗传变异是可行的。邹喻苹等(1999)采用RAPD标记分析了牡丹组各野生类群间的遗传关系。Hosoki等(1997)和Zheng等(2002)分别对日本和中国的栽培牡丹品种的亲缘关系进行了RAPD分析,这些研究为深入探讨牡丹种与品种间的亲缘关系提供了技术保障和理论基础。
在我国4个主要的牡丹栽培品种群中,中原牡丹形成历史最久,园艺化最早,品种最多,是全国各地牡丹精品的荟萃和代表(李嘉珏,1998)。本试验选取了牡丹组11个野生类群和12个中原牡丹品种进行了RAPD-PCR扩增,并构建遗传聚类树状图对牡丹种和品种间的亲缘关系进行了研究,旨在丰富该组植物系统学研究内容,为中国牡丹种质资源的保护和合理地开发利用提供有价值的资料。
1 材料与方法 1.1 供试材料选取6个牡丹种,按来源分为11个野生类群。12个品种采自山东菏泽曹州牡丹精品园,每个品种选取3~5个单株混合。供试牡丹种、品种的名称和生物学特性见表 1。
|
|
由于牡丹酚类物质含量多,基因组DNA分离方法在原有基础(Zheng et al., 2002)上略有改进,在CTAB提取缓冲液中加入2% PVP,降低酚类物质干扰;在加入100 μL TE溶解DNA沉淀3 h后,加入1V(体积)苯酚/氯仿/异戊醇(V:V:V=25:24:1)抽提2~3次,上清液中加入1/4V 3 mol·L-1 NaAc和2V无水乙醇,轻摇混匀,12 000 r·min-1离心5 min,重新洗涤沉淀,真空干燥后,再溶于100 μL TE,0.8%琼脂糖凝胶,电流20 mA左右,电泳1 h后检测。用UV-120紫外分光光度计测定DNA浓度,于-20℃保存备用。
1.3 PCR反应及引物筛选PCR反应体系为20 μL(Zheng et al., 2002), 扩增程序为94℃预变性2 min,94℃变性1 min,36℃退火40 s,72℃延伸1 min,40个循环;最后72℃延伸10 min。扩增产物含0.5 μg·mL-1 EB,1.5%琼脂糖凝胶电泳4 h,在UV-2000紫外分析仪观察结果并照相。分别取23个模板DNA 5μL混合,从Sangon生产的210个10 bp的RAPD随机引物中,筛选出有3~5条以上清晰条带的32个引物,其中有17个单引物和1个组合引物(裴颜龙等,1995;Klein-lankhorst et al., 1991)的重复性好,多态性较高(表 2),最终用于供试材料的RAPD反应及数据分析。
|
|
用DL-2000作为分子量Marker,根据Marker各片段的迁移距离和分子质量,建立回归方程,量出所有记录条带的迁移距离,根据方程计算其碱基数。“有”带记为“1”,并记录产物的迁移距离,求出产物大小;“无”带记为“0”,获得原始二元矩阵。按下列公式计算任意2个分类群(OTU:operational taxonomic unit)间遗传距离D(Genetic distance):S=2Nxy/(Nx+Ny),D=-lnS,式中,S是相似性系数,Nxy为X和Y 2个OTU经RAPD扩增反应碱基对相同的片段数,Nx、Ny分别代表X、Y扩增条带的总数,得到相似系数矩阵,利用UPGMA法进行聚类分析(黄原,1994)。
2 结果与分析 2.1 不同引物扩增结果的多态性分析不同碱基序列的引物及其在牡丹组植物中扩增反应的结果列于表 2。选出17个单引物和1个组合引物扩增的23个DNA样本进行分析,结果表明:选用的18个引物,扩增出的产物谱带从6条(S417)到20条(S13+31)不等,平均每个引物扩增11条带,共产生191条扩增产物带,其中153条具有多态性,占80.1%,充分体现了该组植物丰富的遗传多样性。扩增产物的长度从116 bp(S13+31)至2 040 bp(S13)不等,说明该组植物表现多态性的范围较宽。分析不同引物扩增各样本DNA产物,可见多态性带多数集中于400~1 000 bp,说明供试样本使用S系列引物扩增的片段较小。而在500~800 bp范围内共有带增多,比如S31-500、S428-600和S193-755处,这一区段的条带可作为牡丹组植物的共有条带。
2.2 RAPD特异标记在有些引物扩增的产物中,仅个别样本在某一片段长度处有条带出现,这种其它样本所不具备的带可作为该种材料的特异标记(戴思兰等,1998)。受试的18个引物中,由11个引物产生25条带可作为部分样本的特异标记列于表 3。滇牡丹(P. delavayi)的特异标记较多,共有14个,这是因为供试材料中只有滇牡丹属于肉质花盘亚组(subsect. Delavayanae),而其他材料则属于革质花盘亚组(subsect. Vaginata), 这与2个亚组的分化较明显有关。革质花盘亚组的紫斑牡丹、卵叶牡丹和矮牡丹,以及4个栽培品种:‘胡红’、‘大棕紫’、‘似荷莲’和‘青龙卧墨池’,各有1个,‘景玉’和‘首案红’各有2个特异标记,经试验产生的这些RAPD特异标记与材料的表型和生理性状有关(表 1),可有助于牡丹组植物的分类与鉴定工作。
|
|
将RAPD扩增谱带的统计数据利用UPGMA构建遗传聚类树状图(图 1)。在遗传距离0.2处,各牡丹种和品种单独为一个遗传聚类组。在遗传距离0.6处,滇牡丹和‘赵粉’分别独立出来,革质花盘亚组各种和11个牡丹品种聚为一组,下分Ⅰ和Ⅱ 2个聚类组。Ⅰ聚类组中,‘似荷莲’、‘紫二乔’、‘大棕紫’和‘首案红’聚为i组,4个品种花型和花色等某些性状相似,比如4个品种都是紫红色系列,其中‘紫二乔’和‘大棕紫’都为蔷薇型品种等。‘景玉’和‘青龙卧墨池’,‘姚黄’、‘藕丝魁’和‘胡红’,‘凤丹白’、‘朱砂垒’和杨山牡丹分别聚类后,再聚为ⅱ类。其中‘姚黄’、‘藕丝魁’和‘胡红’均是皇冠型品种,亲缘关系相对较近。杨山牡丹与‘凤丹白’和‘朱砂垒’2个品种的亲缘关系最近,这是由于‘凤丹白’是杨山牡丹驯化了的栽培品种,而‘朱砂垒’则在形态、生理特征上如重瓣率低(荷花型),雌、雄蕊正常,生长力强,结实能力强等与两者相近。矮牡丹、紫斑牡丹、四川牡丹和卵叶牡丹聚为Ⅱ类,尽管来自不同的类群,但聚类分析表明革质花盘亚组的8个野生类群间亲缘关系相对较近。从形态学上分析,各野生牡丹的株高1.0~1.5 m,花瓣为9~12枚,花色大部分为白色,花型都是单瓣型,叶柄也都具凹槽等,表型性状相似;另外自然状态下的各野生类群,人为选择力度薄弱,也必然保持了许多优良的生理性状,比如对恶劣环境的适应能力,对病虫害较强的抵抗能力,以及强结实能力等,这是各野生类群所特有的、共同的性状,因此,各野生种间亲缘关系相对较近,遗传多样性也比栽培品种丰富,这也能启迪育种工作者,应在保护野生资源的基础上,开发野生种的利用潜力,引入优良的基因资源,提高栽培牡丹种内杂合度,培育新品种。

|
图 1 芍药属牡丹组23个OTUs聚类分析树状图 Fig. 1 Dendrogram of RAPD cluster analysis for 23 OTUs of Paeonia sect. Moutan Ⅰ、Ⅱ、ⅰ和ⅱ为各遗传聚类组的代码. Ⅰ、Ⅱ、ⅰ and ⅱ stand for every genetic clustering groups. |
分布在聚类组Ⅰ和Ⅱ中的5个野生种与栽培品种的遗传距离分别为:矮牡丹0.57,紫斑牡丹0.54,卵叶牡丹0.51,四川牡丹0.49,杨山牡丹0.48,按亲缘关系由近到远排列为:杨山牡丹、四川牡丹、卵叶牡丹、紫斑牡丹和矮牡丹。可以看出矮牡丹与栽培品种间亲缘关系相对较远,这与洪德元等(1999)提出的观点一致,但最远与最近的遗传距离差别甚小,因此可以证明中原牡丹的复杂化多元起源,革质花盘亚组的5个野生种对中原栽培牡丹品种群形成的影响程度大小相近。利用聚类图分析表明肉质花盘亚组的滇牡丹与供试其他分类群亲缘关系较远,即牡丹组2个亚组分化明显,这为2个亚组的划分提供了分子水平的证据。另外也表明滇牡丹与牡丹品种间的亲缘关系较远,这与以前研究报道提出的观点相一致(喻衡,1989;王莲英,1997;李嘉珏,1998),即肉质花盘亚组与栽培牡丹的起源无关。但是试验中也发现,除了所有样本的共有条带外,滇牡丹还与部分品种扩增出了共有带,比如S19-679处是滇牡丹与‘藕丝魁’、‘紫二乔’、‘青龙卧墨池’、‘赵粉’和‘景玉’的共有带,S193-438是滇牡丹与‘赵粉、S193-600处是滇牡丹与‘胡红’的共有带,这些位点可以作为栽培品种与滇牡丹的同质谱带。
3 讨论 3.1 关于中原牡丹起源的分析通过历史记载、形态学和细胞学研究(王莲英,1997;李嘉珏,1998;于玲等,1997),中原牡丹属多元起源,主要野生种是矮牡丹、紫斑牡丹和杨山牡丹,是否受四川牡丹和卵叶牡丹的影响还不明显。本次RAPD-PCR试验表明:革质花盘亚组的5个野生种与牡丹栽培品种间亲缘关系较近,从遗传距离远近看,四川牡丹和卵叶牡丹与牡丹品种间的亲缘关系也比较近,进一步证明了中原牡丹的复杂起源。具体体现在除了与其他野生种共同与栽培品种扩增的共有条带以外,个别引物扩增出了四川牡丹和卵叶牡丹与不同品种的共有带,比如S19-1884处,只有四川牡丹、卵叶牡丹、‘胡红’和‘似荷莲’有带;S19-1245处,又与‘胡红’、‘朱砂垒’、‘首案红’和‘大棕紫’4个品种有共有带;S13+31-444处,又是与‘首案红’和‘大棕紫’的共有带。此外S19-526处,只有四川牡丹与‘胡红’、‘藕丝魁’、‘青龙卧墨池’和‘景玉’有带,这些都可作为四川牡丹和卵叶牡丹参与中原牡丹形成的特异标记。因此,从试验结果分析看,中原牡丹不仅受矮牡丹、紫斑牡丹和杨山牡丹的影响,而且与四川牡丹和卵叶牡丹之间也存在着广泛的基因交流。
3.2 RAPD分析中组合引物的应用本试验中,尝试运用组合引物进行RAPD随机扩增,将多态性高的S13与单态性较高的S31引物组合在一个反应体系同时扩增,结果也发现了组合后不是2个单引物扩增带简单地综合,而是既有带的消失,又有新带的出现(图 2)。比如23个分类群对S31扩增,无特异标记,S13也只有3个,而S13+31却产生了7个标记(表 3),这可能是组合后2个引物同时与同一个模板结合,序列不同结合位点不同,扩增的中间产物又可以互相利用,作为下一轮扩增的模板,因此会有新带的产生。这样在以后的试验中,可以只筛选较少的单引物,互相组合用于试验,提高了随机引物的利用率,节省了研究费用。
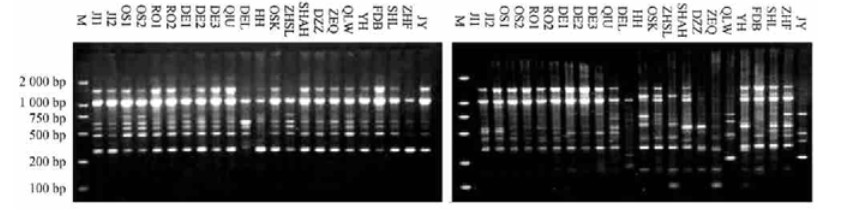
|
图 2 引物S31(左)和S13+31(右)对芍药属牡丹组23个OTUs的RAPD扩增图谱 Fig. 2 RAPD amplification profiles generated by primer S31(Left) and S13+31(Right) for 23 OTUs of Paeonia sect. Moutan Marker M为DL-2000分子量。 M indicates DL-2000 molecular weight. |
戴思兰, 陈俊愉, 李文彬. 1998. 菊花起源的RAPD分析. 植物学报, 40(11): 1053-1059. DOI:10.3321/j.issn:1672-9072.1998.11.012 |
洪德元, 潘开玉. 1999. 芍药属牡丹组的分类历史和分类处理. 植物分类学报, 37(4): 351-368. |
黄原编著.分子系统学——原理、方法及应用(中国博士专著——农业领域).北京:中国农业出版社, 1994:161-170
|
李嘉珏. 1998. 中国牡丹起源的研究. 北京林业大学学报, 20(2): 22-26. DOI:10.3321/j.issn:1000-1522.1998.02.005 |
刘淑敏, 王莲英, 吴涤新, 等. 1987. 牡丹. 北京: 中国建筑出版社, 1-67.
|
裴颜龙, 邹喻萍, 尹蓁, 等. 1995. 矮牡丹与紫斑牡丹RAPD分析初报. 植物分类学报, 33(4): 350-356. |
王莲英主编.中国牡丹品种图志.北京:中国林业出版社, 1997:1-49
|
于玲, 李嘉珏, 何丽霞. 1997. 甘肃紫斑牡丹与中原牡丹类染色体的比较研究. 园艺学报, 24(1): 79-83. DOI:10.3321/j.issn:0513-353X.1997.01.016 |
喻衡. 1989. 中国名花丛书——牡丹. 上海: 上海科学技术出版社, 16-37.
|
张益民, 张赞平, 王世端. 1988. 芍药属叶片解剖结构的研究. 河南农业大学学报, 22(1): 117-126. |
邹喻苹, 蔡美琳, 王子平. 1999. 芍药属牡丹组的系统学研究——基于RAPD分析. 植物分类学报, 37(3): 220-227. |
Hosoki T, Kimure D, Hasegawa R, et al. 1997. Classification of Japanese tree peony cultivars by random amplified DNA (RAPD) analysis. J Jpn Soc Hortic Sci, 66: 393-400. DOI:10.2503/jjshs.66.393 |
Klein-Lankhorst R M, Vermunt R, Liharska W T, et al. 1991. Isolation of molecular markers for tomato (L.esculentum) using random amplified polymorphic DNA(RAPD). Theor Appl Genet, 83: 108-114. DOI:10.1007/BF00229232 |
Welsh J, McClelland M. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nuc Acd Res, 18: 7213-7218. DOI:10.1093/nar/18.24.7213 |
Williams J G K, Kubelik A V, Levak K J, et al. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nuc Aci Res, 18: 6531-6535. DOI:10.1093/nar/18.22.6531 |
Zheng Guosheng, Chen Xiangming, Meng Li. 2002. RAPD-PCR Analysis ongenetic relationships between cultivars of tree peony. Agricultural Science in China, 1(7): 792-797. |
 2004, Vol. 40
2004, Vol. 40




