文章信息
- 李志真, 陈坚, 谢一青, 郑伟文, 陈启锋.
- Li Zhizhen, Chen Jian, Xie Yiqing, Zheng Weiwen, Chen Qifeng.
- Frankia菌遗传多样性的rDNA IGS RFLP研究
- DIVERSITY OF ACTINORHIZAL FRANKIA BY rDNA IGS RFLP ANALYSIS
- 林业科学, 2003, 39(专刊1): 187-193.
- Scientia Silvae Sinicae, 2003, 39(专刊1): 187-193.
-
文章历史
- 收稿日期:2003-03-21
-
作者相关文章
2. 福建省农业科学研究院生物技术中心 福州 350003;
3. 福建农林大学 福州 350002
2. Biotechnology Center, Fujian Academy of Agricultural Sciences Fuzhou 350002;
3. Fujian Agriculture and Forestry University Fuzhou 350003
Frankia是一类能与非豆科树木共生结瘤固氮的放线菌。关于其属的分类目前比较确认, 对于种水平的划分, 虽然从可溶性蛋白类型(张道海等, 1991; Benson et al., 1983; Gardes et al., 1987a)、同功酶类型(Gardes et al., 1987b; 张道海等, 1989)、脂肪酸(Weeler et al., 1986)、血清(Baker et al., 1981; 孙慧君等, 1991)、宿主特异性(Baker, 1987; Torrey et al., 1989)、DNA同源性(Fermamdex et al., 1989; Akimov et al., 1992)、基因组(Dobritsa, 1985)和质粒的限制酶切图谱(Simonet et al., 1985)等诸多方面进行了分析, 但由于研究的菌株数量有限, 且培养过程中菌株的生物学特性易发生变异, 因而迄今为止仍没有一种有效的种的分类方法。近年来, 16S rDNA及其16S ~ 23S rDNA间隔序列作为一个分子标记, 广泛应用于各种微生物的分子遗传差异和分类鉴定研究(韦革宏等, 1999; 冯瑞华, 2000; Clawson et al., 1998; 1999)。尤其是16S ~ 23S rDNA间区, 比16S rDNA本身具有更强的高变性, 具有长度和序列上的多态性, 被认为是区分菌种、鉴定亲缘关系和了解遗传多样性的一种新方法(韦革宏等, 1999; 焦振泉等, 2001; Rouvier et al., 1996)。国内对沙棘(Hippophae rhamnoides)、黄果沙枣(Elaeagnus oxycarpa)、美洲赤杨(Alnus rugosa)、色赤杨(A.tinctoria sarg)、细枝木麻黄(Casuarina cunninghamiana)、短枝木麻黄(C.equisetifolia)等12个Frankia纯培养菌株和沙棘、色赤杨、毛赤杨(A.incana)、冬赤杨(A.nepalensis)等4种放线菌结瘤植物根瘤Frankia内进行了16S DNA扩增及酶切图谱分析, 认为Frankia菌株间存在着丰富的遗传多样性(刘忠等, 2001; 吴少慧等, 2001)。本文旨在利用16S ~ 23S rDNA间区rRNA操纵子基因(rrn)为分子指标, 对来自福建不同地区及广州的木麻黄、赤杨、杨梅(Myrica rubra)、胡颓子(Elaeagnus spp.)等几种放线菌结瘤植物共生Frankia进行PCR扩增和RFLP分析, 以其进一步了解Frankia的遗传差异和种群多样性, 为Frankia的分类鉴定提供参考。
1 材料与方法 1.1 供试菌株供试Frankia菌株21株, 其中木麻黄菌株12株, 杨梅菌株7株, 四川桤木和福建胡颓子(Elaeagnus oldhami)菌株各1株。菌株9550和2215分别由中国林业科学研究院热带林业研究所康丽华研究员和中科院微生物研究所刘志恒研究员惠赠, 其余均分离自福建放线菌结瘤植物, 各菌株及宿主、来源见表 1。
|
|
供试根瘤为细枝木麻黄、粗枝木麻黄、山神木麻黄(C.collina)、杨梅、四川桤木(Alus cremastogyne)、江南桤木(A.traboculosa)等6种, 其中粗枝木麻黄、细枝木麻黄、山神木麻黄根瘤采自海边沙地, 杨梅、四川桤木根瘤采自山地红壤, 江南桤木根瘤来自沼泽地, 海拔约900 m。采集的根瘤保存于-18 ℃冰箱直至使用。
1.3 引物及限制性内切酶引物为FGPS809ac和FGPL132', 其核苷酸序列为:5'-GGGGTCCGTAAGGGTC-3'和5'-CCGGGTTTCCCCATTCGG-3' (Rouvier et al., 1996), 由上海生物工程公司合成。扩增区域为16S ~ 23S rDNA间隔区的核糖体操纵子部分序列(rrn区)。限制性内切酶NciⅠ、RsaⅠ、MspⅠ、NdeⅡ, 系Promiga公司的产品。
1.4 菌株及根瘤DNA的提取参照彭源东等(1997)的CTAB提取方法, 并略加改进。离心收集BAP液体培养4 ~ 8周后菌体(约60 mg), 于研浆器中匀浆, 用TE8(Tris 50 mmol·L-1, EDTA 20 mmol·L-1, pH8.0)溶液洗涤1次, 之后用TNE溶液(Tri·HCl 10 mmol·L-1, NaCl 100 mmol·L-1, EDTA 1mmol·L-1, pH8.0)再洗涤2次, 离心收集菌体悬浮液, 加入300 μL 2 ×CTAB提取缓冲液(CTAB 2%, Tri·HCI 100 mmol·L-1, EDTA 20 mmol·L-1, NaCl 1.4 mol·L-1, pH8.0), 充分摇匀, 70 ℃水浴3 h。随后加入等体积的氯仿:异戊醇(24:1)溶液, 混匀, 7 000 ×g离心3 min, 移取上清液至新EP管, 加2 10体积的CTAB沉淀缓冲液(CTAB 5%, NaCl 0.35mmol·L-1), 65 ℃水浴10 min。混合物加入以酚:氯仿:异戊醇(24:23:1)溶液, 13 000 ×g离心10 min, 如此抽提2次后, 加入1 10体积3 mol·L-1 KAc, 以无水乙醇沉淀DNA, 干燥, 以TE(Tris 10 mmol·L-1, EDTA 1 mmol·L-1)溶液保存于-20 ℃。同时取2 μL于0.8%琼脂糖凝胶电泳检测。
根瘤内生菌DNA的提取参照Rouvier方法(Rouvier et al., 1996), 选取幼嫩根瘤瘤瓣(约1 ~ 10 mg), 以30% H2O2消毒根瘤表面, 洗净, 剥去表皮, 取根瘤尖端的含菌组织, 于300 μL提取缓冲液〔Tris (pH8) 100 mmol·L-1, EDTA 20 mmol·L-1, NaCl1.4 mol·L-1, CTAB 2%, 1% PVPP〕中研磨, 匀浆液置于65 ℃水浴60 min, 7 000 ×g离心10 min, 除去植物组织, 混合物以等体积酚氯仿等抽提, 以无水乙醇沉淀DNA, 干燥, 加入等体积聚乙醇-氯化钠混合液纯化(聚乙醇20%, NaCl 2.5 mol·L-1), 最后DNA在-20 ℃TE溶液中保存。
1.5 PCR扩增PCR扩增反应总体积为50 μL, 其中包括2 μL模板DNA(Frankia基因组DNA), 0.2 mmol·L-1引物、0.2 mmol·L-1dNTP, Taq酶(Promiga公司产品)1U。扩增反应在MJ PTC100扩增仪上进行:95 ℃变性3 min, 然后以94 ℃ 1 min, 50 ℃1 min, 72 ℃2 min程序循环扩增35次, 最后72 ℃延伸7 min。PCR产物在1.5%琼脂糖凝胶上电泳, EB染色, 于Bio-Rad Doc 2000凝胶成像仪上检查扩增效果。
1.6 RFLP分析根据内切酶的酶切位点及Rouvier的试验结果(Rouvier et al., 1996), 本试验选用4种内切酶NciⅠ、RsaⅠ、MspⅠ、NdeⅡ对PCR产物进行酶切。酶切反应体系均为5 μL, 反应温度为37 ℃, 酶切时间为2 h。酶切产物在1.5%的琼脂糖凝胶上电泳, 染色, 照像, 检测。
1.7 聚类分析对应每一个酶切图谱照片, 不同菌株间迁移率相同的带视为同一性状。酶切片段以Bio-Rad Quantity one软件进行带型分析, 用UPMGA方法构建聚类分析图。
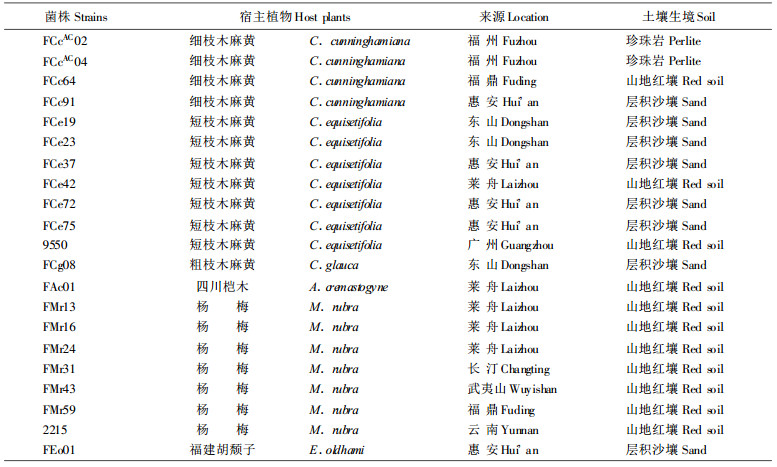
2 结果与分析 2.1 Frankia的16S ~ 23S rDNA rrn区特异带的扩增采用CTAB法从21株供试Frankia菌上均提取到DNA。用引物FGPS989ac和FGPL132'扩增Frankia菌16S ~ 23S rDNA rrn区, 12株木麻黄、4株杨梅和1株桤木等17株菌株获得一条稳定、清晰的特异条带, 片段约1 067 bp左右, 见图 1。对杨梅FMr31、FMr59、2215和福建胡颓子FEo01等9个菌株的DNA进行了多次扩增, 但未能获得rrn区域的扩增带。
|
图 1 木麻黄、杨梅、桤木Frankia 16S ~ 23S rDNA间区序列扩增 Fig. 1 Amplified Frankia rrn region of 16S~ 23S rDNA for C.spp., M.rubra, A.cremastogyne |
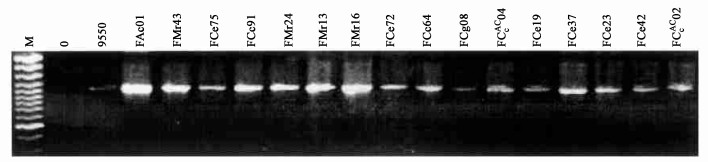
17个菌株扩增特异片断经4种内切酶酶切后结果见图 2。在NciⅠ内切酶的作用下, 17株Frankia菌扩增产物获得4条酶切片断, 分为2种酶谱类型。在这2种酶谱类型中, 3条小分子片断大小一致, 第4条片断存在差异。桤木菌株FAc01和杨梅菌株FMr43、FMr24、FMr13、FMr16的第4条片断大小约510bp, 其余12株木麻黄菌株的第4条酶切片断较小, 约为250 bp。RsaⅠ酶切扩增产物后, 17株菌株均获得同样大小的3条片断, 为同一种酶谱类型。用MspⅠ和NdeⅡ酶切, 供试菌株的扩增产物同样分成2种酶谱类群, 木麻黄菌株共有一种酶切图谱, 杨梅和桤木菌株为另一种酶切图谱, 这2种内切酶作用后各个酶切片段均较小。
|
图 2 NciⅠ、RsaⅠ、MspⅠ、NdeⅡ酶切Frankia 1 067 bp特异片段图谱 Fig. 2 The fingerprints generated after NciⅠ、RsaⅠ、MspⅠ、NdeⅡ digestion of amplified Frankia 1 067 bp length sequence from strains of Casuarina、Alnus、Myrica |
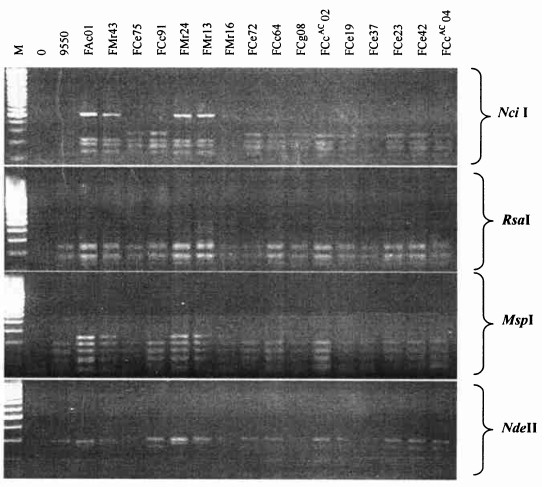
为了直观反映菌株间的16S ~ 23S rDNA基因的遗传关系, 将参试菌株的4种酶切图谱以UPMGA方法进行聚类分析, 构建了相似性树状图, 结果见图 3。
|
图 3 Frnakia 16S~ 23SrDNA间区PCR扩增产物酶切图谱分析聚类树状图 Fig. 3 The clustering of the products of 16S~ 23S rDNA spacer in Frankia by PCR |
从图 3可以看出, 17株供试Frankia菌可以分为2个类群:木麻黄类群和杨梅-桤木类群。来自福建东部福鼎、中部惠安和南部东山和广州等不同地点的细枝木麻黄、短枝木麻黄以及粗枝木麻黄的12株共生Frankia菌亲缘关系比较近, 同属于一个类群, 来自武夷山和莱舟的4个杨梅菌株和来自莱舟的桤木菌株在80%的相似性水平聚类为另一类群。各类群菌株之间存在一定的差异, 如在木麻黄类群中, 来自惠安的短枝木麻黄菌株FCe37与来自东山的FCe23相似性极高, 而与同一地点的FCe75和FCe72差异较大, 来自东山的粗枝木麻黄菌株FCg08则与来自惠安的短枝木麻黄FCe72相似性高。在杨梅-桤木类群中, 来自莱舟的杨梅菌株FMr24与同一地点的四川桤木菌株FAc01相似性在92%以上, 而与同一地点的杨梅FMr16相似性水平只有80%, 说明在同一类群中的同一地点Frankia菌也有较大差异性。
2.4 根瘤内Frankia的RFLP分析对生长于福建的细枝木麻黄、粗枝木麻黄、山神木麻黄、杨梅、四川桤木和江南桤木根瘤进行了DNA的提取, 均获得DNA提取物。以引物FGPS989ac和FGPL132'进行16S ~ 23S的rrn区域扩增, 6种放线菌结瘤植物均获得一条大小为1 067 bp的扩增片段。
选择内切酶NciⅠ进行酶切, 获得图 4结果。细枝木麻黄、粗枝木麻黄和山神木麻黄的扩增带酶切后均获得4条酶切片段, 片段大小相同。杨梅、四川桤木和江南桤木获得3条酶切片段, 第1、2条片段相同, 分别为152 bp和348 bp, 在第3条片段上存在着差异, 四川桤木为542 bp, 杨梅和江南桤木的为769 bp。酶切谱带聚类分析结果(图 5)表明, 6种放线菌结瘤植物共生Frankia可分成2个类群:3种木麻黄的Frankia遗传相似性水平在90%以上, 聚为一个类群, 杨梅和四川桤木、江南桤木聚类为另一类群。在杨梅-桤木类群中, 四川桤木根瘤内Frankia与杨梅、江南桤木的根瘤内生菌亲缘关系较远(相关系数约为0.64 ~ 0.87), 可再分成两个亚群, 四川桤木为一个亚群, 杨梅和江南桤木同属另一个亚群。值得注意的是, 杨梅根瘤与四川桤木根瘤采自同一地点, 其内生菌却差异较大, 而与沼泽地生长的江南桤木的内生菌亲缘关系近得多, 这说明了在不同的生境中存在着亲缘关系相近的Frankia, 同时在同一地点也存在着亲源关系远的Frankia类群。
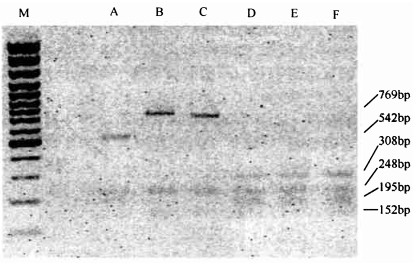
|
图 4 6种放线菌结瘤植物根瘤内生菌1 067 bp扩增产物的NciⅠ酶切图谱 Fig. 4 The fingerprints generated after NciⅠ digestion of amplified Frankia 1 067 bp length sequence from nodules of 6 species of actinorhizal plants A :四川桤木A.cremastogyne; B :杨梅M.rubra; C :江南桤木A.trabecu-losa; D :粗枝木麻黄C.glauca; E :山神木麻黄C.collina; F :细枝木麻黄C.cunninghamiana |
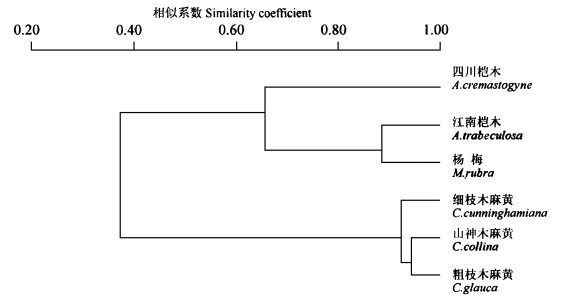
|
图 5 6 种放线菌结瘤植物根瘤内生菌相似性树状图 Fig. 5 Phylogenetic tree analysis of Frankia from nodules of 6 species of actinorhizal plants |
本研究在国内首次以16S ~ 23S PCR-RFLP方法对福建及广州的木麻黄、杨梅、桤木和福建胡颓子共生Frankia菌的遗传多样性进行了分析。引物FGPS989ac和FGPL132'可以扩增全部的供试木麻黄、桤木以及部分杨梅Frankia菌株16S ~ 23S rDNA rrn区域, 但是无法扩增杨梅菌株FMr31、FMr59、2215和福建胡颓子菌株FEo01。该引物为Bosco等人设计, 用于扩增木麻黄和桤木接种类群Frankia的rrn区域, 但不能扩增来自木麻黄根瘤上的无效菌株和非典型性菌株(Rouvier et al., 1996)。本试验结果也说明了它能有效地区分可木麻黄Frankia类群, 同时还适合部分杨梅Frankia菌株, 但对另一些杨梅菌株和胡颓子菌株无效, 因此利用16S ~ 23S间区了解杨梅和胡颓子Frankia类群菌株的遗传多态性, 尚需要进一步设计和筛选引物。
本试验酶切结果说明NciⅠ、MspⅠ和NdeⅡ等3种内切酶能较好地区别供试17株Frankia菌的差异性, 其中NciⅠ酶切后木麻黄类群菌株和杨梅、桤木类群菌株的酶切片段大小差异明显, 可以认为NciⅠ是木麻黄Frankia 16S ~ 23S扩增的判别性内切酶, 该结论与Rouvier等(1996)的试验结果一致。RsaⅠ酶切后17个菌株所形成的片段数及大小均相同, 说明该内切酶无法区别这些菌株的多态性。
Frankia纯培养菌株的PCR-RFLP研究结果说明来自福建不同地区和广州的木麻黄共生Frankia菌同源性程度很高, 为同一种类群, 这与Fernandez等(1989)用DNA相关性分析法、Nazaret等(1991)用16S rDNA序列扩增、Rouvier等(1966)用PCR-RFLP和彭源东等(1998)用RAPD研究方法的结果一致, 但Rouvier等(1966)同时又指出, 实际上存在于细枝木麻黄、短枝木麻黄根瘤内Frankia菌的遗传多样性非常高, 有5种PCR-RFLP类群。从福建省木麻黄根瘤Frankia的PCR-RFLP分析结果看, 与福建不同种木麻黄共生的Frankia菌只有一个类群, 遗传多样性相对低。
不少研究表明杨梅是最古老的放线菌结瘤植物, 与其共生的Frankia有多个分支, 较为混乱, 一部分内生菌与木麻黄-桤木类群相似, 一部分则具有胡颓子类群的遗传特性(Huguet et al., 2001)。本研究经多次试验未能获得部分杨梅和胡颓子菌株的PCR产物, 暗示这些菌株可能不同于木麻黄Frankia菌。由于菌株FEo01的宿主植物福建胡颓子生长在木麻黄林分内, 由此推论在木麻黄林地土壤中有不同类群的Frankia存在, 而宿主植物对与其共生的Frankia菌具有选择性。木麻黄菌株FCcAC 02和FCcAC 04是从四川桤木根瘤匀浆液接种细枝木麻黄所形成的根瘤上分离所得的, PCR-RFLP的研究表明它们与四川桤木菌株FAc01分属不同的Frankia类群, 这暗示接种木麻黄的四川桤木内生菌或者接种根瘤土壤上所附着的Frankia与FAc01菌不同。从Frankia纯培养和根瘤的PCR-RFLP结果也能充分表明, 存在于福建省土壤中的Frankia菌差异大, 至少有2个类群, 遗传多样性是比较丰富的。本研究结果同时说明了16S ~ 23SrDNA PCR-RFLP方法作为一种分子指标, 能很好地区别Frankia菌群的遗传多态性。
冯瑞华. 2000. 用AFLP技术和16S rDNA PCR-RFLP分析毛苜宿根瘤菌的遗传多样性. 微生物学报, 40(4): 341-344. |
焦振泉, 刘秀美. 2001. 细菌分类与鉴定的新热点:16S~23S rDNA间区. 微生物学通报, 28(1): 85-89. DOI:10.3969/j.issn.0253-2654.2001.01.022 |
刘忠, 吴少慧, 周斌, 等. 2001. 应用ARDRA技术研究Frankia菌多样性. 微生物杂志, 21(1): 20-23. |
彭源东, 柯丹兵, 张忠泽. 1997. Frankia基因型DNA提取及其PCR带型. 微生物学杂志, 17(3): 1-5. |
彭源东, 张忠泽. 1998. Frankia菌的遗传多样性的RAPD研究. 应用生态学报, 9(1): 59-63. DOI:10.3321/j.issn:1001-9332.1998.01.013 |
孙慧君, 丁鉴, 吴阳, 等. 1991. 不同寄主来源的Frankia血清学关系. 微生物杂志, 11(1): 82. |
韦革宏, 朱铭莪. 1999. 分子生物学新方法在根瘤菌分类中的应用. 西北农业大学学报, 27(2): 84-89. DOI:10.3321/j.issn:1671-9387.1999.02.018 |
吴少慧, 张惠文, 熊智, 等. 2001. ARDRA对植物根瘤内生放线菌Frankia多样性的研究. 应用生态学报, 12(6): 883-886. DOI:10.3321/j.issn:1001-9332.2001.06.020 |
张道海, 苏凤岩, 丁鉴, 等. 1989. Frankia菌同功酶图谱分析和分类. 微生物杂志, 9(3): 15-21. |
张道海, 苏凤岩, 丁鉴, 等. 1991. Frankia菌全细胞可溶性蛋白图谱分析及分类. 微生物杂志, 11: 38-42. |
Akimov V N, Dobritsa S V. 1992. Grouping of Frankia strains on the basis of DNA relatedness. Syst Appl Microbiol, 15: 372-379. DOI:10.1016/S0723-2020(11)80210-9 |
Baker D R, Pengelly W L, Hanna D G. 1981. Immunochemical analysis of relationships among the isolated Frankia (Actinomycetales). Int J Syst Bacteriol, 31: 148-151. DOI:10.1099/00207713-31-2-148 |
Baker D. 1987. Relationships among pure cultured strains of Frankia based on host specificity. Physiol Plant, 70: 245-248. DOI:10.1111/j.1399-3054.1987.tb06139.x |
Benson D R, Hanna D. 1983. Frankia diversity in an alder stand as estimated by SDS-PAGE of whole cell protein. Can J BOT, 61: 2919-2923. DOI:10.1139/b83-325 |
Clawson M L, Margarita C, Benson D R. 1998. Diversity of Frankia strains in root nodules of plants from the families Elaeagnaceae and Rhamnaceae. Appl Enviorn Microbiol, 64(9): 3539-3543. |
Clawson M L, Benson D R. 1999. Natural diversity of Frankia strains in actinorhizal root nodules from promiscuous hosts in the family Myricaceae. Appl Enviorn Microbiol, 65(10): 4521-4527. |
Dobritsa S V. 1985. Restriction analysis of the Frankia spp.genome. FEMS Microbiol Lett, 29: 123-128. DOI:10.1111/j.1574-6968.1985.tb00846.x |
Fermamdex M P, Meugnier H, Grimont P A D, et al. 1989. Deoxyrobonucleic acid relatedness among members of the genus Frankia. Int J Syst Bacteriol, 39: 424-429. DOI:10.1099/00207713-39-4-424 |
Gardes M, Lalonde M. 1987a. Identification and subgrouping of Frankia strains using sodium dodecyl sulfate-polyacrylamide gel electrophoresis. Physiol Plant, 70: 237-244. DOI:10.1111/j.1399-3054.1987.tb06138.x |
Gardes M, Bousquet J, Lalonde M. 1987b. Isozyme variation among 40 Frankia strains. Appl Environ Microbiol, 53: 1596-1603. |
Huguet V, Batzli J M, Zimpfer J F, et al. 2001. Diversity and specificity of Frankia strains in nodules of sympatric Myrica gale, Alnus incana, and Shepherdia canadensis determined by rrs gene polymorphism. Appl Environm Microbiol, 67(5): 2116-2122. DOI:10.1128/AEM.67.5.2116-2122.2001 |
Nazaret S, Cournoyer B, Normand P, et al. 1991. Phylogenetic relationship among Frankia genomic species determined by use of amplified 16S rDNA sequences. J Bateriol, 173: 4072-4078. |
Rouvier C, Prin Y, Reddell P, et al. 1996. Genetic diversity among Frankia strains nodulating members of the family Casuarinaceae in Autralia revealed by PCR and restriction fragment length polymorphism analysis with crushed root nodulaes. Appl Environm Microbiol, 62(3): 979-985. |
Simonet P, Normand P, Moiroud A, et al. 1985. Restriction enzyme digestion patterns of Frankia plasmids. Plant and Soil, 87: 49-60. DOI:10.1007/BF02277647 |
Torry J G, Racette S. 1989. Specificity among the Casuarinaceae in root nodulation by Frankia. Plant and Soil, 118: 157-164. DOI:10.1007/BF02232802 |
Weeler C T, Hooker J E, Cros A, et al. 1986. The improvement and utilization in forestry of nitrogen fixation by actinorhizal plants with special reference to Alnus in Scotland. Plant and Soil, 90: 393-406. DOI:10.1007/BF02277411 |
 2003, Vol. 39
2003, Vol. 39