文章信息
- 李淑娴, 尹佟明, 邹惠渝, 黄敏仁.
- Li Shuxian, Yin Tongming, Zou Huiyu, Huang Minren.
- 利用分子标记技术对林木近缘种进行遗传鉴别的研究
- DEVELOPING SPECIES-SPECIFIC MOLECULAR MARKERS TO DISTINGUISH CLOSELY RELATED TREE SPECIES
- 林业科学, 2003, 39(3): 129-135.
- Scientia Silvae Sinicae, 2003, 39(3): 129-135.
-
文章历史
- 收稿日期:2001-01-02
-
作者相关文章
长期以来,林木种子的真实性鉴定,一直是种苗生产、贸易中的突出问题(高捍东等,1995)。农作物品种的真实性鉴定方面已有大量的研究,也有较为成熟的技术。国际种子检验协会(ISTA),已经出版了《品种鉴定检验手册》;北美官方种子检验者协会(AOSA)也出版了《品种纯度检验手册》。这些检验规程的鉴定程序主要包括种子形态、快速测定、生长室测定和种子蛋白电泳等方法,其中所依赖的主要是同工酶技术。随着分子生物学的发展,对遗传背景一致的品种鉴别也有了更为成熟可靠的技术。但林业生产中,大多数树种的品系化或无性系化程度较差,而且在造林过程中,特别是在大规模的荒山绿化及飞播造林中,又多是使用种子,一些亲缘关系较近的种,如松属的某些种及落叶松属的某些种,其种子甚至幼苗的形态都非常相似,往往难以根据形态对其进行区分,所以在种的水平上进行遗传鉴别是林业生产中常常遇到的问题。林业生产周期长,真假混杂的种子和苗木给生产带来的后果更加不堪设想和难以补救,发展一整套实用、快速、准确的遗传鉴别技术是一项亟待解决的研究课题。
传统种的划分主要是根据形态性状。但若仅根据形态性状进行分类,一些近缘种的区分十分困难,甚至自相矛盾(Morgenstern et al., 1964; Manley, 1971; Gordon, 1976; Fowler et al., 1988)。例如某些种,在天然分布上存在重叠区域,在生殖上也不存在隔离,同时种内的遗传变异较大,因此许多形态性状用于种的分类时并不十分可靠(Gordon, 1976; Fowler et al., 1988)。另外,有些形态性状的可塑性较大,有的性状的差异仅在一定的生长发育阶段才表现出来(Gordon, 1976; Donoghue et al., 1992),这些都限制了表型性状在种的区分上的可靠性。交配系统也是影响种变异水平的重要因子(Grant, 1975; Hamrick et al., 1990),另外进化过程中的历史因子(如地质,气候等)对种的变异水平及变异分布也有重要影响。地理及生殖隔离是影响种间分化的最重要因素,对于大多数物种而言,即使在分布上存在重叠,在生态位上也存在显著的差异,如分布在美国的黑云杉和红云杉(Morgenstern et al., 1964; Gordon, 1976),再如中国黄山的马尾松和黄山松,这都显示了种在系统分类方面具有相对的稳定性。分子生物学的研究结果表明,基因的进化可以反映物种的进化,并且不同的基因由于变异频率不同,从而在不同分类等级上具有一定的保守性(Chase et al., 1993; Johnson et al., 1995; 汪小全等,1997)。另外由于生殖隔离、地理隔离或生态位上的差异,不同种在进化过程中固定了不同的基因,其中某些被固定的基因在种的水平上具有高度的保守性,这些差异通过DNA指纹技术可以比较容易的发现(Furman et al., 1997)。
利用分子标记技术对林木和作物的品种(无性系)进行鉴别,已有大量的研究报道,如Cheliak和Pitel(1983)利用同工酶技术进行山杨无性系的鉴别,Torres等(1990)利用RAPD技术鉴别不同玫瑰品种,陈洪等(1996)利用SRFA法构建水稻DNA指纹图谱。尹佟明等(1998)利用AFLP标记技术对南京林业大学引种和选育的美洲黑杨优良无性系进行了鉴别。在DNA水平上对品种进行鉴别,要求使用的标记具有高度的灵敏性,标记检测的DNA区域往往为不表达的核基因间隔区或串联重复区,因为这些区域的变异频率极高,所获得的标记许多具有个体特异性。在所有标记中,SSR标记是品种鉴别方面最灵敏、最可靠的。
如何利用分子标记在种的水平上对物种进行区分,一直是生物学家感兴趣的一个研究领域。分子标记技术产生以来,许多科学家在这个问题上进行了探索,Eckert(1990)利用同工酶技术对红云杉和黑云杉进行了种一级水平的区分,高捍东等(1995)也利用同工酶技术对松属几个种的鉴别进行了探索,但获得的标记均不具有100%种的特异性,同时这种技术也仅局限于实验室的研究中,很难在实际工作中推广应用。DNA水平上的标记技术产生以来,这方面的研究有了更多的报道。利用RAPD标记技术,Perron等(1995)对云杉属几个种进行了鉴别,Furman等(1997)获得了中美洲和墨西哥几个松属树种种一级水平上的特异RAPD谱带,并利用这些标记研究了这几个种的遗传关系。Bobola等利用RFLP标记技术(1992a, 1992b,1996)对红云杉和黑云杉进行了鉴别,共使用了5个rDNA重复区探针,2个叶绿体基因组探针和3个线粒体基因组探针,虽然获得的标记不具有100%种的特异性,但通过细胞器和核基因组RFLP标记的组合,可以对红云杉和黑云杉进行可靠的区分,然而种一级水平特异的RFLP标记无论在建立过程中还是推广应用过程中都是相当费时、费钱的,因此很难应用到实际检测工作中。Perron等人(1995)对红云杉和黑云杉进行研究,获得了所有检测样本4个种一级水平上100%保守的RAPD标记。但RAPD标记对反应条件及DNA质量等相当敏感(Ellsworth et al., 1993; Muralidharan et al., 1993; Michili et al., 1994; Vos et al., 1995),所获得的标记难以重复,在不同的实验室间应用时尤其如此,这也限制了RAPD标记在实际检测工作中的应用。因此要获得简单、可靠、在实际工作中易于推广应用且能够在种一级水平上进行鉴别的分子标记,需要在原理和方法上进行进一步的探索。
利用分子标记对种进行鉴别,要求获得的标记位点在进化上具有一定的保守性,同时具有一定的变异速率,即在种间具有多态性而在种内具有高度的保守性。根据上面介绍的情况,有两种类型的候选基因或DNA区段:(1)在进化上有同源性的基因位点或DNA区段;(2)物种在进化过程中固定的特异基因位点或DNA区段。选择哪些基因位点对树种进行鉴别主要取决于以下两点:(1)基因位点在所研究树种中的变异频率;(2)基因的结构域对基因变异的可预测程度。目前已对林木不同树种的许多基因进行了序列测定,相关信息可以在GENEBANK或相关数据库中检索,同时也有大量相关文献可供参考(Olmatead et al., 1994; Steele et al., 1994; Li et al., 1997;1999;汪小全等,1997)。因此根据相关DNA序列的信息发展不同树种种特异的分子标记具有较好的研究基础。
1 根据不同树种的同源基因家族发展种特异的单核苷酸多态性标记(SNPs)不同物种的某些基因在进化过程中有相同的起源,从而形成了不同物种间同源的基因家族,这些基因的进化过程可反应物种的进化过程。进化速率的差异使这些基因在不同分类等级水平上的保守性有所不同。在研究植物系统发育过程中,通常选用的基因位点包括叶绿体基因组的16S rRNA, 23S rRNA, psbA, psbD, psaB, psbC, psaA, rbcL, atpB, ndhA, atpA, ndhD, rpoB, rpoCI, ndhA(包含一个大的内含子),rpoA, ndhF, rpoC2,matK基因等(汪小全等,1997),上述基因中,matK基因是叶绿体基因组蛋白编码基因中变异速率最快的基因之一(Olmatead et al., 1994),和其它叶绿体基因相比,不同物种间的编码蛋白所具有的氨基酸序列的一致性相对较低(如烟草和水稻之间一致性为59%)。该基因位于trnK基因(UUU)的内含子中,长约1 500bp,编码一种成熟酶(Sugita et al., 1985),一般用于科内、属间、甚至种间关系的研究。在裸子植物中,matK基因每位点的变异为rbcL基因的3.4倍(Johnson et al., 1995)。一般系统学研究中,基因的变异要求在所研究的分类等级中具有5%~15%的变异,但在种的区分上只要基因序列存在单碱基的差异就可进行区分。据Hilu和Liang(1997)报道,matK基因在弯扭松和黑松间的核苷酸序列存在1.1%的差异。Gernano和Klein(1999)利用该基因序列差异产生的单核苷酸多态性标记(single nucleotide polymorphisms, SNPs)对黑云杉、红云杉和白云杉进行鉴别,结果表明在红云杉和黑云杉之间仅有0.1%的变异,白云杉和红云杉之间的变异为0.3%,白云杉和黑云杉之间的变异为0.4%,虽然变异频率很低,在该基因区仍获得了6个种特异的SNPs标记。matK基因的变异程度在松属树种中相对较高,所以matK基因在林木不同种的鉴别,尤其是在松属树种的鉴别中有较大的应用潜力。
另外叶绿体基因组的内含子(intron)区和基因间隔区(inter genic spacers, IGS)比编码区存在更多的变异(Taberlet et al., 1991; Demesure et al., 1995; Perezde la Rosa et al., 1995),这为寻找种的特异分子标记提供了一个丰富来源。在所有陆生植物中,叶绿体基因组的基因顺序相对较为保守(Olmstead et al., 1994),如松属的叶绿体基因顺序与云杉属和铁杉属近似(White et al., 1993)。目前,日本已完成了黑松叶绿体全基因组的测序工作(Wakasugi et al., 1994)。叶绿体基因组基因顺序的保守性及已获得的黑松叶绿体全基因组序列,为我们合成PCR引物来寻找松属树种不同种间叶绿体基因间隔区的差异提供了有利条件。Gernano和Klein在鉴别白云杉、红云杉和黑云杉的过程中,对trnK基因5′端的内含子进行了序列测定,同时也检测了rpl33-psaJ-trnP基因的间隔区,分别获得了4个和3个种特异的SNPs标记。松属树种在相应区段的核苷酸变异频率还要高些,黑松(GenBank X57097;Lidholm et al., 1991)与弯扭松(GenBank D17510;Tsudzuki et al., 1992)的trnK内含子区存在1%的差异,与matk基因的变异接近,因此trnK基因内含子区和叶绿体基因组的基因间隔区也适于用来产生种特异的SNPs标记。
叶绿体基因组是单亲遗传的,同时进化上的保守性相对较高,而核基因是双亲遗传的,由核基因组产生的种特异分子标记可以补充由叶绿体产生的种特异分子标记的不足(Soltis et al., 1992)。高等植物的核糖体DNA重复域包含18S、5.8S和26S基因,这3个基因被内转录间隔区(internal transcribed spacer, ITS)ITS-1和ITS-2分隔。其中ITS-1和ITS-2的变异速率比5.8S、18S和26S基因要快得多(Hamby et al., 1992; Baldwin et al., 1995)。ITS在被子植物中的长度变异很小,且扩增片段长度较小,一般小于700bp,所以ITS区在被子植物的系统学研究中应用很广,Baldwin等(1995)推断ITS区在被子植物的科内、属间、种间甚至种内发育关系的研究中有重要应用价值。但在裸子植物中,ITS的变异十分复杂,仅ITS-1区的长度变异就可相差几kb,在欧洲云杉的同一个体中,Karvonen(1993)发现不同ITS-1拷贝的长度可相差约500bp。ITS在裸子植物中的拷贝数比被子植物要高的多,如在红云杉中,拷贝数高达106(Bobola et al., 1992b),另外ITS不同重复间可能存在杂合性差异(Bobola et al., 1992b; Beech et al., 1993; Karvonen et al., 1993; Liston et al., 1999)。所以ITS不适合于裸子植物高分类等级的系统学研究,但ITS区在裸子植物种的分类等级上相对保守,Liston等(1999)利用3′端ITS区(5.8S,ITS-2和ITS-1约200bp片段)研究了松属47个种的系统发育关系,结果表明ITS区是松属最丰富的DNA区段之一,所以该区段是获得松属树种种特异性分子标记的一个重要来源。Liston等(1999)通过研究指出,由于ITS-1比5.8S,ITS-2有更丰富的变异,所以增加ITS-1区的信息,有望解决松属近缘种之间的关系。所以在松属树种鉴别研究中,ITS区是获得种特异分子标记的一个最为丰富的来源。ITS区拷贝数及位点杂合性对系统学研究带来许多影响,但对发展种特异分子标记影响较小。
以上介绍的是已知基因序列、基因位置与性质的情形。根据已知的DNA序列信息,可发展单核苷酸多态性(single nucleotide polymorphisms,SNPs)标记,用于在种的水平上鉴别物种,Gernano等(1999)在这方面已进行了成功的尝试,他们利用matK基因、trnK基因内含子、叶绿体基因间隔区和ITS区序列发展的种特异SNP标记,成功的对亲缘关系较近的云杉属的3个种(白云杉、红云杉和黑云杉)进行了区分,其中2个SNPs标记可将黑云杉从白云杉和红云杉中区分开来,这2个标记在所检测样品中的种内保守性达96%~100%。另有5个SNPs标记可将白云杉从黑云杉和红云杉中区别开来,这些标记在所检测样品中的种内保守性均达100%,他们利用获得的标记对植物分类学家提供的匿名样品的鉴别准确率达100%。他们的研究还表明,由上述序列差异转化的SNPs标记在检测过程中比其它标记(如RFLP和RAPD)更省力,并可直接应用于大量样品的检测工作中。由上述种的保守序列转化的种特异SNPs标记有以下优点:检测的基因位点的差异、基因所处位置及基因的性质是已知的;SNPs标记一旦获得,就可通过简单、廉价的手段检测,具有简单、快速、可靠且成本低的优点;可直接应用于大量样品的实际检测工作中,易于推广应用。
SNPs检测手段主要有以下3种:1.等位特异性PCR(allele-specific PCR, ASPCR)(Okayama et al., 1989; Sommer et al., 1989;Wu et al., 1989); 2.单链构相多态性电泳(single strand conformation polymorphism,SSCP)(Shefifeld et al., 1993); 3.限制性内切酶酶切(PCR-RFLP)(Landegren et al., 1998)。这3种技术均是基于PCR基础上的,所以只要少量样品即可满足分析需要。
上面介绍的matK基因、trnK基因内含子、叶绿体基因间隔区(rpl33-psaJ-trmP)和ITS区序列(图 1)在许多不同树种中已进行了大量的研究(Bobola et al., 1992b;Beech et al., 1993;Karvonen et al., 1993;Liston et al., 1999;Wakasugi et al., 1994; Lidholm et al., 1991;Tsudzuki et al., 1992;Hilu et al., 1997)。许多种上述区段的核苷酸序列也已经测定,可直接在相关的DNA序列数据库中查找比对,发现SNPs并设计相应的引物。由于所利用的DNA区段在种内具有校高的保守性,所以获得的SNPs标记在种内保守性较高,通过上面介绍的检测手段对所获得的SNPs标记在种内的标准样品中进行验证,就可发展出用于实际检验中的种特异SNPs标记。所以在不同树种种子的鉴别研究中,对上述序列信息的开发利用,将是一个极有开发前景的研究方向。
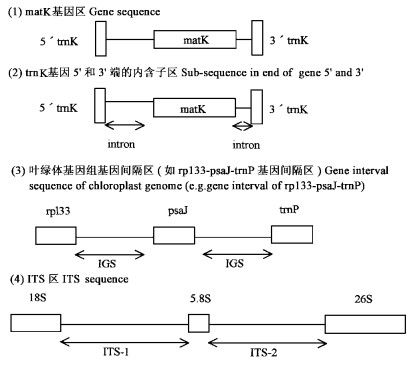
|
图 1 开发种特异分子标记可利用的DNA区段 Fig. 1 The available DNA sequence for developing species-specific markers |
种特异分子标记还有一个重要来源,即物种在进化过程中固定的特异基因位点或DNA区段。利用分群法(bulked segregate analysis, BSA)(Michelmore et al., 1996)结合RAPD (Williams et al., 1990)或AFLP(Vos et al., 1995)标记技术可比较容易地获得这些种特异的扩增谱带(Furman et al., 1997)。利用上述方法在不同树种中筛选种特异扩增谱带的大致过程如下:每个种取不同种源的样本40~50株,种子发芽后的幼苗混合提取DNA,建立种的初级基因池(gene pool),通过RAPD或AFLP扩增,寻找在各基因池间有差异的谱带。然后每个种分种源各取约10株样本混合提取DNA,建立各个种的亚基因池(subgene pool),在亚基因池间验证在初级基因池间获得的差异谱带的可重复性及在种内的保守性。最后,对各个种不同种源的种子分单粒(每个种源检测样本数大于30)进行验证,从而最终确定所获得的差异谱带是否为种特异的谱带。按此法,我们对松属的5个种进行了种特异RAPD谱带的筛选,并获得了预期的研究结果。下图所示为BSA法和RAPD标记相结合获得的马尾松与黄山松相区别的马尾松的种特异的RAPD谱带(图 2)。RAPD扩增谱带OPAKl7-200为马尾松区别于黄山松的种特异的RAPD谱带(样品来源于本校林木种子检验中心标本室收集的标准样品)。Furman等(1997)利用同样的方法获得了中美洲和墨西哥的几个松属树种种特异的RAPD谱带,并利用这些标记研究这几个种的遗传关系。
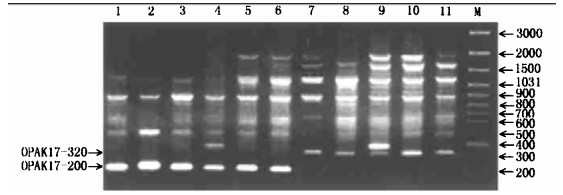
|
图 2 马尾松区别于黄山松的种特异RAPD谱带 Fig. 2 The species-specific RAPD band which distinguish Pinus massoniana from Pinus taiwanensis 1~6为6个不同种源的马尾松各取10个样本建立的马尾松的亚基因池,7~11为5个不同种源的黄山松各取10个样本建立的黄山松的亚基因池,M为标准分子量。 1~6 are subgene pools of P.massoniana established by each 10 samples from 6 provenances, 7~11 are subgene pools of P.taiwanensis established by each 10 samples from 5 provenances, M is the standard molecular weight. |
虽然利用分群法结合RAPD或AFLP标记技术可以快速获得种特异的分子标记,但由于这些标记本身的局限性,还难以将获得的标记直接应用于实际检验中。要在实际检验过程中能够应用和推广,必须对获得的标记进行转化。这一过程是将获得的RAPD谱带或AFLP谱带经过克隆、测序,然后根据测得的序列合成特异的PCR引物,从而对相应的DNA区段进行扩增,由此而产生的标记称为序列标记位点(sequence tag site,STS,由特征谱带转化)或序列特征扩增域(sequence characterized amplified region,SCAR,由与特定性状相连锁的特征谱带转化)。STS或SCAR标记的发展主要受标记辅助选择育种(marker aided selection, MAS)研究的推动和影响。育种学家们希望将获得的与某一性状相连锁的分子标记直接应用到实际的选育过程中,而SCAR和STS标记具有简单、快速、稳定性好且可以同时对大量样品进行检测的优点(贾继增,1996),可满足上述要求。近几年来有许多这方面的研究报道,如Bradeen和Simon(1998)将获得的与胡萝卜芯的颜色相连锁的AFLP标记转化为SCAR标记,Myburg等(1998)把与抗小麦蚜虫基因(Dn2)相连锁的RAPD标记转化为SCAR标记用于标记辅助选择育种研究。Geffroy等(1998)把与Phaseolus vulgaris中抗碳疽病基因Co-2相连锁的RAPD标记转化为SCAR标记。印度的Alok等则将VAM细菌(Gigaspora margarita and Glomus intradices)的特异RAPD谱带转化为SCAR标记用于植物受VAM细菌侵染的检测。STS或SCAR标记是一种可直接应用于实际的育种程序或种子与苗木的检验工作中的标记。将获得的种特异RAPD谱带转化为STS或SCAR标记后,就可以应用于实际的检验工作中。在上述研究中需要注意原始材料来源的可靠性及遗传上的纯度,要避免采集种间的天然杂种及可能发生基因渐渗的材料。研究和开发根据表型难以区分的林木近缘种的种特异分子标记具有重要意义:(1)可提高种子和苗木的真实性鉴定水平,突破传统的形态鉴定模式。(2)可用于裁决种子调拨中的真实性纠纷,为林木种子监督检验部门提供科学的检测手段,有利于种子生产、销售环节的监督和管理。(3)为打假提供了科学依据,避免假种子浪费土地、贻误造林季节。(4)填补我国《林木种子检验规程》的空白。同时发展不同树种种特异的分子标记,为林木近缘种的系统发育、分类、种质资源保存等的研究提供了更为丰富的资料和信息。
陈洪, 王振山, 朱立煌, 等. 1996. SRFA法构建水稻DNA指纹图谱. 生物工程学报, 12(3): 266-269. |
高捍东, 沈永宝, 苑兆和, 等. 1995. 用过氧化物同功酶分析鉴定松属种子的真实性. 中南林学院学报, 15(1): 30-33. |
贾继增. 1996. 分子标记种质资源鉴定的分子标记育种. 中国农业科学, 29(4): 1-10. |
汪小全, 洪德元. 1997. 植物分子系统学近五年的研究进展概况. 植物分类学报, 35(5): 465-480. |
尹佟明, 孙晔, 易能君, 等. 1998. 美洲黑杨无性系AFLP指纹分析. 植物学报, 40(8): 778-780. DOI:10.3321/j.issn:1672-9072.1998.08.015 |
Baldwin B G, Sanderson M J, Porter J M, et al. 1995. The ITS region of nuclear ribosomal DNA:a valuable source of evidence on angiosperm phylogeny. Ann.Mo Bot Gard, 82: 247-277. DOI:10.2307/2399880 |
Beech R N, Strobeck C. 1993. Structure of the intergenic spacer region from the ribosomal RNA gene family of White spruce (Spruce glauca). Plant Mol Biol, 22: 887-892. DOI:10.1007/BF00027373 |
Bobola M S, Eckert R T, Klein A S, et al. 1992a. Restriction fragment variation in the nuclear ribosomal DNA repeat unit within and between P.rubens and Picea mariana. Can J For RES, 22: 255-263. DOI:10.1139/x92-033 |
Bobola M S, Eckert R T, Klein A S, et al. 1992b. Five major nuclear ribosomal repeats represent a large and variable fraction of the genomic DNA of Picea rubens and Picea mariana. Mol.Biol Evol, 9: 125-137. |
Bobola M S, Eckert R T, Klein A S, et al. 1996. Using nuclear and organelle DNA markers to discriminate among Picea rubens, Picea mariana, and their hybrids. Can J For Res, 26: 433-443. DOI:10.1139/x26-049 |
Bradeen J M, Simon P M. 1998. PCR-based Markers from AFLP Bands. Plant & Animal Genome Ⅵ Meeting-January: 18-22. |
Chase M W, Soltis D E, Morgan D, et al. 1993. Phylogenetics of seed plants:an analysis of nucleotide sequences from the plastid gene rbdL. Ann MoBot Gard, 80: 528-580. DOI:10.2307/2399846 |
Cheliak M W, Pitel J A. 1983. Electrophoretic identification of clones in trembling aspen. Can J For Res, 14: 740-743. |
Demesure B, Sodzi N, Petit R J, et al. 1995. A set of universal primers for amplification of polymorphic non-coding regions of itochondrial and chloroplast DNA in plants. Mol Ecol, 4: 129-131. DOI:10.1111/j.1365-294X.1995.tb00201.x |
Donoghue M J, Sanderson M J.The suitability of molecular and morphological evidence in reconstructing plant phylogeny.In: Soltis P S.Soltis D E, Doyle J J(eds)Molecular systematics of plants.Chapman and Hall.New York, 1992: 340-368
|
Eckert T T. 1990. Genetic variation in red spruce pollution response, Final report. Spruce-Fir Research Cooperative, ortheast Forest Experiment Station, Radnor, Pa. |
Ellsworth D L, Rittenhouse K D, Honeycutt R L. 1993. Artifactual variation in randomly amplified polymorphic DNA banding patterns. Biotechniques, 14: 214-217. |
Fowler D P, Park Y S, Gordon A G. 1988. Genetic variation of red spruce in the Maritimes. Can J For Res, 18: 703-709. DOI:10.1139/x88-107 |
Furman B J, Grattapaglia D, Dvorak W S, et al. 1997. Analysis of genetic relationships of central american and mexicanpines using RAPD markers that distinguish species. Molecular Ecology, 6: 321-331. DOI:10.1046/j.1365-294X.1997.00183.x |
Geffroy V, Creusot F, Falquet J, et al. 1998. A family of LRR sequences in the vicinity of the Co-2 locus for anthracnose resistance in Phaseolus vulgaris and its potential use in marker-assisted selection. Theoretical and Applied Genetics, 96: 494-502. DOI:10.1007/s001220050766 |
Gernano J, Klein A S. 1999. Species-specific nuclear and chloroplast single nucleotide polymorphisms to distinguish Picea glauca, P.mariana and P.rubens. Theor Appl Genet, 99: 37-49. DOI:10.1007/s001220051206 |
Gordon A G. 1976. The taxonomy and gentics of Picea rubens and its relationship to Picea mariana. Can J Bot, 54: 781-813. DOI:10.1139/b76-084 |
Grant V. 1975. Genetics of flowering plants. New York.NY:Columbia university Press: 514. |
Hamby R K, Zimmer E A.Ribosomal RNA as a phylogenetic tool in plant systematics.In: Soltis P S.Soltis D E.Doyle J J(eds).Molecular systematics of plants.Chapman and Hall, New York.1992: 50-91
|
Hamrick J L, Godt M J.Allozyme diversity in plant species.In AHD.Brown.M T.Clegg, A.L.Kahler, and B.S.Weir, (eds).Plant population genetics, breeding and genetic resource, Sinauer, Sunderland, M A.1990: 43-63
|
Hilu K W, Liang H. 1997. The matK gene:sequence variation and application in plant systematics. Am J Bot, 84: 830-839. DOI:10.2307/2445819 |
Johnson L A, Soltis D E. 1995. Phylogenetic inference in Saxi-fragaceae sensu stricto and Gilia(Polemoniaceae)using matK sequences. Ann Mo Bot Gard, 82: 149-175. DOI:10.2307/2399875 |
Karvonen P, Savolainen O. 1993. Variation and inheritance of ribosomal DNA in Pinus sylvestris L.(Scots pine). Heredity, 71: 614-622. DOI:10.1038/hdy.1993.186 |
Landegren U, Nilsson M, Kwork P Y. 1998. Reading bits of genetic information:methods for single-nucleotide polymorphism analysis. Genome Res, 8: 769-776. DOI:10.1101/gr.8.8.769 |
Li J H, Bogle A L, Klein A S. 1997. Interspecific relationships and genetic divergence of the disjunct genus Liquidambar(Hamamelidaceae). Rhodora, 99: 229-240. |
Li J H, Bogle A L, Klein A S.Phylogenetic ralationships of the Hamamelidaceae: evidence from the nucleotide sequences of the plastid gene matK.Plant Syst Evol.(in press)
|
Lidholm J, Gustafsson P. 1991. A three-step model for the rearrangement of the chloroplast trnK-psbA region of the gymnosperm Pinus contorta. Nucleic Acids Res, 19: 2881-2887. DOI:10.1093/nar/19.11.2881 |
Liston A, Robinson W A, Pinero D, et al. 1999. Phylogenetics of Pinus(Pinaceae)based on nuclear ribosomal DNA internal transcribed spacer region sequences. Mol Phyl Evol, 11: 95-109. DOI:10.1006/mpev.1998.0550 |
Manley S A M. 1971. Identification of red, black and hybrid spruce. Can Dep Environ For Can For Serv, Ottawa, Canada. |
Michelmore R W, Paran I, Kesseli R V. 1996. Identification of markers linked to disease ressistant gene by bulked segregation population. Proc, Natl Acad Sci, USA, 88: 9828-9832. |
Michili M R, Bova R, Pascale E, et al. 1994. Reproducible DNA fingerprinting with the random amplified polymorphic DNA (RAPD) method. Nucleic Acids Res, 22: 1921-1922. DOI:10.1093/nar/22.10.1921 |
Morgenstern E K, Farrar J L. 1964. Introgressive hybridization in red spruce and black spruce. Technical Report No.4.Faculty of Forestry, University of Toronto, Toronto.Ontario: 1-46. |
Muralidharan K, Wakeland E K. 1993. Concentration of primer and template qualitatively affects products in random amplified polymorphic DNA PCR. Biotechniques, 14: 362-364. |
Myburg A A, Cawood M, Wingfield B D. 1998. Development of RAPD and SCAR markers linked to the Russian wheat aphid resistance gene Dn2 in wheat. Theoretical and Applied Genetics, 96: 1162-1169. DOI:10.1007/s001220050852 |
Okayama H, Curiel D T, Brantly M L. 1989. Rapid, non-radioactivity detection of mutations in the human genome by allele-specific amplification. J Lab Clin Med, 114: 105-113. |
Olmatead R G, Palmer J D. 1994. Chloroplast DNA systematics:a review of methods and data analysis. Am J Bot, 81: 1205-1224. DOI:10.1002/j.1537-2197.1994.tb15615.x |
Perezde la Rosa J, Harris S A, Farjon A. 1995. Noncoding chloroplst DNA variation in Mexican pines. Theor.Appl.Genet, 91: 1101-1106. |
Perron M, Gordon A G, Bousquet J. 1995. Species-specific RAPD fingerprints for the closely related Picea mariana and P.rubens. Theor.Appl.Genet, 91: 142-149. DOI:10.1007/BF00220871 |
Shefifeld V C, Beck J S, Kwitek A E. 1993. The sensitivity of single-strand conformation polymorphism analysis for the detection of single base substitutions. Genomics, 16: 325-332. DOI:10.1006/geno.1993.1193 |
Soltis P S.Doyle J J, Soltis D E.Molecular data and polyploid evolution in plants.In: Soltis P S.Soltis D E, Doyle J J(eds), Molecular systematics of plants.Chapman and Hall, New York, 1992: 177-201
|
Sommer S S, Cassady J D, Sobell J L. 1989. A novel method for detecting point mutations of polymorphisms and its application to population screening for carriers of phenylketonuria. Mayo Clin Proc, 64: 1361-1372. DOI:10.1016/S0025-6196(12)65378-6 |
Steele K P, Vilgalys R. 1994. Phylogenetic analysis of Polemoniaceae using nucleotide sequences of the plastid gene matK. Sys Bot, 19: 126-142. DOI:10.2307/2419717 |
Sugita M, Shinozaki K, Sugiura M. 1985. Tobacco chloroplast tRNA Lys(UUU) gene contains a 2.5-kilobase-pair intron:an open reading frame and a conserved boundary sequence in the intron. Proc Natl Acad Sci USA, 82: 3557-3561. DOI:10.1073/pnas.82.11.3557 |
Taberlet P, Gielly L, Pautou G, et al. 1991. Universal primers for amplification of three non-coding regions of chloroplast DNA. Plant Mol Biol, 17: 1105-1109. DOI:10.1007/BF00037152 |
Torres A M. 1993. Identifying rose cultivars using random amplified polymorphic DNA markers. HortScience, 28(4): 333-334. DOI:10.21273/HORTSCI.28.4.333 |
Tsudzuki J, Nakashima K, Tsudzuki T, et al. 1992. Choproplast DNA of black pine retains a residual inverted repeat lacking rRNA genes:nucleotide sequences of trnQ, rtnK, psbA and trnH and the absence of rps16. Mol Gen Genet, 232: 206-214. |
Vos P, Hogers R, Bleeker M, et al. 1995. AFLP:A new technique for DNA fingerprinting. Nuc Aci Res, 23: 4407-4414. DOI:10.1093/nar/23.21.4407 |
Wakasugi T, Tsudzuki J, Ito S. 1994. Loss of all ndh genes as determined by sequencing the entire chloroplast genome of the black pine Pinus thunbergii. Proc Natl Acad Sci USA, 91: 9794-9798. DOI:10.1073/pnas.91.21.9794 |
White E E, Watkins R F, Fowler D P. 1993. Comparative restriction site maps of chloroplast DNA of Picea abies, Picea glauca, Picea mariana, and Picea sitchensis. Can J For Res, 23: 427-435. DOI:10.1139/x93-060 |
Williams J G K, Kubelik A R, Livak K J. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nuc Aci Res, 18: 6531-6535. DOI:10.1093/nar/18.22.6531 |
Wu D Y, Ugozzoli L, Pal B K, et al. 1989. Allele-specific enzymatic amplification of beta-globin genomic DNA for diagnosis of sickle cell anemia. Proc Natl Acad Sci USA, 86: 2757-2760. DOI:10.1073/pnas.86.8.2757 |
 2003, Vol. 39
2003, Vol. 39
