文章信息
- 邱英雄, 傅承新, 何云芳.
- Qiu Yingxiong, Fu Chengxin, He Yunfang.
- 乐昌含笑不同类型鉴定的ISSR-PCR分析浙江省林业厅种苗管理站杭州310029
- IDENTIFICATION OF MICHELIA TSOI TYPES USING ISSR-PCR MARKER ASSAYS
- 林业科学, 2002, 38(6): 49-52.
- Scientia Silvae Sinicae, 2002, 38(6): 49-52.
-
文章历史
- 收稿日期:2001-08-27
-
作者相关文章
2. 浙江省林业厅种苗管理站 杭州 310029
2. Management Station of Seed and Seedling, Zhejiang Forestry Bureau Hangzhou 310029
20世纪90年代以来, DNA分子标记技术的出现为在DNA分子水平上鉴定果树品种提供了强有力的工具(贾继增, 1996; 栾雨时等, 1999; Hu et al., 1999)。Zietkiewicz等(1994)提出了锚定SSR的新策略, 简称ISSR。ISSR类似于RAPD, 但利用包含重复序列并在3’或5’锚定的单寡聚核苷酸引物对基因组进行PCR扩增。它可用于品种鉴定和居群遗传学研究。近年来ISSR分析技术在品种鉴定、种质资源和遗传多样性研究中得到广泛应用(钱韦等, 2000; Gilbert et al., 1999; Prevost et al., 1999)。
乐昌含笑(Michelia tsoi Dandy)又名南方白兰花, 木兰科含笑属, 是南方常绿阔叶林的主要组成树种之一。水平分布西起贵州西北部, 东抵福建北部, 北至江西北部, 南达广东北部, 以南岭山系地区为中心分布区, 一般分布在海拔300~1 500 m的深山沟谷两侧的常绿阔叶林中。中国林科院亚热带林业研究所从上世纪70年代后期, 对乐昌含笑的自然分布区进行实地考察, 并在浙江长乐林场进行了引种试验, 共收集6个种源, 35个家系。通过10 a优株选育和大批量繁殖实验、品种和新类型的比较实验, 选育出6个株型、叶型和花期差异大的类型(盛能荣等, 1996)。本研究在已有工作的基础上, 利用ISSR分析技术对乐昌含笑的6种类型进行确定, 对新类型在DNA水平的遗传变异进行了分析, 并筛选出分辨率高、重复性好, 而且能扩增出特有条带的引物。
1 材料和方法 1.1 植物材料试验所用的材料为乐昌含笑的6个变型(表 1)。
|
|
采取新鲜叶片, 硅胶彻底干燥, 称取0.1 g干叶, CTAB法(Doyle et al., 1987)提取各类型总DNA, 用1.0%琼脂糖电泳检测DNA质量, 图 1为乐昌含笑的6种类型的DNA电泳结果, DNA的纯度和浓度通过紫外吸收法测定(邹喻苹等, 2001)。
|
图 1 乐昌含笑6个类型的基因组DNA电泳结果 Fig. 1 The electrophoresis result of genome DNAof 6 Michelia tsoi types |
ISSR-PCR扩增反应是在英国Hybaid公司生产的SPRT001型PCR仪上进行, 反应条件确定为:25 μL的PCR反应液内含:模板约60 ng, 1Utaq酶, 1.5 mmol·L-1 MgCl2, 4种dNTP各0.25mmol·L-1, 0.3 μmol·L-1引物, 0.5 mmol·L-1亚精胺, 2%甲酰胺。PCR扩增条件:94℃预变性5 min, 然后进行45个循环:94℃变性1 min, 52~55℃复性45 s, 72℃延伸2 min, 循环结束后72℃延伸5 min。PCR产物在2.0%琼脂糖(0.5 μg·mL-1)上电泳, EPSON紫外自动成像仪照相。
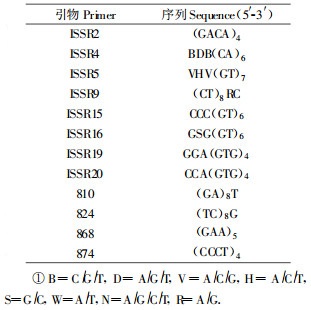
1.4 引物筛选ISSR引物是根据加拿大British Columbia大学公布的序列设计, 由上海生工公司合成。从30个引物中选出12个扩增条带较多、信号强、背景清晰的引物用于ISSR-PCR反应(表 2), 变性温度根据引物的Tm值变化1~3℃, ISSR16, ISSR19和ISSR2的变性温度均确定为52℃。
|
|
电泳图谱的每条带(DNA片段), 均为一个分子标记(Marker), 代表一个引物的结合位点。根据各分子标记的迁移率及其有无统计所有的二元数据; 有带(显性)记作1, 无带(隐性)记为0, 强带和弱带的赋值均为1。对于多态位点, 仅在重复实验中能稳定出现的差异带用于数据分析。
1.5.2 数据的统计分析方法利用DPS统计软件的欧氏可变类平均法构建类型之间的分子系统树(唐启义等, 1997), 并计算两个类型之间的遗传距离。
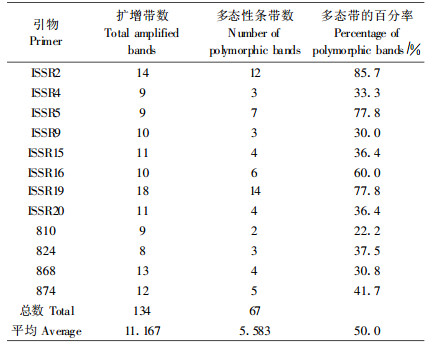
2 结果与分析 2.1 DNA扩增结果和基因组多样性程度分析表 3结果表明, 所选用的12个引物对所有样品进行PCR扩增, 共计扩增出134条DNA条带, 其中, 多态性DNA条带67条, 占总带数的50%, 每个引物扩增的DNA带的数目在2~14条, 平均5.583条。扩增出的DNA带的大小在300~2 500bp。
|
|
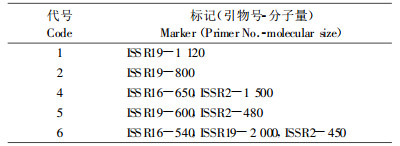
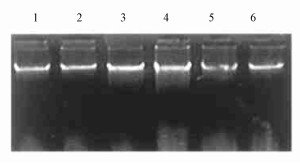
通过30个引物的筛选, 检测了乐昌含笑的基因组134个位点, 大部分类型均有特有条带产生, 可作为重要的分子性状用于早期品种或类型鉴定和早期性状预选。6种类型仅大叶稠密型无特有条带, 其它5种类型能根据其特有条带可各自区分开(图 2)。有特有条带的类型占供试类型的83.6% (表 4)。
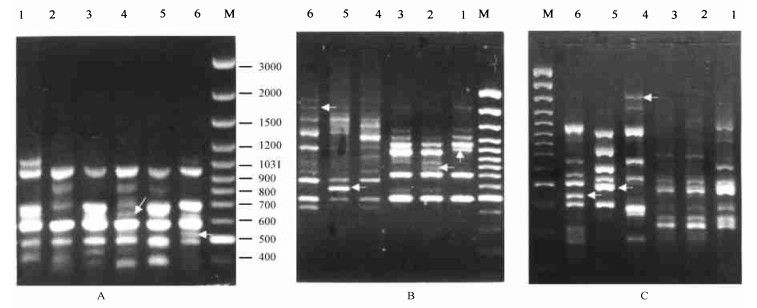
|
图 2 乐昌含笑6个类型的ISSR-PCR扩增带谱(箭头示特异性扩增产物) Fig. 2 ISSR-PCRprofiles of 6 M.tsoi types (Arrows indicate specific amplification products) 1~6为类型代码, M:DNA标准分子量Number 1~6 stand for types.Mindicates DNA standard molecular weight.A.ISSR16, B.ISSR19, C.ISSR2. |
|
|
从图 3可知, 乐昌含笑的6个类型在遗传距离约为3.6处被明显分为两大类, 其中, 细叶稠密型、皱叶紧密型和大叶稠密型聚为一类, 而枝叶稀疏、普通和早花型聚为一类。枝叶稀疏和普通型之间的遗传距离最近2.83, 其次是皱叶紧密型和大叶稠密型, 遗传距离为3.16。

|
图 3 乐昌含笑6个类型的ISSR聚类分析树状图 Fig. 3 Dendrogram of ISSR cluster analysis for 6 types of M.tsoi |
乐昌含笑选育的6种类型在叶形、株型和花期等性状方面出现了明显的变异。如:细叶稠密型的树冠圆锥状, 分枝角度中等; 叶倒卵状椭圆形; 叶排列稠密; 顶芽、嫩枝、侧芽具细短毛。皱叶紧密型的树冠呈卵形, 分枝角度小; 叶倒卵形, 叶面波皱明显。大叶稠密型的树冠呈卵圆形, 分枝角度中等; 叶片长圆状倒卵形, 先端尖头钝, 叶面波皱明显。枝叶稀疏型的树冠松散, 分枝角度大; 树皮灰白色; 叶面平展。普通型的树冠呈卵圆形, 分枝角度中等; 树皮褐色; 叶片长圆状倒卵形, 叶缘微皱。早花型的树冠紧密; 嫩枝、芽、叶柄具褐色短毛; 叶倒卵形; 始花期早。经过ISSR分析, 这6种类型在DNA水平也出现了一定程度的遗传分化, 因此可以作为新类型应用于园林绿化树种。
3.2 ISSR分析技术用于类型或品种鉴定的可行性本实验的研究结果表明, ISSR技术在对乐昌含笑6种类型鉴定的研究中, 具备分辨率高和多态性强的特点, 多态性条带百分率达50%。从30个引物中筛选出的引物ISSR2、ISSR16和ISSR19在乐昌含笑的5种类型中产生了特有条带。对于不同类型的ISSR扩增的特有条带, 还可进行切胶、回收, 以及克隆和测序, 通过序列和软件分析, 设计出一对互补到ISSR片段两端的引物, 将其转化为品种和类型的特异引物。由于不同引物的扩增带型差异较大, 因此筛选引物非常有必要。本实验筛选的能扩增特有条带的引物在生产实践中具有较高的应用价值, 可为以后鉴定和检测乐昌含笑类型或品种的真实性、品种的苗期鉴定提供客观、准确的技术保证。
ISSR技术的操作过程简单, 快捷。但有3点值得注意:一要保证模板DNA的质量。二对于不同的引物的退火温度应根据引物的Tm值要略有变动。三是ISSR扩增的带型背景较强, 应在PCR反应体系中增加一些化学物质, 使背景颜色减弱, 条带清晰。ISSR引物的长度一般都在15~24 bp, 反应的退火温度52~55℃比RAPD的36~40℃高, 因此对PCR反应的敏感性低于RAPD; ISSR引物中包含有一定长度的重复序列, 与它结合的目标序列在DNA复制的过程中存在滑动和不均等交换现象, 使得它们在不同品种或个体间的重复次数存在较大差异, 更易于导致引物结合位点和两结合位点间的片段长度产生差异(Gilbert et al., 1999; 钱韦等, 2000)。因此ISSR在反应的稳定性和检测DNA遗传变异能力方面较RAPD分析技术更高。在ISSR反应体系中加入2%甲酰胺能够降低由于引物滑动而引起的背景模糊, 加入2%~4%DMSO能提高反应的特异性(Joshi et al., 2000)。
综上所述, ISSR分析技术将在DNA水平上, 为检测和鉴定品种苗木的真实性、登录新品种、保护育种产权提供科学和准确的技术保障。
贾继增. 1996. 分子标记种质资源鉴定和分子标记育种. 中国农业科学, 29(4): 1-10. |
栾雨时, 安利佳, 黄百渠, 等. 1999. 用SSR探针进行番茄品种的DNA指纹分析. 园艺学报, 26(1): 1-4. DOI:10.3321/j.issn:0513-353X.1999.01.001 |
钱韦, 葛颂, 洪德元. 2000. 采用RAPD和ISSR标记探讨中国疣粒野生稻的遗传多样性. 植物学报, 42(7): 741-750. DOI:10.3321/j.issn:1672-9072.2000.07.017 |
盛能荣, 刘昭息, 丁林. 1996. 乐昌含笑的分布与引种. 浙江林业科技, 16(3): 24-30. |
唐启义, 冯明光. 1997. 实用统计分析及其计算机处理平台. 北京: 中国农业出版社.
|
邹喻萍, 葛颂, 王晓东. 2001. 系统与进化植物学中的分子标记. 北京: 科学出版社.
|
Doyle J J, Doyle J L. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem Bull, 19: 11-15. |
Gilbert J E, Lewis R B, WilkinsonM J, et al. 1999. Developing an appropriate strategy to assess genetic variability in plant germplasm collections. Theor Appl Genet, 98: 1125-1131. DOI:10.1007/s001220051176 |
Hu J, Quiros C F. 1999. Identification of broccoli and cauliflower cultivars with RAPD markers. Plant Cell Reporter, 10: 505-511. |
Joshi S P, Gupta V S, Aggarwal R K, Ranjekar P K, Brar D S. 2000. Genetic diversity and phylogenetic relationship as revealed by inter simple sequence repeat (ISSR)polymorphism in the genus Oryza. Theor Appl Genet, 100: 1311-1320. DOI:10.1007/s001220051440 |
Prevost A, Wilkinson M J. 1999. A new system of comparing PCR primers applied to ISSR fingerprinting of potato cultivars. Theor Appl Genet, 98: 107-112. DOI:10.1007/s001220051046 |
Zietkiewicz E, Rafalski A, Labuda D. 1994. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification. Genomics, 20: 176-183. DOI:10.1006/geno.1994.1151 |
 2002, Vol. 38
2002, Vol. 38