文章信息
- 曾东方, 罗信昌.
- Zeng Dongfang, Luo Xinchang.
- 应用RAPD分析快速鉴定外生菌根蘑菇分离物的真伪
- IDENTIFICATION OF ISOLATES FOR ECTOMYCORRHIZAL MUSHROOMS BY DNA FINGERPRINTING COMPARISON
- 林业科学, 2001, 37(6): 59-65.
- Scientia Silvae Sinicae, 2001, 37(6): 59-65.
-
文章历史
- 收稿日期:2000-09-25
-
作者相关文章
在外生菌根菌的分类及其菌丝体分离物的鉴定方面, 人们主要根据子实体的形态解剖特征确定菌种(黄亦存等, 1992)。有一些菌根菌不产生子实体或尚未找到子实体时, 就无法进行菌种的鉴定。即使是从外生菌根蘑菇子实体上分离到纯培养, 也有可能是由杂菌污染造成的。因此遵循柯赫法则(Koch' s Postulate)进行回接试验是比较理想的鉴定方法。一些外生菌根菌的纯培养目前难以获得出菇试验的鉴定, 寻求DNA亲菌鉴定的报道归纳为三种方法。其一是使用专一性探针, 进行分子杂交, 该技术较早地用于蜡蘑(Laccaria sp.)的鉴定(Gardes等, 1991), 后来也被用于乳牛肝菌(Suillus sp.) (Bruns等, 1993)、毒蝇鹅膏(Amanita muscaria)等(Kreuzinger等, 1996)的鉴定。其二是利用rDNA ITS区段的RFLP鉴定外生菌根菌的种类, 例如Farmer等(1998)对67种外生菌根菌的鉴定。其三是依据外生菌根菌某个特定基因的序列分析顺应, 目前已有80属层菌纲的担子菌的线粒体大亚基rDNA部分序列得以测定并公开发表(Bruns等, 1998)。RAPD(Random Amplified Polymorphic DNA)指纹技术相对上述分子杂交与基因序列分析方法而言更为方便快速, 我们用腐生食用真菌香菇、平菇作试验, 使用子实体RAPD指纹标准鉴定分离菌丝体, 结果与人工栽培出菇试验鉴定一致, 为食用菌尤其是共生食用菌分离菌株的真伪鉴定提供了遗传依据(曾东方, 2000)。前人对真菌进行RAPD分析, 一般是在供试真菌分类地位明确的情况下, 采用菌丝体作为试验材料, 研究各真菌菌株的DNA多态性。但使用子实体RAPD指纹鉴定共生真菌组织分离菌丝体的真伪尚未见报道。本文报道使用RAPD指纹技术快速鉴定外生菌根蘑菇分离物的结果, 具有重要的理论价值与实践意义。
1 材料和方法 1.1 外生菌根蘑菇的采集与分离在针阔混交林地里, 采集刚出土的新鲜外生菌根蘑菇的子实体, 即时带回室内当日分离。分离用培养基包括如下5种:马铃薯葡萄糖琼脂培养基(PDA) (方中达, 1979); 马铃薯葡萄糖酵母膏培养基(PDAY):在PDA里添加0.5 %酵母膏; 马铃薯葡萄糖土壤滤液培养基(PDAS):在PDA里添加松林土壤煮沸滤液20 %; 马铃薯葡萄糖麦麸滤液培养基(PDAW):在PDA里添加麦麸煮沸滤汁20 %; 马丁氏培养基(方中达, 1979)。试验子实体及分离菌株编号见表 1。
|
|
称取各供试子实体各100 mg, 刮取分离菌株斜面培养基上的菌丝体各1支试管, 分别用液氮研磨并装入1.5 mL EP管中。加入提取缓冲液(Tris-HCl, pH8.0, 50mmol·L-1, EDTA, pH8.0, 50mmol·L-1; NaCl, 1.4mol·L-1; CTAB, 3 %; α-巯基乙醇, 3 %; PVP, 3 %) 750 μL, 充分混匀, 置60 ℃水浴20 min。加入约700 μL的氯仿异戊醇(24 1), 轻摇30 sec。12000r·min-1室温离心5 min。收集上清液, 再用氯仿异戊醇萃取1 ~2次。接着在收集的上清液中加入2倍以上的无水乙醇, 静置于-20 ℃冰箱中30 min以上。然后3000 r·min-1离心3min。倾去上清液, 加入适量冰冷的80 %乙醇淌洗, 在卫生纸上沥干。最后在每支EP管中加入TER缓冲液(Tris-HCl, pH8.0, 10 mmol·L-1; EDTA, pH8.0, 1mmol·L-1; RNase, 10 μg·mL-1)约50 μL, 小心溶解, 再置60 ℃水浴20 min。即为各样品的总DNA溶液。取DNA溶液5 μL进行0.8 %琼脂糖水平电泳, 检测总DNA的质量, DNA分子完好的样品即可贮备于4 ℃冰箱中。近期不用的总DNA溶液, 放在-20 ℃冰箱保存。用时按Sambrook et al. (1989)介绍的方法进行定量, 然后稀释至所需要的浓度。
1.3 RAPD-PCR分析(1) 25 μL RAPD-PCR反应组分:
PCR buffer 1×
MgCl2 2mmol·L-1
dNTPs 100μmol·L-1
Primer 0.4μmol·L-1
Taq DNA polymerase 1u
Template DNA 10ng(对照反应不加)
P.H2O(超纯水) 定容至25μL
Mineral oil 覆盖30μL
(2) PCR循环参数:
在Thinker Series Ⅱ型PCR基因体外扩增仪上, 本试验采用如下PCR循环参数:

|
(3) 随机引物:
RAPD-PCR随机引物编号及其序列顺序(5′→3′)是: S61 : TTCGAGCCAG, S62 : GTGAGGCGTC, S64 : CCGCATCTAC, S69 : CTCACCGTCC, S72 : TGTCATCCCC, S74 : TGCGTGCTTG, S80 : ACTTCGCCAC, S121 : ACGGATCCTG, S129 : CCAAGCTTCC, S132 : ACGGTACCAG。
1.4 DNA指纹的数字转化与统计分析把分子量不同的DNA条带作为多态性状, 分子量相同的条带作为一个相同性状, 有性状记为1, 无性状记为0, DNA指纹图谱即转化为数字矩阵。用NTSYS-PC软件中的SIMQUAL程序计算DNA指纹间的相似系数, 按UPGMA法构建树状图。
2 结果与分析 2.1 外生菌根蘑菇组织分离菌丝体 2.1.1 点柄乳牛肝菌Suillus granulatus(Fr.) Kuntze以菌盖、菌柄内组织, 在PDA、PDAY、PDAS、马丁氏4种培养基上培养15d, 结果发现20块菌柄组织在4种培养基上无一萌发生长; 5支PDA试管里的菌盖分离物全部萌动生长出相同的白色菌丝菌落, 且扩展较慢, 见图 1。菌盖分离物在PDAS上也见萌动, 菌落与PDA上相同, 但长势很差。把PDA上的菌落扩大繁殖, 仍然是在PDA上生长较好, 而在PDAS上长势差, 扩展慢。其它菌盖组织在PDAY、马丁氏培养基上, 全部未生长萌发成菌丝, 也未见杂菌污染。结果表明:点柄乳牛肝菌的菌丝体分离, 以菌盖组织在PDA培养基上容易分离到纯培养, 在其它供试培养基上难以生长; 而菌柄则不适宜作分离部位。
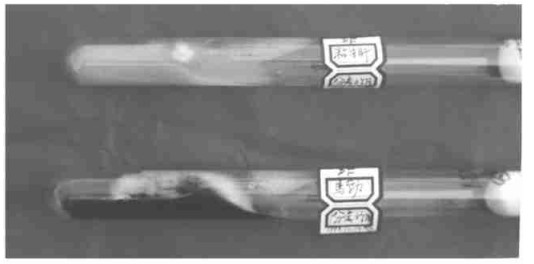
|
图 1 乳牛肝菌(上)和马勃(下)分离菌株 Fig. 1 Isolates from Suillus (upper) and Scleroderma (below) mushrooms |
以黑色担孢子粉在PDAS、PDAW培养基上培养30 d, 10支斜面试管全部长出一致的菌丝体菌落, 即菌丝白色, 生长缓慢并分泌色素, 使培养基变成黑褐色(见图 2)。

|
图 2 与红菇相关的分离菌株 Fig. 2 Isolates related with Russula mushroom |
委托鉴定单位提供的12个大红菇子实体标本与8支试管斜面培养物(分离培养过程不详), 列见表 1与图 2。其中HC1~10据称是分离自大红菇子实体, 但不是表 1列出的供试子实体, HA1据称是分离自大红菇相关菌根。
2.2 牛肝菌与分离菌株之间RAPD指纹比较分析把各供试引物制备的RAPD指纹图谱(如图 3是其中一个引物S62之试验结果)转换成分子数据, 进行电脑统计运算, 得到牛肝菌及其分离菌株间的DNA相似系数矩阵, 进一步绘制成牛肝菌与分离菌株之间的类缘关系树状图, 见图 4。
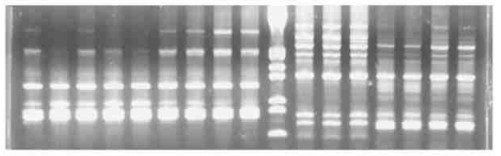
|
图 3 随机引物S62介导的牛肝菌、马勃子实体与分离菌株的RAPD谱 Fig. 3 RAPD patterns of Suillus and Scleroderma mushrooms and isolates by primer S62 从左至右(From left to right): NB1、NB2、NB3、NB4、NB5、NM1、NM2、NM3、NM4、λDNA/E+H、MB1、MB2、MB3、MM1、MM2、MM 3、MM4。 |

|
图 4 牛肝菌与其分离菌株的类缘关系 Fig. 4 Relationships between Suillus mushrooms and isolates |
由图 4可知, 牛肝菌子实体NB1、NB3、NB4与对应的分离菌株NM1、NM2、NM3、NM4之DNA指纹完全相同, 在试验所有引物RAPD-PCR分析中无不同的结果, 表明试验通过组织分离法制得的菌丝体培养物确系牛肝菌的分离菌株。供试牛肝菌NB2、NB5指纹相同, 但与NB1、NB3、NB4类的相似系数为0.939, 说明它们分属2类居群的子实体。
2.3 马勃与分离菌株之间RAPD指纹比较分析RAPD指纹图谱如图 3右部所示其一引物之试验结果, 转换成分子数据, 绘制马勃与分离菌株之间的类缘关系树状图见图 5。

|
图 5 马勃与其分离菌株类缘关系 Fig. 5 Relationships between Scleroderma mushrooms and isolates |
由图 5可知, 马勃子实体MB1、MB2、MB3的DNA指纹相同, 因而在树状图中聚为一类; 分离菌株MM2、MM3、MM4的DNA指纹相同, 在图中聚为一类, 然后与分离菌株MM1在0.979相似水平聚为一类; 最后3个子实体与4个分离菌株在0.755相似水平上聚在一起。
在RAPD-PCR指纹图谱中, S69、S64、S62介导的子实体与分离菌株之DNA指纹主带相同, 分离菌株均有一条小分子带未扩增, 见图 3所示照片; S80、S121、S72介导的子实体与分离菌株之DNA指纹则完全相同。仅见S129介导的子实体与分离株之DNA指纹相差较大。但总的数据统计分析结果表明, 子实体与分离株之DNA相似水平为0.755, 根据Kurtman(1987)相似系数在0.7以上的菌株同种的观点, 可以判定本试验所获得的4株分离菌株是马勃的分离物。
2.4 福建供试红菇分离物的DNA鉴定结果试验获得的RAPD图谱(如其中之一随机引物S129试验结果见图 6)显示, 供试红菇子实体与分离物之间的DNA指纹相差很大, 共有DNA条带很少, 说明二者DNA相似性很低, 判断供试菌株与子实体之间存在极大的DNA异质性, 因而认为供试培养物并非供试红菇的纯培养物或分离菌株。

|
图 6 随机引物S129介导的供试红菇子实体与待鉴定分离物的RAPD谱 Fig. 6 RAPD pattern of Russula sp. mushroom and isolates tested by primer S129 从左至右(From left to right): λDNA/E+H、HB3、HB4、HB5、HB6、HB7、HB8、HB9、HB10、HB11、HB12、HC1、HC4、HC5、HC6、HC8、HC9、HC10、HA1。 |
RAPD是以PCR(Polymerase Chain Reaction)技术为基础的随机扩增多态性DNA(Random Amplifed Polymorphic DNAs)。在RAPD-PCR反应中, 不同物种的基因组内与引物相匹配的碱基序列空间位置和数目都可能不同, 所以扩增产物的大小和数量也可能不同, 获得的DNA指纹图谱充分展现出基因组DNA的多态性(Williams et al., 1990)。大量研究证明, RAPD具有个体、群体、亚种、种等各层次水平的特异性, 在动物、植物、人和微生物的遗传多样性检测、基因定位、品系(克隆)鉴定、法医鉴定、系统演化、遗传图谱的构建等方面广泛应用。
真菌RAPD研究报导广泛见于群体分型, 如大丽轮枝孢菌(Verticillium dahliae)的不同致病菌系的RAPD(Ramsay et al., 1996)。RAPD也可揭示真菌属种的遗传变异, 如我们实验室利用RAPD对木耳属(Auricularia)内不同木耳种的分类(阎培生等, 2000)。对于真菌个体的不同组织, 人们可以根据遗传学原理判断它们的DNA应该是均一性的(homogeneity)。我们对食用蘑菇香菇、平菇的子实体进行组织分离, 获得的菌丝体进行人工栽培产生了香菇、平菇新的子实体, 再组织分离获得新的菌丝体, 根据柯赫法则, 这些菌丝体就是香菇、平菇的真正分离物。随后进行RAPD分析, 12个引物制备的DNA指纹图谱重复性很好地揭示, 不同品种的香菇的DNA相似系数在0.886~0.986之间, 不同品种的平菇的DNA相似系数在0.779~0.976之间, 但不论香菇还是平菇, 子实体及其组织分离菌丝体则毫无例外地出现了完全相同的DNA指纹图谱(即相似系数为1.000), 从分子水平上证明供试香菇、平菇的子实体组织与自身分离菌丝体为一个无性克隆系的两种不同表达形态, 这就为以子实体RAPD指纹图谱鉴定分离菌丝体的真伪提供了理论依据1)。许多珍贵的共生食用蘑菇如松口蘑、美味牛肝菌等的菌丝体分离, 就腐生食用菌香菇、平菇而言是比较困难的, 尤其是目前这些纯培养菌种还不能由人工栽培出菇, 对分离菌株寻求DNA亲菌鉴定势在必然。在本研究中, 供试牛肝菌的分离菌丝体与其来源子实体的DNA指纹相同, 这与我们先前用香菇、平菇的RAPD分析结果类似, 因而鉴定它确为牛肝菌分离菌株。供试马勃分离菌丝体与其来源子实体的DNA指纹图谱有3个引物完全相同, 有4个引物出现多态性DNA, 但总的数学统计得出的DNA相似系数在75 %以上, 把它判定为马勃分离菌株符合真菌分子系统学原理(Kurtzman, 1987; 曾东方, 1999)。我们认为:由于马勃分离菌丝体来自减数分裂产生的担孢子(单核)的萌发, 其基因拷贝数较少, PCR扩增的带较少, 故而与子实体之RAPD指纹相似系数低于本试验中牛肝菌的值。另一个供试真菌红菇的斜面培养菌丝体与供试子实体的DNA指纹图谱差异极大(照片备索), DNA相似系数极低, 因而判断该分离菌株与红菇子实体亲缘关系很远。
1) 曾东方, 腐生与共生食用菌菌丝体分离、培养及其DNA鉴定研究.博士学位论文.武汉:华中农业大学, 2000, 1~118
3.2 RAPD用于共生食用菌物种鉴定的技术范围RAPD技术对DNA样品需要量少、不需探针杂交与放射性自显影, 比RFLP(Restriction Fragment Length Polymorphism)方便快捷, 但RAPD是显性标记, 不能区分纯合和杂合型, RFLP是共显性标记, 可区分纯合与杂合交型。RAPD是基于基因组DNA序列顺序的多态性, 因而被称作为简易的序列分析, 这比全序列顺序的测定要经济得多。
正如前述, RAPD用于物种鉴定不存在理论上的问题, 但技术操作上还是有要求的。尽管RAPD分析用的DNA需要量少, 制备容易, 但供试DNA样品不能被外源DNA污染, 否则影响鉴定结果。在本研究中, 子实体是鉴定的标准, 其DNA制备必须完整、均一。待鉴定的分离菌株必须按微生物学技术给予纯化, 如果供试样品混有杂菌或其它生物DNA, 即使供试样品中存在目标菌株, RAPD鉴定可能把它与杂菌判作另一种生物。
外生菌根菌在植树造林中具有重要作用, 人们对菌根进行分离, 从中培养出菌根菌, 就可制备接种制剂。对外生菌根进行分类鉴定是一个实践上含有挑战性的课题, 有一些菌根菌一时难以找到它的子实体, 就无法进行形态学上的分类与鉴定(黄亦存等, 1990)。如同本研究试验一样, 如果制备了各种外生菌根菌的RAPD指纹图谱库, 是可以对分离物迅速进行鉴定的。但是, 如果菌根难以分离, 而且菌根中存在多种菌根菌, 需要设计特定外生菌根菌的的专一性探针, 就可以直接取菌根合体制备DNA样品, 采用分子杂交技术即可鉴定菌根菌的种类及组成(Bruns et al., 1993), 进而判断菌根的类型。
方中达. 1979. 植病研究方法. 北京: 农业出版社, 83, 123.
|
黄亦存, 沈崇尧, 裘维蕃. 1992. 外生菌根的形态学、解剖学及分类学研究进展. 真菌学报, 11(3): 169-181. |
阎培生, 罗信昌, 周启. 2000. 利用RAPD技术对木耳属菌株进行分类鉴定的研究. 菌物系统, 19(1): 29-33. DOI:10.3969/j.issn.1672-6472.2000.01.007 |
曾东方. 1999. 真菌分子系统学的产生和发展. 中国食用菌, 18(2): 3-4. |
Bruns T D, GardesM. 1993. Molecular tools for the identification of ectomycorrhizal fungi-taxon-specific digonucleotide probes for suilloid fungi. Molecular Ecology, 2: 233-242. DOI:10.1111/j.1365-294X.1993.tb00013.x |
Bruns T D, Szaro T M, Gardes M, et al. 1998. A sequence database for the identification of ectomycorrhizal bsidiomycetes by phylogenetic analysis. Molecular Ecology, 7: 257-272. DOI:10.1046/j.1365-294X.1998.00337.x |
Farmer D J, Sylvia DM. 1998. Variation in the ribosomal DNA internal transcribed spacer of a diverse collection of ectomycorrhizal fungi. Mycological Research, 102: 859-865. DOI:10.1017/S0953756297005601 |
Gardes M, White T J, Fortin J, et al. 1991. Identification of indigenous and introduced symbiotic fungi in ectomycorrhizae by amplification of nuclear and mitochondrial ribosomal DNA. Canadian Journal of Botany, 69: 180-190. DOI:10.1139/b91-026 |
Kreuzinger N, Podeu R, Gruber F, et al. 1996. Identification of some ectomycorrhizal basidiomycetes by PCR amplification of their gpd genes. Applied and Environmental Microbiology, 62: 3432-3438. |
Kurtzman C P. 1987. Impact of nucleic acid comparisons on the classification of fungi. Proceedings of the Indian Academy of Science, 97: 185-201. |
Ramsay J R, Multani D S, Lyon B R. 1996. RAPD-PCR identification of Verticillium dahliae isolateswith differential pathogenicity on cotton. Australia Journal of Agriculture Research, 47(5): 681-693. DOI:10.1071/AR9960681 |
Williams J G K, Kubelic A R, Livak K J, et al. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nuc.Acid.Res., 18: 6531-6533. DOI:10.1093/nar/18.22.6531 |
 2001, Vol. 37
2001, Vol. 37

