文章信息
- 夏铭, 周晓峰, 赵士洞.
- Xia Ming, Zhou Xiaofeng, Zhao Shidong.
- 天然蒙古栎群体遗传多样性的RAPD分析
- RAPD ANALYSIS ON GENETIC DIVERSITY OF NATURAL POPULATIONS OF QUERCUS MONGOLICA
- 林业科学, 2001, 37(5): 126-133.
- Scientia Silvae Sinicae, 2001, 37(5): 126-133.
-
文章历史
- 收稿日期:2000-03-09
-
作者相关文章
2. 东北林业大学森林资源与环境学院 哈尔滨 150040;
3. 中国科学院地理科学与资源研究所 北京 100101
2. College of Forest Resource and Environment, Northeast Forestry University Harbin 150040;
3. Institute of Geographical Science and Resources, Chinese Academy of Sciences Beijing 100101
蒙古栎(Quercus mongolica Fisch)为壳斗科、栎属植物, 主要分布区包括中国的华北、东北、内蒙古东部, 朝鲜半岛, 俄罗斯远东和蒙古的连续分布区及日本桦太、北海道的间断分布区(吴晓春, 1993)。蒙古栎是我国东北的典型植被类型———红松阔叶林的主要伴生树种之一, 同时在红松林被干扰破坏后是形成次生阔叶林面积较大的主要树种, 也是分布区超过北纬45°的唯一栎属植物。近年来, 蒙古栎的分布区和群体数量呈扩张趋势, 其原因除人为干扰外, 可能涉及气候变化、物种特性、种内遗传多样性、种间互作等等。对蒙古栎群体进行遗传多样性的研究可以从遗传的角度为其群体的变化提供直接的依据。
RAPD技术作为一种简便、快速、易行的分子标记技术, 近年来被广泛地应用于木本植物遗传多样性和遗传结构的研究中, 已经对辽东栎(Quercus liaotungensis)(恽锐等, 1998), 矮牡丹(Paeonia suffruticosa)和紫斑牡丹(P.rockii)(裴颜龙等, 1995), 蔷薇属(Rosa)(Millan et al., 1996)大青杨(Populus ussuriensis)(苏晓华等, 1997), 茶树(Camellia)(Wachira et al., 1995), 墨西哥丁香(Gliricidia sepium)(Chalmers et al., 1992), 欧洲山杨(Populus tremuloides)(Yeh et al., 1995), 青杨(Populus cathayana Rehd)(李宽钰等, 1997), 椴树(Morone saxatilis)(Bielawski et al., 1997), 柠条(Caragana spp.)(魏伟等, 1999)等树种进行了报道。
本研究用RAPD技术对分布于中国东北境内的4个典型蒙古栎天然群体的遗传多样性、群体内和群体间的遗传变异进行了研究, 为合理解释、预测蒙古栎群体变化趋势, 合理开发、利用植物资源提供科学依据。
1 材料和方法 1.1 实验材料所有实验样品采自中国境内的典型分布区, 分别为黑龙江省帽儿山实验林场、龙江山泉林场、黑河平山林场和虎林五泡林场的天然林。采样时分单株随机取带有芽苞的枝条或带有萌动幼芽的枝条, 所有样品采自成年树, 每两个个体的间距大于500 m, 每个群体样本数为20个。采集的样品装于塑料袋中, 放一张湿滤纸以保持湿度, 于0 ℃~4 ℃条件下带回实验室。对未萌动的芽在温室中进行水培, 待发芽后采集新长出的嫩芽, 保存于液氮中, 对已萌动的芽, 直接保存于液氮中, 用于DNA提取。
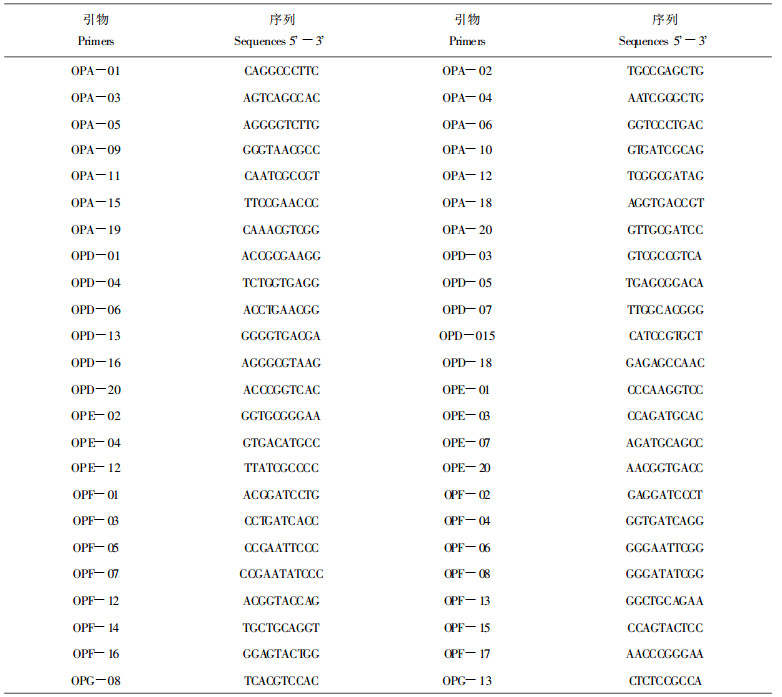
1.2 实验方法〈1〉DNA的提取 4个群体各20份样品, 采用十六烷基三甲基溴化胺(CTAB)方法, (Doyle et al., 1990), 稍做修改后提取基因组DNA, 通过测定紫外光吸收确定DNA浓度和纯度, 通过琼脂糖电泳检查DNA的完整性, -20 ℃保存备用。〈2〉引物的筛选 随机引物采用Operon公司的引物系列A、D、E、F、G, 经预备试验选择扩增产物稳定、重复性好的引物对所有个体的基因组DNA进行扩增, 共用引物48个, 序列见表 1。〈3〉RAPD扩增及产物分离 参照Williams等的方法(Williams et al., 1990), 经条件优化后确定反应体系如下:总体积20 μL, 包括40 ng基因组DNA, 15 ng随机引物, 200 μmol·L-1 dATP、dCTP、dGTP、dTTP, 1 unit TaqDNA聚合酶, 1.5 mmol·L-1 MgCl2。反应程序为:95 ℃预变性5 min, 40次热循环, 条件为94 ℃、1 min, 36 ℃、1 min, 72 ℃、1 min, 最后在72 ℃下延伸7 min, 所有反应在美国M.J.公司生产的PTC-200热循环仪中进行。扩增产物在1.2%的琼脂糖凝胶中电泳分离, 溴化乙锭染色, 透射紫外灯下观察并拍照。
|
|
〈1〉多态位点比率 在某一特定位点上, 若扩增片段出现的频率小于0.99, 则此位点称为多态位点。多态位点比率就是在所有检测到的位点中多态位点所占的比例。〈2〉Shannon表型多样性指数 根据King和Schaal的方法(King et al., 1989), 利用Shannon表型多样性指数来计算群体内和群体间的遗传多样性, 计算公式为:

|
式中Pi为表型频率, 即某一扩增带出现的频率。H可以有两种水平的遗传多样性, Hpop为群体内的遗传多样性, Hsp为种内总的遗传多样性。〈3〉RAPD标记共享度和遗传距离 根据Nei和Li的方法(Nei et al., 1979), 计算RAPD片段共享度(F)和遗传距离(D)公式为:
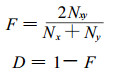
|
式中, Nxy为x、y两个个体或两个群体共同拥有的RAPD标记数; Nx、Ny和x、y两个个体或两个群体各自拥有的RAPD标记数。
2 结果与分析用48个随机引物对4个天然蒙古栎群体共80个个体的基因组DNA进行了RAPD分析, 每个引物检测到的位点数在2~12之间, 扩增DNA片段长度介于300~2500之间。48个引物共检测到344个可重复的位点, 平均每个引物检测到的位点数为7.17。
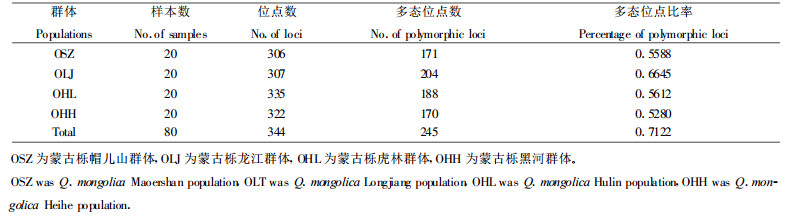
2.1 多态位点比率4个蒙古栎群体的总位点数、多态位点数及多态位点比率见表 2。
|
|
在帽儿山群体中, 48个引物检测到306个位点, 其中多态位点171个, 占55.88%;龙江群体中总位点数307个, 多态位点204个, 多态位点比率达到66.45%;虎林群体中共检出335个位点, 多态位点188个, 占全部位点的56.12%;黑河群体中总位点数322个, 多态位点170个, 多态位点比率为52.80%。在总计80个蒙古栎个体中, 检测到的总位点数为344个, 多态位点245个, 种内多态位点比率为71.22%。可以看到, 龙江群体的多态位点比率明显高于其他3个群体, 黑河群体多态位点比率最低, 虎林群体与帽儿山群体基本相同, 仅相差0.24%。
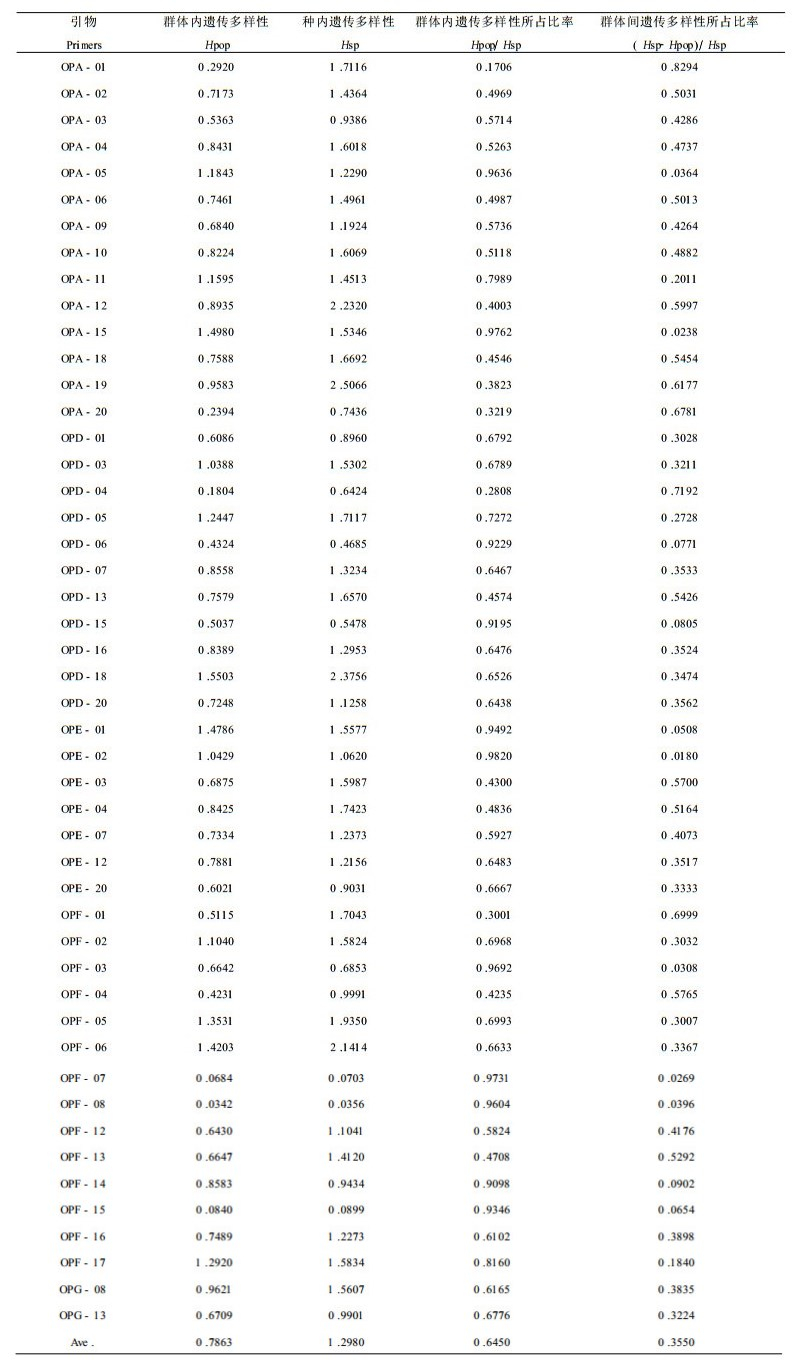
2.2 用Shannon表型多样性指数计算蒙古栎遗传多样性用Shannon指数计算了4个天然蒙古栎群体的群体内、群体间的遗传多样性, 以及各自在总变异中所占的比例, 见表 3、表 4。由表 3可见, 不同引物检测到的群体内遗传多样性有较大差异, 变化范围从0~2.2275。4个蒙古栎群体中, 龙江群体内的平均遗传多样性水平最高(0.9611), 帽儿山群体和虎林群体比较接近, 平均遗传多样性分别为0.7946和0.7711, 黑河群体的遗传多样性最低(0.6185)。可以看出, 蒙古栎不同群体间的遗传多样性存在较大差异。
|
|
|
|
从表 4可以看到, 蒙古栎群体内平均遗传多样性为0.7863, 种内平均遗传多样性为1.2980。在种内总遗传变异中, 大部分存在于群体内(64.50%), 群体间遗传变异占总变异的35.50%, 即蒙古栎群体间的遗传分化达到35.50%。
2.3 蒙古栎群体内、群体间的遗传相似度和遗传距离通过计算群体内个体间和群体间的RAPD片段共享度, 可以得到群体内和群体间的遗传相似度和遗传距离, 结果见表 5、表 6。
|
|
|
|
由表 5、表 6可见, 蒙古栎群体间遗传相似度以帽儿山与龙江两群体间最大(0.8981), 虎林与黑河群体间最小(0.8570), 其他从大到小依次为龙江与虎林群体(0.8833)、龙江与黑河群体(0.8703)、帽儿山与黑河群体(0.8587)、帽儿山与虎林群体(0.8585)。群体内遗传相似度以黑河群体最高(0.9164), 龙江群体最低(0.8984)。相反地, 群体间的遗传距离则以虎林与黑河群体间最大(0.1430), 帽儿山与龙江群体间最小(0.1019), 群体间平均遗传距离为0.1290。群体内遗传距离最大的是龙江群体(0.1016), 最小的是黑河群体(0.0836), 群体内平均遗传距离为0.0962。
2.4 蒙古栎群体的特征谱带在对蒙古栎的RAPD电泳图谱进行统计时, 发现了只在某一特定群体中出现, 而在其他群体中无扩增的特征谱带。蒙古栎虎林群体的特征谱带有两条, 分别为OPA-191100(图 2)、OPF-05500(图 3)。黑河群体的特征谱带有一条, 为OPA-20600(图 4)。这些条带在特定群体中百分之百出现, 而在其他群体中检测不到, 因而可以作为各自群体的特异RAPD标记。
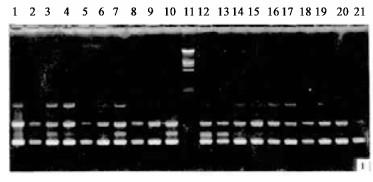
|
图 1 OPA-01扩增蒙古栎虎林群体的RAPD电泳图谱 Fig. 1 RAPD electrophoretic profiles of OHL amplified with primer OPA-01 泳道11为分子量标准(21227, 5149, 4973, 4268, 3530, 2027, 1904, 1581, 1375, 947, 831 bp) Line 11 was molecular weight standards. |

|
图 2 OPA-19扩增蒙古栎虎林群体的RAPD电泳图谱 Fig. 2 RAPD electrophoretic profiles of OHL amplified with primer OPA-19 箭头所示的OPA-191100为该种群的特异谱带 The arrow showed that OPA-191100 was special band of OHL. |
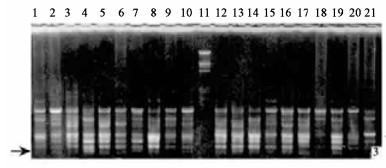
|
图 3 OPF-05扩增蒙古栎虎林群体的RAPD电泳图谱 Fig. 3 RAPD electrophoretic profiles of OHL amplified with primer OPF-05 箭头所示的OPF-05500为该种群的特异谱带 The arrow showed that OPF-05500 was special band of OHL. |

|
图 4 OPA-20扩增蒙古栎黑河群体的RAPD电泳图谱 Fig. 4 RAPD electrophoretic profiles of OHH amplified with primer OPA-20 箭头所示的OPA-20600为该种群的特异谱带 The arrow showed that OPA-20600 was special band of OHH. |
本研究利用48个引物对4个蒙古栎天然群体进行RAPD研究, 结果表明, 各群体内的多态位点比率介于52.80%~66.45%之间, 种内多态位点比率为71.22%(表 2)。在对辽东栎两个群体的DNA多态分析中, 12个引物共检测到205个位点, 多态位点占79%(恽锐等, 1998)。对矮牡丹和紫斑牡丹的RAPD研究中, 多态位点比率分别为22.5%和27.6%(裴颜龙等, 1995), 这两种牡丹均为我国3级保护植物, 其资源稀缺的原因之一可能就是较低水平的遗传多样性。对四种蔷薇属植物的RAPD分析中(Millan et al., 1996), 10个引物共获得75条扩增片段, 其中多态片段占了83%, 从以上的对比可以看到, 蒙古栎种内的DNA多态水平是较高的。
Shannon表型多样性指数的统计表明, 蒙古栎群体内遗传多样性介于0.6185~0.9611之间, 平均遗传多样性为0.7863, 种内总的遗传多样性为1.2980, 群体间遗传多样性占总遗传多样性的35.50%(表 3, 表 4)。在对其他多年生异交木本植物的RAPD研究中, Shannon指数估算的遗传多样性群体间遗传分化各有不同。大青杨7个群体遗传多样性为0.271~0.392, 平均为0.310(苏晓华等, 1997)。欧洲山杨8个天然群体内的平均遗传多样性为0.65, 群体间遗传多样性占总多样性的2.6%(Yeh et al., 1995)。茶树3个品种的群体内平均遗传多样性为1.71, 群体间遗传分化为30%(Wachira et al., 1995)。墨西哥丁香的平均群体内遗传多样性为1.194, 种内总变异为2.976, 群体间遗传分化达到59.9%(Chalmers et al., 1992)。同以上树种相比, 蒙古栎种内的遗传多样性较为丰富, 群体间的遗传分化处于中等水平。
从蒙古栎群体的多态位点比率和遗传多样性来看, 龙江群体内的遗传多样性水平最高, 明显高于其他群体, 黑河群体的遗传多样性最低, 帽儿山群体和虎林群体遗传多样性水平比较接近, 呈现出中间高, 南北低的趋势。从各群体的分布来看, 帽儿山位于蒙古栎分布的中心区, 龙江则接近蒙古栎分布区的西部边缘。一般地, 一个物种的边缘分布区中生境较为复杂, 环境压力也相对较大, 导致种内发生遗传变异的频率升高, 从而使边缘区群体内积累较多的遗传变异以适应复杂的环境。我们在蒙古栎中检测到的遗传多样性分布正说明了这一点。
从蒙古栎群体间的遗传距离来看, 最大遗传距离存在于黑河与虎林群体之间(0.1430), 最小遗传距离发生在龙江与帽儿山群体之间(0.1019)。地理距离最远的龙江与虎林群体间遗传距离为0.1167, 地理上最近的龙江与黑河群体间遗传距离为0.1297, 地理距离较近的帽儿山与虎林群体的遗传距离却达到0.1415。可见, 蒙古栎群体间的遗传距离与地理距离相关性不大。从纬度来看, 纬度相差较大的黑河与帽儿山群体、黑河与虎林群体的遗传距离分别达到0.1413和0.1430, 而纬度相差较小的黑河与龙江群体的遗传距离为0.1297, 似乎表明纬度的差异与遗传距离存在正相关; 但纬度差异最小的虎林与帽儿山群体的遗传距离却达到0.1415, 反映出蒙古栎群体间的遗传分化格局的复杂性, 这种复杂性可能与各群体的种源、进化过程以及群体间的基因交流等多种因素有关, 这一点有待进一步的研究。
根据对已有研究结果的统计(Hamrick et al., 1990), 长寿命异交木本植物中存在丰富的遗传变异, 其中大部分变异存在于群体内, 本研究得到的蒙古栎遗传变异及分布与以上结果是一致的。
近年来, 蒙古栎的分布区有较为明显的扩展, 这与蒙古栎种内较为丰富的遗传多样性和较强的生态适应性密切相关, 也同人类活动的干扰有较大关系。人类对林木的过度砍伐和生产活动导致的生境恶化, 使大量山地变为无林地, 随之而来的水土流失使林地的立地条件变得很差, 多数针、阔树种都难以生长, 只有蒙古栎凭借很强的适应能力在破坏后的迹地上逐步形成次生栎林。可以说, 生态环境的破坏客观上为蒙古栎群体的扩展提供了可能, 而蒙古栎较高水平的遗传多样性则是其群体的稳定存在和发展的根本保障。
李宽钰, 黄敏仁, 杨自湘, 等. 1997. 青杨的遗传分化. 植物学报, 39(8): 753-758. |
裴颜龙, 邹喻苹, 尹蓁, 等. 1995. 矮牡丹与紫斑牡丹RADP分析初报. 植物分类学报, 33(4): 350-356. |
苏晓华, 张绮纹, 郑先武, 等. 1997. 利用RAPD分析大青杨天然群体的遗传结构. 林业科学, 33(6): 504-511. DOI:10.3321/j.issn:1001-7488.1997.06.004 |
魏伟, 王洪新, 胡志昂, 等. 1999. 毛乌素沙地拧条群体分子生态学初步研究:RAPD证据. 生态学报, 19(1): 16-22. |
吴晓春.蒙古栎地理变异的研究(黑龙江省自然科学基金研究报告).哈尔滨, 1993
|
恽锐, 钟敏, 王洪新. 1998. 北京东灵山辽东栎群体DNA多样性的研究. 植物学报, 40(2): 169-175. DOI:10.3321/j.issn:1672-9072.1998.02.013 |
Bielawski J P, Pumo D E. 1997. Random amplified polymorphic DNA(RAPD)analysis of Atlantic Coast striped bass. Heredity, 78: 32-40. DOI:10.1038/hdy.1997.4 |
Chalmers K J, Waugh R, Sprent J I, et al. 1992. Detection of genetic variation between and within population of Gliricidia sepium and G. maculata using RAPD markers. Herdity, 69: 465-472. DOI:10.1038/hdy.1992.151 |
Doyle J J, Doyle J L. 1990. Isolation of plant DNA from fresh tissue. Focus, 12(1): 13-15. |
Hamrick J L and Godt M J W.Allozyme diversity in plant species.In A D H Brown, M T Clegg, A L Kahler and B S Weir (eds.)Plant Population Genetics, Breeeding, and Genetic Resources.Sunderland: Sinauer, 1990, 43~63
|
King L M, Schaal B A. 1989. Ribosomal-DNA variation and distribution in Rudbeckia missouriensis. Evolution, 43(5): 1117-1119. DOI:10.1111/j.1558-5646.1989.tb02557.x |
Millan T, Osuna F, Cobos S, et al. 1996. Using RAPDs to study phylogenetic relationships in Rosa. Theor.Appl.Genet., 92: 273-277. DOI:10.1007/BF00223385 |
Nei M, Li W H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc.Natl.Acad.Sci.USA, 76: 5269-5273. DOI:10.1073/pnas.76.10.5269 |
Wachira F N, Waugh R, Hackett C A, Powell W. 1995. Detection of genetic diversiyt in tea(Camellia sinensis)using RAPD markers. Genome, 38: 201-210. DOI:10.1139/g95-025 |
Williams J G K, Kubelik A R, Livak K J, et al. 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research, 18: 6531-6535. DOI:10.1093/nar/18.22.6531 |
Yeh F C, Chong D K X, Yang R C. 1995. RAPD variation within and among natural populations of trembling aspen(Populus tremuloides Michx.)from Alberta. The Jounal of Heredity, 86(6): 454-459. DOI:10.1093/oxfordjournals.jhered.a111620 |
 2001, Vol. 37
2001, Vol. 37