文章信息
- 高捍东, 黄宝龙.
- Gao Handong, Huang Baolong.
- 板栗主要栽培品种的分子鉴别
- GENETIC ANALYSIS AND IDENTIFICATION OF MAIN CULTIVATED VARITIES OF CHESTNUT (CASTANEA MOLLISSIMA) BY RAPD MOLECULAR MARKERS
- 林业科学, 2001, 37(1): 64-71.
- Scientia Silvae Sinicae, 2001, 37(1): 64-71.
-
文章历史
- 收稿日期:2000-05-12
-
作者相关文章
板栗是经济价值很高的经济树种,栽培历史悠久,资源丰富。传统的板栗品种划分方法主要依据其形态学特征、生物学特性和经济性状,其中又以经济性状为主,即以花序、总苞和果实的形态为主要依据,难以准确鉴别一些形态相近的品种。木本植物结实周期长,仅仅依据营养器官的形态特征难以在早期准确地鉴定品种,生产上因伪劣品种或品种混杂造成损失的现象时有发生,因此准确鉴别品种显得尤为重要和迫切,同时对新品种的审定、注册、保护和管理,建立准确、可靠的品种特异性检测方法,具有一定的理论意义和较高的实践价值。
与其它分子标记方法相比,此方法具有独到的优点。应用RAPD分析农作物品种纯度的研究十分活跃,被广泛应用于玉米、大豆、小麦、棉花、水稻和红三叶种子的品种鉴定(傅家瑞,1994;李文彬,1995;余四斌等,1996;陈洪等,1996)。
园艺学上也用RAPD标记进行品种鉴定,已报道的蔷薇属(Millan et al., 1996)、梅(Shimada et al., 1994;1996;刘青林,1999)、洋李(Gregor, 1994)、百合属(Wilkie et al., 1993)、柿(罗正荣等,1995)、茶叶(陈亮等,1998)等等。
RAPD研究也被广泛应用于美洲黑杨无性系鉴定(阿根廷Silvia Cortizo et al., 1996)、构建马尾松的分子标记连锁图(尹佟明,1997)、尾叶桉和细叶桉无性系研究(甘四明等,1999)、芒果品种鉴定(Adato et al., 1995;徐碧玉,1998)。
本研究应用RAPD分子标记手段,从DNA水平上研究板栗品种的遗传多样性,建立了板栗品种RAPD标记的标准程序,以及板栗品种的DNA指纹数据库,为板栗育种提供理论依据,并为板栗品种鉴别提供了准确、可靠的标准方法。
1 材料与方法 1.1 实验材料46个板栗品种的带叶幼枝采自江苏省溧阳市龙潭林场国家林业局板栗良种基地,冰冻保存,供提取DNA用。该基地地处31°10′25″~31°17′24″N;119°21′18″~ 119°32′06″E之间,已建立品种收集圃、良种采穗圃、良种繁育圃、丰产示范园、良种对比试验园。各品种名称及产地见图 2。
|
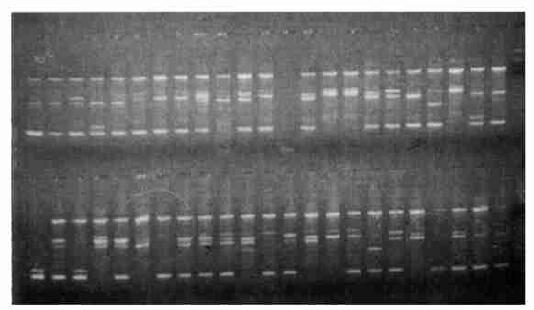
图 1 应用引物S28扩增的46个板栗品种RAPD谱带 Fig. 1 RAPD fingerprints of 46 chestnut varieties genomic DNA amplified with S28 primer 上下两行最右一条为标准分子量标记(100 bp DNA Ladder Plus); 其余从右上方自右向左依此为1~46号样品的谱带。 The rightest lane in up and low lines is the standard molecular marker (100 bp DNA Ladder Plus). The fingerprints refer to varieties 1 to 46 from up right to left. |
|
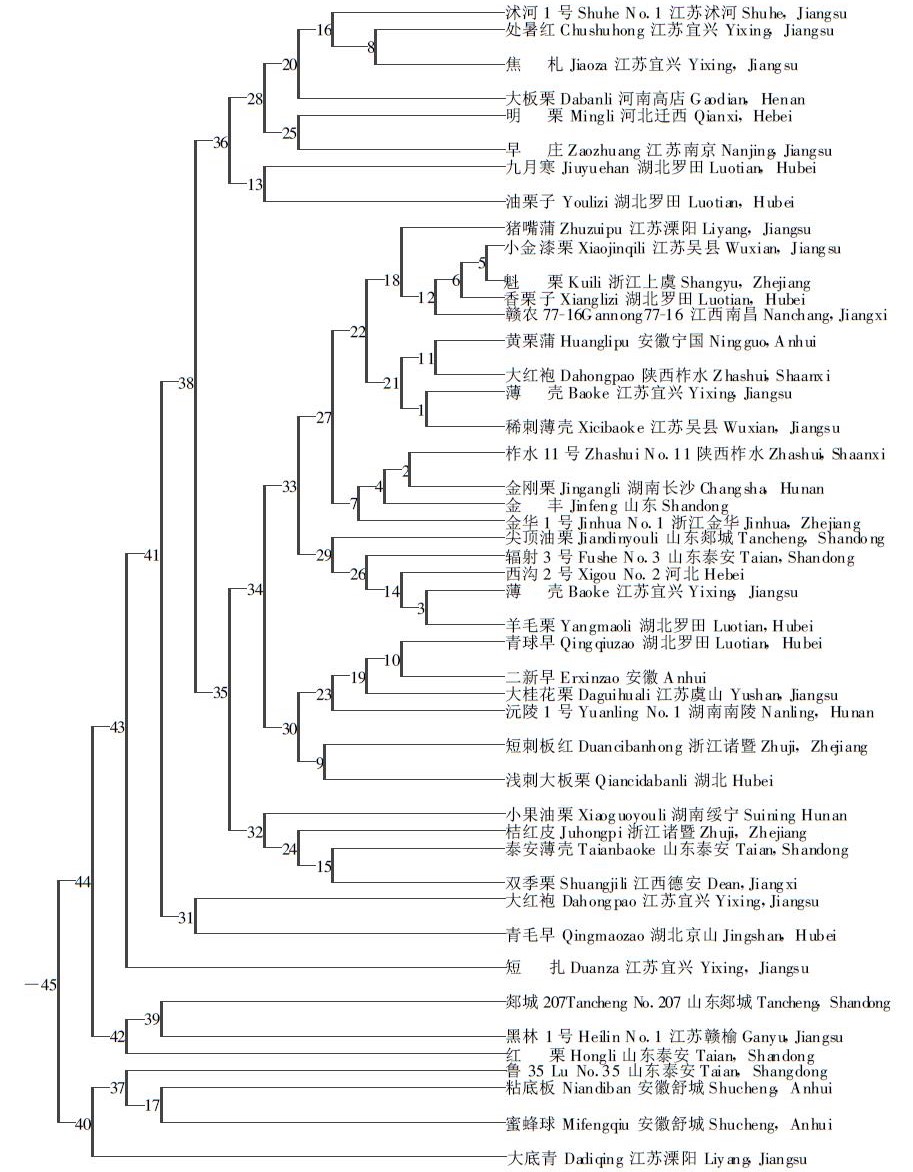
图 2 46个板栗品种的系统聚类图 Fig. 2 UPGMA dendrograms of 46 chestnut varieties based on the RAPD band pattern data |
DNA扩增仪为美国珀金—埃尔默(PERK IN-ELMER)公司生产的2400型。
用于扩增反应的10 mer引物为S系列S1~S100,共100种。引物及dNTP(10 mmol)购自加拿大Sangon独资上海分公司。标准分子量标记(100 bp DNA Ladder Plus)由立陶宛生产。Taq酶购自南京生达生物工程中心。10×PCR缓冲液等试剂均自行配置。DNA的提取与纯化采用Murray等(1990)提出的CTAB法加以改进。采用标准λDNA凝胶比色法测定样品DNA的浓度,并调节样品DNA浓度于3~30 ng/mL,一般为15 ng/mL。PCR反应体系为:总体积为20 μL。组成是:模板DNA (DNA template) 2 μL;引物(primer,10 pmol)2 μL;缓冲液(Buffer, 100 μmol/L Tris-HCl pH 8.3),500 mmol KCl,15 mmol MgCl2)2 μL;重蒸水(ddH2O)11 μL;酶稀液2 μL;0.2 μL Taq DNA聚合酶(Tag DNA Polymerase,1单位)。采用200 μL薄壁反应管;扩增反应程序为:94 ℃预变性2 min,然后执行38个扩增循环,每个循环中,94 ℃变性30 s, 40 ℃复性(退火)30 s,72 ℃延伸90 s,最后72 ℃延伸7 min。扩增产物的检测采用1 %琼脂糖凝胶板,点样后在1倍TBE缓冲液中电泳,电压100 V(电流约为5 mA)。凝胶两侧同时加有标准分子量的标记(DNA Ladder Marker),用以确定各分子标记的分子量大小。
电泳凝胶经溴化乙锭(EB)染色后,在紫外透射分析仪上观察谱带,采用快速成像系统摄影。快速成像相纸为Polaroid (ISO 3000/36°)。
1.3 数据处理由各品种DNA扩增产物谱带,记录多态性谱带的RAPD标记数据,根据Nei' s(1972, 1978)的方法,计算各品种间的遗传距离(Genetic distance)。根据DNA扩增结果的多态性谱带数据,用PHYLIP(3.5版本)的NEIGHBOR程序改进软件计算品种间的相似性,并建立UPGMA (Unweighted Pair Group Means Analysis)系统分类树。
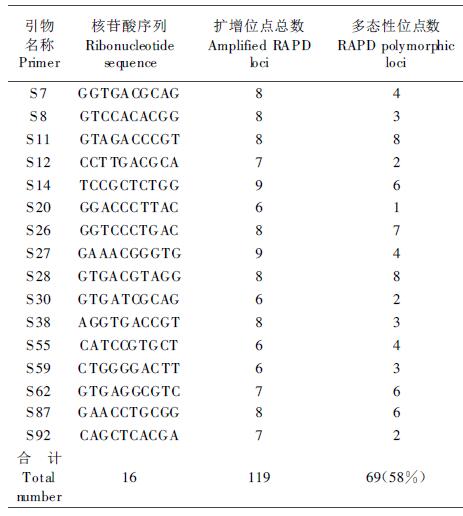
2 结果与分析 2.1 扩增结果46个板栗品种样品的DNA,用经筛选后能产生特异谱带的16个引物进行扩增反应,位点总数为119个,产生69个多态位点。板栗品种RAPD分析所用引物及其扩增谱带数如表 1。各品种的基因组DNA扩增的具有特异谱型,即品种指纹。其中,应用引物S28扩增的46个板栗品种RAPD谱带见图 1。
|
|
46个品种用16个引物随机扩增的69个特异位点的谱型,即为品种的DNA指纹,据此可以从分子水平上作品种真实性鉴定。
为采用最少的引物组合,即最优化的特异位点,来准确有效地鉴定品种。为此对特定的引物组合,计算品种间的遗传距离,从而确定这些引物组合对某一品种鉴别的特异性。逐步分析结果发现16个引物标记中,采用引物S28、S87、S11这3个引物与S59、S27或S30之一的引物组合,便可准确鉴定所研究的46个板栗品种。品种指纹库如表 2。
|
|
品种间遗传距离最大值为0.8001,即品种20与品种32(品种代号及名称详见图 2),亦即安徽舒城的粘板底与河北的西沟2号。品种间遗传距离最小值为0.0910,为品种8与品种9,即江苏宜兴的薄壳与江苏吴县西山的稀刺薄壳,据漂阳市龙潭林场进行的初步生物学观察,这两个品种差别很微小,只是稀毛薄壳的栗蒲外生刺密度稍稀,尚待进一步分析其密度的数量指标差异。
根据DNA随机扩增结果的多态性谱型分析数据,基于品种间的Nei氏遗传距离,构建品种间的UPGMA系统分类树,如图 2。具有相同起源和分布的品种,如安徽宁国的九月寒和油栗子、安徽舒城的粘底板和蜜蜂球、山东郯城的郯城207和江苏赣榆的黑林1号都被聚类到一起,归入相应的一类。其中也有相似经济性状的品种被聚成一类,如浙江诸暨的短刺板红与湖北的浅刺大板栗。
3 结论(1) 本研究发现各品种在分子水平上差异很大。RAPD分析快速、相对简便、可靠性极高。品种DNA指纹数据库及其标准化是品种审定、特异性检验、新品种专利保护和利用的重要依据。这些研究方法和结果对植物种质资源遗传多样性的分析、植物新品种保护、以及种苗质量促裁检验等具有理论意义和实践价值。
(2) 本研究发现分子分类能够有效地揭示形态相似、经济性状相近或部分地理种源相同的品种间的近缘关系。但与传统分类方法中按板栗栽培的地理区域划分的6大品种群并不完全一致。其原因主要是,一方面由于本研究所采用的46个品种主要来自长江流域品种群,这是本研究取样方面的局限。另一方面,板栗品种存在同名异物现象,特别是近10多年来各地相互引种,存在品种溯源不清的现象。这种现象使得分子分类的部分结果有待进一步分析它们的形态特长和经济性状。
陈洪. 1996. 杂交水稻汕优63杂种纯度的RAPD鉴定. 科学通报, 41(9). |
陈亮, 等. 1998. 15个茶叶品种遗传多样性的RAPD分析. 茶叶科学, 18(1): 21-27. |
傅家瑞. 1994. 利用RAPD分析技术鉴定品种纯度的研究. 种子, (22). |
甘四明, 施季森, 白嘉雨, 等. 1999. 尾叶桉和细叶桉无性系的RAPD指纹图谱构建. 南京林业大学学报, 23(1): 11-14. DOI:10.3969/j.issn.1000-2006.1999.01.003 |
李文彬, 周小梅, 张健, 等. 1995. 水稻三系及其杂种的DNA指纹图谱分析. 农业生物技术学报, 3(2). |
刘青林, 陈俊愉. 1999. 梅花亲缘关系RAPD研究初报. 北京林业大学学报, 21(2): 81-85. DOI:10.3321/j.issn:1000-1522.1999.02.015 |
罗正荣, 光森敬三, 杉浦明. 1995. 应用RAPD技术进行柿种类和品种鉴定. 日本园艺学会杂志, 64: 535-541. |
苏晓华. 1995. 用RAPD技术估测柳树种及无性系的变异. 林业科学, 31(3): 211-214. |
徐碧玉, 金志强, 彭世清, 等. 1998. 海南主栽芒果品种基因组DNA的RAPD分析. 热带作物学报, 19(3): 33-37. |
尹佟明, 黄敏仁, 王明庥, 等. 1997. 利用RAPD标记和单株树大配子体构建马尾松的分子标记连锁图谱. 植物学报, 39(7): 607-612. |
余四斌, 徐才国. 1996. 用RAPD鉴定水稻种子纯度的初探. 种子, 5: 56-57. |
Adato A, Sharon D, Lari U. 1995. Application of DNA fingerprints for identification and genetic analysis of Mango (Mangifera indica L. genotypes. J. Amer. Hort. Sci., 120(2): 259-264. DOI:10.21273/JASHS.120.2.259 |
Gregor D, Hartman W, Stosser R. 1994. Cultivar identication in Prunus domestica using PARD. Acta Hortic, 359: 33-44. |
Millan T, Osuna F, Cobos S, et al. 1996. Using RAPDS to study phylogenetic relationships in Rosa. TAG, 92(2): 273-277. DOI:10.1007/BF00223385 |
Nei Masatosiii. Genetic distance between populations. The American naturalist, 1972, Vol.106 No. 949
|
Shimada T, Haji t, Yamaguchi T, et al. 1994. Classification of mume (Prunus mume Sieb. Et Zucc.) by RAPD assay. J Japan Soc Hort Sci, 63(3): 543-551. DOI:10.2503/jjshs.63.543 |
 2001, Vol. 37
2001, Vol. 37