文章信息
- 张星耀, 赵仕光, 吕全, 贾秀贞, 刘会香.
- Zhang Xingyao, Zhao Shiguang, Lü Quan, Jia Xiuzhen, Liu Huixiang.
- 树木溃疡病病原真菌类群分子遗传多样性研究Ⅱ——Botryosphaeria属28S rDNA-PCR-RFLP和RAPD解析
- MOLECULAR GENETIC DIVERSITY OF PATHOGENIC FUNGAL GROUP CAUSING TREE CANKER Ⅱ-28S rDNA-PCR-RFLP AND RAPD ANALYSIS OF BOTR YOSPHAERIA SPP.
- 林业科学, 2000, 36(2): 75-81.
- Scientia Silvae Sinicae, 2000, 36(2): 75-81.
-
文章历史
- 收稿日期:1999-10-14
-
作者相关文章
2. 山东农业大学林学院 泰安 271000
2. Forestry College of Shangdong Agriculture University Taian 271000
Botryosphaeria Ces.et de Not. (无性阶段是Dothiorella Sacc)属引起的树木溃疡病是目前我国人工林的重大生物灾害之一(张星耀, 1998a), 它分布于我国广大区域, 包括东北、华北、江淮、江西、西北等地区。该属真菌可危害近200个杨树种、杂交种和无性系, 造成生长量的损失和死亡(向玉英, 1987)。除了杨树外, 还危害核桃、柳树、梧桐、刺槐、苹果、石榴、桃树以及雪松等。较大的寄主范围和地理分布区域促使病原菌发生分化, 是产生分子遗传多样性的机制之一(张星耀, 1999)。
研究该病原真菌的分子遗传多样性, 对于掌握病原菌对寄主的适应性、致病性和地理分化具有重要意义, 为抗病育种和溃疡病的可持续控制提供理论依据, 同时为溃疡病病原真菌类群的分子系统积累原始数据。
1 材料和方法 1.1 供试菌供试菌为Botryosphaeria属内2个种的30个菌株(见表 1), 地理分布区域包括西北暖温带半干旱半湿润季风气候区, 东北温带干旱、半干旱季风型大陆性气候区, 江淮平原北亚热带湿润区, 华北湿润、半湿润暖温带区南部的黄淮平原和华北湿润、半湿润地区北部; 寄主包括杨树的多个品种、槐树、桃树、石榴、雪松以及苹果等。虽然供试菌株并没有涵盖所有寄主及地理区域, 但从整体范围上看具有代表性。在野外具有典型症状的寄主上采得病组织后, 采用一般的组织分离法和菌落边缘菌丝纯化法得到菌株(方中达, 1979)。在PSA (马铃薯200g, 蔗糖20g, 琼脂20g, 水1000mL)平面培养基上培养, 并据在培养基上产生的子实体对菌株的分类地位进行形态解剖鉴定。鉴定后的菌株进一步人工接种至寄主枝条, 证实其致病性, 并根据在寄主接种部位出现典型症状的组织上产生的子实体, 再次对菌株进行形态解剖鉴定。经上述过程最后确定分类地位的菌株进行进一步单分生孢子分离纯化, 纯化后的菌株用于分子遗传多样性研究的试验。试验用菌株保存于PSA斜面培养基上。使用前在PSA平面培养基上培养10d左右, 随后在菌落边缘取直径3mm的菌饼接种至液体培养基(马铃薯200g, 蔗糖20g, 水1000mL)中, 27℃、静置、无光照培养10d后收获菌丝。收获后的菌丝经灭菌无离子水3次洗净后保存于-20℃备用。
|
|
DNA提取方法主要参考Zhu Heng等的方法(1993), 并作部分改进。具体方法如下:将0.1 g菌丝剪断, 加500μL提取缓冲液(100 mM Tris-HCl, pH9.0;40 mM EDTA, pH8.0), 100μL 10%SDS, 300μL氯化苄, 充分混匀, 50℃培育1 h, 每5 min充分混匀1次。加300μL 3 M醋酸钠, pH5.0, 冰中保持15 min, 然后离心(11000 rpm, 4℃, 20 min), 取上清液。加入1/2体积的饱和酚, 抽提2次, 加2/3体积的冷异丙醇, 混匀, 放置冰箱中1 h以上。离心(12000 rpm, 10 min), 去上清液。70%冷乙醇洗涤2~3次, 每次10 min并离心(12000 rpm)。充分干燥, 去除乙醇(阴干)。加适量(约100μL) TE溶解, 并加入RNaseA (20μg/mL), 37℃处理45 min后, 置-20℃保存备用。
1.3 28S rDNA的PCR扩增试验采用在真菌、植物和动物的rDNA扩增中广泛使用的2种引物:LROR (5'-ACCCGCTAACT-TAAGC-3')和LR7 (5'-TACTACCACCAAGATCT-3') (张星耀, 1998b; Reader U et al., 1985; Vilgalys R et al., 1990), 委托华美生物技术公司(北京)合成, 在PCR仪(Perkin-Elmer DNA Thermal Cycler480)上扩增。反应总体积为100μL, 其中模板DNA50 ng左右, 10×的Mg2+10μL, 10×的PCR反应缓冲液10μL, dNTP (10mM) 4μL, Tag酶5U, 以灭菌无离子水补足100μL。反应条件为:94℃预变性4min以后, 94℃变性1 min、52℃退火1 min和72℃延伸2 min为一个循环, 共计45个循环后, 72℃保持10 min。为防止扩增过程中高温引起反应液蒸发, 于反应液表面加入20μL矿物油。相同条件下设无模板DNA的扩增反应为对照。扩增产物在1.6%的琼脂糖(Promega产品)凝胶(含有5μL 1%的溴化乙锭/100 mL)上、1×TBE缓冲液、电压80 V、3 h电泳分离, 紫外灯下观察、照相记录。选用华美公司Ladder 100 bp的PCR marker作为分子量标记。
1.4 内切酶筛选和酶解反应通过用PvuⅡ, RsaⅠ, Sau3AⅠ, TagⅠ, NruⅠ, MspⅠ, BssHⅡ, HaeⅢ, EcoRⅠ, HpaⅡ, PstⅠ, EcoRV, Hind Ⅲ, BamHⅠ, HhaⅠ等15种内切酶作酶解试验, 其中6种酶有切点(表 2), 用于RFLP解析。
|
|
酶解反应总体积为25μL, 其中DNA扩增产物10μL, 限制性内切酶2μL, 相应的缓冲液2μL, 灭菌无离子水11μL, 最适反应温度完全酶解(1 h以上)后, 加入EDTA溶液使终浓度为10 mM, 终止反应。酶解产物在1.6%琼脂糖凝胶(含有5μL 1%的溴化乙锭/100 mL)上、1×TBE缓冲液、80V电压、3 h电泳分离, 紫外灯下观察、照相记录。选用华美公司Ladder 100bp的PCR marker作为分子量标记。
从DNA提取, 经PCR扩增至酶切及电泳分离, 整个试验的重复次数大于2。
1.5 RAPD分析的DNA随机扩增及其引物的选择实验中所用引物由Operan Tech.公司合成, Tag酶由中国科学院遗传研究所生产。
扩增条件 采用25μL反应体系。包括2.5μL缓冲液(100 mM Tris-HCl, 15 mM MgCl2, 50 mM KCl), 1μL的随机引物, 0.5μL的dNTP (10 mM), 0.2μL的Tag酶(5U/μL), 1μL的模板DNA和19.8μL的灭菌双蒸水, 反应体系中最后加入20μL矿物油以防止反应液蒸发。
反应程序 94℃预变性4 min后进入循环:94℃变性45 sec, 39℃退火1 min, 72℃延伸1 min 40 sec, 共45个循环, 然后在72℃延伸10min结束反应。
扩增反应在PCR扩增仪(Perkin-Elmer DNA Thermal Cycler 480)上进行。相同条件下设无模板DNA的扩增反应为对照。扩增产物在1.6%的琼脂糖(Promega产品)凝胶(含有5μL 1%的溴化乙锭/100 mL)上、1×TBE缓冲液、电压80 V、3 h电泳分离, 紫外灯下观察, 照相记录。选用华美公司Ladder 100 bp的PCRmarder作为分子量标记。
从DNA提取, 经PCR随机扩增至电泳分离, 整个试验的重复次数大于2。
使用40个引物(KITs:OPK1~20和OPR1~20)进行DNA的随机扩增, 其中13个引物(见表 3)具有重复性好、特异性高且谱带强、适合判读、有较高多态性的表现, 用于RAPD分析。
|
|
根据某一条谱带在样品中出现和不出现, 出现赋值为“1”、不出现赋值为“0”, 统计各菌株的扩增谱带数, 并依据公式S=2Nab/ (na+nb), 计算样品间的相似系数S。公式中Nab代表两样品间共有的谱带数, na代表a样品的总谱带数, nb代表b样品的总谱带数。
用非加权平均连锁法(UPGMA)分析供试菌株间的亲缘关系。相似系数的计算和系统聚类分析均由NTSYS (Version 1.72, 1995)分析软件自动完成。
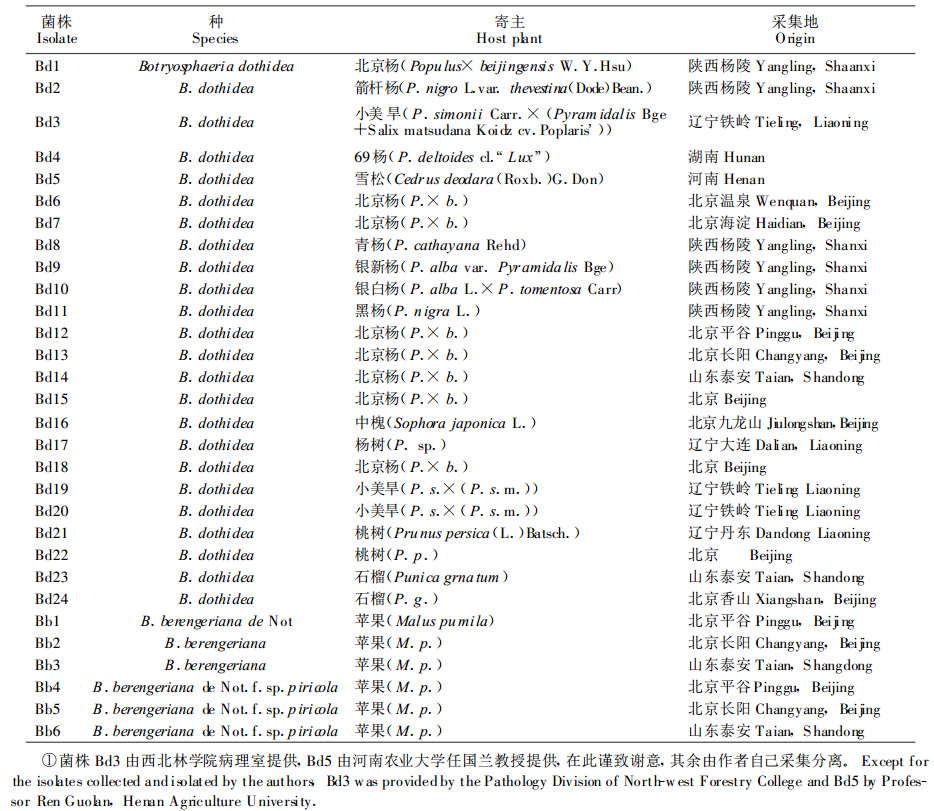
2 结果与分析 2.1 28S rDNA扩增结果所有供试菌株在上述28S rDNA的PCR扩增体系和条件下均得到稳定的一条谱带, 分子量为1380 bp。供试菌株的扩增图谱如图版 1所示。
|
图版Ⅰ Plate Ⅰ 1.供试菌株28S rDNA的扩增结果。2.Tag Ⅰ对供试菌株28S rDNA的酶切图谱。小部分菌株, 其余菌株谱带相向。下同。3. Rsa Ⅰ对供试菌株28S rDNA的酶切图谱。4. Hae Ⅲ对供试菌株28S rDNA的酶切图谱。5. Sau3A Ⅰ对供试南株28S rDNA的酶切图谱。6. Hpa Ⅱ对供试菌株28S rDNA的酶切图谱。7.Msp Ⅰ对供试菌株28S rDNA的酶切图谱。 1. PCR result of 28S rDNA of isolates 2. Ihe bands of isolates digested by Tag Ⅰ Part of isolates showed. the rest are same with them. It is same as following 3. The bands of isolates digested by Rsa Ⅰ The bands of islates digested by Hae Ⅲ. 5. The bands of isolates digested by San3A Ⅰ 6. Ihe bands of isolates digested by Hpa Ⅱ 7 The bands of isolates digested by Msp Ⅰ |
6种内切酶对供试菌株28S rDNA的酶切图谱如图版1所示, 每种内切酶对于30个菌株的酶切谱带都表现出完全一致性(表 4), 表明试验所用内切酶不足以区分Botryosphaeria属内部28S rDNA的差异。
|
|
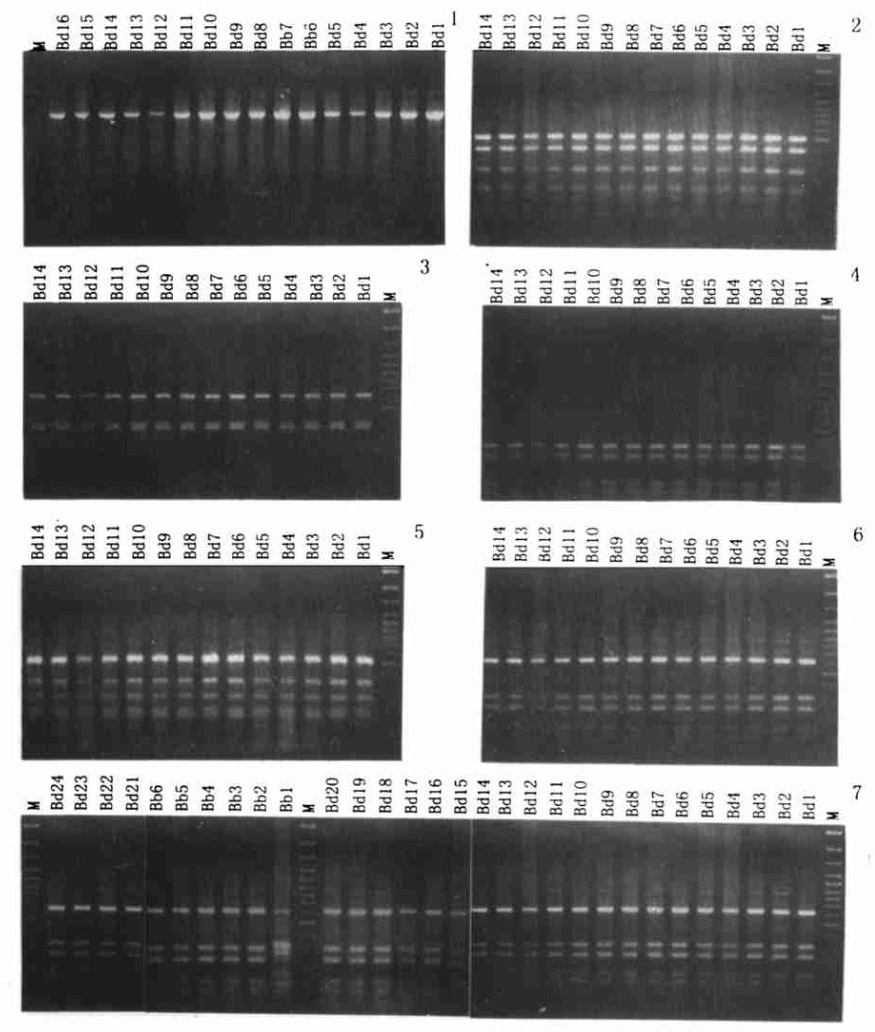
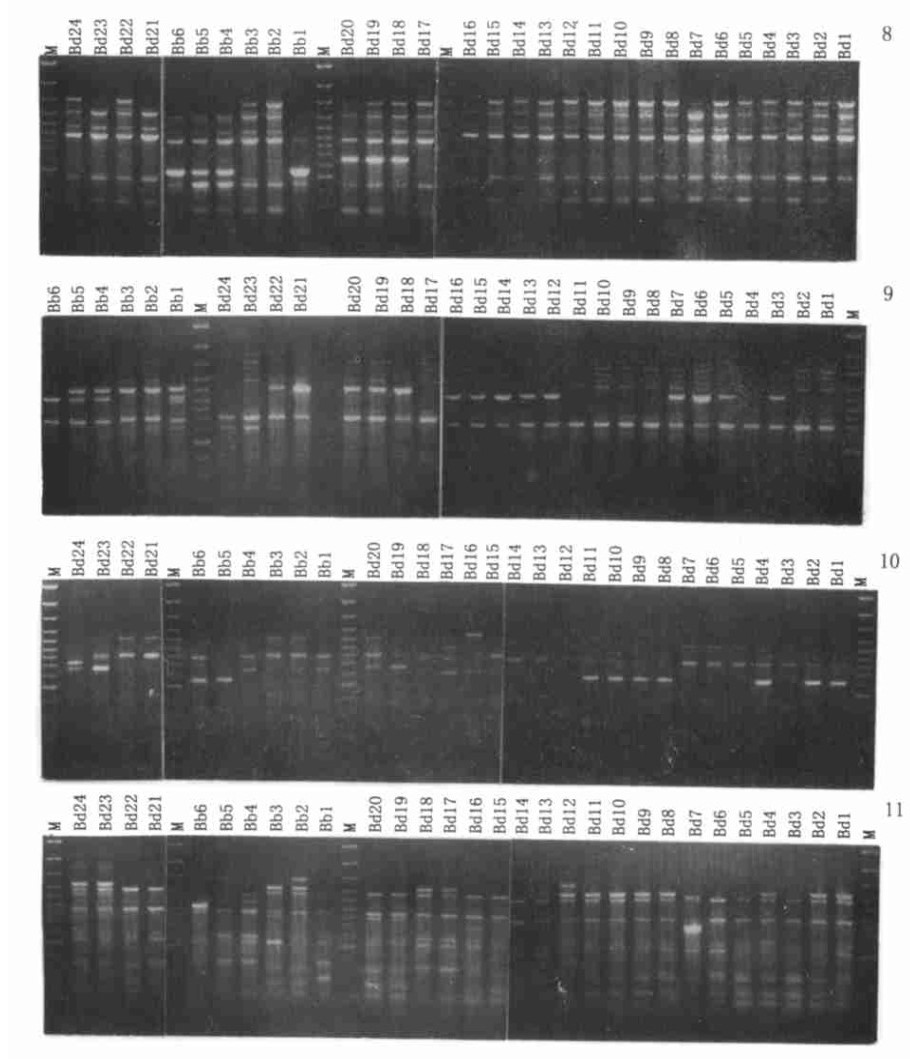
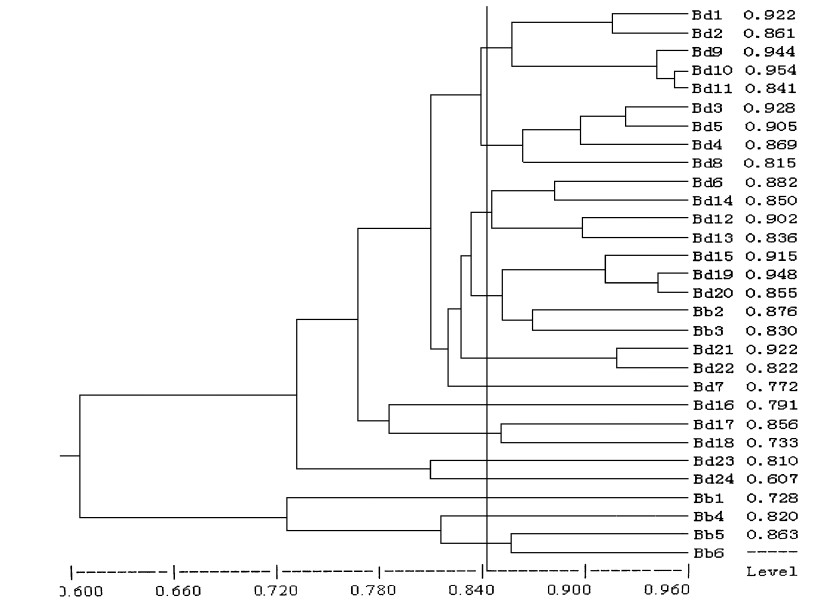
使用13个引物进行RAPD扩增, 得到153条谱带(图版 2), 其中142条出现多态性, 占总带数的92.81%。对所用谱带进行聚类分析, 得到相似系数矩阵(表 5)、树状聚类图(图 1)。
|
图版Ⅱ Plate Ⅱ 8.R08对供试菌株的扩增图谱。M为分子量标记, 从上到下分子量依次是:3000, 2000, 1500, 1200, 1031, 900, 800, 700, 600, 500, 400, 300, 200, 100(bp)。其余同。9.K16对供试菌株的扩增图谱。10.R14对供试菌株的扩增图谱。11.R11对供试菌株的扩增图谱。 8. The amplified bands of isolates by R08. The molecular weight of each bands of marker(M) from the top down is 3000, 2000, 1500, 1200, 1031, 900, 800, 700, 600, 500, 400, 300, 200, 100(bp). It is same with the rest. 9 The amplified bands of isolates by K16. 10. The amplified bands of isolates by R14. 11. The amplified bands of isolates by R11. |
|
|
|
图 1 供试菌株RAPD聚类UPGMA树状图 Fig. 1 UPGMA cluster dendrogram based on RAPD data from 30 isolates using 13 different primers |
根据0.845的相似系数水平可以将供试的30个菌株分为13类。其中来自陕西杨陵不同杨树品种上的菌株Bd1、Bd2、Bd9、Bd10、和Bd11聚为一类, 表现出病原菌的地理分化(来自同一地区的菌株表现出更强的亲缘性), 另外有来自于相同寄主的菌株聚为一类, 表现出明显的寄主分化, 如分别来自北京和山东的北京杨上的菌株Bd6、Bd12、Bd13和Bd14聚为一类, 分别来自辽宁、北京的桃树上的两菌株Bd21和Bd22聚为一类, 以及来自中槐的菌株Bd16单独成为一类。但是在表现出地理分化、寄主分化的同时, 也有不表现分化的情况。同样来自陕西杨陵的菌株Bd8与其余5个聚在一起的陕西菌株中最大的相似系数也仅为0.882, 被聚在另外一类中, 而与分别来自于辽宁、河南、湖南的菌株Bd3、Bd5、Bd4聚为一类; 同样来自北京杨上的菌株Bd12、Bd15、Bd7、Bd18分属于不同类群, 没有表现出寄主分化, 以及石榴上的山东菌株Bd23和北京菌株Bd24之间的相关系数仅为0.810, 分属于两个类群。同一地区小美旱上的3个菌株Bd3、Bd19和Bd20中, 后两者亲缘关系十分接近, 相关系数达0.948, 而Bd3则聚在另外一个较远的类群中。
Bd5是引起雪松溃疡病的菌株, 30个菌株中唯一来自于裸子植物寄主上的。从系统聚类树中可以明显看出它与其它所有阔叶树上的菌株没有表现出明显差异, 甚至体现出的多态性只是局限于B.dothidea种内, 与另外一个种B.berengeriana处于亲缘关系较远的两个类群中。表明B.dothidea不仅能够寄生被子植物, 还能寄生裸子植物, 有非常广阔的寄主范围。
3 结论和讨论 3.128S rDNA分析结果表明该编码区段不适宜表现Botryosphaeria属下分类单位的分子多样性。这与该区段进化速率相对慢, 序列高度保守有关, 它只适宜于进行属、科乃至目水平上的分类比较。
3.2引物K16和R14可以作为鉴别陕西杨陵菌株(Bd1、Bd2、Bd8、Bd9、Bd10和Bd11)和湖南菌株(Bd4)与其它菌株区别的特异性引物。R14的扩增图谱中, 这两个地区的菌株共有一条600 bp的特殊谱带, 而在K16的扩增谱带中这些菌株与其它菌株相比又恰好缺少一条1000 bp的谱带。
3.3目前认为苹果干腐病菌B.berengeriana与杨树溃疡病菌B.dothidea是同属一属的两个种, 苹果轮纹病菌B.berengeriana de Not.f.sp.piricola为苹果干腐病菌的专化型。本试验分析结果与这种分类状况基本一致, 表明它们分类地位的合理性。苹果轮纹病菌Bb4、Bb5和Bb6有较高的亲缘关系。而苹果干腐病菌Bb1、Bb2和Bb3并没有表现出紧密的亲缘关系, Bb2和Bb3聚在一起, 与其它溃疡病菌共同形成一类, 与Bb1只在0.607的相似系数上聚为一类, 种间的相似性大于种内相似性。是不是由于苹果干腐病菌的分化或不同病原物引起苹果干腐形成这种现象, 有待深入研究。
3.4B.dothidea种群内部存在着较为丰富的RAPD多态性, 这种多态性能够在一定程度上反映出菌株的地理分化、寄主分化, 但同时也有不表现地理分化、寄主分化的大量多态性存在。溃疡病菌在大范围内主要依靠人为传播, 受到地理、社会和经济等多方面的影响。溃疡病菌有潜伏侵染的特性, 人为传播的主要方式是苗木调运。苗木的调运也受树种的地区适应性影响和限制, 使得不同地区溃疡病菌株在地区间的传播也受到限制, 随着时间的延长, 则可能形成适应当地气候条件的菌株, 最后产生致病力和形态上的分化, 在DNA的水平上也表现出了一致性和多态性。可以据此推断病原菌的起源。该种具有广阔的寄主范围, 不但可以寄生被子植物, 还可以以裸子植物为寄主产生致病性。
方中达. 1979. 植病研究法. 北京: 农业出版社, 127-132.
|
向玉英. 1987. 杨树病害及其防治. 北京: 中国林业出版社, 55-60.
|
张星耀, 赵仕光, 朴春根, 等. 1999. 树木溃疡病病原真菌类群分子遗传多样性研究. Ⅰ.小穴壳菌属、疡壳孢属、壳囊孢属、盾壳霉属分类地位的分子证明. 林业科学, 35(3): 34-40. DOI:10.3321/j.issn:1001-7488.1999.03.007 |
张星耀.森林生物灾害控制的新世纪策略及其实施基础.中国林业科学院编, 面向21世纪的林业-可持续发展全球战略下的林业科学技术.北京: 中国农业科技出版社, 1998a: 341~345
|
张星耀. 1998b. 基于培养性质模糊解析和28S rDNA PCR RFLP解析的外担子菌属真菌的分类学研究. 林业科学, 34(4): 59-71. |
Reader U, Breda P. 1985. Rapid preparation of DNA from filamentous fungi. Lett. Appl. Microbiol., 1: 17-20. DOI:10.1111/j.1472-765X.1985.tb01479.x |
Vilgalys R, Hester M. 1990. Rapid genetic identification and m apping of enzymatically amplified ribosomal DNA from several Ceyptoccus species. J. Bacteriol, 172: 4238-4268. DOI:10.1128/jb.172.8.4238-4246.1990 |
Zhu Heng, Qu Feng, Zhu Lihuang. 1993. Isolation of genomic DNAs from plants, fungi and bacteria using benzy 1 chloride. Nucleic Acids Research, 21(22): 5279-5280. DOI:10.1093/nar/21.22.5279 |
 2000, Vol. 36
2000, Vol. 36