文章信息
- 尤勇, 洪菊生.
- You Yong, Hong Jusheng.
- 杉木rDNA ITS -1区的克隆及序列测定
- THE CLONING AND SEQUENCING OF THE rDNA INTERNAL TRANSCRIBED SPACER 1 REGIONS OF CHINESE FIR
- 林业科学, 2000, 36(1): 121-124.
- Scientia Silvae Sinicae, 2000, 36(1): 121-124.
-
文章历史
- 收稿日期:1998-05-25
-
作者相关文章
编码核糖体RNA的基因是由一些高度重复序列组成的多基因家族。rRNA基因以串联重复形式存在于染色体上, 每个细胞中的拷贝数目在1000~10, 000之间, 由于rRNA基因中包含了进化速度不等的编码区、非编码转录区和非转录区, 因而可以选择其中保守程度不同的片段作分子标记研究植物的系统发育及遗传变异。
ITS是核糖体DNA中介于18s和5.8s之间(ITS-1)以及5.8s和26s (ITS-2)之间的转录间隔区, 目前已研究过的被子植物ITS-1一般小于700bp (Alvarez-Bulylla et al., 1995), 而裸子植物的ITS-1远大于1000bp, 由于该片段长度不大且其进化速度快, 加上协同进化(concertedevolution), 使得该片段在基因不同重复单元间十分一致, 适于作为探讨科内、属间及属下种间系统关系的标记。
杉木属(Cunninghamia Lanceolata Hook)是我国南方主要用材、造林树种, 其栽培历史悠久, 分布区域广泛生态环境多样对于杉木属的系统分类研究长期以来学者们从杉木的化石形态结构染色体等水平作了大量工作, 形成了多种观点(中国植物志编辑委员会, 1978; 郝景盛, 1950; 叶培忠, 1979; 王德银, 1982; 吴中伦, 1983)。目前大多学者认为有杉木属有一个种(吴中伦, 1983)杉木(而且形成了许多的观点, Cunninghamia Lanceolata ((Lamb.) Hook)和一个变种台湾的峦大杉(Cunninghamiakonishii Hayata)。
本实验对于杉木的ITS-1序列进行了克隆和测序, 为杉木ITS序列的研究做了最基础的铺垫, 也为更进一步研究杉木属的系统进化创造了条件。由于杉木属rDNA的研究尚未见报道, 本文在引物设计时借鉴了已研究过植物的ITS-1上下游序列。
1 材料和方法 1.1 样品的采集实验材料从全国杉木种源基因库中采样, 它们分别是:福建建瓯、四川德昌。从树木上采集到的新鲜针叶经液氮处理后, 放置在-70℃冰箱内保存。
1.2 杉木总DNA提取按文献的方法(尤勇, 1998)进行。
1.3 ITS-1 序列扩增及克隆根据已发表的植物18SrRNA和5.8SrRNA的序列合成了引物1和引物2。
引物1:AGAAGTCGTAACAAGGTTTCCGTAGG
引物2:GATGCGAGAGCCGAGATAACCGTTG
其中引物1与18SRNA基因3'段序列相同, 引物2同5.8SRNA5'端序列互补, 引物1和2的结合位点及PCR扩增ITS-1区位置见图 1
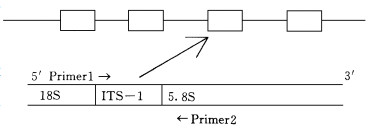
|
图 1 引物及其扩增区域 Fig. 1 Primers and their amplification regions |
ITS-1序列的扩增及补平PCR反应体系总体积为25μL, 其中dTTP、dCTP、dGTP和dATP各100μM, 引物1和引物2乃各50pmol, 模板DNA200ng, 10×PCR反应缓冲液2.5μL, Taq耐热DNA聚合酶2.5μL, 反应液上覆盖40μL石蜡油。反应的条件为:97℃5min→95℃5min→加入Taq DNA聚和酶→94℃1min, 56℃1min, 72℃2min, 35个循环→反应产物在72℃中补平10min。
PCR产物在1.0%琼脂糖凝胶中电泳检查, 并用Geneclean Kit回收, 取500 ng扩增产物用T4DNA聚和酶补平。
补平反应体系为10μL, 具体成分如下:T4DNA ligase l μL (SuluL), 1μL 1.25 mMdNTP, 1μL 10×buffer (A), 7μL PCR产物。补平条件为37℃保温15min, 然后放入65℃的水浴锅中灭活T4DNA ligase。
ITS-1序列的克隆 取上述补平反应液与经HindⅡ完全酶切并纯化的pUC19以3:1的比例在T4DNA连接酶作用下连接, 连接产物用常规方法转化大肠杆菌JM83菌株, 插入片段经EcoRI-HindⅢ双酶切予以确认。
DNA序列测定 序列测定采用Sangger双脱氧法(Roger, 1987), 在LKB Model 2010序列仪上进行。
2 结果与分析 2.1 杉木ITS-1序列的克隆本实验比较了模板的质量及浓度, 扩增的程序等因素对ITS-1序列的扩增的影响。ITS-1序列的扩增对于模板质量要求较高, 带有杂质或部分降解的模板都不能扩增出清晰ITS-1条带。在反应程序的设计上, 延长模板变性的时间至10min, 同时将Taq酶的加入放在模板变性以后有利于ITS-1条带的特异扩增。不同植物ITS-1扩增的退火温度也不相同, 56℃的温度更适合杉木ITS-1序列的扩增。
扩增的ITS-1序列回收后, 必须补平, 这样有利于同质粒载体的连接。连接产物转化时, 本实验使用了DH5α和JM83菌株两种大肠杆菌, 转化结果基本相同, 由于JM83菌株生长较DH5α菌株快, 且阳性克隆易选择所以最后选择了菌株。
2.2 杉木ITS-1序列的测定利用DNA序列测定仪测定了杉木两个种源的杉木ITS-1序列(见图 2)。杉木ITS-1序列的长度为273bp, 两个种源的ITS-1序列长度以及碱基组成完全相同。该结果说明杉木ITS-1序列在杉木两种源间比较保守, 不存在变异。从杉木ITS-1序列中可以看出A、T、C、G四种碱基的使用频率分别是27.5%、23%、21.6%、27.9%, 碱基G的使用频率略高, 但总体上看4种碱基的使用频率基本一致, 不象水稻、大豆等植物G/C含量那么高。由于杉木ITS-1序列G/C含量不是太高, 所以其ITS-1序列在测定时较G/C含量高的植物容易。另外序列分析的结果显示, 杉木ITS-1序列中存在[AAAG]n和[TTG]n的短重复序列。

|
图 2 杉木(Cunninghamia Lanceolata Hook)不同种源ITS-1序列排列阵(Sequence Alignment) Fig. 2 The ITS-1 Sequence alignment of Chinese fir from differert provenance |
ITS序列在被子植物系统学上应用时间虽然较短, 但已取得许多可喜的进展, ITS序列的分析被应用于许多被子植物的分类学及系统学研究上(李承森, 1997)。裸子植物的ITS序列的测定和应用研究较少, 而且对ITS序列能否作为一种分子标记应用于裸子植物的系统演化及遗传变异上尚存在不同的观点, 有的学者认为裸子植物ITS序列变异较大、序列长度较长, 不适合作研究裸子植物系统演化及遗传变异的标记(李承森, 1997), 也有的学者认为ITS序列可应用于裸子植物系统学及遗传变异的研究, 并且已做了深入的探讨(Vining, 1998; Markos et al., 1998), 如Karvonen的研究结果认为不同裸子植物的ITS-1拷贝的长度不同, 可相差约500bp, 有的甚至相差几千个碱基(Karvonenn et al., 1994)。Alvarez-Buylla通过研究裸子植物7个科25个属的ITS发现裸子植物ITS明显长于被子植物, ITS的平均长度为1500, 其中雪松属(Cedrus)、粗榧属(Cephalotaxus)、落羽杉属(Taxodim)的为1000bp, 云杉属(Picea)的可长达3400bp (Alvarez-Bulylla et al., 1995); Karvonenn通过研究挪威云杉发现其长度变异主要存在于ITS-2部分, 而且认为ITS长度的变异对于云杉的系统学研究是一个有利的工具(Karvonenn et al., 1994)。Alvarnez通过研究两个松科亚属的15个代表种发现, ITS序列的变异主要存在ITS-2部分, 并且认为TIS-2的序列变异便于揭示较高分类级别的系统进化关系。从以上几例可以看出ITS-1和ITS-2分别适合于不同分类层次的系统研究。对于松科来讲, 其ITS-2部分的变异较大, 而ITS-1部分的变异较小。本实验只是克隆和测定了杉木两种源的ITS-1序列。从实验取材上讲, 两个杉木种源分别来自福建和四川两个省, 分别位于杉木产区的东西边缘, 种源的生存环境存在极大的差异(于永福, 1995)。全国杉木种源协作组曾将德昌种源列为特殊生态类型下的种源, 建瓯种源与德昌种源在生长状况材性抗逆性等方面存在较大差异建瓯种源被认定为生产力优良种源, 而德昌种源为生长劣势种源(洪菊生, 1994)。德昌种源还被有的学者认定为杉木属的一个新种(王德银等, 1982)。从实验的结果看两种源间不存在ITS-1序列上的差异, 至于杉木ITS2部分以及整个杉木的ITS情况有待更进一步的研究。
对于杉科ITS序列的研究至今尚未见报道, 从已报道的松科(Pinaceae)、粗榧科(Cephalotaxaceae)、红豆杉科(Taxaceae)的ITS来看, ITS的序列长度变异也很大, 从雪松1000bp到云杉的3400bp不等。由于杉木整个ITS序列尚未测定, 现在还不能断定其ITS的长度情况, 单从其ITS-1的长度可以说其较已知裸子植物的序列要短一些要了解更多的情况须借助更多杉科序列的测定.
郝景盛. 1950. 中国裸子植物志. 北京: 人民出版社.
|
洪菊生, 等. 1994. 全国杉木地理种源试验专刊. 林业科学研究, 7: 130-144. |
李承森等编.植物科学进展.第一卷.北京: 高等教育出版社, 1997: 22 ~ 23
|
王德银, 刘和林. 1982. 杉木属新种及新变种. 植物分类学报, 20(2): 210-232. |
叶培忠.杉科树木属间杂交初步研究.南京林产工业学院学报, 1979
|
尤勇, 等. 1998. 杉木DNA的简便提取方法. 林业科学研究, 11(1): 111-113. |
吴中伦. 1983. 杉木. 北京: 中国林业出版社.
|
于永福. 1995. 杉科植物的起源、演化及其分布. 植物分类学报, 33(4): 362-389. |
中国植物志编辑委员会.中国植物志第七卷, 北京: 科学出版社, 1978
|
Alvarez-Buylla, Elena 1 Listion. The nuclear ribosome DNA internal transcribed spacer of gymnosperms is significantly longer than angiosperm s. Plant Genome Ⅲ, January, 1995
|
Karvonen P, Szmidt A E. 1994. Length variation in the internal transcribed spacers of ribosomal DNA in Picea abies and related species. Theoretical and Applied Genetics, 89: 969-974. |
Markos, Staci and Bruce G, Baldw in. Use of the external transcribed spacer(ETS)of nrDNA in phylogenetics studies of young lineages: An exam ple from Lessingia(Compositae, Astereae). Systematic Section/ american Society of Plant Taxonomists Abstract Index, 1998, Annual Meeting of the Botanical Society of America, 2 ~ 6, August, 1998, Baltimore, M D
|
Rogers S O.Ribosomal RNA genes in plants: variability in copy num ber and in the intergenic spacer. Plant Mo1 Bio1, 1987, 17: 2852
|
Vining, Thomas F. Nuclear ribosomal DNA (nrDNA)internal transcribed spacer1(ITS1) subrepeats and phlogeny of Pinaceae. Systematic Sec-tion/ American Society of Plant Taxonomists Abstract Index, 1998, Annual Meeting of the Botanical Society of America, 2 ~ 6, August, 1998, Baltimore, M D
|
 2000, Vol. 36
2000, Vol. 36
