文章信息
- 崔廷涛, 潘一展, 高瑞, 邵云华, 李向东, 时呈奎, 赵文军, 朱水芳.
- Cui Tingtao, Pan Yizhan, Gao Rui, Shao Yunhua, Li Xiangdong, Shi Chengkui, Zhao Wenjun, Zhu Shuifang
- 荆条丛枝病植原体的分子检测及鉴定
- Molecular Detection and Identification of the Phytoplasma Associated with Chaste Tree Witches'- Broom
- 林业科学, 2009, 45(6): 162-165.
- Scientia Silvae Sinicae, 2009, 45(6): 162-165.
-
文章历史
- 收稿日期:2008-05-12
-
作者相关文章
2. 商丘职业技术学院 商丘 476100;
3. 中国检验检疫科学研究院动植物检疫研究所 北京 100029
2. Shangqiu Vocational and Technical College Shangqiu 476100;
3. Institute of Animal and Plant Quarantine, Chinese Academy of Inspection and Quarantine Beijing 100029
植原体(Phytoplasma)是一类无细胞壁,迄今不能人工培养,存在于植物筛管细胞中的类似植物病原细菌的原核生物。据估计,植原体可在98个科,几百种植物上引起病害(Lee et al., 2000),主要症状包括丛枝、黄化、花变叶、花器退化等,严重时使植株提早衰老,直至整株枯死。
PCR、RFLP、核酸杂交等技术的发展极大地推动了植原体的研究。16S rRNA基因、16S-23S基因间隔区(16S-23S spacer region,SR)、核糖体蛋白(ribosomal protein)基因rp、延伸因子Tu(elongation factor Tu)基因tuf等均是植原体进化中高度保守的基因组序列,已经被广泛用于植原体的检测和鉴定(Lim et al., 1989;Lee,1993;Lee et al., 1993;Schneider et al., 1997;Lee et al.,1998;Tian et al., 2000;Abou-Jawdah et al., 2002;Harrison et al., 2002;Gao et al., 2008),其中以16S rRNA基因的应用最多(车海彦等,2006)。Lee等(1998)根据植原体16S rRNA基因的RFLP分析将植原体34个典型的株系划分为14个组32个亚组,其中翠菊黄化组(16SrI)变异最大、分布最广。2004年,国际比较菌原体学研究计划(IRPCM)植原体/螺原体工作组建议建立一个新属‘Candidatus (Ca.) Phytoplasma’,制定了相应的分类标准,综合相关研究把植原体划分成26个候选种(The IRPCM Phytoplasma/Spiroplasma Working Team,2004)。2007年,Wei等(2007)根据16S rRNA序列的RFLP分析结果将植原体划分为28个组。
荆条(Vitex negundo var.heterophylla,英文名:chaste tree),马鞭草科, 落叶灌木,叶和果实可入药,花是优良的蜜源植物。荆条耐寒、耐旱,多生长于山地阳坡的干燥地带,形成灌丛,或在盐碱沙荒地与蒿类植物自然混生。荆条对荒地护坡和风沙防治等有一定作用,可开发利用为裸露坡面生态修复的乡土护坡植物,因而极具开发价值。为了证明泰山上近年发生的荆条丛枝病是否与植原体有关,笔者应用巢式PCR技术及16SrRNA序列比较和分析等手段,对该病的病原进行了分子检测和鉴定。
1 材料与方法 1.1 试验材料荆条健株和丛枝病株均采自泰山。Mini-DNA Rapid Purification Kit购自北京博大泰克生物基因技术有限公司,克隆载体pMD18-T Easy Vector及分子生物学试剂购自TaKaRa公司。
1.2 电镜超薄切片观察参照文献(Gao et al., 2008)中的方法,取表现丛枝的荆条植株嫩茎,切成1 mm ×10 mm的长条,经2.5%戊二醛和1%锇酸双重固定,乙醇逐级脱水,环氧丙烷置换,Epon812渗透包埋,LKB-2188型超薄切片机切片,经醋酸双氧铀和柠檬酸铅双重染色后,置JEM-1200 EX Ⅱ型透射电镜下观察。
1.3 荆条丛枝植原体的分子检测1) 植物总DNA的提取 分别取0.1 g感病及健康荆条植株的幼嫩部分及韧皮部组织,采用改良的CTAB法(漆艳香等,2004)提取植株总DNA。2)PCR扩增及产物的电泳检测参照Lee等(1993)和Gundersen等(1996)设计的植原体16S rRNA通用引物R16mF2/ R16mR1和R16F2n/ R16R2的序列合成引物。第1轮PCR以植株总DNA为模板,以R16mF2(5′-CAT GCA AGT CGA ACG GA-3′)和R16mR1(5′-CTT AAC CCC AAT CAT CGA C-3′)为引物。反应循环为:94 ℃预处理3 min;94 ℃ 30 s,55 ℃ 1 min,72 ℃ 1.5 min,共35个循环;最后72 ℃延伸10 min。第2轮PCR以第1轮PCR扩增产物的50倍稀释液为模板,以R16F2n (5′-GAA ACG ACT GCT AAG ACT GG-3′)和R16R2(5′-TGA CGG GCG GTG TGT ACA AAC CCC G-3′)为引物,退火温度提高至60 ℃,其余条件同第1轮PCR。PCR产物在0.8%琼脂糖凝胶中电泳检测。
1.4 PCR产物纯化,克隆及序列测定PCR产物的纯化采用Mini-DNA Rapid Purification Kit,具体操作参照说明书进行。纯化后的PCR产物与pMD18-T Easy Vector于4 ℃连接过夜。将连接产物加到100 μL DH5α感受态细胞中,冰浴30 min,42 ℃水浴热击90 s,冰浴2 min,加入800 μl LB液体培养基,37 ℃ 220 r·min-1轻摇45 min。菌液经8 000 r·min-1离心2 min,留沉淀及少量上清,混匀,涂布于含50 mg·mL-1氨苄青霉素的LB平板上, 倒置培养皿于37 ℃培养12~16 h。挑选筛选平板上的白色菌落到LB液体培养基中振荡培养,采用碱裂解法提取少量重组质粒DNA,经菌落PCR和电泳检测鉴定后,选含有正确插入片段的重组质粒,送北京博尚生物技术有限公司进行序列测定。
1.5 16S rRNA序列分析利用DNAStar和MEGA 4.0软件比较所得序列与其他植原体16S rRNA序列的一致率,并构建系统进化树(Gao et al., 2008)。
2 结果与分析 2.1 感病植株的症状表现丛枝症状先从荆条的个别枝条上出现。发病枝条叶片变小,颜色变浅或黄化,枝条节间缩短,分枝变细,密集丛生,形成典型的丛枝症状(图版Ⅰ-A,B)。病株矮化明显,多年生的枝条上有顶枯症状。

|
图版Ⅰ Plate Ⅰ |
对表现丛枝的荆条嫩茎进行超薄切片、染色后在电镜下观察,结果在其韧皮部筛管细胞中发现典型的植原体细胞(图版Ⅰ-C,D),大小在400~800 nm,主要形态为有圆形、椭圆形、哑铃形等,均具有明显的单位膜结构。在健康植株韧皮部筛管中未观察到植原体细胞。
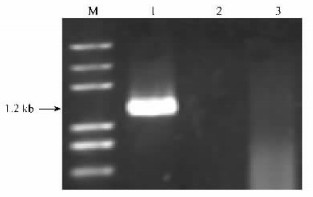
2.3 植原体的PCR检测利用巢式PCR技术从表现丛枝症状的荆条总DNA中扩增到了约1.2 kb的片段,与预期大小相符,而健康植株和双蒸水对照中均未扩增出片段(图 1),这说明该病的病原是植原体。将此植原体命名为荆条丛枝病植原体(chaste tree witches'-broom phytoplasma,CTWB)。
|
图 1 荆条丛枝植原体16S rRNA基因PCR扩增电泳图 Figure 1 PCR amplification of 16S rRNA gene of phytoplasma associated with chaste tree witches'- broom M: DNA分子量标准V DNA marker DL2000; 1:病株Diseased chaste tree; 2:双蒸水作为空白对照Double distilled water as blank control; 3:健康植株DNA from healthy chaste tree as negative control. |
通过测定来自2个PCR反应的各1个克隆的序列获得了CTWB 16S rRNA基因的核苷酸序列(EU709819)。该基因含有1 243个核苷酸,G+C含量为46.39%。CTWB与16S rI组植原体16S rRNA基因的序列一致率均在99.0%以上,而与其他组植原体的一致率低于97.0%(表 1)。CTWB与玉米丛矮植原体(Maize b ushy stunt,序列号AY265208)16S rRNA基因的序列一致率最高,达到99.8%,2者仅在第77,324,445位核苷酸有差异,分别是G-T,T-C,A-G的碱基替换。
|
|
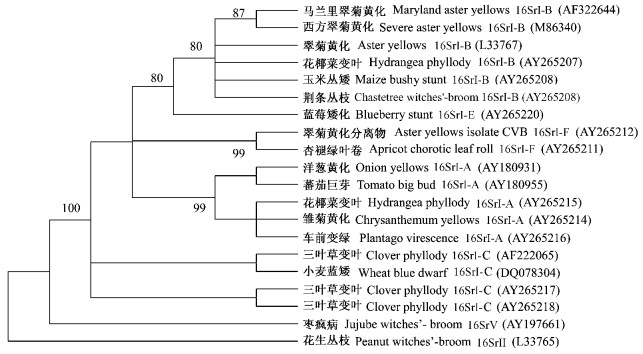
在根据16S rRNA基因序列构建的系统发育进化树(图 2)中,CTWB与翠菊黄化组(16S rI组)的植原体聚为1群,表明CTWB为16S rI组的成员。而且,CTWB与玉米丛矮、马里兰翠菊黄化等16S rI-B亚组的植原体形成1个共同的分枝,说明该植原体属于16S rI-B亚组。
|
图 2 利用邻接法构建的基于16S rRNA序列的系统发生树 Figure 2 Phylogenetic tree based on 16S rRNA sequences of chaste tree witches'- broom and related phytoplasmas constructed with the Neighbor-Joining method |
综合16SrRNA基因序列一致率和系统进化树分析的结果,CTWB属于16S rI-B亚组。
3 结论与讨论本研究通过电镜观察和PCR检测证实了荆条丛枝病是由植原体引起的,并根据16S rRNA基因序列一致率和系统进化树分析结果,证明该植原体属于翠菊黄化组(16SrI)的B亚组,明确其分类地位。
迄今为止,荆条多处于半野生状态,关于荆条病害发生的报道很少。本研究是关于荆条植原体病害的首次报道。荆条丛枝病是泰山周围近年出现的一种新病害,但发生非常普遍,局部地区发病率达30%。该病害在山东其他地区和和河南等地也有发生,严重影响我国的林业生产和环境改善。鉴于荆条在我国分布广泛,具有重要的药用和经济价值,对防止水土流失和防风固沙具有重要的生态意义,需要尽快研究明确其昆虫介体种类和传播特点、发生规律和防治措施等。
目前已报道了700多种植物自然感染植原体病害(Lee et al., 2000),而新的植原体病害种类还在不断出现(Parada et al., 2006;Rott et al., 2007;Arocha et al., 2008;Fránová et al., 2008;Kamińska et al., 2008)。我国报道的植原体病害有70多种(李永等,2005)。目前,枣疯病、泡桐丛枝病和小麦蓝矮病等植原体病害在我国的的发生危害依然非常严重,新病害也不断被发现(安凤秋,2006;蔡红,2007)。绣线菊丛枝病(高瑞等,2007)、玫瑰丛枝病(Gao et al., 2008)、榆树黄化病(朱天生等,2008)等在中国的发生,还发现了樱桃绿瓣、石榴带化、国槐带化和榆树丛枝等植原体病害(高瑞,2008)。因此,需要密切关注植原体病害,尤其是新病害或新病害记录在我国的发生情况,尽快制定合适的控制策略和方法,以减轻它们对我国农业和林业生产可能造成的危害。
蔡红. 2007. 云南植原体株系及相关病害的多样性研究. 云南农业大学植物保护学院博士学位论文.
|
车海彦, 罗大全. 2006. 植原体病害的检测方法研究进展. 华南热带农业大学学报, 12(3): 69-73. DOI:10.3969/j.issn.1674-7054.2006.03.016 |
高瑞. 2008. 山东省六种植原体病害的分子检测和鉴定. 山东农业大学植物保护学院硕士学位论文. http://cdmd.cnki.com.cn/Article/CDMD-10434-2008206719.htm
|
高瑞, 李向东, 王洁, 等. 2007. 绣线菊丛枝病病原的分子鉴定. 林业科学, 43(11): 72-75. |
李永, 田国忠, 朴春根, 等. 2005. 我国几种植物植原体的快速分子鉴别与鉴定的研究. 植物病理学报, 35(4): 293-299. DOI:10.3321/j.issn:0412-0914.2005.04.002 |
漆艳香, 谢艺贤, 张辉强, 等. 2004. 植原体DNA提取方法的改良. 生物技术通报, 4: 44-46. |
朱天生, 潘一展, 崔廷涛, 等. 2008. 榆树黄化病枝原体的分子检测与鉴定. 植物病理学报, 38(4): 401-406. DOI:10.3321/j.issn:0412-0914.2008.04.011 |
Abou-Jawdah Y, Karakashian A, Sobh H, et al. 2002. An epidemic of almond witches'-broom in Lebanon: classification and phylogenetic relationships of the associated phytoplasma. Plant Disease, 86(5): 477-484. DOI:10.1094/PDIS.2002.86.5.477 |
Arocha Y, Bekele B, Tadesse D, et al. 2008. First report of a 16SrII group phytoplasma associated with die-back diseases of papaya and citrus in Ethiopia. Plant Pathology, 57(2): 1039. |
Fránová J, Pibylová J, Petrzik K. 2009. Purple coneflower with reddening and phyllody: a new host of clover phyllody phytoplasma. European Journal of Plant Pathology, 123: 85-90. DOI:10.1007/s10658-008-9320-3 |
Gao R, Zhang G M, Lan Y F, et al. 2008. Molecular characterization of phytoplasma associated with rose witches'-broom in China. Journal of Phytopathology, 156(2): 93-98. DOI:10.1111/jph.2008.156.issue-2 |
Gundersen D E, Lee I M. 1996. Ultrasensitive detection of phytoplasmas by nested-PCR assays using two universal primer pairs. Phytopathologia Mediterranea, 35: 144-151. |
Harrison N A, Womack M, Carpio M L. 2002. Detection and characterization of a lethal yellowing (16SrIV) group phytoplasma in Canary Island date palms affected by lethal decline in Texas. Plant Disease, 86(6): 676-681. DOI:10.1094/PDIS.2002.86.6.676 |
Kamińska M, Śliwa H. 2008. First report of 'Candidatus Phytoplasma mali' in oriental lilies and its association with leaf scorch in Poland. Plant Pathology, 57(2): 363. |
Lee I M, Davis R E, Gundersen-Rindal D E. 2000. Phytoplasma: phytopathogenic mollicutes. Annual Review of Microbiology, 54: 221-255. DOI:10.1146/annurev.micro.54.1.221 |
Lee I M, Gundersen-Rindal D E, Davis R E, et al. 1998. Revised classification scheme of phytoplasmas based on RFLP analyses of 16S rRNA and ribosomal protein gene sequences. International Journal of Systematic Bacteriology, 48: 1153-1169. DOI:10.1099/00207713-48-4-1153 |
Lee I M, Hammond R W, Davis R E, et al. 1993. Universal amplification and analysis of pathogen 16S rRNA for classification and identification of mycoplasma-like organisms. Phytopathology, 83(8): 834-842. DOI:10.1094/Phyto-83-834 |
Lee I M. 1993. Use of mycoplasmalike organisms (MLO) group specific oligonucleotide primers for nested-PCR assays to detect mixed-MLO infections in a single host plant. Phytopathology, 84(6): 559-566. |
Lim P O, Sears B B. 1989. 16S rRNA sequence indicates that plant-pathogenic mycoplasma-like organisms are evolutionarily distinct from animal mycoplasmas. Journal of Bacteriology, 171: 5901-5906. DOI:10.1128/jb.171.11.5901-5906.1989 |
Parada R Y, Castro S, de Serrano R, et al. 2006. First report of a phytoplasma associated with Spondias purpurea (Jocote de Corona) in El Salvador. Journal of General Plant Pathology, 72: 40-42. DOI:10.1007/s10327-005-0241-0 |
Rott M, Johnson R, Masters C, et al. 2007. First report of bois noir phytoplasma in Grapevine in Canada. Plant Disease, 91(12): 1682. |
Schneider B, Gibb K S, Seemüller E. 1997. Sequence and RFLP analysis of the elongation factor Tu gene used in differentiation and classification of phytoplasmas. Microbiology, 143: 3381-3389. DOI:10.1099/00221287-143-10-3381 |
The IRPCM Phytoplasma/Spiroplasma Working Team. 2004. 'Candidatus Phytoplasma', a taxon for the wall-less, non-helical prokaryotes that colonize plant phloem and insects. International Journal of Systematic and Evolutionary Microbiology, 54: 1243-1255. DOI:10.1099/ijs.0.02854-0 |
Tian J B, Guo H P, Bertaccini A, et al. 2000. Molecular detection of jujube witches' broom phytoplasmas in micropropagated jujube shoots. Hortscience, 35(7): 1274-1275. |
Wei W, Davis R E, Lee I M, et al. 2007. Computer-simulated RFLP analysis of 16S rRNA genes: identification of ten new phytoplasma groups. International Journal of Systematic and Evolutionary Microbiology, 57: 1855-1867. DOI:10.1099/ijs.0.65000-0 |
 2009, Vol. 45
2009, Vol. 45