文章信息
- 程丽莉, 黄武刚, 周志军, 刘建立, 王艳梅, 苏淑钗, 翟明普.
- Cheng Lili, Huang Wugang, Zhou Zhijun, Liu Jianli, Wang Yanmei, Su Shuchai, Zhai Mingpu
- 欧洲榛微卫星对我国榛属种质资源的分析
- Genetic Diversity of Six Corylus Species in China Detected with Microsatellite Isolated from Corylus avellana
- 林业科学, 2009, 45(2): 22-26.
- Scientia Silvae Sinicae, 2009, 45(2): 22-26.
-
文章历史
- 收稿日期:2008-02-01
-
作者相关文章
2. 北京市昌平林业局 北京 102200;
3. 北京林业大学 北京 100083
2. Changping District Forest Department Beijing 102200;
3. Beijing Forestry University Beijing 100083
榛子是世界四大坚果之一,以其独特的风味和丰富的营养深受消费者喜爱,发展前景广阔。然而,目前世界上只有4个相当局限的地区(土耳其、意大利、西班牙及美国)存在商业性欧洲榛(Corylus avellana)的生产(麦克丹尼尔斯, 1989)。我国的榛育种工作已经持续了近40年,在此期间,通过平榛(Corylus heterophylla)与欧洲榛种间远缘杂交及野生平榛实生选优,已获得了多个抗寒、大果型的榛品种(张宇和等,2005)。鉴于平欧榛种间杂交仍然是我国未来榛子育种的主要手段,探讨欧榛SSR在中国榛属植物中应用的可行性将对我国榛属遗传资源研究、农业性状分析、品种DNA指纹鉴别以及辅助育种具有重要作用。
简单重复序列(simple sequence repeat, SSR),也称微卫星(microsatellite)DNA或短串联重复序列(short tandem repeat, STR)(Amos et al., 1996; Debrauwer et al., 1997),广泛存在于真核基因组中(Toch et al., 2000)。作为共显性分子标记,SSR因具有多态性高、试验操作简单、稳定重复性好、DNA用量少等优点,很快地从多种分子标记中脱颖而出,目前已广泛应用于欧洲榛的基因型鉴定(Galderisi et al., 1999)、遗传图谱构建(Mehlenbacher et al., 2006)、目的基因定位(Pomper et al., 1998)等领域。
本研究利用欧洲榛微卫星引物(Bassil et al., 2005a; Gökirmak et al., 2005)扩增我国榛属6个种的样本,明确欧洲榛微卫星用于我国榛属植物遗传多样性研究的可行性,并在此基础上对微卫星位点在榛属种间的多态性进行了初步分析。
1 材料与方法 1.1 试验材料及来源于春季枝条萌发后野外采集榛属植物叶片(表 1),带回实验室后用蒸馏水冲洗干净,在滤纸上吸干水分,液氮速冻处理,放入-80 ℃冰箱保存。
|
|
采用CTAB法提取高质量的榛子基因组DNA(王艳梅等,2007)。在1%琼脂糖凝胶电泳(北京六一仪器厂,DYY-Ⅲ型)检测DNA的完整性,用紫外分光光度计(Eppendorf Biophotometer 6131)测定DNA的浓度和纯度。
1.2.2 引物合成及来源引物从已公布的欧洲榛SSR引物(Bassil et al., 2005a; Gökirmak et al., 2005)中筛选,引物序列及目标片段特征见表 2,由上海生物工程技术服务有限公司(Sangon)合成。
|
|
SSR扩增在Biometra公司的T1 Theromcycler PCR仪上进行。25 μL反应体系含有:10 mmol·L-1Tris-HCl(pH 8.3),1.5 mmol·L-1MgCl2,0.2 mmol·L-1dNTPs,0.2 μmol·L-1引物,1.0 U Taq DNA聚合酶,50~100 ng的模板DNA(上述药品均来自华美生物工程公司)。扩增程序为:94 ℃预变性3 min,94 ℃变性30 s,51~56 ℃复性40 s,72 ℃延伸40 s,共40个循环,最后在72 ℃延伸8 min。
1.2.4 扩增产物银染检测扩增产物用聚丙烯酰胺凝胶电泳检测。在扩增产物中加入等体积的变性载样缓冲液(98%甲酰胺,10 mmol·L-1 EDTA,0.025%溴酚蓝和二甲苯青兰),混匀,95 ℃变性5 min后,立即放入冰中,上样量为5 μL,6%变性聚丙烯酰胺凝胶电泳,60 W电泳2.5 h,银染染色。染色方法:乙醇固定5 min,双蒸水漂洗60 s,硝酸氧化5 min,双蒸水漂洗60 s,硝酸银中染色30 min,双蒸水漂洗60 s,碳酸钠+ 400 μL·L-1甲醛+ 0.2 mg·L-1硫代硫酸钠溶液中显色,显色后用10%乙酸固定。1.2.5数据分析读取电泳图谱上微卫星位点的等位基因,获得共显性标记数据矩阵,应用Cervus2.0软件计算等位基因的频率。
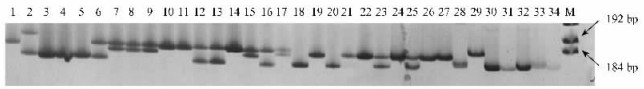
2 结果与分析 2.1 微卫星属内扩增同源性及有效性随机选用6个种的试材对已发表的18个SSR引物进行筛选,结果表明所有引物在目标区域内都能得到扩增产物(图 1),扩增片段大小在124~234 bp之间,但不同引物的扩增效率及清晰度有所差异。经过对比分析,选取扩增条带清晰、重复性好,且多态性高的11个引物对我国榛属植物进行SSR分析。
|
图 1 引物CAC CAT6扩增结果 Figure 1 PCR amplification and electrophoresis of the SSR CaDCAT6 in 6 Corylus species 1.藏刺榛Corylus ferox var.thibetica; 2~3.滇榛C. yunnanensis; 4.华榛C.chinensis; 5~15.川榛C. heterophylla var. sutchuenensis; 16~27.毛榛C. mandshurica; 28~34.平榛C. heterophylla. |
为了确定微卫星的同源性,对PCR产物进行了克隆测序。测序结果表明从6个种得到的PCR产物中都含有重复序列,表现出长度单位的变化,微卫星侧翼区域高度保守,仅有藏刺榛在CACC28位点与榛属其他5个种在41 bp处显示出较大差异,多出了一个GCT(AG)2序列,增加的碱基与邻近(AG)2形成(AG)2GCT(AG)2短重复序列(图 2);同时在该位点还发现(GAA)n序列附近存在着另外一个(CTG)n重复序列,该微卫星在榛属种间和种内n的变化在3~4重复单位之间,表现出长度的多态性,而研究者没有给出相关的报道(Bassil et al., 2005b)。

|
图 2 引物CAC C28扩增产物的序列图 Figure 2 Aligment of nucleotide sequences by the primer pair of CAC C28 |
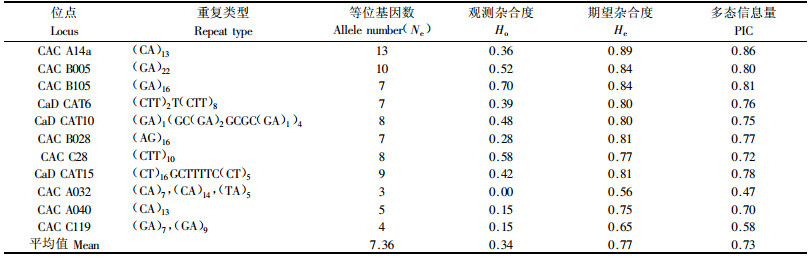
11个引物在榛属6个种的34样本中共扩出81个等位基因,不同基因座位拥有的等位基因数量差异较大,在4~13之间,平均每位点的等位基因数目为7.36个。不同位点的PIC(polymorphism informative content)值在0.47~0.86之间,平均PIC值为0.73 (表 3),充分说明了榛属植物遗传背景的复杂性和多样性。微卫星本身的重复单位数的变异是形成微卫星标记多态性的基础,具有长重复序列的位点给出较多等位基因数。这些数据表明在不同微卫星位点上,榛属植物表现出较高的多态性水平,利用SSR分子标记方法是可以检测榛属植物的种间亲缘关系及多样性水平的(李淑娴等,2003)。
|
|
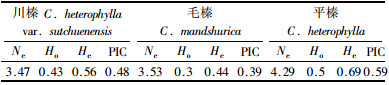
对于具有商业潜力的平榛、毛榛、川榛样本的遗传多样性分析(表 4)表明:平榛的遗传多样性最高(0.59),其次为川榛(0.48),毛榛最低(0.39),3个种的遗传多样性均低于从欧榛品种中得到的结果(Bassil et al., 2005a)。
|
|
18个欧洲榛微卫星引物成功地跨种扩增表明上述基因座在榛属种间具有高度的保守性。从微卫星侧翼得到的引物可跨植物的种、属甚至科进行扩增已多有报道(Guilford et al., 1997; Huang et al., 1998),周涵韬等(2002)曾经利用2对甘蓝型油菜(Brassica napus)微卫星引物对禾本科(Gramineae)、十字花科(Cruciferae)、芸香科(Rutaceae)和锦葵科(Malvaceae)作物的DNA进行扩增并得到扩增产物。然而,微卫星侧翼的保守性并不能保证扩增产物具有重复序列,因为SSRs是一种复杂的位点,受很多不同突变或进化压力的影响,对PCR产物的杂交或DNA测序成为进行种间SSR分析先期检验条件(Huang et al., 1998; Bassil et al., 2005a)。本研究中对PCR产物DNA测序证明了欧洲榛SSR基因座位中微卫星序列在我国榛属6个种内的保守性,以及等位基因在电泳中的大小与重复单位的增减相关。藏刺榛在形态学特征及生境上均明显地区别于榛属其他种(匡可任等,1979),而在CAC C28位点41 bp处多出的GCT(AG)2序列显示出藏刺榛与榛属其他种在DNA水平上的差异,然而该突变是否为刺榛组特有有待进一步研究。
11对欧洲榛微卫星引物都可从中国榛属植物中扩增出有效片段,位点拥有等位基因数量明显高于同工酶,而相对于RAPD标记(Miaja et al., 2001),其共显性的特性使其在研究榛野生资源遗传多样性和居群遗传结构方面具有更大的优势(贾继增,1996)。
微卫星的重复单位数的变异是微卫星标记多态性的基础,目前对微卫星位点的等位基因判别主要基于电泳后PCR产物大小的差异。在引物设计过程中,较短的重复序列(n≤4或5)更容易被忽略(Katti et al., 2001)。而关于引起三碱基重复序列发生滑动复制的最小重复单位或碱基数目,不同的研究给出不同的结果(Borstnik et al., 2002; Rocha et al., 2002; Ross et al., 2003)。本研究对CAC C28位点的分析测序结果表明具有重复单位的三碱基微卫星(CTG)3就可能产生长度的变化,这一结果较Lai等(2003)提出的产生滑动复制的最短三碱基重复序列小了1个拷贝,而支持Klevytska等(2001)对三碱基微卫星的最小重复数量的划分标准。对于短重复序列多态性的忽视,导致在分析CAC C28位点的等位基因时产生偏差,偏差源于拥有同等长度片段的个体间含有不同拷贝数目的(CTG)n和(GAA)n微卫星(图 2),进而导致在遗传多样性分析中的数据模糊。因此,在对目标微卫星进行引物设计时,应避开其附近的短重复序列,否则应考虑该重复序列可能产生长度变化。
大部分欧洲榛微卫星位点在我国榛属中拥有较多的等位基因,而在平榛、毛榛和川榛的样本中等位基因数量明显少于欧洲榛种内等位基因数量(Gökirmak et al., 2005; Bassil et al., 2005a)。有研究表明种群内等位基因数量受样本量的影响较大(闫路娜等,2004);Ellegren等(1995)认为微卫星应用于其他物种时,微卫星重复单位数量趋于减少,而短重复序列提供的信息较少;此外,本文的结果也反映了位点的等位基因数量在野生群体与受人类长期选择的品种群之间的差异。
物种自身的生物学特性在很大程度上影响其整个基因组的进化和遗传变异的格局(Hamrick et al., 1990)。在野生状态下,平榛、毛榛和川榛以根蘖繁殖为主,种子繁殖的植株较少(戚继忠等,1996),产生遗传变异机会较少,故这3个种的PIC值总体较低,而与取样数量无关;物种的分布范围也是影响遗传多样性水平的主要因子之一,自然分布范围广的物种通常比分布范围小的物种具有更高的遗传变异水平,本研究中平榛的分布范围最为广泛,且分布区域内生态条件多样,在长期的进化过程中,有可能形成丰富的遗传变异,微卫星标记分析显示其遗传变异水平明显高于其他2个种(表 4)。所以在遗传改良工作中,将平榛作为亲本培育适应其生境范围的杂种榛是我国榛子良种化的有效途径之一。可见多态位点百分比在一定程度上可以指导育种工作者,选择遗传基础较宽的群体作为育种材料,以获得差异较大的优良品种,为基因资源的保存和进一步利用打下良好基础。
通过种间杂交将欧洲榛的大果型和平榛较强的适应性结合在一起的育种实践,已使我国向榛生产品种化迈出坚实的一步。岳新丽等(2003)分别对平榛和欧榛的2个品种及平欧杂种榛的78个品种(系)的亲缘关系作了初步探测,结果表明,远缘杂交丰富了杂交种的遗传基础,杂种榛各品种(系)间均存在明显的遗传差异,说明杂种榛作为我国榛品种遗传改良的种质资源具有较高的价值。为了进一步培育出能适应我国不同自然环境的榛品种,利用欧洲榛与我国榛属中具商业潜力的种进行杂交将是一条主要的育种途径。欧洲榛微卫星引物在榛属种间的成功转移及位点在相关种内表现出的高多态性将为分子手段在榛子遗传育种实践中的进一步应用打下良好基础。
贾继增. 1996. 分子标记种质资源鉴定和分子标记育种. 中国科学, 29(4): 1-10. |
匡可任, 郑斯绪, 李沛琼, 等. 1979. 中国植物志:二十一卷. 北京: 科学出版社, 47-50.
|
麦克丹尼尔斯L H. 1989.坚果栽培.朱金兆, 查多禄, 魏康年, 译.北京: 中国林业出版社, 158-162.
|
戚继忠, 赫永福, 李克元, 等. 1996. 榛子种群空间格局及其动态研究. 吉林林学院学报, 12(2): 99-103. |
王艳梅, 程丽莉, 翟明普, 等. 2007. 中国榛属植物DNA提取与SSR初步分析. 河南师范大学学报, 35(2): 129-132. |
闫路娜, 张德兴. 2004. 种群微卫星DNA分析中样本量对各种遗传多样性度量指标的影响. 动物学报, 50(2): 279-290. |
岳新丽, 季兰. 2003.榛子遗传育种研究进展//郗荣庭.第三届全国干果生产与科研进展学术研讨会: 干果研究进展.北京: 中国农业科学技术出版社, 110-115. http://www.wanfangdata.com.cn/details/detail.do?_type=conference&id=4884663
|
张宇和, 柳鎏, 梁维坚, 等. 2005. 中国果树志:板栗榛子卷. 北京: 中国林业出版社, 194-198.
|
周涵韬, 郑文竹, 周以□, 等. 2002. 不同作物间共用SSR引物的初步研究. 厦门大学学报:自然科学版, 41(1): 89-91. |
Amos W, Rubinstzein D C. 1996. Microsatellites are subject to directional evolution. Nat Genet, 12(1): 13-14. DOI:10.1038/ng0196-13 |
Bassil N V, Botta R, Mehlenbacher S A. 2005a. Microsatellite markers in hazelnut: isolation, characterization, and cross-species amplification. J Amer Soc Hort Sci, 130(4): 543-549. |
Bassil N V, Botta R, Mehlenbacher S A. 2005b. Additional microsatellite markers of the European Hazelnut. Acta Horticulturae, 686: 105-110. |
Borstnik B, Pumpernik D. 2002. Tandem repeats in protein coding regions of primate genes. Genome Res, 12(6): 909-915. DOI:10.1101/gr.138802 |
Debrauwer E H, Gendrel C G, Lechat S, et al. 1997. Differences and similarities between various tandem repeat sequences: minisatellites and microsatellites. Biochimie, 79(9/10): 577-586. |
Ellegren H, Primmer C R, Sheldon B C. 1995. Microsatellite evolution: directionality or bias in locus selection. Nature Genet, 11: 360-362. DOI:10.1038/ng1295-360 |
Galderisi U, Cipollaro M, DI Bernardo G, et al. 1999. Identification of hazelnut (Corylus avellana) cultivars by RAPD analysis. Plant Cell Reports, 18: 652-655. DOI:10.1007/s002990050637 |
Gökirmak T, Mehlenbacher S A, Bassil N V. 2005. Investigation of genetic diversity among European hazelnut (Corylus avellana) cultivars using SSR markers. Acta Horticulturae, 686: 141-147. |
Guilford P, Prakash S, Zhu J M, et al. 1997. Microsatellites in Malus×domestica (apple): abundance, polymorphism and cultivar identification. Theor Appl Genet, 94: 249-254. DOI:10.1007/s001220050407 |
Hamrick J L, Godt M J W. 1990. Allozyme diversity in plant species//Brown A H D, Clegg M T, Kahler A L, et al. Plant population genetics, breeding and genetic resources. Sinauer, Sunderland: Massachusetts Press, 43-63.
|
Huang W G, Cipriani G, Morgante M, et al. 1998. Microsatellite DNA in Actinidia chinensis: isolation, characterization, and homology in related species. Theor Appl Genet, 97: 1269-1278. DOI:10.1007/s001220051019 |
Katti M V, Ranjekar P K, Grpta V D. 2001. Differential distribution of simple sequence repeats in eukaryotics genome sequences. Mol Biol Evol, 18: 1161-1167. DOI:10.1093/oxfordjournals.molbev.a003903 |
Klevytska A M, Price L B, Schupp J M, et al. 2001. Identification and characterization of variable-number tandem repeats in the Yersinia pestis genome. J Clin Microbiol, 39(9): 3179-3185. DOI:10.1128/JCM.39.9.3179-3185.2001 |
Lai Y L, Sun F Z. 2003. The relationship between microsatellite slippage mutation rate and the number of repeat units. Mol Bio Evol, 20: 2123-2131. DOI:10.1093/molbev/msg228 |
Mehlenbacher S A, Brown R N, Nouhra E R, et al. 2006. A genetic linkage map for hazelnut (Corylus avellana L.) based on RAPD and SSR markers. Genome, 49: 122-133. DOI:10.1139/g05-091 |
Miaja M L, Vallania R, Me C, et al. 2001. Varietal characterization in hazelnut by RAPD markers. Acta Horticulturae, 556: 247-250. |
Pomper K W, Azarenko A N, Bassil N V, et al. 1998. Identification of random amplified polymorphic DNA (RAPD) markers for self-incompatibility alleles in Corylus avellana L.. Theor Appl Genet, 97: 479-487. DOI:10.1007/s001220050920 |
Rocha E P, Matic I, Taddei F. 2002. Over-representation of repeats in stress response genes: A strategy to increase versatity under stressful conditions. Nucleic Acids Res, 30(9): 1886-1894. DOI:10.1093/nar/30.9.1886 |
Ross C L, Dyer K A, Erez T, et al. 2003. Rapid divergence of microsatellite abundance among species of Drosophila. Mol Biol Evol, 20(7): 1143-1157. DOI:10.1093/molbev/msg137 |
Toch G, Gaspari Z, Jurka J. 2000. Microsatellites in different eukaryotic genomes: survey and analysis. Genome Research, 10: 967-981. DOI:10.1101/gr.10.7.967 |
 2009, Vol. 45
2009, Vol. 45