文章信息
- 胥猛, 潘惠新, 张博, 王树东, 黄敏仁.
- Xu Meng, Pan Huixin, Zhang Bo, Wang Shudong, Huang Minren
- 林木遗传改良中的分子生物学研究进展
- Molecular Biology Applied in the Improvement Process of Forest Trees
- 林业科学, 2009, 45(1): 136-143.
- Scientia Silvae Sinicae, 2009, 45(1): 136-143.
-
文章历史
- 收稿日期:2008-01-08
-
作者相关文章
2. 国家林业局竹子研究开发中心 杭州 310012
2. China National Bamboo Research Center Hangzhou 310012
在过去几十年间,林木生物技术研究取得了重要进展,不仅有一批性状优良的转基因林木进入了田间试验、环境释放阶段,而且在遗传基因组学和关联遗传学方面的发展也有望对林木遗传改良带来新的突破。在未来10年内,世界木材需求量预计增长20%,而全球森林覆盖率以每年940万hm2的速度减少(Fenning et al., 2002)。伴随公众对天然林过度开发和生物多样性损失的持续关注(Wimp et al., 2004),各国政府和国际社会为推进森林资源可持续发展做出了不懈努力,但林木育种是一个缓慢的过程,林木生长周期长,许多重要经济性状仅在轮伐年龄才能得以完全有效地评定。同时,传统林木遗传改良策略主要考虑遗传增益,而杂交育种所获得的遗传增益随着育种世代的增加逐渐减少(卢孟柱,2003)。得益于对基因和功能关系的深入理解,人们可以通过2个主要的途径加速林木遗传改良:一是运用基因工程技术,将新的基因转入已经驯化的优良个体中达到种质创新的目的;其次是在育种程序中充分利用林木遗传基因组学和关联遗传学的研究成果,有效开发林木种质资源的遗传多样性(Boerjan,2005)。
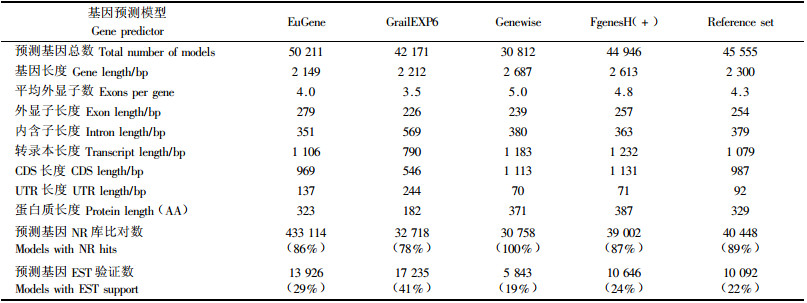
1 杨树全基因组测序计划林木独特的生物学特性,生长和休眠的季节性循环、幼年期与成熟期的发育变化、木材形成,以及对极端环境条件的适应性等,在1年生模式植物拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)中是难以进行深入研究的。杨树作为林木模式物种,生长迅速,无性繁殖容易,种间可杂交,组织培养和遗传转化技术较为成熟,并具有适中的基因组大小(Jansson et al., 2007; Groover,2005; Taylor,2002)。美国能源部基因组联合研究所(DOE Joint Genome Institute,JGI)与多家研究机构(DOE Oak Ridge National Laboratory; Ghent University; Belgium; GenomeCanada; Umea Plant Science Centre)于2002年启动了杨树全基因组测序计划,该计划以毛果杨(Populus trichocarpa)雌株无性系Nisqually-1为材料,采用全基因组鸟枪法(a whole-genome shotgun approach)进行全基因组测序。迄今,已进行了约为基因组全长8.6倍的基因组序列测定,完成了约480 Mbp的测序任务,基因组覆盖度达98.6%,预测到杨树基因组有4.5万多个可能的蛋白质编码基因(表 1)(Tuskan et al., 2006)。之前,科学家已经完成了拟南芥和水稻的基因组测序,杨树是第3个开展全基因组测序的高等植物,并且是第1个完成全基因组测序的多年生木本植物。
|
|
在系统分类学中杨树属于Rosids(蔷薇)分支中的EurosidⅠ(真蔷薇Ⅰ)单元,拟南芥属于Rosids分支中EurosidⅡ(真蔷薇Ⅱ)单元,在进化过程中2个物种距离较近。系统分类学地位有利于两物种进行比较生物学分析,基于杨树与拟南芥全基因组水平间的比较研究能揭示更多的物种进化与发育信息。在杨树基因组的4.5万多个蛋白质编码基因中,大约12%(5 248)的基因在拟南芥基因组中没有检索到相似性(E≤e×10-3),而拟南芥基因组中也有9%(2 321)的基因在杨树基因组中没有相似序列。通过BLAST双向比对发现,杨树和拟南芥基因组中有13 019对直系同源基因,平均相互覆盖度达93%,其中11 654对基因的序列一致性超过90%,仅有156对基因覆盖度低于50%。杨树比拟南芥有更多的蛋白质编码基因,相对于拟南芥每个基因平均有1.4~1.6个同源拷贝;然而,相关的蛋白质结构域频率在2个基因组中是相似的。杨树基因组中还存在丰富的诸如木纤维生物合成、分生组织发育、抗病以及代谢运输等相关基因。核酸替换、串连基因倍增和染色体重组在杨树基因组进化过程中持续发生(Tuskan et al., 2006)。通过对杨树基因组内8 000对旁系同源基因的分析,推测杨树基因组在进化过程中至少发生了3次基因组复制事件(Tuskan et al., 2006)。第1次复制事件发生于被子植物分化时期,而发生在1亿~1.2亿年前的第2次基因组加倍事件使杨树和拟南芥走上了不同的进化道路,最近一次基因组复制发生在距今6 000万~6 500万年之间,直接导致杨柳科的分化。基因组的复制为杨树提供了更多的基因拷贝,从而有利于进化出更多新的功能基因。
杨树全基因组序列信息也将为其他林木基因组研究提供有价值的参考,使依据林木功能基因进化上的保守性及相似性发现并鉴定顺式调控元件成为可能。全基因组序列间的比较与各种分子遗传学方法相结合将有力地促进比较生物学的发展(Jansson et al., 2007)。林木基因组学研究的根本动力在于认识林木的多样性和环境适应性,探索森林生产力的调控机制(Brunner et al., 2004)。目前,植物生长发育和抗逆等相关基因的基本生物学功能已经在拟南芥、水稻等1年生模式植物中进行了精确、有效的分析研究。因此,对于林木分子生物学研究而言,其主要挑战是如何针对多年生林木物种独特的解剖学、生理学及遗传学属性设计科学可行的试验方案,选择适宜的试验材料,从而在分子水平上深入剖析林木生长和发育规律。杨树全基因组测序的完成,不仅是对多年生木本植物基因组的首次破译,也是林木分子生物学研究的“一个全新科学时代的开端”。
2 林木分子生物学资源在杨树全基因组测序计划进行的同时,林木EST测序计划的开展也极大地促进了人们对林木基因组信息的洞悉。由瑞典皇家科学院(Royal Institute of Technology,Sweden)推动的杨树EST测序项目(The Swedish Populus Genome Project)已经获得了一系列基因突变的杨树无性系和杨树不同发育时期的cDNA微阵列,从中得到超过20万条EST序列,已经拼接了约2.5万条单一基因(unigene),其中包含了1 000条全长cDNA序列(Brunner et al., 2004;Bhalerao et al., 2003a; Hertzberg et al., 2001)。加拿大哥伦比亚杨树基因组项目(Genome British Columbia)分别构建了均一化的cDNA文库(normalized cDNA libraries)和富集cDNA文库(enriched cDNA libraries),获得了10万条EST序列和5 000条全长cDNA信息。此外,EST测序计划也在其他几个树种中开展,如桦树(Betula spp.)、松树(Pinus ssp.)(Kirst et al., 2003)、桉树(Eucalyptus ssp.)(Paux et al., 2004)、云杉(Picea spp.)、栎树(Quercus spp.)和刺槐(Robinia pseudoacacia)。在林木EST测序和基因组测序开展的同时,众多林木功能基因组学研究工具如DNA微阵列已经发展起来。这些分子生物学资源包括,瑞典开发的含有2.5万个点的cDNA微阵列,法国研制的2.7万个点的商业化PICME cDNA微阵列(http://www.picme.at/),以及加拿大开发的15 400个点的Treenomix cDNA微阵列(Ralph et al., 2006)。随着杨树基因组序列的公布,杨树寡核苷酸全基因组微阵列已经或正在被Affymetix、NimbleGen和Operon等公司商业化开发。
基因组序列信息和基因表达谱芯片技术是深入开展林木功能基因组研究不可或缺的资源,然而对林木表型性状和生物学过程的全面分析仍离不开代谢组和蛋白组水平的研究。当前,对林木代谢组学还知之甚少,仅少量文献报道了林木的代谢谱(Jonsson et al., 2004; Morreel et al., 2004a;2004b; Morris et al., 2004)。林木蛋白组学研究也刚刚起步(Plomion et al., 2003;2004)。有关海岸松(Pinus pinaster)木材形成的蛋白质组学研究已见报道(Plomion et al., 2004),该研究揭示了175个已知功能蛋白质的特性。林木蛋白质组学研究正处于其初期,但来源于杨树基因组序列的45 555个基因模型的注释将有助于质谱数据的蛋白质预测。同时,遗传工程在林木遗传改良中已初见成效。杨树较小的基因组及其较高的转化效率有利于构建基因陷阱、增强子陷阱和激活标签文库(Groover et al., 2004),这将便利和加速林木基因功能研究。Busov等(2003)通过在矮化激活的株系中过量表达赤酶素氧化酶,成功分离鉴定了首个林木发育调控相关基因的激活标签。另外,转座子标签也运用在林木基因功能研究中(Fladung et al., 2004)。这些技术手段有力地拓展了林木分子生物学研究的深度和广度。伴随现代分子生物学的深入发展和林木基因组计划的全面实施,日益丰富的林木分子生物学资源将为林木独特生物学特性的分子基础研究提供技术条件。
3 林木典型生长发育现象的分子基础林木典型生长发育性状,既是科学理论研究的热门话题,又是再生资源可持续开发利用的焦点。探索这些生物学性状的分子基础,将加速其生产应用。当前,微阵列和反向遗传学技术在林木分子生物学研究领域得到广泛运用,林木独特生物性状的分子基础研究取得一定的进展,转基因林木的改良机理也得到深入研究。从而加速了基因工程在林木遗传改良中的应用,搭建了林木分子育种发展平台。林木基因工程在可持续林业经济中彰显出独特魅力,大大缩短林木育种周期,强化林业经营的可预见性,创造出用常规遗传育种方法难以产生的新种质、新材料,为规模化种植和产业化开发创造了条件。
3.1 次生生长与应力材的形成植物分子生物学的发展主要以草本模式植物为研究对象,在分子水平上研究植物的生长、发育、繁殖、抗逆等生命过程。但构成木材的次生维管组织为木本植物所特有,成为林木分子生物学的主要研究内容之一。林木生物学家迫切需要基于草本模式生物的研究基础,以木本植物为研究对象,探索林木次生维管系统形成和发育的分子基础,挖掘控制维管形成层活动的关键基因,以最终实现人为调控这些基因的表达,改良树木材性。木材形成取决于维管形成层活动,受控于生长季节的长短。越来越多的研究运用基因表达谱技术探索杨树维管束形成层的活动,聚焦木材形成的分子机理。传统的组织解剖学与现代的DNA微阵列技术结合,构建了研究木材形成机理的良好平台,揭示了形成层区6个解剖学上相似的细胞层在转录水平上存在显著的基因表达差异(Uggla et al., 2002)。同时相关研究也表明,在木质部和韧皮部分化过程中存在各自的阶段性标记基因,形成层的活动受关键基因调控(Schrader et al., 2004b)。林木次生生长是一个复杂的生理发育过程,也涉及对环境信号的响应。应力材的形成也同样受到越来越多的关注(Pilate et al., 2004; Plomion et al., 2003)。当树干倾斜,木本被子植物在倾斜树干上方形成应拉木,反之木本裸子植物在其下方形成应压木。这样的木材组织在解剖学和化学组成上都存在差异,给木材加工工艺带来麻烦。在应压木形成过程中,一类上调表达基因编码阿拉伯半乳聚糖蛋白,这些基因可能在应拉木典型次生细胞壁凝胶层(G层)的形成中起到特殊作用(Déjardin et al., 2004; Lafarguette et al., 2004)。在杨树、松树和桉树的相关研究中,转录和蛋白质谱也鉴定出许多与次生生长和环境胁迫相关的基因,这些基因在早晚材形成(Le Provost et al., 2003; Egertsdotter et al., 2004)、幼龄材与成熟材发育(Le Provost et al., 2003)、秋叶衰老(Bhalerao et al., 2003b; Andersson et al., 2004)、木质部分化(Lorenz et al., 2002)、不定根发生(Kohler et al., 2003)、创伤和病害感染(Smith et al., 2004)、水分胁迫(Dubos et al., 2003)、低温胁迫(Renaut et al., 2004),以及外生菌根共生(Martin et al., 2004)等过程中有显著的表达变化。这些研究均集中于“野生型”林木的生长发育性状,可以预见遗传修饰过的转基因树木也是研究相关性状的常规材料。进行转基因植株的转录谱分析,也是鉴定相关改良特性基因调控的有效方式(Israelsson et al., 2003)。此外,一年生模式植物拟南芥,在短日照条件下也能从维管束形成层产生一定的次生木质部,也是研究木材形成分子机理的较好系统(Chaffey et al., 2002; Ko et al., 2004)。
植物分子生物学的发展主要以草本模式植物为研究对象,在分子水平上研究植物的生长、发育、繁殖、抗逆等生命过程。但构成木材的次生维管组织为木本植物所特有,成为林木分子生物学的主要研究内容之一。林木生物学家迫切需要基于草本模式生物的研究基础,以木本植物为研究对象,探索林木次生维管系统形成和发育的分子基础,挖掘控制维管形成层活动的关键基因,以最终实现人为调控这些基因的表达,改良树木材性。木材形成取决于维管形成层活动,受控于生长季节的长短。越来越多的研究运用基因表达谱技术探索杨树维管束形成层的活动,聚焦木材形成的分子机理。传统的组织解剖学与现代的DNA微阵列技术结合,构建了研究木材形成机理的良好平台,揭示了形成层区6个解剖学上相似的细胞层在转录水平上存在显著的基因表达差异(Uggla et al., 2002)。同时相关研究也表明,在木质部和韧皮部分化过程中存在各自的阶段性标记基因,形成层的活动受关键基因调控(Schrader et al., 2004b)。林木次生生长是一个复杂的生理发育过程,也涉及对环境信号的响应。应力材的形成也同样受到越来越多的关注(Pilate et al., 2004; Plomion et al., 2003)。当树干倾斜,木本被子植物在倾斜树干上方形成应拉木,反之木本裸子植物在其下方形成应压木。这样的木材组织在解剖学和化学组成上都存在差异,给木材加工工艺带来麻烦。在应压木形成过程中,一类上调表达基因编码阿拉伯半乳聚糖蛋白,这些基因可能在应拉木典型次生细胞壁凝胶层(G层)的形成中起到特殊作用(Déjardin et al., 2004; Lafarguette et al., 2004)。在杨树、松树和桉树的相关研究中,转录和蛋白质谱也鉴定出许多与次生生长和环境胁迫相关的基因,这些基因在早晚材形成(Le Provost et al., 2003; Egertsdotter et al., 2004)、幼龄材与成熟材发育(Le Provost et al., 2003)、秋叶衰老(Bhalerao et al., 2003b; Andersson et al., 2004)、木质部分化(Lorenz et al., 2002)、不定根发生(Kohler et al., 2003)、创伤和病害感染(Smith et al., 2004)、水分胁迫(Dubos et al., 2003)、低温胁迫(Renaut et al., 2004),以及外生菌根共生(Martin et al., 2004)等过程中有显著的表达变化。这些研究均集中于“野生型”林木的生长发育性状,可以预见遗传修饰过的转基因树木也是研究相关性状的常规材料。进行转基因植株的转录谱分析,也是鉴定相关改良特性基因调控的有效方式(Israelsson et al., 2003)。此外,一年生模式植物拟南芥,在短日照条件下也能从维管束形成层产生一定的次生木质部,也是研究木材形成分子机理的较好系统(Chaffey et al., 2002; Ko et al., 2004)。
3.2 林木开花调控机制与季节性休眠林木在进入生殖生长(第一次开花)之前的幼年期的生长发育可持续若干年甚至几十年,控制多年延迟开花的分子机制有待进一步深入研究。杨树FT基因(PtFT1)的表达机制可能是杨树开花调控的重要决定因素(Bohlenius et al., 2006),雄株杂种山杨(Populus tremula×Populus tremuloides)转化株35S::PtFT1经过4周的生长发育就能直接形成类似花的结构,而正常植株开花要8~20年。研究表明,杨树PtFT1基因同时涉及调控林木的开花延迟、秋季的驻芽形成和生长抑制。在进入生殖生长的成年树木中,PtFT1的表达受光周期控制继而调控树木生长发育,即春季和早夏长日照诱导开花,秋季短日照促进驻芽形成(bud set)和抑制生长(Bohlenius et al., 2006)。林木CO/FT基因通过响应光周期的变化而调控林木生长发育的现象,揭示了林木在生态和进化上的“权衡”机制,即多数林木是如何在生存和生长之间做出权衡调控的(Weiser,1970; Horvath et al., 2003; Howe et al., 2003),这也是多年生林木物种适应新的地理环境和当前气候变化的关键。关于这一机制的了解将对今后的林木育种计划起着重要的作用,诱导早期开花的技术也使得加速林木育种进程成为可能。生长和休眠的季节性循环是林木生物学的又一个重要方面,林木生物学家们正致力于揭示其分子机制。在林木中发现的CO/FT调控机制,揭示了植物“休眠”的本质,以便重新认识这种季节性和周期性的生理活动。开花时间和分生组织发育方向是区别1年生、2年生和多年生生命形式的重要标志。多年生林木物种,与其他植物的关键区别在于它们具有响应环境信号而中止或恢复生长发育的能力,并且这种能力是季节性和条件性的。植物的生长发育在很大程度上决定于分生组织的活动。分生组织分化活动影响着植物营养生长和开花特性,而这些都是植物界多样性生活策略的组成部分。多年生植物首先至少具备一个“不确定”的分生组织,以便在后续的生长季中恢复生长。而这种多年生植物的“不确定”分生组织已经被广泛认知(Battey,2000; Thomas,2000),该分生组织具备在非最适季节生存的能力。在生存的不同层面,休眠是多年生植物生长发育的关键策略之一。在生长季节末,受光周期变化,休眠施加于所有的分生组织。通过对形成层分生组织的基因表达谱分析,发现在休眠过程中形成层的转录组发生了广泛的表达变化,同时鉴定出了部分影响相关活动的调控基因(Schrader et al., 2004a)。顶端分生组织的休眠感应是通过芽的形成诱导的。PtABI3是在芽发育中的一个转录因子(Rohde et al., 2002),在临界日照条件展叶过程中临时表达。反向遗传学实验也表明这个转录因子在芽形成的相关发育阶段扮演重要角色。受益于多年生林木开花调控模式CO/FT的发现,休眠机制研究有了突破性的进展。研究证实,杨树中CO/FT模式在短日照条件下调控树木休眠。当毛果杨的FT1和CO基因在转基因的杂种山杨中过量表达时,植株在短日照条件下并不会停止生长(Bohlenius et al., 2006)。通过RNAi技术抑制PtFT1基因的表达后导致生长停止和驻芽形成,此时已经不再依赖于日照长度。很明显FT是一种多重调节因子,响应环境信号调控分生组织的转换,如控制分生组织在花器官与驻芽间的定向分化(Rohde et al., 2007)。
3.3 转基因林木改良特性林木遗传工程仍是加速林木改良的一个重要途径。其中一个主要的优势在于通过遗传工程手段定向改造后的优良性状能被稳定保持。当前,在遗传工程中大量的工作致力于改良树木中木质素的含量和组分以强化纸浆工艺过程中木质素的分离(Baucher et al., 2003)。这些转基因树木能提高纸浆的生产量,从而减少生产成本,更重要的是减少纸浆工艺对环境的生态冲击。F5H在杨树中的过量表达降低木质素含量,改善了木质素分离和漂白工艺,而纤维质量仍保持不变或更好。相似地,CAD在木质素单体生物合成中有重要功能,抑制CAD的表达能显著改善木材的质量。从来源于转基因树木的纸浆中除去木质素只需要极少的化学品,且得到了较高的纸浆产量(Pilate et al., 2002)。通过用2个载体共转化,一个旨在增加F5H的表达,另一个用于减少4-coumarate CoA-ligase的表达——得到预期的结合效应:木质素量减少,纤维素增加。这项研究表明,通过堆垛转基因(stacking transgenes)策略,针对一个基因型同时进行多个性状的定向改良是可行的(Halpin et al., 2003)。除质量性状之外,产量性状也是基因工程改造的潜在对象,杨树中几个与产量性状相关的基因已经显示出其应用前景。GS是一个涉及氮同化的关键性酶,通过对3年生转基因杨树的测定,GS过量表达植株高增加41%,树干增粗36%(Jing et al., 2004)。拟南芥内源葡聚糖酶CEL1基因在杨树中的过量表达也能显著增加树高、叶面积、树干直径和纤维素/半纤维素含量(Shani et al., 2004; Park et al., 2004)。相似地,山葵过氧化物酶(horseradish peroxidase)在杨树中的过量表达也明显增强氧化抵抗力,提高树木生长量,而这一结果可能是由于改变了转基因植株的抗坏血酸盐/脱水抗坏血酸盐的水平导致的,该物质被认为在细胞分裂和伸长中扮演重要角色(Kawaoka et al., 2003)。此外,林木基因工程也关注林木在病虫害防御、生物补救、环境胁迫响应等过程中相关性状的定向改造。由于1年生模式植物存在转基因沉默的现象,越来越多的学者开始关注多年生转基因树木改良性状的稳定性,来源于大田试验的结果表明目的基因在转基因林木连续生长发育年份是稳定表达的(Meilan et al., 2002; Hawkins et al., 2003)。因此,改良性状的稳定性对于转基因林木商业应用而言应当不是首要考虑的。在全世界范围内,有超过210个田间试验种植转基因林木,绝大多数为杨树、松树、枫香树(Liquidambar)和桉树,仅中国就种植有300~500 hm2约140万株抗虫的转基因杨树(Halpin et al., 2003; FAO,2004)。
4 林木遗传基因组学和关联遗传学通过对转录本和蛋白质表达谱分析能洞悉林木复杂的发育程序和鉴定感兴趣的基因,进一步结合反向遗传学手段可揭示相关基因的生物功能。当前,林木分子生物学研究面临的巨大挑战是如何从这些复杂的数据里提取那些能改良特定性状的基因。遗传基因组学(Kirst et al., 2004)和关联遗传学(Neale et al., 2004)能揭示天然群体中表型变化的分子基础,是有效的林木分子育种策略。所谓遗传基因组学,就是在构建分子标记连锁图谱的基础上,结合分离群体内表型变异性状,采用DNA微阵列技术分析该群体内每个个体的全基因组表达谱,将每个基因的表达水平作为数量性状,分析定位该基因的表达QTL(eQTL)。它为研究复杂性状的分子机理和调控网络提供全新的手段。关联遗传学是以现有群体或种质资源为材料,通过检测群体内连锁位点间等位基因的非随机关联来解析复杂性状,阐明与个体表型变异有关的候选基因及其等位基因。
在过去数年间,许多研究者借助于与QTL连锁的DNA分子标记分析追踪特定性状,构建了大量的高密度遗传图谱,一系列农业重要性状的QTL被定位。但是,在林木育种中分子标记辅助选择(MAS)的潜力是有限的,除非这个标记就是基因本身,遗传重组导致每代的性状与标记连锁逐渐减少,对于具备较长世代和较大基因组的远缘繁殖物种——林木进行图位克隆也是难以实现的。目前几个策略用于鉴定潜在于QTLs中的基因。一个是关联遗传学策略,进行特定性状相关基因的遗传图谱定位与QTLs关联分析。例如,18个与木质素合成和细胞壁结构相关的候选基因已经图谱定位在火炬松(Pinus taeda)上,其中一些基因与木材性状QTLs共定位(Brown et al., 2003)。然而,关联遗传分析中可能存在大量连锁产生于虚假的杂交,以致候选基因在决定性状中的作用没有任何肯定的结论。另一个策略就是遗传基因组学,在一个作图谱系中结合表型的QTL分析与基因表达水平的QTL(eQTL)分析,筛选鉴定相关基因在决定性状中的作用(Kirst et al., 2004)。这个策略首次应用于一个桉树回交群体,直径生长相关QTLs与木质素相关基因的eQTL共定位,暗示生长和木质素特性受同一位点控制。一个SAMS基因被定位于直径生长相关QTL上。在玉米中SAMS的下调导致木质素含量减少,因此在桉树中推测SAMS是木质素形成和树木生长发育的调控基因。同时,一个鉴定产量候选基因的类似策略用于海岸松(Pinus pinaster)的蛋白质组水平,生物生产量QTL被发现与GS基因共定位,蛋白质数量QTL(PQTL)控制GS基因的丰度(Plomion et al., 2004)。尽管如此,这些候选基因与性状存在的关联性有可能仅因为在作图谱系上是连锁的。图谱定位候选基因和eQTL等位基因缺乏说服力的问题,可以用自然群体通过关联遗传学得以解决。在自然群体中较长的进化历史打破了标记和基因之间的连锁(Neale et al., 2004),在群体中存在大量等位基因的变异。在桉树、松树和杨树中一系列候选基因的测序,证实存在这样的等位基因变异,包括单核苷酸多态性(single nucleotide polymorphisms,SNPs)和插入/缺失(insertion-deletion polymorphisms,Indels)多态也很容易被发现(Poke et al., 2003; Gill et al., 2003; Brown et al., 2004; Ingvarsson,2005)。在火炬松种质中,连锁不平衡(LD)按基因物理长度的次序衰减;在欧洲山杨(Populus tremula)中,LD仅延伸几百个碱基对,这些表明了关联遗传学在鉴定重要性状相关基因上的潜力(Brown et al., 2004; Ingvarsson,2005)。
5 展望林木本身独特的生物学特性极大地限制了其遗传改良进程,而常规的林木遗传改良手段多集中于种质资源收集、引种驯化以及杂交育种等研究。现代分子生物学技术的迅速发展给林木遗传改良研究带来了新的机遇和挑战。林木分子生物学是一门有着广泛应用前景的学科,那些在草本模式植物中不易开展的分子生物学领域已经或正在林木中深入和拓展。转基因林木稳定的改良特性揭示了生物技术在林木遗传改良研究中具有巨大潜力。加强林木分子育种研究,不仅有理论意义,而且有潜在应用价值。当前林木分子生物学研究刚刚起步,林木遗传改良的分子基础研究仍然较为薄弱,对于多年生林木的诸多生物学性状的基因表达调控机制尚不清楚。就林木分子生物学研究而言,选择适宜的试验材料和技术手段对于解决相关问题将起到重要作用。近来杨树基因组测序计划的完成及其全基因组芯片的开发将为林木分子生物学研究提供新思路,势必加速多年生林木的遗传改良进程,优化林木遗传育种程序。
卢孟柱. 2003. 次生维管系统发育的分子基础研究. 中国农业科技导报, 5(2): 14-17. DOI:10.3969/j.issn.1008-0864.2003.02.004 |
Andersson A, Keskitalo J, Sjödin A, et al. 2004. A transcriptional timetable of autumn senescence. Genome Biol, 5: R24.1-R24.13. DOI:10.1186/gb-2004-5-9-241 |
Battey N H. 2000. Aspects of seasonality. J Exp Bot, 51: 1769-1780. DOI:10.1093/jexbot/51.352.1769 |
Baucher M, Petit-Conil M, Boerjan W. 2003. Lignin:genetic engineering and impact on pulping. Crit Rev Biochem Mol Biol, 38: 305-350. DOI:10.1080/10409230391036757 |
Bhalerao R, Nilsson O, Sandberg G. 2003a. Out of the woods:Forest biotechnology enters the genomic era. Curr Opin Biotechnol, 14: 206-213. DOI:10.1016/S0958-1669(03)00029-6 |
Bhalerao R, Keskitalo J, Sterky F, et al. 2003b. Gene expression in autumn leaves. Plant Physiol, 131: 430-442. DOI:10.1104/pp.012732 |
Boerjan W. 2005. Biotechnology and the domestication of forest trees. Current Opinion in Biotechnology, 16: 159-166. DOI:10.1016/j.copbio.2005.03.003 |
Bohlenius H, Huang T, Charbonnel-Campaa L, et al. 2006. CO/FT regulatory module controls timing of flowering and seasonal growth cessation in trees. Science, 312: 1040-1043. DOI:10.1126/science.1126038 |
Brown G R, Bassoni D L, Gill G P, et al. 2003. Identification of quantitative trait loci influencing wood property traits in loblolly pine (Pinus taeda L.) Ⅲ:QTL verification and candidate gene mapping. Genetics, 164: 1537-1546. |
Brown G R, Gill G P, Kuntz R J, et al. 2004. Nucleotide diversity and linkage disequilibrium in loblolly pine. Proc Natl Acad Sci, 101: 15255-15260. DOI:10.1073/pnas.0404231101 |
Brunner A M, Busov V B, Strauss S H. 2004. Poplar genome sequence:Functional genomics in an ecologically dominant plant species. Trends Plant Sci, 9: 49-56. |
Busov V B, Meilan R, Pearce D W, et al. 2003. Activation tagging of a dominant gibberellin catabolism gene (GA 2-oxidase) from poplar that regulates tree stature. Plant Physiol, 132: 1283-1291. DOI:10.1104/pp.103.020354 |
Chaffey N, Cholewa E, Regan S, et al. 2002. Secondary xylem development in Arabidopsis: a model for wood formation. Physiol Plant, 114: 594-600. DOI:10.1034/j.1399-3054.2002.1140413.x |
Déjardin A, Leplé J C, Lesage-Descauses M C, et al. 2004. Expressed sequence tags from poplar wood tissues—a comparative analysis from multiple libraries. Plant Biol, 6: 55-64. DOI:10.1055/s-2003-44744 |
Dubos C, Plomion C. 2003. Identification of water-deficit responsive genes in maritime pine (Pinus pinaster Ait.) roots. Plant Mol Biol, 51: 249-262. DOI:10.1023/A:1021168811590 |
Egertsdotter U, van Zyl L M, Mackay J, et al. 2004. Gene expression during formation of earlywood and latewood in loblolly pine:expression profiles of 350 genes. Plant Biol, 6: 654-663. DOI:10.1055/s-2004-830383 |
Fenning T M, Gershenzon J. 2002. Where will the wood come from? Plantation forests and the role of biotechnology. Trends Biotechnology, 20: 291-296. DOI:10.1016/S0167-7799(02)01983-2 |
Fladung M, Deutsch F, Hönicka H, et al. 2004. T-DNA and transposon tagging in aspen. Plant Biol, 6: 5-11. DOI:10.1055/s-2003-44745 |
Food and Agricultural Organization (FAO). 2004. Preliminary review of biotechnology in forestry, including genetic modification. Forest Genetic Resources Working Paper FGR/59E. Rome: Forest Resources Development Service, Forest Resources Division.
|
Gill G P, Brown G R, Neale D B. 2003. A sequence mutation in the cinnamyl alcohol dehydrogenase gene associated with altered lignification in loblolly pine. Plant Biotechnol J, 1: 253-258. DOI:10.1046/j.1467-7652.2003.00024.x |
Groover A T. 2005. What genes make a tree a tree?. Trends Plant Sci, 10: 210-214. DOI:10.1016/j.tplants.2005.03.001 |
Groover A, Fontana J R, Dupper G, et al. 2004. Gene and enhancer trap tagging of vascular-expressed genes in poplar trees. Plant Physiol, 134: 1742-1751. DOI:10.1104/pp.103.034330 |
Halpin C, Boerjan W. 2003. Stacking transgenes in forest trees. Trends Plant Sci, 8: 363-365. DOI:10.1016/S1360-1385(03)00155-9 |
Hawkins S, Leplé J C, Cornu D, et al. 2003. Stability of transgene expression in poplar:a model forest tree species. Ann For Sci, 60: 427-438. DOI:10.1051/forest:2003035 |
Hertzberg M, Aspeborg H, Schrader J, et al. 2001. A transcriptional roadmap to wood formation. Proc Natl Acad Sci, 98: 14732-14737. DOI:10.1073/pnas.261293398 |
Horvath D P, Anderson J V, Chao W S, et al. 2003. Knowing when to grow:signals regulating bud dormancy. Trends Plant Sci, 8: 534-540. DOI:10.1016/j.tplants.2003.09.013 |
Howe G T, Aitken S N, Neale D B, et al. 2003. From genotype to phenotype unraveling the complexities of cold adaptation in forest trees. Can J Bot, 81: 1247-1266. DOI:10.1139/b03-141 |
Ingvarsson P K. 2005. Nucleotide polymorphism and linkage disequilibrium within and among natural populations of European aspen (Populus tremula L., Salicaceae). Genetics, 169: 945-953. DOI:10.1534/genetics.104.034959 |
Israelsson M, Eriksson M E, Hertzberg M, et al. 2003. Changes in gene expression in the wood-forming tissue of transgenic hybrid aspen with increased secondary growth. Plant Mol Biol, 52: 893-903. DOI:10.1023/A:1025097410445 |
Jansson S, Douglas C J. 2007. Populus:A model system for plant biology. Annu Rev Plant Biol, 58: 435-458. DOI:10.1146/annurev.arplant.58.032806.103956 |
Jing Z P, Gallardo F, Pascual M B, et al. 2004. Improved growth in a field trial of transgenic hybrid poplar overexpressing glutamine synthetase. New Phytol, 164: 137-145. DOI:10.1111/j.1469-8137.2004.01173.x |
Jonsson P, Gullberg J, Nordström A, et al. 2004. A strategy for identifying differences in large series of metabolomic samples analyzed by GC/MS. Anal Chem, 76: 1738-1745. DOI:10.1021/ac0352427 |
Kawaoka A, Matsunaga E, Endo S, et al. 2003. Ectopic expression of a horseradish peroxidase enhances growth rate and increases oxidative stress resistance in hybrid aspen. Plant Physiol, 132: 1177-1185. DOI:10.1104/pp.102.019794 |
Kirst M, Johnson A F, Baucom C, et al. 2003. Apparent homology of expressed genes from wood-forming tissues of loblolly pine (Pinus taeda L.) with Arabidopsis thaliana. Proc Natl Acad Sci, 100: 7383-7388. DOI:10.1073/pnas.1132171100 |
Kirst M, Myburg A A, De Léon J P G, et al. 2004. Coordinated genetic regulation of growth and lignin revealed by quantitative trait locus analysis of cDNA microarray data in an interspecific backcross of eucalyptus. Plant Physiol, 135: 2368-2378. DOI:10.1104/pp.103.037960 |
Ko J H, Han K H, Park S, et al. 2004. Plant body weight-induced secondary growth in Arabidopsis and its transcription phenotype revealed by whole-transcriptome profiling. Plant Physiol, 135: 1069-1083. DOI:10.1104/pp.104.038844 |
Kohler A, Delaruelle C, Martin D, et al. 2003. The poplar root transcriptome:analysis of 7000 expressed sequence tags. FEBS Lett, 542: 37-41. DOI:10.1016/S0014-5793(03)00334-X |
Lafarguette F, Leplé J C, Déjardin A, et al. 2004. Poplar genes encoding fasciclin-like arabinogalactan proteins are highly expressed in tension wood. New Phytol, 164: 107-121. DOI:10.1111/j.1469-8137.2004.01175.x |
Le Provost G, Paiva J, Pot D, et al. 2003. Seasonal variation in transcript accumulation in wood-forming tissues of maritime pine (Pinus pinaster Ait.) with emphasis on a cell wall glycine-rich protein. Planta, 217: 820-830. DOI:10.1007/s00425-003-1051-2 |
Lorenz W W, Dean J F D. 2002. SAGE profiling and demonstration of differential gene expression along the axial developmental gradient of lignifying xylem in loblolly pine (Pinus taeda). Tree Physiol, 22: 301-310. DOI:10.1093/treephys/22.5.301 |
Martin F, Duplessis S, Kohler A, et al. 2004. Exploring the transcriptome of the ectomycorrhizal symbiosis//Kumar S, Fladung M. Molecular genetics and breeding of forest trees. New York: Food Products Press, 81-109.
|
Meilan R, Auerbach D J, Ma C, et al. 2002. Stability of herbicide resistance and GUS expression in transgenic hybrid poplars (Populus spp.) during four years of field trials and vegetative propagation. HortScience, 37: 277-280. |
Morreel K, Ralph J, Kim H, et al. 2004a. Profiling of oligolignols reveals monolignol coupling conditions in lignifying poplar xylem. Plant Physiol, 136: 3537-3549. DOI:10.1104/pp.104.049304 |
Morreel K, Ralph J, Lu F, et al. 2004b. Phenolic profiling of caffeic acid O-methyltransferase-deficient poplar reveals novel benzodioxane oligolignols. Plant Physiol, 136: 4023-4036. DOI:10.1104/pp.104.049312 |
Morris C R, Scott J T, Chang H M, et al. 2004. Metabolic profiling:a new tool in the study of wood formation. J Agric Food Chem, 52: 1427-1434. DOI:10.1021/jf034688l |
Neale D B, Savolainen O. 2004. Association genetics of complex traits in conifers. Trends Plant Sci, 9: 325-330. DOI:10.1016/j.tplants.2004.05.006 |
Park Y W, Baba K, Furuta Y, et al. 2004. Enhancement of growth and cellulose accumulation by overexpression of xyloglucanase in poplar. FEBS Lett, 564: 183-187. DOI:10.1016/S0014-5793(04)00346-1 |
Paux E, Tamasloukht M, Ladouce N, et al. 2004. Identification of genes preferentially expressed during wood formation in Eucalyptus. Plant Mol Biol, 55: 263-280. DOI:10.1007/s11103-004-0621-4 |
Pilate G, Déjardin A, Laurans F, et al. 2004. Tension wood as a model for functional genomics of wood formation. New Phytol, 164: 63-72. DOI:10.1111/j.1469-8137.2004.01176.x |
Pilate G, Guiney E, Holt K, et al. 2002. Field and pulping performances of transgenic trees with altered lignification. Nat Biotechnol, 20: 607-612. DOI:10.1038/nbt0602-607 |
Plomion C, Bahrman N, Costa P, et al. 2004. Proteomics for genetic and physiological studies in forest trees: application in maritime pine//Kumar S, Fladung M. Molecular genetics and breeding of forest trees. New York: Food Products Press, 53-79.
|
Plomion C, Pionneau C, Baillères H. 2003. Analysis of protein expression along the normal to tension wood gradient in Eucalyptus gunnii. Holzforschung, 57: 353-358. |
Poke F S, Vaillancourt R E, Elliott R C, et al. 2003. Sequence variation in two lignin biosynthesis genes, cinnamoyl CoA reductase (CCR) and cinnamyl alcohol dehydrogenase 2 (CAD2). Mol Breed, 12: 107-118. DOI:10.1023/A:1026006828721 |
Ralph S, Oddy C, Cooper D, et al. 2006. Genomics of hybrid poplar (Populus trichocarpa×deltoides) interacting with forest tent caterpillars (Malacosoma disstria):normalized and full-length cDNA libraries, expressed sequence tags, and a cDNA microarray for the study of insect-induced defences in poplar. Mol Ecol, 15: 1275-1297. DOI:10.1111/j.1365-294X.2006.02824.x |
Renaut J, Lutts S, Hoffmann L, et al. 2004. Responses of poplar to chilling temperatures:proteomic and physiological aspects. Plant Biol, 6: 81-90. DOI:10.1055/s-2004-815733 |
Rohde A, Bhalerao R P. 2007. Plant dormancy in the perennial context. TRENDS in Plant Science, 12(5): 217-223. DOI:10.1016/j.tplants.2007.03.012 |
Rohde A, Prinsen E, De Rycke R, et al. 2002. ABI3 impinges on growth and differentiation of embryonic leaves during bud set in poplar. Plant Cell, 14: 1885-1901. DOI:10.1105/tpc.003186 |
Schrader J, Moyle R, Bhalerao R, et al. 2004a. Cambial meristem dormancy in trees involves extensive remodelling of the transcriptome. Plant J, 40: 173-187. DOI:10.1111/tpj.2004.40.issue-2 |
Schrader J, Nilsson J, Mellerowicz E, et al. 2004b. A high-resolution transcript profile across the wood-forming meristem of poplar identifies potential regulators of cambial stem cell identity. Plant Cell, 16: 2278-2292. DOI:10.1105/tpc.104.024190 |
Shani Z, Dekel M, Tsabary G, et al. 2004. Growth enhancement of transgenic poplar plants by overexpression of Arabidopsis thaliana endo-1, 4-b-glucanase (Cel1). Mol Breed, 14: 321-330. DOI:10.1023/B:MOLB.0000049213.15952.8a |
Smith C M, Rodriguez-Buey M, Karlsson J, et al. 2004. The response of the poplar transcriptome to wounding and subsequent infection by viral pathogen. New Phytol, 164: 123-136. DOI:10.1111/j.1469-8137.2004.01151.x |
Taylor G. 2002. Populus:Arabidopsis for forestry. Do we need a model tree?. Ann Bot, 90: 681-689. DOI:10.1093/aob/mcf255 |
Thomas H. 2000. Annuality, perenniality and cell death. J Exp Bot, 51: 1781-1788. DOI:10.1093/jexbot/51.352.1781 |
Tuskan G A, DiFazio S, Jansson S, et al. 2006. The genome of black cottonwood, Populus trichocarpa (Torr & Gray ex. Brayshaw). Science, 313: 1596-15604. DOI:10.1126/science.1128691 |
Uggla C, Sundberg B. 2002. Sampling of cambial region tissues for high resolution analysis//Chaffey N J. Wood formation in trees: cellular and molecular biology techniques. London: Taylor & Francis, 215-228.
|
Weiser C J. 1970. Cold resistance and injury in woody plants. Science, 169: 1299-1278. DOI:10.1126/science.169.3952.1299 |
Wimp G M, Young W P, Woolbright S A, et al. 2004. Conserving plant genetic diversity for dependent animal communities. Ecol Lett, 7: 776-780. DOI:10.1111/ele.2004.7.issue-9 |
 2009, Vol. 45
2009, Vol. 45