文章信息
- 郭凌飞, 邹明宏, 曾辉, 杜丽清, 陆超忠.
- Guo Lingfei, Zou Minghong, Zeng Hui, Du Liqing, Lu Chaozhong.
- 澳洲坚果ISSR-PCR反应体系的建立与优化
- Establishment and Optimization of ISSR-PCR System in Macadamia
- 林业科学, 2008, 44(5): 160-164.
- Scientia Silvae Sinicae, 2008, 44(5): 160-164.
-
文章历史
- 收稿日期:2007-04-16
-
作者相关文章
2. 华南热带农业大学园艺学院 儋州 571737
2. College of Horticulture, South China University of Tropical Agriculture Danzhou 571737
澳洲坚果(Macadamia spp.)属山龙眼科(Proteaceae)澳洲坚果属(Macadamia)常绿乔木。原产于澳大利亚昆士兰州东南部和新南威尔士州北部、南纬25°—31°之间的沿海亚热带雨林。其种仁富含不饱和脂肪酸、蛋白质、糖等,营养价值高,风味十分独特,有“干果皇后”之称;其副产品也有多种用途,如果壳可合成活性炭(Ahmadpour et al., 1997),果皮可混作家畜饲料等。我国自1979年起引入澳洲坚果,经过近20年的发展,至2005年,全国栽培面积已达3 516.53 hm2,其中,仅云南就达3 149.00 hm2。目前我国澳洲坚果的研究主要集中于栽培、生理等方面,分子生物学方面的研究较少,而国外在分子水平上的研究主要采用同工酶(Vithanage et al., 1992;Aradhya et al., 1998)、RAPD(Vithanage et al., 1998)、STMS(Vithanage et al., 1998)、AFLP(Steiger et al., 2003)、RAMiFi(Peace et al., 2004)、RAF(Peace et al., 2005)等技术。
Zietkiewicz等(1994)提出的简单序列重复区间(Inter Simple Sequence Repeat,ISSR)DNA标记技术,结合了SSR和RAPD的优点,具有模板需要量少,多态性丰富,成本低,操作简单,稳定性高等优点,而较之AFLP又具技术要求较低的特点。近年来ISSR已广泛应用于品种鉴定(Ammiraju et al., 2001)、种质资源和遗传多样性研究(Lia,2001)等领域。本研究采用单因素试验法,对体系中的各因素进行筛选,建立并优化澳洲坚果的ISSR反应体系,为研究澳洲坚果的遗传多样性奠定基础。
1 材料与方法 1.1 材料15份供试澳洲坚果(Macadamia integrifolia)品种(表 1)均取自中国热带农业科学院南亚热带作物研究所澳洲坚果种质资源圃。选取无病虫害的叶片,采摘洗净后于-70 ℃保存。反应体系的优化以HAES246为模板完成,甲酰胺浓度的优化以O.C.和HAES660为模板完成。
|
|
所用10×PCR buffer(含Mg2+)、Taq DNA聚合酶、marker(DL2000)购于大连宝生物,核酸荧光染料GoldView购于北京赛百盛,dNTPs购于上海生工,引物由上海生工合成。甲酰胺为国产分析纯。
1.3 DNA提取参照Doyle等(1987)的方法并加以改良,所提取的基因组DNA用核酸蛋白仪(Eppendorf)检测浓度,稀释到10 ng·μL-1,于-20 ℃保存备用。
1.4 ISSR体系优化及PCR扩增程序参照Zietkiewicz等(1994)所创立的ISSR原始扩增程序并略做修改,采用加拿大哥伦比亚大学UBC公司公布的引物序列,选用引物UBC-844[(CT)8RT]对澳洲坚果ISSR反应体系中的Taq DNA聚合酶、模板DNA、dNTPs、引物、Mg2+的浓度以及退火温度和循环次数逐一做多水平试验。PCR扩增在梯度PCR仪(Eppendorf)上完成,扩增产物用1.6%琼脂糖凝胶(含1 μL GoldView/25 mL)电泳分离,电泳缓冲液为0.5×TBE,结束后在GelDoc EQ凝胶成像系统(Bio-Rad)上拍照观察。各因素水平见表 2。
|
|
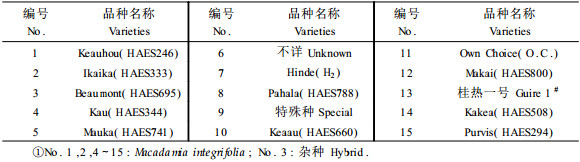
在ISSR反应中,Taq DNA聚合酶对ISSR扩增结果具有显著影响。Taq DNA聚合酶的浓度过高会造成浪费,且易产生非特异性扩增产物的积累;过低会降低新链的合成效率,从而影响试验结果。如图 1所示,在25 μL的反应体系中,Taq DNA聚合酶从0.5~2 U均有扩增产物。当Taq DNA聚合酶的含量为0.5 U时,扩增谱带较弱。而在1~2 U时,所得谱带较为相似,清晰程度也近似。综合经济角度和实际观测结果,确定1 U为本试验中Taq DNA聚合酶的最佳用量。
2.2 模板DNA浓度对ISSR扩增的影响模板DNA浓度对ISSR-PCR扩增的结果有一定的影响。浓度过低,扩增产物不稳定或无扩增产物;浓度过高可能增加非特异性产物。由图 1可见,澳洲坚果的ISSR反应体系对模板DNA浓度的变化不敏感,在所选浓度范围内均可扩增出谱带,但在10 ng时扩增出的谱带略为模糊,而含量为20~40 ng时扩增的谱带稳定,清晰可辨。考虑到结果的稳定性以及样品使用量,在澳洲坚果ISSR-PCR反应体系中选用20 ng作为适宜浓度。
|
图 1 Taq DNA聚合酶、模板DNA、dNTPs浓度、引物、Mg2+浓度对ISSR-PCR的影响 Figure 1 The effect of different concentrations of five factors on ISSR-PCR 1~4.Taq DNA聚合酶浓度Taq DNA polymerase: 0.5,1,1.5,2 U; 5~8.模板DNA Template DNA: 10,20,30,40 ng·(25 μL)-1; 9~12.dNTPs浓度Concentration of dNTPs: 0.1,0.15,0.2,0.25 mmol·L-1; 13~16.引物浓度Concentration of primer:0.25,0.5,0.75,1 μmol·L-1; 17~20. Mg2+浓度Mg2+ concentration: 1,1.5,2,2.5 mmol·L-1. |
dNTPs是ISSR反应的原料,浓度过高会导致PCR错配,使扩增出现非特异性扩增;过低将影响合成效率,甚至会因过早的消耗而使产物单链化,影响扩增效果(卢圣栋,1999)。本试验针对dNTPs设计了4个浓度梯度,结果(图 1)表明:浓度为0.1~0.2 mmol·L-1时,扩增出的谱带较为一致,但在0.15 mmol·L-1时条带较之0.1 mmol·L-1时清晰,而在0.2 mmol·L-1时谱带趋于弱化。当dNTPs的浓度达到0.25 mmol·L-1时,扩增谱带显著减少。因此可以确定,在该体系中dNTPs的最佳用量为0.15 mmol·L-1。
2.4 引物浓度对ISSR-PCR反应的影响引物浓度对PCR的带型可产生显著影响。浓度过低不能扩增,太高易产生新的位点(邹喻萍等,2001)。在本研究中,发现不同浓度引物对扩增出的谱带数量和强弱影响较为显著。如图 1所示,当引物浓度增至0.5 μmol·L-1时,扩增不完全,条带数量少,亮度低;当浓度升至0.75~1 μmol·L-1时,出现非特异性谱带。这是由于引物浓度过高会促进引物错误引导非特异性产物的合成。而在0.25 μmol·L-1时,扩增效率最高,所得谱带清晰易辨。
2.5 Mg2+浓度对ISSR-PCR反应的影响通常认为Mg2+的适宜浓度范围在1.5~2.5 mmol·L-1之间。Taq DNA聚合酶是Mg2+依赖性酶,对Mg2+的浓度非常敏感。选择合适的Mg2+浓度,对PCR反应至关重要。引物与模板的双链杂交体的解链与退火温度受二价阳离子的影响,特别是Mg2+浓度能影响反应的特异性和扩增片段的产率(卢圣栋,1999)。当反应体系中Mg2+的浓度过高时会增强背景干扰,过低将降低扩增产率。由图 1可见,在澳洲坚果的ISSR反应体系中,随着Mg2+浓度的增大,扩增谱带的数量及亮度呈上升趋势。当Mg2+浓度为2.0~2.5 mmol·L-1时,电泳条带一致,但以2.5 mmol·L-1时的带型最佳,亮度最好。而浓度为1.0 mmol·L-1时,带型模糊且不稳定。因此选用2.5 mmol·L-1作为澳洲坚果ISSR反应体系中Mg2+的最佳浓度。
2.6 退火温度对ISSR反应的影响在同一PCR体系中,退火温度不同,产生错配的程度也不同。一般较低的退火温度能保证引物与模板结合的稳定性,而在允许的范围内选择较高的退火温度则可减少引物与模板DNA之间的非特异性结合(冯富娟等,2004)。因此,有必要对各个引物的最佳退火温度进行优化筛选。
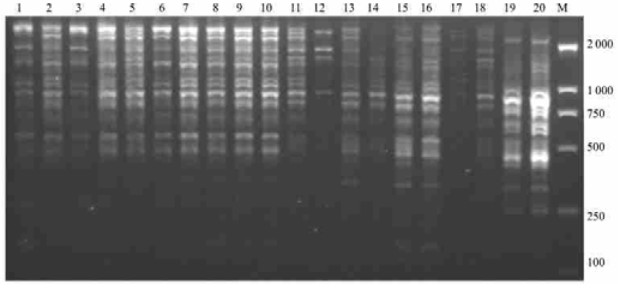
根据上述5个因子的影响程度确定澳洲坚果最佳的ISSR反应体系,25 μL的反应体系包括:Taq DNA聚合酶1 U;模板DNA 20 ng;dNTPs 0.15 mmol·L-1;引物0.25 μmol·L-1;Mg2+ 2.5 mmol·L-1。在此基础上进行退火温度的优化。根据退火温度理论值计算公式Tm=4(G+C)+2(A+T)计算出引物UBC-844的理论退火温度为52 ℃。但在本试验中,当退火温度为57.9 ℃时,引物UBC-844扩增出的谱带最清晰,在理论值52 ℃左右得到的扩增谱带较为模糊,并有部分条带缺失。图 2A的结果表明,退火温度低于56.5 ℃和高于57.9 ℃时扩增出的谱带较弱,并有部分谱带扩增不出。虽然在56.5 ℃时也扩增出了与57.9 ℃相似的谱带,但所得带型不如57.9 ℃时获得的清晰。

|
图 2 UBC-844不同退火温度、循环数及不同甲酰胺浓度对澳洲坚果ISSR扩增的影响 Figure 2 The effect of different annealing temperatures, cycles and concentrations of formamide of UBC-844 on ISSR amplification of macadamia A.1~12.退火温度Annealing temperature: 51.1,51.3,51.8,52.7,53.8,55.1,56.5,57.9,59.2,60.3,61.1,61.5 ℃;B.1~5.循环数Cycles: 25,30,35,38,40;C.1~2,3~4,5~6,7~8,9~10,11~12.甲酰胺浓度Concentration of formamide: 0%,0.4%,0.8%,1.2%,1.6%,2.0%. |
PCR的循环次数决定了产量,循环次数太少,产物少或者根本没有产物。次数太多产物也不会有所增加,反而会引发非特异性条带的产生。在本试验中,分别设计了25、30、35、38、40个循环数以探究其对ISSR反应的影响。结果(图 2B)表明:循环数为25时,几乎无法扩增出可辨谱带。而在30个循环时,产物不够稳定,部分带型较弱。35~40个循环时,扩增结果一致,由此可见,在本试验中35个循环已达到要求。
2.8 甲酰胺浓度的选择一般认为在反应体系中加入2%的甲酰胺能有效地使背景颜色减弱,谱带清晰(Joshi et al., 2000)。如图 2C所示,加入甲酰胺可在一定程度上起到降噪的作用。当体系中甲酰胺的浓度为0.4%~0.8%时,扩增谱带的数量差异不大,但背景干扰相对不加甲酰胺的对照较弱。而高于0.8%时,一些弱带已无法扩增出。在实际应用中,由于甲酰胺的使用可导致弱带的消失,因此是否使用应加以斟酌。
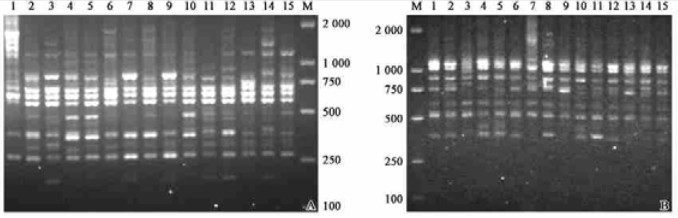
2.9 ISSR反应体系稳定性检测随机选择引物UBC-835、UBC-855对部分澳洲坚果种质进行扩增,以检测所确立的ISSR反应体系的稳定性。结果(图 3)表明,所选引物可在15份澳洲坚果种质上扩增出清晰、重复性好的谱带,证明本试验所建立的体系稳定可靠。
|
图 3 引物UBC-835和UBC-855对15份种质的扩增结果 Figure 3 The result of amplification of primer UBC-835 and UBC-855 on 15 varieties of macadamia A.UBC-835 (退火温度Annealing temperature:50.6 ℃);B.UBC-855 (退火温度Annealing temperature:50.4 ℃) |
虽然ISSR标记技术结合了SSR、RAPD等技术的优点,但它同样是一种基于PCR的分子标记技术,必然会受到Taq DNA聚合酶含量、模板DNA浓度与纯度、引物、dNTPs和Mg2+浓度以及引物退火温度等因素的影响(邹喻萍等,2001)。本试验也印证了这一点。因此有必要在进行ISSR分析之前对反应体系进行优化。虽然一般认为ISSR技术对模板DNA的质量要求较低,但杨传平等(2005)研究发现未经纯化的模板DNA可扩增出的谱带数量明显少于纯度高的模板DNA,因此笔者认为高质量的基因组DNA是获得ISSR反应稳定结果的前提。引物由于碱基序列的长短不同而具有不同的退火温度,同种引物在不同植物样品中的退火温度也不相同。穆立蔷等(2006)在优化紫椴(Tilia amurensis)种质的ISSR体系中发现引物UBC-844和UBC-835的最佳退火温度分别为50.2 ℃和51.4 ℃。而在本研究中,经优化确定UBC-844的最佳退火温度为57.9 ℃,UBC-835为50.6 ℃,差距较大。可能是物种不同造成模板DNA的特性差异;另一方面则可能是设备差异所造成。此外,Taq DNA聚合酶直接关系到扩增反应的成功与否。由于不同厂家之间的产品质量存在很大差异,甚至是同一厂家不同批次的产品都可能存在差异,为确保试验的准确性,应尽可能选择同一厂家同一批次的产品。
相比正交试验而言,单因素试验设计可直观快速地表现出各因子对反应体系的影响,但不具正交试验能体现各因素间互作效应的特性。因此,可考虑将正交试验与单因素设计相结合,同时体现二者的优势。
冯富娟, 王凤友, 刘彤. 2004. 红松ISSR-PCR实验系统影响因素. 植物学通报, 21(3): 326-331. DOI:10.3969/j.issn.1674-3466.2004.03.010 |
卢圣栋. 1999. 现代分子生物学技术. 2版. 北京: 中国协和医科大学出版社, 458-463.
|
穆立蔷, 刘赢男, 冯富娟, 等. 2006. 紫椴ISSR-PCR反应体系的建立与优化. 林业科学, 42(6): 26-31. |
杨传平, 潘华, 魏志刚, 等. 2005. 白桦ISSR-PCR反应体系的优化. 东北林业大学学报, 33(6): 1-3. DOI:10.3969/j.issn.1000-5382.2005.06.001 |
邹喻萍, 葛颂, 王晓东. 2001. 系统进化植物学中的分子标记. 北京: 科学出版社, 36-97.
|
Ahmadpour A, Do D D. 1997. The preparation of activated carbon from macadamia nutshell by chemical activation. Carbon, 35(12): 1723-1732. DOI:10.1016/S0008-6223(97)00127-9 |
Ammiraju J S S, Dholakin B B, Santra D K, et al. 2001. Identification of inter simple sequence repeat(ISSR)markers associated with seed size in wheat. Theor Appl Genet, 102: 726-732. DOI:10.1007/s001220051703 |
Aradhya M K, Yee L K, Zee F T, et al. 1998. Genetic variability in macadamia. Genetic Resources and Crop Evolution, 44: 19-32. |
Doyle J J, Dickson E E. 1987. Preservation of plant samples for DNA restriction endonuclease analysis. Taxon, 36: 715-722. DOI:10.2307/1221122 |
Joshi S P, Gupta V S, Aggarwal R K, et al. 2000. Genetic diversity and phylogenetic relationship as revealed by inter simple sequence repeat (ISSR) polymorphism in the genus Oryza. Theor Appl Genet, 100: 1311-1320. DOI:10.1007/s001220051440 |
Lia G S. 2001. Genetic variation and colonal diversity of Psammochloa villosa (Poaceae) detected by ISSR markers. Ann Bot, 87: 585-590. DOI:10.1006/anbo.2001.1390 |
Peace C P, Allan P, Vithanage V, et al. 2005. Genetic relationships amongst macadamia varieties grown in South Africa as assessed by RAF markers. S Afr J Plant Soil, 22(2): 71-75. DOI:10.1080/02571862.2005.10634684 |
Peace C P, Vithanage V, Neal J, et al. 2004. A comparison of molecular markers for genetic analysis of macadamia. Journal of Horticultural Science & Biotechnology, 79(6): 965-970. |
Steiger D L, Moore P H, Zee F, et al. 2003. Genetic relationships of macadamia cultivars and species revealed by AFLP markers. Euphytica, 132: 269-277. DOI:10.1023/A:1025025522276 |
Vithanage V, Hardner C, Peace C P, et al. 1998. Progress made with molecular markers for genetic improvement of macadamia. Acta Horticulturae, 461: 199-207. |
Vithanage V, Winks C W. 1992. Isozymes as genetic markers for macadamia. Scientia Horticulturae, 49: 103-115. DOI:10.1016/0304-4238(92)90147-5 |
Zietkiewicz E, Rafalski A, Labuda D. 1994. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification. Genomics, 20: 176-183. DOI:10.1006/geno.1994.1151 |
 2008, Vol. 44
2008, Vol. 44