文章信息
- 黄久香, 黄妃本, 许涵, 李意德, 庄雪影.
- Huang Jiuxiang, Huang Feiben, Xu Han, Li Yide, Zhuang Xueying.
- 海南岛青梅AFLP标记的遗传多样性
- Genetic Diversity of Vatica mangachapoi in Hainan Island Revealed by AFLP
- 林业科学, 2008, 44(5): 46-52.
- Scientia Silvae Sinicae, 2008, 44(5): 46-52.
-
文章历史
- 收稿日期:2007-03-12
-
作者相关文章
2. 中国林业科学研究院热带林业研究所 广州 510520
2. Institute of Tropical Forestry, CAF Guangzhou 510520
遗传多样性是物种长期生存和保持进化潜力的物质基础。对于濒危植物的保育,无论是就地保育地点的选择,还是迁地保育的取样策略,不仅需要考虑植物的宏观生态学特性,同时也需要考虑植物的遗传多样性和遗传结构特点(Morin, 2000; Clarke et al., 2000; Frankham et al., 2002)。现有研究表明,居群数量少、个体数量低的特有或稀有物种通常具有较低的遗传多样性,遗传变异下降或低的遗传多样性是这些物种难以适应急剧变化环境而导致濒危乃至灭绝的原因之一(Ellstrand et al., 1993);但也有一些研究揭示,即使同是濒危物种,其遗传多样性水平和遗传结构也会有很大的不同(黄宏文,1998;Mattew et al., 2000)。因此,开展珍稀濒危物种居群遗传结构研究是制定有效保育措施的重要基础(Schemske et al., 1994; Oostermeijer et al., 2003)。
利用分子标记方法来研究特有濒危植物的遗传多样性,是保育生物学研究的一个热点(Frankham et al., 2002; Zhang et al., 2003)。比较常用的分子标记技术包括蛋白质水平的同工酶和等位酶,以及DNA水平的RFLP、RAPD、SSR、ISSR、AFLP和DNA序列测定。其中,AFLP是近年才发展起来的分子标记技术(Vos et al., 1995),该技术结合了RFLP技术的高重复性和RAPD技术的简便快速的特点,通过对基因组DNA的限制性酶切片段进行选择性扩增来探索DNA多态性,具有重复性好和多态性高的特点,目前已被广泛应用于珍稀植物的遗传多样性分析等研究(Zhang et al., 2003; Cao et al., 2006; 李善文等, 2007)。
青梅(Vatica mangachapoi)是龙脑香科(Dipterocarpaceae)常绿乔木,树高可达30 m,胸径可达1.2 m。因其树皮呈青灰色,故又称青皮。该种具有翅果,其果翅是由宿存萼片发育而成。该种自然分布于中国、越南、泰国、菲律宾和印度尼西亚等地,在我国仅分布在海南岛,以霸王岭、猕猴岭、尖峰岭较为集中。青梅的垂直分布范围较广,从海拔3~4 m至1 000 m均有出现,但以海拔200 ~ 500 m较为普遍,为海南岛热带低地雨林的优势种、热带常绿季雨林的表征种和建群种(杨小波等,2005;王发国等,2005;李意德等,2006;陈伟等,2006)。青梅主干通直,材质优良,但由于半个世纪以来的高强度砍伐利用,海南岛青梅群落及居群数量急剧萎缩。该种已被国务院批准的《国家重点保护野生植物名录(第一批)》列为国家二级保护植物(于永福,1999)。
有关青梅外貌及分布特点和群落结构的研究已有报道(胡玉佳,1991;梁淑群等,1993;1994;方洪等,2004;杨小波等,2005;李意德等,2006)。其中,陈伟等(2006)根据生境条件将海南岛青梅林划分为湿润性青梅林、季节性青梅林和单优青梅林。青梅自然居群分子生物学水平的研究尚缺。本研究利用AFLP标记来分析海南岛青梅自然居群的遗传多样性和遗传结构,以期为其现存居群保育措施的制订提供依据,为我国热带亚热带季雨林建群种的保育提供参考。
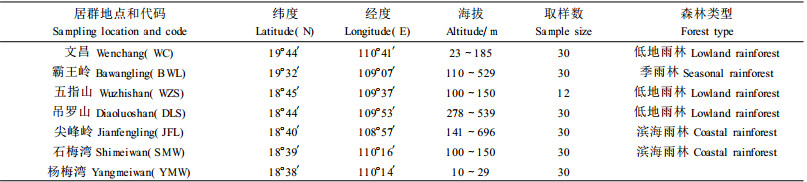
1 材料和方法 1.1 居群采样为了保证取样的代表性,共采集了海南岛文昌、霸王岭、五指山、吊罗山、尖峰岭、杨梅湾和石梅湾(陵水)共7个自然居群的材料,除五指山仅采到12个样品外,其他居群均各取30个样品(表 1,图 1)。其中,石梅湾和杨梅湾为海南岛东部滨海雨林,文昌、吊罗山、五指山和尖峰岭为低地雨林, 霸王岭为季雨林。个体间的采样间距在50 m以上,所采新鲜叶片迅速放入装有变性硅胶的封口袋中进行快速干燥,之后转移至超低温冰箱保存备用。
|
|
采用改良的CTAB法提取叶片总DNA (Doyle, 1991)。通过0.8%的琼脂糖电泳检测DNA质量,-20 ℃保存备用。从上海基因公司(Gene Company Limited)提供的AFLP引物中筛选出6对能获得多态性高、重复性好的清晰位点的引物组合用于7个居群的PCR扩增。基本步骤如下:1)酶切和连接:采用20 μL反应体系,其中模板DNA 4 μL(50 ng·μL),Adapter 2.0 μL,EcoRI/MseI 2.0 μL,10×Reaction buffer 2.0 μL,10 mmol·L-1 ATP 2.0 μL,T4 Ligase 1.0 μL,AFLP-Water 7.0 μL。混匀,离心数秒,37 ℃保温5 h,8 ℃保温4 h,4 ℃过夜。2)预扩增:25 μL反应体系包括模板DNA 2.0 μL,Pre-ampmix 1.0 μL,10 mmol·L-1 dNTPs 1.0 μL,10×PCR buffer 2.5 μL,5 U·μL-1Taq DNA polymerase 0.5 μL,ddH2O 18.0 μL。扩增程序为94 ℃ 2 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 80 s, 共30个循环;最后72 ℃ 5 min。用TE将预扩增产物按1:20稀释,作为选择性扩增模板。3)选择性扩增:筛选出在预扩增中6对扩增效果好的引物组合(E-AAC/M-CAG;E-AAC/M-CTA;E-AAC/M-CTC;E-AAG/M-CTA;E-ACA/M-CTC;E-AGG/M-CTA;其中MseI引物用FAM荧光素标记)应用于正式扩增。25 μL反应体系包括预扩增稀释样品DNA 2.0 μL,10×PCR buffer 2.5 μL,10 mmol·L-1 dNTPs 0.5 μL,5 U·μL-1 Taq DNA polymerase 0.5 μL,ddH2O 19.5 μL。混匀,离心数秒。按下列参数PCR循环:第1个循环的扩增参数为94 ℃ 30 s,65 ℃ 30 s,72 ℃ 80 s;以后每个循环温度递减0.7 ℃,扩增12个循环;再按下列参数扩增23个循环,即94 ℃ 30 s,55 ℃ 30 s,72 ℃ 80 s。
PCR扩增在Gene Amp PCR System 9600 PCR仪(Perkin-Elmer)中进行,选择性扩增产物在ABI 377自动测序仪(ABI)6%聚丙烯酰胺凝胶上电泳2.5 h并自动采集电泳图谱。
1.3 数据分析一般认为,低频率等位基因多为有害的,且只存在少数世代,而与生态适应相关、应长期保存的基因多为频率大于5.00%的中高频等位基因(Frankham et al., 2002)。为了减少实验误差,本研究仅将清晰无误且频率大于5.00%的电泳位点转换成0/1矩阵用于数据分析。
用PopGene 32软件计算以下各遗传多样性指数(Yeh et al., 1999):1)多态位点数(number of polymorphic loci)和多态位点百分率(percentage of polymorphic loci);2)观测等位基因数Na(observed number of alleles)和有效等位基因数Ne(effective number of alleles);3) Nei's基因多样性指数H(Nei's gene diversity), Shannon多态性信息指数I(Shannon information index);4)总遗传多样性Ht (total gene diversity)和居群内多样性Hs(population gene diversity);5)遗传距离D(genetic distance)(Nei, 1978)。
居群间的遗传分化使用ARLEQUIN 2.0 (Schneider et al., 2000)中的AMOVA来计算;根据Nm=(1-Fst)/4Fst计算基因流Nm(gene flow)(Wright,1931);用NTSYS2.1软件对7个居群进行非加权算术平均法聚类(unweighted pair group with arithmetic average,UPGMA)(Rohlf, 2000)。居群遗传距离与地理距离相关性采用TFPGA (Tools for Population Genetic Analysis)软件的Mantel test评估(Miller, 1997)。
2 结果与分析 2.1 青梅的遗传多样性应用6对引物组合,从海南岛7个青梅自然居群192株个体的DNA样本中共扩增出频率大于5.00%的位点777个,其中多态性位点774个,多态性比例P高达99.61%;观测等位基因数Na和有效等位基因数Ne分别为1.996 1和1.421 8;Nei's基因多样性指数H为0.266 1,Shannon多态性信息指数I为0.420 4;总基因多样性Ht为0.268 4,居群内基因多样性Hs为0.215 2。居群水平平均多态性位点数和多态性比例为617和79.45%;Na和Ne分别为1.794 5和1.351 1;H和I平均值分别为0.215 3和0.335 2(表 2)。
|
|
多态位点百分率(P)是目前应用较为广泛的多样性指标,在7个自然居群中,P的变化范围在68.73%~84.17%之间。按所检测的多态位点丰富程度,7个居群的排序为:霸王岭(BWL)>文昌(WC)>杨梅湾(YMW)>尖峰岭(JFL)>吊罗山(DLS)>石梅湾(SMW)>五指山(WZS)(表 2)。
Nei's基因多样性指数(H)与Shannon多态性信息指数(I)分析结果趋势一致,多样性最高的是文昌居群(H=0.241 9,I=0.372 2),最低的是尖峰岭居群(H=0.186 0,I=0.297 5)(表 2),7个居群多样性指数依次如下:文昌(WC)>霸王岭(BWL)>杨梅湾(YMW)>五指山(WZS)>吊罗山(DLS)>石梅湾(SMW)>尖峰岭(JFL),与多态性位点百分率的排序稍有差别。
2.2 青梅居群的遗传结构根据AMOVA计算结果,海南岛7个青梅居群的遗传分化为0.198 0,即在青梅总的遗传变异中,居群间变异为19.80%,80.20%的遗传变异存在于居群内的个体之间;基因流Nm为1.012 7。这些结果反映了青梅各自然居群内存在着丰富的遗传变异,居群间存在一定的遗传分化。
2.3 居群间遗传距离和地理距离在7个居群中,吊罗山和杨梅湾居群间的遗传相似性最高,遗传距离最小;霸王岭和五指山居群间遗传相似性最低,遗传距离最大,分别位于聚类分支的最外端和次外端(图 2,表 3)。

|
图 2 海南岛青梅基于Nei's遗传距离的UPGMA聚类图 Figure 2 UPGMA dendrogram based on Nei's genetic distance of Vatica mangachapoi on Hainan Island |
|
|
1 000次随机抽样的Mantel test检测表明,居群间遗传距离和空间距离的相关性不明显(P=0.230 0,r2=0.232 5)。
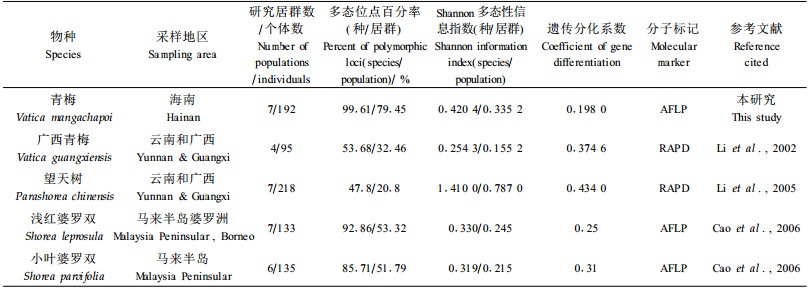
3 讨论 3.1 海南岛青梅的遗传多样性青梅属主要分布于东南亚和中国西南部至南部,全球约65种,我国有3种,其中青梅的分布面积最广,为海南岛与泰国、越南、马来西亚、印度尼西亚和菲律宾共有种;另外2种分别是西藏、印度和缅甸共有的西藏青梅(Vatica lanceifolia)和仅分布于广西和云南的广西青梅(V. guangxiensis),后两者分布区均较窄。目前,有关龙脑香科植物遗传多样性的研究不多,只有娑罗双属(Shorea)(Ng et al., 2006; Cao et al., 2006)、望天树(Parashorea chinensis)和广西青梅(Li et al., 2002;2005)。与2种印度尼西亚广布种——浅红婆罗双(Shorea leprosula)与小叶婆罗双(S. parvifolia)的AFLP遗传多样性(Cao et al., 2006)和2种中国特有植物——广西青梅与望天树的RAPD遗传多样性相比,无论是物种水平还是居群水平,海南岛青梅遗传多样性均较高(表 4)。
|
|
海南岛青梅具有丰富的遗传多样性,这与其自然分布区特点及其生物学、生态学特性是相一致的。
从地理分布特点看,青梅在全球分布范围较广,在海南岛曾有广泛的分布,但由于近半个世纪人为砍伐和破坏,自然居群不断缩小,目前呈片断化分布(杨小波等,2005;陈伟等,2006),但还保留了较丰富的遗传多样性。从生态学特性看,青梅是海南典型热带雨林群落上层林冠的优势种和关键种(胡玉佳,1991;杨小波等,2005;李意德等,2006)。作为林冠层优势立木,青梅具有获取营养和有利繁殖的优势。而且,从青梅在海南岛的生境看,该种在海拔3 ~ 4 m至1 000 m都能见到。青梅林大都生长在红壤中,但在海南岛石梅湾海滨营养贫乏的滨海砂土上也生长着青梅林,反映了该种具有很强的生态适应性(杨小波等,2000)。这些生态学特点和表现与其较高的遗传多样性和遗传适应性是一致的。
研究表明,异交和种子易传播扩散的物种其遗传多样性较高(Clarke et al., 2000;Frankham et al., 2002)。从青梅的生物学和形态学特性看,青梅具圆锥花序,每花序具多数花,柱头高出花药,具有避免自花传粉的结构,且花期长,盛花期来访昆虫频繁;青梅种子成熟后,翅状的宿存花萼能凭借风力传播(李意德等,2006)。青梅在春夏季开花,这也是海南岛的台风季节,台风威力巨大,有利于青梅种实的传播。由此可见,青梅的生物学、形态学特点及其生态环境均有利于其遗传多样性的发展和保持。
3.2 海南岛青梅居群遗传分化与基因流遗传结构是一个物种最基本的特征之一,它受突变、迁移、选择和基因流的共同作用,同时还与物种的进化历史和生物学特性有关。许多研究结果证明,植物类群的分类地位、生活型、分布范围、分布地区、繁育系统、种子扩散方式、生殖模式、演替阶段等因素都会影响到植物居群的分化(Ellstrand et al., 1993; 黄宏文,1998;Mattew et al., 2000)。
青梅种子寿命较短,成熟后只能保持几天的活力,超过70 h基本死亡(刘文明等,1993),但在青梅自然群落中,母树周围通常拥有丰富的青梅幼苗(颜文洪等,2004),反映了青梅种子能在林下自然萌发。但野外调查也发现,群落中能健康成长的青梅幼树和小树较少,反映了在幼苗与幼树之间的生长阶段存在瓶颈环节,可能是受光照及水分在时间和空间上的限制(胡玉佳,1991;陈伟等,2006)。本研究揭示了80.20%的青梅遗传变异存在于居群内,与同科植物相比,其居群内遗传变异较高,居群间遗传分化较小(表 4);与Nybom(2004)基于AFLP标记研究所得出的植物居群平均遗传分化(ϕst=0.35 ±0.18,Gst=0.21±0.14)相比,海南岛青梅居群间的遗传分化(Fst=0.198 0)低于这个水平。认为,造成海南岛青梅目前遗传分化水平的原因除了与其生物学、生态学特性及生境条件有关外,还可能与近世纪来人类破坏活动和本研究取样有关。
本研究所涉及的吊罗山、五指山、文昌和尖峰岭居群均属于湿润性群落;霸王岭居群属于季节性青梅林;石梅湾和杨梅湾居群为滨海单优青梅林(图 1)。从UPGMA聚类图来看,位于海南岛西北部霸王岭和海南岛中部的五指山青梅居群与其他居群的遗传距离较大,而位于海南岛东部及东南部的青梅居群间的遗传距离较小,特别是2个地理位置相近、均生长于滨海沙土上的青梅居群(SMW,YMW)并没有首先聚在一起,而是与位于湿润区的居群聚在一起(图 2),反映了滨海区的青梅居群与湿润区居群所在的群落在历史上曾经相连,只是由于近世纪的人类活动才导致了其居群在生理上的隔离和基因上的分化(梁淑群等,1993;1994;陈伟等,2006)。
3.3 保育建议森林群落多样性的保护,首先要保护好关键种(keystone species),因为一个关键种的兴衰不仅将直接导致10 ~ 20个相关物种的兴衰,而且又与该地区物种的遗传多样性具有积极的相关性(Christian et al., 2006)。青梅是热带常绿季雨林生态系统的关键种,对生态系统的健康与持续发展将起到非常重要的作用,但人类干扰已导致海南岛青梅许多自然居群萎缩和生境片段化,群落结构正在衰退(杨小波等,2005;方洪等,2004;颜文洪等,2004)。特别是文昌、五指山、石梅湾和杨梅湾等几个青梅所在群落面积较小,人为活动频繁。如果现有群落的片断化趋势不能得到有效控制,青梅居群数量的萎缩必然会加剧居群内的近交(inbreeding)及遗传漂变(genetic drift),从而导致更多遗传多样性的丧失和更大的遗传分化(Naito et al., 2005)。
青梅的迁地保护已获得初步成功(杨清等,1995;孟令曾等,2005)。根据作者的研究结果,目前海南岛的青梅具有较高的遗传多样性,东西部居群间存在一定的遗传分化。因此,在迁地保育时,应尽可能收集各居群的种子,同一居群也应选择环境异质性差异较大地段的个体,以保存其最大遗传资源。而对于就地保育措施,应当注意保留全岛不同生境和不同区域的居群,特别是一些特殊生境的个体,即不仅需要保护西部、中部、北部和南部的低地居群,而且也需要保护东部的滨海居群。此外,根据青梅生物学和生态学特性,在林下进行适度人工稀疏等措施,改善林下光照环境,促进群落下层青梅幼苗、幼树的生长,或在林缘或林窗进行人工造林,都是促进现有居群更新和恢复的有效途径。
青梅为优良的热带用材树种,且该树种生长快,易繁殖栽培。在注重青梅物种资源和遗传多样性保育的同时,也应注重发挥其在经济和生态方面的利用价值。建议加强青梅良种选育研究,应用优良种源进行人工造林,扩大青梅的分布面积,不仅可以达到保存青梅种质资源的目的,同时也可促进海南岛热带雨林及其生态系统的恢复和发展。
陈伟, 蒋菊生, 兰国玉. 2006. 海南青梅林生态系统的分布规律、存在问题与管理对策. 热带农业科学, 26(6): 67-70. DOI:10.3969/j.issn.1009-2196.2006.06.019 |
方洪, 李意德, 罗文, 等. 2004. 尖峰岭国家级自然保护区青皮林资源及其垂直分布特征. 热带林业, 32(4): 43-46, 38. DOI:10.3969/j.issn.1672-0938.2004.04.013 |
胡玉佳. 1991. 海南岛青梅种群结构的研究. 中山大学学报:自然科学版, 30(2): 91-97. |
黄宏文. 1998. 保育遗传学与植物遗传资源的保育策略. 武汉植物学研究, 16(4): 346-358. |
李意德, 方洪, 罗文, 等. 2006. 海南尖峰岭国家级保护区青皮林资源与乔木层群落学特征. 林业科学, 42(1): 1-6. |
李善文, 张有慧, 张志毅, 等. 2007. 杨属部分种及杂种的AFLP分析. 林业科学, 43(1): 35-41. |
梁淑群, 林英, 杨小波, 等. 1993. 海南万宁礼纪青梅林. 海南大学学报:自然科学版, 11(4): 1-9. |
梁淑群, 林英, 杨小波, 等. 1994. 海南万宁礼纪青梅林(续). 海南大学学报:自然科学版, 12(1): 14-19. |
刘文明, 宋学之. 1993. 失水对青皮种子劣变的影响. 林业科学研究, 6(2): 162-166. |
孟令曾, 许再富. 2005. 中国龙脑香科植物受胁状况及迁地群落保护探讨. 广西植物, 25(1): 8-13. DOI:10.3969/j.issn.1000-3142.2005.01.002 |
王发国, 叶华谷, 邢福武, 等. 2005. 海南岛亚地带界线划分的探讨. 北京林业大学学报, 27(2): 54-58. DOI:10.3321/j.issn:1000-1522.2005.02.011 |
颜文洪, 胡玉佳. 2004. 海南石梅湾青皮林最小取样面积与物种多样性研究. 生物多样性, 12(2): 245-251. DOI:10.3321/j.issn:1005-0094.2004.02.004 |
杨清, 肖来云, 普正和. 1995. 青梅的迁地保护研究. 热带林业, 23(3): 126-129. |
杨小波, 胡荣桂. 2000. 热带滨海沙滩上森林植被的组成成分与土壤性质的研究. 生态学杂志, 19(4): 6-11. DOI:10.3321/j.issn:1000-4890.2000.04.002 |
杨小波, 吴庆书, 李跃列, 等. 2005. 海南北部地区热带雨林的组成特征. 林业科学, 41(3): 19-24. DOI:10.3321/j.issn:1001-7488.2005.03.004 |
于永福. 1999. 中国野生植物保护工作的里程碑——国家重点保护野生植物名录(第1批). 植物杂志, (5): 3-11. |
Cao C P, Finkeldey R, Siregar I Z, et al. 2006. Genetic diversity within and among populations of Shorea leprosula Miq. and Shorea parvifolia Dyer (Dipterocarpaceae) in Indonesia detected by AFLPs. Tree Genetics & Genomes, 2: 225-239. |
Christian W, Fritz B, Hans-Rolf G. 2006. Is there a trade-off between species diversity and genetic diversity in forest tree communities?. Plant Ecology, 185(2): 151-161. |
Clarke G M, Young A G. 2000. Introduction: genetics, demography and the conservation of fragmented populations//Young A G, Clarke G M. Genetics, demography and viability of fragmented populations. Cambridge: Cambridge University Press, 1-6.
|
Doyle J. 1991. DNA protocols for plants-CTAB total DNA isolation//Hewitt G M, Johnston A, Young J P. Molecular techniques in taxonomy. Berlin: Springer, 283 -193.
|
Ellstrand N C, Elam D R. 1993. Population genetic consequences of small population size: implications for plant conservation. Annual Review of Ecology and Systematics, 24: 217-242. DOI:10.1146/annurev.es.24.110193.001245 |
Frankham R, Ballou J D, Briscoe D A. 2002. Introduction to conservation genetics. Cambridge: Cambridge University Press.
|
Li Qiaoming, Xu Zaifu, He Tianhua. 2002. Ex situ genetic conservation of endangered Vatica guangxiensis (Dipterocarpaceae) in China. Biological Conservation, 106(2): 151-156. DOI:10.1016/S0006-3207(01)00240-3 |
Li Qiaoming, He Tianhua, Xu Zaifu. 2005. Genetic evaluation of the efficacy of in situ and ex situ conservation of Parashorea chinensis (Dipterocarpaceae) in southwestern China. Biochemical Genetics, 43(7/8): 387-406. |
Mattew A G, Pamela S S. 2000. Patterns of genetic variation in rare and widespread plant congeners. American Journal of Botany, 87: 783-792. DOI:10.2307/2656886 |
Miller M P. 1997. Tools for Population Genetic Analysis (TFPGA): Version 1.3. Arizona: Department of Biological Sciences, Northern Arizona University.
|
Morin P A. 2000. Genetic resources: Opportunities and perspectives for the new century. Conservation Genetics, 1(3): 271-275. DOI:10.1023/A:1011561906832 |
Naito Y, Konuma A, Iwata H, et al. 2005. Selfing and inbreeding depression in seeds and seedlings of Neobalanocarpus heimii (Dipterocarpaceae). Journal of Plant Research, 118: 423-430. DOI:10.1007/s10265-005-0245-z |
Nei M.1978. Molecular Evolutionary Genetics. Columbia University Press, New York.
|
Ng K K S, Lee S L, Saw L G, et al. 2006. Spatial structure and genetic diversity of three tropical tree species with different habitat preferences within a natural forest. Tree Genetics & Genomes, 2: 121-131. |
Nybom H. 2004. Comparison of different nuclear DNA markers for estimating intra specific genetic diversity in plants. Molecular Ecology, 13(5): 1143-1155. DOI:10.1111/mec.2004.13.issue-5 |
Oostermeijer J G B, Luijten S H, Den Nijs J C M. 2003. Integrating demographic and genetic approaches in plant conservation. Biological Conservation, 113: 389-398. DOI:10.1016/S0006-3207(03)00127-7 |
Rohlf F J.2000. NTSYS pc2.1: Numerical Taxonomy and Multivariate Analysis System Version 2.1[EB/OL]. New York: Applied Biostatistics Inc. [2000-11-01]. http://www.exetersoftware.com/cat/ntsyspc/ntsyspc.html.
|
Schemske D W, Husband B C, Ruckelshaus M H, et al. 1994. Evaluating approaches to the conservation of rare and endangered plants. Ecology, 75: 584-606. DOI:10.2307/1941718 |
Schneider S, Roessli D, Excoffier L. 2000. Arlequin ver. 2.000: A software for population genetics data analysis. Switzerland: Genetics and Biometry Laboratory, University of Geneva.
|
Vos P, Hogers R, Bleeke R M, et al. 1995. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Research, 23: 4407-441. DOI:10.1093/nar/23.21.4407 |
Wright S. 1931. Evolution in Mendelian populations. Genetics, 16(2): 97-159. |
Yeh F C, Yang R C, Boyle T. 1999. POPGENE. Microsoft Windows-Based Freeware for Population Genetic Analysis Release 1.31. Edmonton: University of Alberta.
|
Zhang D X, Hewitt G M. 2003. Nuclear DNA analyses in genetic studies of populations: practice, problems and prospects. Molecular Ecology, 12(3): 563-584. DOI:10.1046/j.1365-294X.2003.01773.x |
 2008, Vol. 44
2008, Vol. 44