文章信息
- 谭晓风, 陈鸿鹏, 张党权, 曾艳玲, 李魏, 蒋瑶, 谢禄山, 胡孝义, 胡芳名.
- Tan Xiaofeng, Chen Hongpeng, Zhang Dangquan, Zeng Yanling, Li Wei, Jiang Yao, Xie Lushan, Hu Xiaoyi, Hu Fangming.
- 油茶FAD2基因全长cDNA的克隆和序列分析
- Cloning of Full-Length cDNA of FAD2 Gene from Camellia oleifera
- 林业科学, 2008, 44(3): 70-75.
- Scientia Silvae Sinicae, 2008, 44(3): 70-75.
-
文章历史
- 收稿日期:2007-09-24
-
作者相关文章
油茶(Camellia oleifera)是我国南方重要的木本油料树种,用其种子榨取的茶油富含油酸、亚油酸和亚麻酸等不饱和脂肪酸,营养价值较高(胡芳名等, 2006;廖书娟等, 2005)。茶油还是制造优质化妆品的原料,也可作为优质生物柴油使用。在油茶的油脂合成代谢过程中,油酸经硬脂酸脱氢而来,由SAD基因控制;而亚油酸和亚麻酸等多不饱和脂肪酸则是在FAD基因家族的控制下,由油酸经过脱氢形成的。FAD2的主要功能是在油酸的第12和第13位碳原子之间形成双键,生成亚油酸,因此FAD2决定了油酸与亚油酸的比例。2004年,作者以优良无性系湘林1号和湘林4号的近成熟种子构建了高质量的cDNA文库(胡芳名等, 2004;谭晓风等, 2005),并在此基础上又构建了相应的高代表性EST文库(谭晓风等, 2006),从中发现了1条FAD2基因片段,经分析确认为非全长的cDNA序列。分离克隆该基因对于揭示油茶油脂合成的基本规律具有重大的理论意义,对于油茶的分子育种具有重大的实用价值。为得到该基因的全长cDNA序列,采用5′RACE技术(Chenchik et al., 1998)和交错延伸PCR技术开展了以获得油茶FAD2基因全长cDNA的研究。
1 材料与方法 1.1 材料2006年9月下旬,湘林1号油茶的近成熟果实采自湖南省林业科学研究院油茶采穗圃,洗净后于-75 ℃超低温冰箱保存。油茶近成熟种子的cDNA文库为作者所在实验室构建,其相应的EST文库和大肠杆菌DH5α也为作者所在实验室保存。pMD18-T质粒载体、Total RNA Purification System、SMART RACE Kit、Puprep Gel Extraction Kit等分别购自TaKaRa、Invitrogen、Clontech、Ambiogen公司,DEPC、H2O2、Tris、EDTA、无水乙醇、EB、β-巯基乙醇等均为分析纯。
1.2 方法 1.2.1 油茶cDNA文库中FAD2基因的分离将油茶cDNA文库中的FAD2基因的克隆进行活化,对重组子进行T3和T7双向测序,并应用软件Vector NTI 9.0分析测序结果,在网上进行BLAST X比对分析。
1.2.2 油茶种子总RNA提取将超低温保存的湘林1号油茶果实迅速取出,去除果壳和种皮,称质量并记录,在液氮中研磨种仁,用Total RNA Purification System提取油茶种子的总RNA。
1.2.3 RACE扩增与克隆鉴定首先根据文库中FAD2基因的cDNA序列,使用软件Primer Premier 5.0设计1个特异的下游引物GSP:5′-GTGGTGACGGCGGTGACTGTATTTC-3′(上海生工公司合成)。参照SMART-RACE kit说明书上的步骤进行逆转录反应。用Puprep Gel Extraction Kit回收RACE产物,并通过TA克隆法将5′RACE产物连接至载体pMD18-T,转化大肠杆菌DH5α。在含有X-gal和IPTG及氨苄青霉素的LB平板上进行蓝白斑筛选,挑取单菌落于含有相同浓度氨苄青霉素的液体LB中,过夜培养。采用碱式裂解法提取质粒DNA,PCR鉴定重组子,最后对重组子测序。
1.2.4 交错延伸PCR扩增油茶FAD2基因全长cDNA根据EST序列和RACE技术获得的部分序列拼接成的FAD2全长cDNA序列,设计交错延伸PCR引物OE-F1和OE-R1,OE-F2和OE-R2,其中OE-F1和OE-R2分别位于全长cDNA序列的两端,OE-R1和OE-F2位于5′ RACE序列与文库中FAD2基因的cDNA序列的重叠区,且有13 bp的重叠,引物由上海生工公司合成,序列如下:
OE-F1: 5′-TCTAATACGACTCACTATAGGGCAAG-3′;OE-R1: 5′-ACACAACCTTGAAGTGCCCAG-3′;OE-F2: 5′-TTCAAGGTTGTGTCCTCACTGGT-3′;OE-R2: 5′-CTACATGTTCATTACATACCAAAGCC-3′。
用OE-F1和OE-R1扩增5′RACE重组质粒DNA,OE-F2和OE-R2扩增文库中的FAD2基因的质粒DNA,所得产物分别命名为T1和T2,2个反应体系见表 1。反应条件为:94 ℃预变性3 min;94 ℃变性30 s,68 ℃退火30 s,72 ℃延伸3 min,35个循环;72 ℃延伸10 min;4 ℃终止反应。
|
|
PCR反应结束后,电泳检测并分别回收T1和T2。在200 μL无菌离心管中加入1.0 μL Pfu,6.0 μL buffer,6.0 μL dNTP Mix,10.0 μL T1,10.0 μL T2,21.0 μL超纯水后,进行交错PCR。反应条件为:94 ℃预变性5 min;94 ℃变性45 s,55 ℃退火1 min,72 ℃延伸70 s,5个循环;72 ℃,延伸10 min;4 ℃ 5 min终止反应。反应结束后,立刻取出离心管,加入3.0 μL OE-F1,3.0 μL OE-R2,混匀,置入PCR仪中,反应条件为:94 ℃预变性5 min;94 ℃变性45 s,60 ℃退火45 s,72 ℃延伸2 min,35个循环;72 ℃延伸5 min;4 ℃终止反应。最后电泳检测并回收。
将交错延伸PCR产物进行回收、克隆、转化、提取质粒DNA、PCR检测等常规分子生物学操作后,再对重组子进行双向测序。
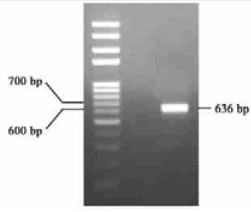
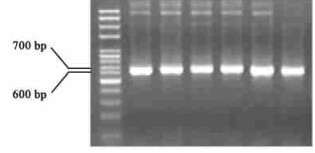
2 结果与分析 2.1 5′RACE产物的克隆与测序RACE反应结束后,对其产物进行电泳检测,获得了图 1的结果。从图中可以看出,DNA条带清晰特异,大小约为640 bp,符合预期目的片段的大小,说明此条带即为RACE的扩增产物。RACE产物转化感受态大肠杆菌后,提取质粒DNA,经PCR检测获得图 2的电泳结果。在图 2中,6个PCR产物在640 bp处均有与RACE产物大小接近的DNA条带,说明随机挑取的6个克隆均为阳性。选取其中1个克隆进行测序,结果显示该片段长636 bp,与原来的cDNA片段重合190 bp,包括5′非编码区203 bp和编码区433 bp,编码N端144个氨基酸残基。通过网上BLAST分析,该片段与其他油料植物的FAD2基因的相似性达到了85%以上,由此确认所得片段为FAD2基因的5′端的部分序列。
|
图 1 RACE产物电泳图 Figure 1 Electrophoresis pattern of 5′ RACE product |
|
图 2 PCR鉴定重组子 Figure 2 Identification of recombinants by PCR |
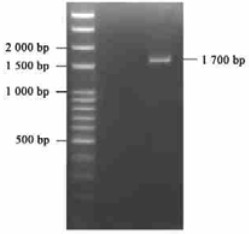
将PCR扩增得到的T1和T2进行电泳检测(图 3)。图 3中,T1条带清晰特异,大小约500 bp,T2在1 200 bp左右处有1条特异条带,这与引物的设计和预期的长度相符。采用交错延伸PCR技术对T1和T2进行重组,获得油茶FAD2基因的全长cDNA,得到图 4的电泳结果。从图 4看出,交错延伸PCR的产物约长1 700 bp,恰好为除去重叠部分以外的T1与T2的长度之和,说明此产物为油茶FAD2基因的全长cDNA。

|
图 3 T1、T2电泳结果 Figure 3 The electrophoresis result of T1 and T2 |
|
图 4 交错延伸PCR产物电泳结果 Figure 4 The electrophoresis result of Overlap Extension PCR |
将5′RACE测序结果与原来文库中的序列进行拼接,得到了1条长1 682 bp的cDNA序列,并且此序列与交错延伸PCR产物的测序结果完全一致,其cDNA序列及推导的氨基酸序列见图 5。序列分析结果表明,该cDNA含有1个1 149 bp的编码框,编码382个氨基酸,5′非编码区长203 bp,3′非编码区长311 bp,有1个长19 bp的polyA尾。FAD2的活性作用位点是特有的3个组氨酸簇(Los et al., 1998; Shanklin et al., 1998)。将油茶FAD2推导的蛋白质序列提交到网上进行结构预测后,发现有3个保守性很高的组氨酸簇——HECGHH、HRHH和HVAHH,分别在此序列的104~109、140~144、316~320的位置,在图 5中分别用红色阴影部分表示。其中第1个和第2个组氨酸簇相隔为30个氨基酸,距离比较短,而第2个与第3个之间的距离较远,这个预测结果与其他不同来源的Δ12-FAD2的氨基酸簇的特征相同,由此可以进一步确认此cDNA序列为Δ12-FAD2的编码序列。

|
图 5 油茶FAD2的cDNA序列与推导的蛋白质序列 Figure 5 cDNA and deduced protein sequences of FAD2 gene from Camellia oleifera ATG是起始密码子,TGA是终止密码子,OE-F1、OE-R1、OE-F2和OE-R2是交错延伸PCR的引物。箭头标注的位置为交错延伸PCR的引物,下划线表示5′RACE产物与文库中FAD2基因cDNA的重叠区,3个组氨酸簇用阴影标注。 ATG is start codon, TGA is stop codon, OE-F1, OE-R1, OE-F2 and OE-R2 are the primers for Overlap Extension PCR. Four arrows mark OE primers, the part underlined is the overlap of RACE product with FAD2 cDNA in the library, and three His clusters are marked by shadow. |
将此序列在NCBI网站上进行Blast X比对分析,并将部分比对结果列于表 2。从表 2中可以看出,油茶的FAD2基因同其他物种的FAD2基因相似性很高,最高达到了88%,并且同油料植物油橄榄(Sesamum indicum)的相似性为87%,推测它们在功能以及结构上可能相似。
|
|
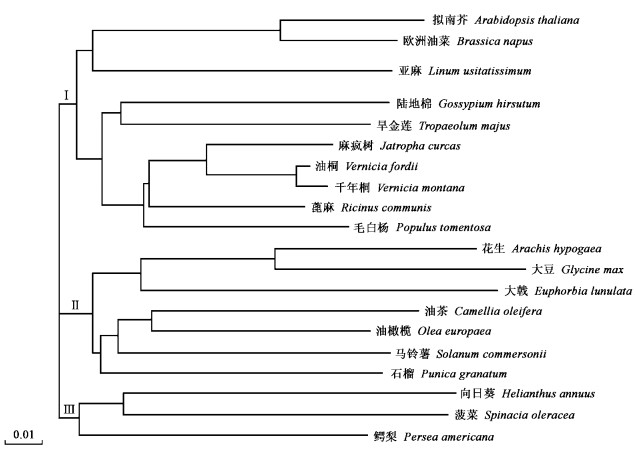
将油茶的FAD2与其他植物的FAD2的蛋白质序列进行亲缘关系分析。结果表明,在FAD2的亲缘关系进化树中,这些物种在遗传进化上主要分为3类,其中,拟南芥、欧洲油菜、亚麻、旱金莲、麻疯树、油桐、千年桐、蓖麻、杨树亲缘关系较近,向日葵、菠菜和鳄梨的亲缘关系较近,花生、大豆、茄子、石榴和油茶等属于第2类,并且油茶与油橄榄的FAD2的亲缘关系最接近(图 6)。
|
图 6 几种植物的FAD2的亲缘关系进化树 Figure 6 Phylogenetic tree based on FAD2 of C. oleifera and other species |
CLONTECH公司的“Touchdown PCR”技术增强了RACE产物的特异性。引物设计是RACE成功的关键。一般采用GC含量(50%~70%)和Tm值(70 ℃以上)较高的引物,才可以进行Touchdown PCR。在用UPM和GSP进行PCR反应时,虽然可能出现非目的条带或无扩增带的情况,但可通过升高或降低退火温度来解决。再经过NUP和NGSP扩增后,扩增产物的特异性会得到进一步的增强。本研究中,通过UPM和GSP扩增即得到非常特异的条带,故省去用NUP和NGSP扩增一步。
交错延伸PCR技术又称重叠延伸PCR,是Horton等(1989)创建的一种技术,并应用该技术成功地构建了一种杂交基因,其原理主要是通过寡聚核苷酸链之间重叠的部分互相搭桥、互为模板进行PCR扩增,从而获得目的DNA基因片段。在重叠延伸PCR反应中,引物的设计是整个试验最重要的一个环节。本研究巧妙地设计了2对引物,尤其是OE-R1与OE-F2有13 bp的反向互补,使得T1和T2有13 bp的重叠区,进而将T1和T2拼接成油茶FAD2基因的全长cDNA。
本研究在已建立的油茶cDNA文库的基础上,根据油茶的FAD2基因片段设计引物,进行5′RACE扩增,获得了油茶FAD2基因cDNA的5′端部分cDNA,利用交错延伸PCR技术将5′RACE扩增片段与原来的cDNA部分片段拼接成油茶FAD2基因的全长cDNA,为油茶FAD2基因的结构和FAD2的功能研究,以及转基因应用奠定了基础。FAD2基因已经从大豆、花生、芝麻等很多植物中克隆得到,但对油茶中FAD2基因的研究本文为首次报道。今后将在此研究的基础上,构建合适的原核表达载体,对目的蛋白进行检测、回收,对其进行酶活性鉴定等。
胡芳名, 谭晓风, 刘惠民, 等. 2006. 中国主要经济树种栽培与利用. 北京: 中国林业出版社, 370-383.
|
胡芳名, 谭晓风, 石明旺, 等. 2004. 油茶种子cDNA文库的构建. 中南林学院学报, 24(5): 1-5. DOI:10.3969/j.issn.1673-923X.2004.05.001 |
廖书娟, 吉当玲, 童荣华. 2005. 茶油脂肪酸组成及其营养保健功能. 粮食与油脂, (6): 7-9. DOI:10.3969/j.issn.1008-9578.2005.06.002 |
谭晓风, 胡芳名, 谢禄山, 等. 2005. 油茶近成熟种子表达的发育相关基因及其分析. 中南林学院学报, 25(4): 17-23. DOI:10.3969/j.issn.1673-923X.2005.04.004 |
谭晓风, 胡芳名, 谢禄山, 等. 2006. 油茶种子EST文库构建及主要表达基因的分析. 林业科学, 42(1): 43-48. |
Chenchik A, Zhu Y Y, Diatchenko L, et al. 1998. Generation and use of high-quality cDNA from small amounts of total RNA by SMART PCR. BioTechniques Books, MA: 305-319.
|
Horton R M, Hunt H D, Ho S N, et al. 1989. Enginnering hybrid gene without the use of restriction enzymes: gene splicing by overlap extention. Gene, 77(1): 61-68. DOI:10.1016/0378-1119(89)90359-4 |
Los D A, Murata N. 1998. Structure and expression of fatty acid desaturases. Biochim Biophys Acta, 1394: 3-15. DOI:10.1016/S0005-2760(98)00091-5 |
Shanklin J, Cahoon E B. 1998. Desaturation and related modifications of fatty acids. Ann Rev Plant Physiol Plant Mol Biol, 49(6): 611-641. |
 2008, Vol. 44
2008, Vol. 44


