文章信息
- 冀宪领, 盖英萍, 马建平, 牟志美.
- Ji Xianling, Gai Yingping, Ma Jianping, Mu Zhimei.
- 桑树景天庚酮糖-1, 7-二磷酸酶基因的克隆、原核表达与植物表达载体的构建
- Cloning and Prokaryotic Expression of the Sedoheptulose-1, 7-Bisphosphatase cDNA from Mulberry and Construction of Plant Expression Vector
- 林业科学, 2008, 44(3): 62-69.
- Scientia Silvae Sinicae, 2008, 44(3): 62-69.
-
文章历史
- 收稿日期:2007-02-27
-
作者相关文章
2. 山东农业大学生命科学学院 泰安 271018
2. College of Life Science, Shandong Agricultural University Tai'an 271018
增加绿色植物光合同化CO2的能力,对提高农作物产量和缓解大气环境中CO2浓度日益升高的威胁有重要意义。因此, 提高光合效率一直是植物学、农业、林业等领域的学者和生产者努力的一个方向(马为民等, 2003)。景天庚酮糖-1, 7-二磷酸酶(SBPase)是卡尔文循环过程中的关键酶,它不可逆地催化景天庚酮糖-1, 7-二磷酸去磷酸化,生成景天庚酮糖-7-磷酸,景天庚酮糖-7-磷酸经过一系列酶促反应转变为CO2的受体核酮糖-1, 5-二磷酸(Rubisco)(Anja et al., 2002)。SBPase活性轻微受抑制, 就会导致卡尔文循环更新能力的减弱、光合活性的下降。因此, SBPase是植物光合作用途径的主要限速酶之一。Miyagawa等(2001)报道, 把聚球藻(Synechococcus sp. PCC7942)中的FBP/SBPase基因转入烟草(Nicotiana tabacum cv. Xanthi)叶绿体中, 在大气中CO2浓度为360 μmol·mol-1条件下,光合效率和Rubisco活性升高,明显提高了转基因烟草光合固定CO2的效率和糖类的累积, 同时也加速了其生长。该研究首次表明了改变一个酶就能显著地提高植物的光合作用。Tamoi等(2006)也证明把SBPase基因转入烟草,转基因植株的生长速度、生长量和光合能力明显高于野生植株。因此,SBPase是一个可以利用基因工程提高光合同化CO2效率的酶。
桑树(Morus)是一种重要的多年生经济林木,桑叶是桑蚕(Bombyx mori)的主要食物来源,桑叶的产量和质量直接影响蚕茧的产量和质量(Jain et al., 2000)。依靠常规的育种方法和栽培措施的改善,已很难使桑叶的产量和质量明显地提高,利用基因工程技术改良桑树品质是当今桑树育种的新方向(潘一乐, 2000)。目前有关桑树SBPase基因的克隆还未见报道。本研究以湖桑32号桑树(Morus alba var. multicaulis)的叶片为材料,利用同源克隆方法和RACE技术成功克隆出SBPase基因全长cDNA,命名为MSBPase并进行了序列分析, 成功地在大肠杆菌中表达了该蛋白;同时构建了MSBPase基因植物表达载体,为利用基因工程提高桑叶的产量和质量提供了有效的候选基因,为深入研究其分子调控机制奠定了基础。
1 材料与方法 1.1 材料叶片取自湖桑32号健康枝条梢部4~6位叶片,液氮速冻后-80 ℃保存备用。质粒载体pMD18-T vector system、TaqDNA聚合酶、dNTP、dCTP和DNA凝胶回收试剂盒、DNA Fragment Purifiction Kit Ver.2.0、反转录酶M-MLV、末端脱氧核糖核酸转移酶(TdT)、X-gal和IPTG均购自大连宝生物工程有限公司。所有引物(表 1)均由上海生物工程公司合成,大肠杆菌(Escherichia coli)菌株DH5α、BL21,原核表达载体pET30a (+)及植物表达载体pBI121由山东农业大学作物生物学国家重点实验室提供。
|
|
取1 g桑树叶片置于预冷的研钵中,加液氮快速研磨成粉末状,利用CTAB法(刘洋等, 2006)提取RNA,以总RNA为模板,以T15为引物,利用M-MLV反转录酶合成cDNA第一链,置-80 ℃保存备用。
1.2.2 MSBPase中间片段的克隆利用BLAST分析NCBI已经登录的SBPase氨基酸序列的保守区域,设计一对兼并引物SBP-1、SBP-2用于扩增MSBPase中间片段。反应条件:94 ℃预变性5 min →(94 ℃变性1 min, 55 ℃退火1 min, 72 ℃延伸1 min),共35个循环后再72 ℃延伸10 min。反应结束后用1.2%的琼脂糖凝胶电泳检查PCR产物,回收目的片段进行克隆测序。
1.2.3 MSBPase的3'RACE根据所克隆到的中间片段测序结果,设计特异引物3SW和3SN,分别与T15和ST作嵌套PCR,扩增目的基因的3'端。取1 μL cDNA做模板,反应条件为:94 ℃预变性5 min →(94 ℃变性1 min, 56 ℃退火1 min, 72 ℃延伸1 min),共35个循环后再72 ℃延伸10 min。反应结束后用1.2%的琼脂糖凝胶电泳检查PCR产物,回收目的片段进行克隆测序。
1.2.4 MSBPase的5'RACE将1.2.1得到的反转录产物,采用DNA Fragment Purifiction Kit Ver.2.0试剂盒纯化,利用TdT进行加尾反应,将反应产物-20 ℃贮存备用。根据已知的中间片段设计特异引物5SW和5SN与G16和GT作嵌套PCR,扩增目的基因的5'端。反应条件为:94 ℃预变性5 min →(94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸1 min),共35个循环后再72 ℃延伸10 min。反应结束后用1.2%的琼脂糖凝胶电泳检测PCR产物,回收目的片段进行克隆测序。
1.2.5 扩增产物的克隆与序列测定用DNA凝胶回收试剂盒回收PCR扩增产物。回收产物与pMD18-T载体进行连接,连接产物转化大肠杆菌DH5α。在含氨苄青霉素的培养基上进行蓝白斑筛选。随机挑取白色菌落,进行菌落PCR鉴定。由上海生物工程公司用测序仪ABIPRISMTM 377 DNA Sequencer进行序列测定。
1.2.6 MSBPase全长序列的获得与序列分析根据已测定片段的序列及其重叠区域,拼出目的基因的全长cDNA,用DNAMAN及DNAClub软件进行序列分析。
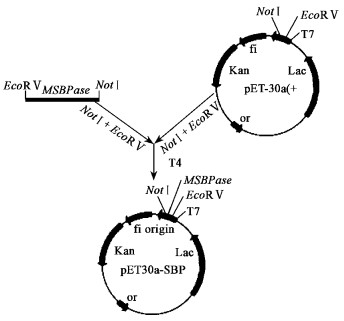
1.2.7 原核表达载体的构建与鉴定根据已克隆的MSBPase核苷酸序列,设计一对引物SBT5和SBT3,扩增MSBPase的编码区,琼脂糖凝胶电泳检测PCR产物,回收目的片段与pMD18-T载体进行连接,连接产物转化大肠杆菌DH5α。筛选阳性克隆测序验证后利用E.Z.N.A.TM Plasmid Mini Kit Ⅰ试剂盒提取重组质粒。利用ECoR V和NotⅠ酶切重组质粒,回收MSBPase片段,将之与同样酶切的载体pET30a (+)连接,转化E.coli BL2l感受态细胞,对重组质粒进行酶切鉴定,选择阳性克隆,命名为pET30a-SBP(图 1)。
|
图 1 原核表达载体pET30a-SBP构建图 Figure 1 Construction of E.coli expression vector pET30a-SBP |
含重组质粒的大肠杆菌BL21菌株接种于5 mL的LB液体培养基中,37 ℃培养过夜,然后按1:25的比例接种l00 mL新鲜的LB液体培养基培养3~4 h,至OD值为0.5左右,加入终浓度1 mmol·L-1的IPTG诱导表达。37 ℃继续培养4 h,收集培养菌体经5 000 r·min-1离心后,弃上清,加入2×SDS-PAGE上样缓冲液混匀。SDS-PAGE电泳检测(10%分离胶,5%浓缩胶)。
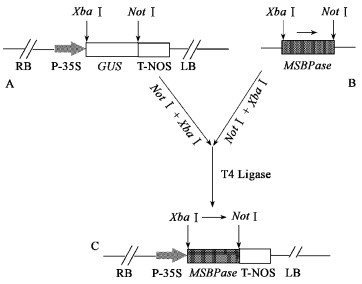
1.2.9 MSBPase植物表达载体的构建与鉴定根据已知的MSBPase核苷酸序列,设计一对引物SBTN5和SBTN3,扩增MSBPase的编码区,琼脂糖凝胶电泳检测PCR产物,回收目的片段与pMD18-T载体进行连接,连接产物转化大肠杆菌DH5α。筛选阳性克隆提取重组质粒。利用XbaⅠ和NotⅠ酶切重组质粒,回收MSBPase片段,与经过同样双酶切的植物表达载体pBI121相连。连接产物转化DH5α大肠杆菌感受态细胞,在含50 mg ·L-1的卡那霉素的LB平板上进行筛选。随机挑取菌落,进行PCR扩增和酶切鉴定,选择阳性克隆,命名为pBI121-SBP(图 2)。
|
图 2 MSBPase植物表达载体构建示意图 Figure 2 Diagram of MSBPase plant expression vector A:空载体pBI121 Plant expression vector pBI121; B:插入MSBPase编码区的pMD18-T载体The pMD18-T with the MSBPase coding region inserted positively; C:在XbaⅠ和NotⅠ位点之间正向插入MSBPase编码区的植物表达载体pBI121 The MSBPase coding region was inserted into the plant expression vector pBI121 at the NotⅠ and XbaⅠ sites positively. |
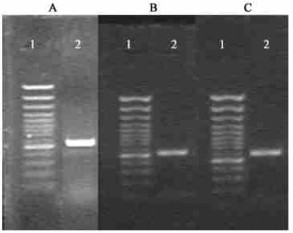
利用一对兼并引物SBP-1和SBP-2,进行RT-PCR,琼脂糖凝胶电泳检测到1条大小约510 bp的条带(图 3A),与所预测的片段大小基本一致。将该片段进行序列测定、同源性比较,发现它与其他植物SBPase基因的核苷酸序列同源性较高,表明在桑树中成功分离到SBPase基因的中间片段。根据已分离到的桑树SBPase基因中间片段设计特异引物3SW和3SN,分别与T15和ST作嵌套PCR,扩增目的基因的3′端,扩增到540 bp左右的片段(图 3B)。对其进行序列分析,表明该片段是目的基因的3′端的序列。根据已分离到的桑树SBPase基因中间片段设计特异引物5SW和5SN分别与G16和GT作嵌套PCR,进行5′RACE,扩增出640 bp左右的片段(图 3C)。进行序列分析表明该片段为目的基因的5′端片段。通过以上三步克隆测序,拼接出桑树SBPase基因(MSBPase)的cDNA全长序列。
|
图 3 桑树SBPase基因MSBPase的扩增 Figure 3 Isolation of MSBPase by RT-PCR from mulberry (A)MSBPase中间片段的PCR扩增Isolation of middle fragment of MSBPase; (B)MSBPase 3′端片段的PCR扩增Isolation of 3′ fragment of MSBPase; (C)MSBPase 5′端片段的PCR扩增Isolation of 5′ fragment of MSBPase; 1:100 bp DNA ladder marker; 2:PCR产物PCR product. |
分离得到MSBPase全长为1 527 bp,阅读框位于133~1 311 bp之间,共编码393个氨基酸(图 4)。将推测出的氨基酸全序列与其他植物同源序列比较发现,该序列与拟南芥(Arabidopsis thaliana)、菠菜(Spinacia oleracea)、小麦(Triticum aestivum)、水稻(Oryza sativa)的SBPase序列高度同源,同源性分别为83.0%、80.9%、75.1%、62.5%(图 4),进一步证明本试验克隆得到的基因为编码桑树SBPase的全长cDNA。由图 4可以看出,在所有的SBPase氨基酸序列中,有5个Cys是保守的,其中的②和③形成CGGT(A/Q)C结构, 这一结构被认为是很多酶的氧化还原活性位点,参与光的调节反应(Fox et al., 1983; Dunford et al., 1998)。由图 4还可以发现,不同植物的SBPase氨基酸序列的N-末端同源性较低,但它们都具有叶绿体蛋白转移肽的共同特征,即含有较多的Ser,较少的Asp、Glu (Fujimori et al., 1998; von Heijne et al., 1989)。因此,由全长cDNA推演出的氨基酸的N-末端可能包含有引导蛋白质由细胞质转移到叶绿体的转移肽序列。利用TARGETP(http://www.cbs.dtu.dk/services/targetp)软件N-末端进行分析,发现其存在一个具有55个氨基酸残基的叶绿体转移肽序列,转移肽的断裂位点应该在Lys55-Ser56区域。MSBPase成熟蛋白应含有338个氨基酸残基,分子量为35~36 ku, 这与前人的研究结果是一致的(Nishizawa et al., 1981; Cadet et al., 1987)。

|
图 4 不同植物来源的SBPase的同源性比较 Figure 4 Alignment of amino acid sequence of MSBPase with those of other plant SBPases 同源比较图用DNAMAN分析软件完成。相同的氨基酸序列用黑色框起;同源的氨基酸序列用灰色框起。序列中的原点代表缺少的氨基酸,保守的半胱氨酸在其上方用数字标出。SBPase的来源为:拟南芥(AAM91137);水稻(AA022559);菠菜(AAB81104);小麦(CAA46507)。 Alignment was performed using the DNAMAN program. Identical amino acids were black shaded and similar amino acids were gray shaded. The dot signs represent the amino acid gaps introduced to maximize similarity. The identical Cys residues were numbered above them. The aligned sequences included those from Arabidopsis thaliana (AAM91137), Oryza sativa (AA022559), Spinacia oleracea (AAB81104), Triticum aestivum (CAA46507). |
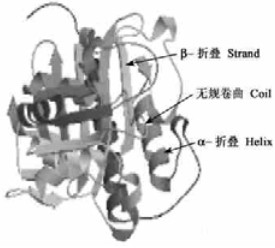
用Antheprot软件对MSBPase编码的蛋白质的基本特性进行分析。结果为:相对分子质量是42.6 ku,等电点为5.85。经信号肽检测证明MSBPase为非分泌性蛋白(non-secretory protein) (http://psort.hgc.jp/form.html)。对MSBPase进行结构预测分析, 结果表明该蛋白富含无规卷曲(coil),高达64.29 %,其次是α-螺旋(helix),为22.19 %,而β-折叠(strand)只有13.52 %(图 5)。MSBPase的2个亚基配对形成一个功能二聚体,每个亚基都是由许多的α-螺旋和β-折叠通过无规卷曲联系在一起折叠形成的(图 6)(http://npsa-pbil.ibcp.fr/cgi-bin/npsa\.automat.pl?page=/NPSA/npsa\.preda.html)。

|
图 5 MSBPase的二级结构预测分析 Figure 5 Secondary structure prediction of MSBPase h:α-螺旋Helix; c:无规卷曲Coil; e:β-折叠Strand. |
|
图 6 MSBPase的三维结构预测 Figure 6 Predicted tridimensional structure of MSBPase |
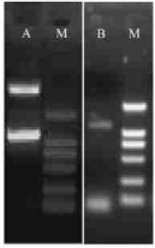
筛选出的阳性克隆的插入序列经测序证明为MSBPase的编码区(数据未附)。利用ECoRⅤ和NotⅠ酶切重组质粒,回收MSBPase片段,将之与同样酶切的载体pET30a (+)连接,转化E.coli BL2l感受态细胞,选择阳性克隆,命名为pET30a-SBP。对pET30a-SBP进行酶切鉴定和PCR分析,均可获得1条1 200 bp左右的片段,长度与MSBPase cDNA编码区片段相同(图 7),经测序证实阅读框没有发生碱基突变和移码错位。因此可以确定目的基因已整合到pET30a (+)载体上。
|
图 7 原核表达载体pET30a-SBP的酶切和PCR鉴定 Figure 7 Identification of expression vector of pET30a-RCT by restriction enzyme digestion and PCR M:DNA分子标准2000 DNA marker 2000; A:酶切产物Restriction enzyme digestion product; B: PCR产物PCR product. |
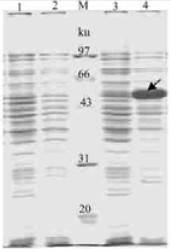
重组质粒pET30a-SBP转入大肠杆菌BL21后,用IPTG诱导其表达,SDS-PAGE结果(图 8)显示,诱导培养后的菌体蛋白与未诱导的对照菌、空载体转化菌相比,有1条非常明显的表达增强蛋白条带,证实所构建的基因重组菌经诱导能够表达外源蛋白。参照相对分子量标准,初步测定目的蛋白相对分子质量约51 ku,与预期相符。目的蛋白组分主要集中于菌体沉淀中,破菌后电泳证实目的蛋白主要以包涵体形式表达。
|
图 8 SDS-PAGE电泳分析融合蛋白MSBPase在大肠杆菌BL21中的表达 Figure 8 SDS-PAGE analysis of the expression of recombinant protein MSBPase in E.coli BL21 1:IPTG诱导的含pET30a (+)的BL21菌体pET30a(+)-transformed BL21with IPTG induction; 2:未经IPTG诱导的含pET30a (+)的BL21菌体pET30a (+)-transformed BL21without IPTG induction; 3:未诱导的含pET30a-SBP的BL21菌体pET30a-SBP transformed BL21 without IPTG induction; 4:IPTG诱导的含pET30a-SBP的BL21菌体pET30a-SBP transformed BL21with IPTG induction; M:标准分子量蛋白Protein marker; 箭头所示为表达的融合蛋白The arrow point to the expression recombinant protein. |
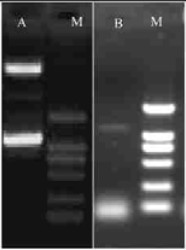
筛选出的阳性克隆的插入序列经测序证明为MSBPase编码区(数据未附)。利用XbaⅠ和NotⅠ酶切重组质粒,回收MSBPase片段,将之与经过同样双酶切的植物表达载体pBI121相连。连接产物转化DH5α大肠杆菌感受态细胞,选择阳性克隆命名为pBI121-SBP。对pBI121-SBP进行酶切和PCR分析鉴定,均可获得1条1 200 bp左右的片段,长度与MSBPase cDNA编码区片段相同(图 9),经测序证实阅读框没有发生碱基突变和移码错位。因此,可以确定目的基因已整合到pBI121载体上。
|
图 9 植物表达载体pBI121-SBP的PCR和酶切鉴定 Figure 9 Identification of plant expression vector of pBI121-SBP by PCR and restriction enzyme digestion M:DNA分子标准2000 DNA marker 2000; A:酶切产物Restriction enzyme digestion product; B:PCR产物PCR product. |
目前已从小麦、拟南芥和菠菜等多种植物中分离得到SBPase基因及其cDNA,不同植物的SBPase包含了不同数目和长度的外显子和内含子(Raines et al., 1992; Willingham et al., 1994; Hahn et al., 1998)。拟南芥和衣藻的SBPase在基因组中都是单拷贝,但由于存在基因复制,在不同倍性的生物体基因组中SBPase的拷贝数较为复杂(Raines et al., 2000)。在对SBPase结构的研究中发现拟南芥SBPase的上游序列中含有2个与Ⅰ-box同源的GATA框,这是一段存在于许多光调节基因启动子区域的保守DNA序列(Willingham et al., 1994; Gilmartin et al., 1990);在小麦的SBPase的启动子内具有一个与核内WF1 DNA结合蛋白作用的结合位点(Katagiri et al., 1992)。通过对该酶基因结构的研究,有助于了解其表达调控机制。SBPase基因的表达调控与卡尔文循环中其他核基因编码的酶一样,主要是在转录水平上进行的,受到光、植物生长发育和己糖水平的调节(Willingham et al., 1994; Jones et al., 1996)。Raines等(1999)认为可能存在一个共同的未知因子,控制着这些基因的转录,但其具体的机制目前还不清楚。利用转基因技术控制植株的SBPase活性,可以进一步研究该酶的作用及其催化活性与植物各种代谢的关系,有助于阐明SBPase的作用机制。本研究成功克隆出SBPase基因全长cDNA,构建了原核表达载体, 成功地在大肠杆菌中表达了该蛋白;同时构建了MSBPase基因植物表达载体,为深入研究该基因的作用机制奠定了基础。
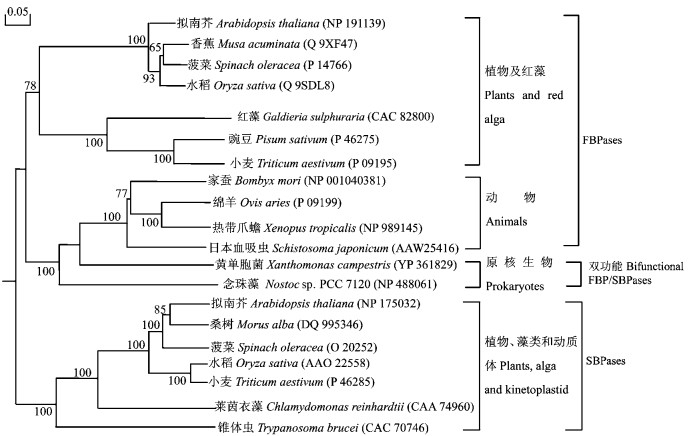
FBPase是调控光合和PO43-循环的关键酶之一,该酶催化一个不可逆的反应,使果糖-1, 6-二磷酸(FBP)转变成果糖-6-磷酸和无机磷酸(Pi)(唐功利等, 2001)。FBPase/SBPase是从聚球藻(Synechococcus sp. PCC7942)中分离出来的一种酶,它能催化FBP或景天庚酮糖-1, 7-二磷酸(SuBP)去磷酸化反应,该酶促反应有助于Calvin循环中原碳受体——核酮糖-1, 5-二磷酸(RuBP)的更新。这一反应在高等植物中是被2个独立的酶FBPase和SBPase所催化(马为民等, 2003)。对GenBank数据库中不同物种来源的SBPase、FBPase和FBPase/SBPase的蛋白质氨基酸序列进行BLAST(http://www.ncbi.nlm.nih.gov/BLAST/)分析发现,所有物种的SBPase基因有一个共同的进化起源。不同物种的SBPase、FBPase和FBPase/SBPase蛋白氨基酸序列具有一定的同源性(28%~32%)(图 10)。这些保守的氨基酸序列可能反映出这3种酶在功能或结构上的相似性,同时也表明这3种酶可能具有共同的进化起源,即SBPase和FBPase可能是由兼有这2种酶功能的FBPase/SBPase经过长时间的进化分化而来(Hannaert et al., 2003),其具体的分化历程以及保守氨基酸残基在维持酶的结构和功能上的作用值得探索和研究。
|
图 10 Neighbor-joining法构建的SBPases、FBPases、FBP/SBPase氨基酸序列系统进化树 Figure 10 Molecular phylogenetic tree of the amino acid sequences of SBPases, FBPases and FBP/SBPases constructed by neighbor-joining method 括号内为所引蛋白氨基酸序列的GenBank登录号。 The GenBank accession numbers for nucleotide sequence data were shown in the brackets. |
刘洋, 何心尧, 马红波, 等. 2006. 用CTAB-PVP法提取棉花各组织总RNA的研究. 中国农业大学学报, 11(1): 53-56. DOI:10.3321/j.issn:1007-4333.2006.01.011 |
马为民, 施定基, 王全喜. 2003. 用基因工程提高光合同化CO2效率的一个关键酶——果糖-1, 6-二磷酸酶. 生物化学与生物物理进展, 30(3): 446. DOI:10.3321/j.issn:1000-3282.2003.03.034 |
潘一乐. 2000. 桑种质资源和桑树育种的研究现状与展望. 蚕业科学, 26(增刊): 1-5. |
唐功利, 杨春松, 鲍建绍, 等. 2001. 丙糖磷酸异构酶、果糖-1, 6-二磷酸醛缩酶及果糖-1, 6-二磷酸酶的共表达. 生物化学与生物物理学报, 33(1): 131-136. |
Anja S, Marco B, Rüdiger H. 2002. Overexpression of the potential herbicide target sedoheptulose-1, 7-bisphosphatase from Spinacia oleracea in transgenic tobacco. Molecular Breeding, 9: 53-61. DOI:10.1023/A:1019297521424 |
Cadet F, Meunier J C, Ferte N. 1987. Isolation and purification of chloroplastic spinach (Spinacia oleracea) sedoheptulose-1, 7-bisphosphatase. Biochem J, 241: 71-74. DOI:10.1042/bj2410071 |
Dunford R P, Catley M A, Raines C A, et al. 1998. Expression of wheat sedoheptulose-1, 7-bisphosphatase in E.coli and affinity purification of functional protein. Protein Expression and Purification, 14: 139-145. DOI:10.1006/prep.1998.0939 |
Gilmartin P M, Sarokin L, Memelink J, et al. 1990. Molecular light switches for plant genes. Plant Cell, 2: 369-378. DOI:10.1105/tpc.2.5.369 |
Fox B S, Walsh C T. 1983. Mercuric reductase: homology to glutathione reductase and lipoamide dehydrogenase. Iodoacetamide alkylation and sequence of the active site peptide. Biochemistry, 22: 4082-4088. DOI:10.1021/bi00286a014 |
Fujimori K S, Tada S K, Ohta D. 1998. Molecular cloning and characterization of the gene encoding N′--[(5′-phosphoribosyl)-formimino]-5-aminoimidazole-4-carboxamide ribonucleotide (BBM Ⅱ) isomerase from Arabidopsis thaliana. Mol Gen Gent, 259: 216-223. DOI:10.1007/s004380050807 |
Hannaert V, Saavedra E, Duffieux F, et al. 2003. Plant-like traits associated with metabolism of Trypanosoma parasites. PNAS, 100: 1067-1071. DOI:10.1073/pnas.0335769100 |
Hahn D, Kaltenbach C, Kuck U. 1998. The Calvin cycle enzyme sedoheptulose-1, 7-bisphosphatase is encoded by a light-regulated gene in Chlamydomonas reinhardtii. Plant Mol Biol, 36: 929-934. DOI:10.1023/A:1005911022601 |
Jain A K, Vincent R M, Nessler C L. 2000. Molecular characterization of a hydroxymethyl-glutaryl-CoA reductase gene from mulberry (Morus alba L.). Plant Molecular Biology, 42: 559-569. DOI:10.1023/A:1006336825226 |
Jones P G, Lloyd J C, Raines C A. 1996. Glucose feeding of intact wheat plants represses the expression of a number of Calvin cycle genes. Plant Cell Environ, 19: 231-236. DOI:10.1111/pce.1996.19.issue-2 |
Katagiri F, Chua N H. 1992. Plant transcription factors—present knowledge and future challenges. Trends in Genetics, 8: 22-27. DOI:10.1016/0168-9525(92)90020-5 |
Miyagawa Y, Tamoi M, Shigeoka S. 2001. Overexpression of a cyanobacterial fructose-1, 6/-sedoheptulose-1, 7-bisphosphatase in tobacco enhances photosynthesis and growth. Nature Biotech, 19: 965-969. DOI:10.1038/nbt1001-965 |
Nishizawa A N, Buchanan B B. 1981. Enzyme regulation in C4 photosynthesis. Purification and properties of thioredoxin-linked fructose bisphosphatase and sedoheptulose bisphosphatase from corn leaves. J Biol Chem, 256: 6119-6126. |
Raines C A, Lloyd J C, Willingham N M, et al. 1992. cDNA and gene sequences of wheat chloroplast sedoheptulose-1, 7-bisphosphatase reveal homology with fructose-1, 6-bisphosphatases. Eur J Biochem, 205: 1053-1059. DOI:10.1111/ejb.1992.205.issue-3 |
Raines C A, Harrison E P, Ölçer H, et al. 2000. Investigating the role of the thiol-regulated enzyme sedoheptulose-1, 7-bisphosphatase in the control of photosynthesis. Physiologia Plantarum, 110: 303-308. DOI:10.1034/j.1399-3054.2000.1100303.x |
Raines C A, Lloyd J C, Dyer T. 1999. New insights into the structure and function of sedoheptulose-1, 7- bisphophatase: an important but neglected Calvin cycle enzyme. Journal of Experimental Botany, 50: 1-8. |
Tamoi M, Nagaoka M, Miyagawa Y, et al. 2006. Contribution of fructose-1, 6-bisphosphatase and sedoheptulose-1, 7-bisphosphatase to the photosynthetic rate and carbon flow in the Calvin cycle in transgenic plants. Plant Cell Physiol, 47: 380-390. DOI:10.1093/pcp/pcj004 |
von Heijne G, Steppuhn J, Herrmann R G. 1989. Domain structure of mitochondrial and chloroplast targeting peptides. Eur J Biochem, 180: 535-545. DOI:10.1111/ejb.1989.180.issue-3 |
Willingham N M, Lloyd J C, Raines C A. 1994. Molecular cloning of the Arabidopsis thaliana sedoheptulose- 1, 7-bisphosphatase gene and expression studies in wheat and Arabidopsis thaliana. Plant Mol Biol, 26: 1191-1200. DOI:10.1007/BF00040699 |
 2008, Vol. 44
2008, Vol. 44