文章信息
- 谢一青, 李志真, 黄儒珠, 肖祥希, 黄勇.
- Xie Yiqing, Li Zhizhen, Huang Ruzhu, Xiao Xiangxi, Huang Yong.
- 武夷山不同海拔光皮桦种群遗传多样性及其与生态因子的相关性
- Genetic Diversity of Betula luminifera Populations at Different Altitudes in Wuyi Mountains and Its Association with Ecological Factors
- 林业科学, 2008, 44(3): 50-55.
- Scientia Silvae Sinicae, 2008, 44(3): 50-55.
-
文章历史
- 收稿日期:2006-12-25
-
作者相关文章
2. 福建师范大学生命科学学院 福州 350007
2. College of Life Science, Fujian Normal University Fuzhou 350007
光皮桦(Betula luminifera)隶属于桦木科(Betulaceae)桦木属(Betula),为亚热带中部桦木属的代表种,是中国特有分布的珍贵阔叶树种(郑万钧,1985;吴子诚等,1996)。由于光皮桦具有生长快、材质优异、适应性强、落叶量大、林内菌根丰富、林下植被多样等特点,不仅是航空、建筑、高级家具的优质用材和优良的纸浆原料,而且对土壤改良和生态环境的改善作用也十分明显,近几年在我国南方地区广为栽种,特别是在裸地荒坡的绿化和恢复生态中应用较广。对光皮桦的生长特性(董建文等,2000)、育苗造林(谢芳等,2000)、群落特征(李建民,2000)和综合利用(周志春等,2003)等方面已有较多研究,但未见其分子生态学方面的报道。本研究选择武夷山国家级自然保护区不同海拔生态环境下的光皮桦野生种群为研究对象,采用RAPD技术对其遗传多样性进行研究,探讨其遗传多样性随环境因子的变化规律,以进一步揭示与光皮桦种群遗传多样性相关联的生态因子和对光皮桦种群遗传分化起决定作用的因素,为光皮桦种质资源的保护、遗传改良和永续利用提供理论依据。
1 材料与方法 1.1 材料试验材料采自福建省武夷山国家级自然保护区黄岗山(117°47′18″ E, 27°51′42″ N),按不同海拔设置含光皮桦组分的4个典型样地,各样地的生态因子如表 1所示。其中,土壤全氮、碳氮比用全碳氮分析仪NC-80测定,土壤有机质含量采用容量分析法测定并经计算获得,海拔由GPS直接测得,气象数据(年降水和年均温)由保护区先锋岭气象观测站资料计算而得。为避免同一家系而产生的试验偏差,每个样地随机选择相邻间距30 m以上的光皮桦成熟植株采样。在植株上采集当年生枝条上的嫩叶,用改进的饱和NaCl-CTAB溶液(李志真等,2006)保存并带回实验室,供DNA提取。
|
|
基因组DNA提取参考Zeng等(2002)的CTAB法并针对光皮桦材料特点进行改良。从改进的饱和NaCl-CTAB溶液保存的光皮桦叶片中提取单株植物基因组总DNA(谢一青等,2006)。所提DNA样品的纯度和含量用紫外吸收和凝胶电泳检测。PCR反应在PCR扩增仪2720型(ABI公司)上进行。扩增反应总体积为20 μL,内含DNA模板30 ng、10×PCR缓冲液2 μL、MgCl22.5 mmol·L-1、引物0.4 μmol·L-1、dNTP 0.2 mmol·L-1、Taq DNA聚合酶1 U。反应程序为:95 ℃预变性300 s;94 ℃变性60 s,40 ℃复性90 s,72 ℃延伸120 s,40个循环;72 ℃延伸300 s。随机引物为Sangon公司生产的10碱基寡聚核苷酸。扩增产物以GeneRuler 100 Ladder(MBI公司)作Marker,经1.5%琼脂糖凝胶电泳、染色后,在UVP凝胶成像系统下观察拍照。将能够扩增出分离、清晰且重复性好的引物用于正式试验。每个引物扩增2次,以保证结果的可重复性。
1.3 数据处理1) 种群遗传多样性指数和分化指数计算 RAPD扩增产物按条带的有无分别赋值,有带的记为1,无带的记为0,建立0/1二元数据;根据所得数据矩阵,利用POPGENE 32软件(Yeh et al., 1999)中dominant(显性)-diploid(二倍体)计算多态位点百分比(PPL)、观测等位基因数(Na)、期望等位基因数(Ne)、种群的Shannon多样性信息指数(I)和Nei’s基因多样性(h)等遗传多样性指数;根据POPGENE 32计算所得的种群平均多样性指数和所有样品总多样性指数,得到Nei’s遗传分化系数[Gst=(hT-hS)/hT]和Shannon遗传分化系数[(IT-IS)/IT],式中,hS和IS分别为种群平均Nei’s和Shannon多样性指数,hT和IT分别为总Nei’s和Shannon多样性指数;同时采用AMOVA-PREP1.01(Miller, 1997)和WINAMOVA软件(Excoffier, 1995)进行分子变异分析(AMOVA),统计种群内和种群间的遗传分布,并做差异显著性检验。
2) 相关性分析用SPSS 11.0中的Pearson相关分析,计算RAPD分析所得光皮桦种群内遗传多样性指数与生态因子间的相关关系;再利用Mantel检验对遗传距离矩阵与海拔距离矩阵(两点之间的垂直距离)、生态因子的分异[将不同样地各种生态因子数据标准化后,计算所得的两两之间的欧式距离(Knapp et al., 1998)]之间的相关关系进行检验。
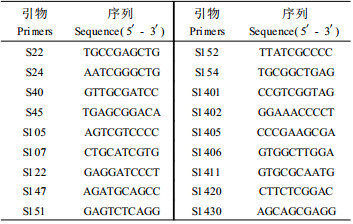
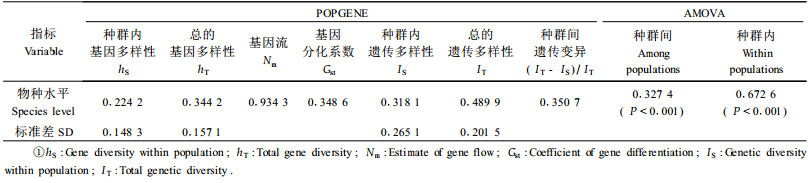
2 结果与分析 2.1 光皮桦种群遗传多样性对Sangon公司的139个10碱基寡核苷酸引物进行筛选,共选择18个扩增条带清晰、重复性好、多态性高的引物(表 2)用于4个不同海拔种群91个光皮桦DNA样品的扩增。18条引物共检测到199个位点,不同引物的扩增位点从8到14不等,位点分子量范围在200~3 000 bp之间。在物种水平,199个位点中多态位点百分比(PPL)占87.44%,总的Shannon多样性指数IT=0.489 9,总的Nei’s基因多样性指数hT=0.344 2(表 3),与同属的西南桦(Betula alnoides)(Zeng et al., 2003)等一些植物的遗传多样性水平相当,也高于一般针阔叶树种的估计值(葛颂,1988),说明武夷山光皮桦具有较高的遗传多样性,这可能与其生物学特性及生活习性有关。光皮桦为雌雄同株的长寿命多年生树种,在相当长的时间内可以保持遗传多样性,且该群落位于保护区内得到了较好的保护,因此其本身可能就具有较高的遗传多样性基础。
|
|
|
|
在种群水平上,不同海拔光皮桦种群的平均遗传多样性统计如下:PPL为60.05%,I=0.318 1,h=0.224 2,Na=1.633 9,Ne=1.389 3(表 3)。但由于不同海拔生境的差异,在PPL、I、h等种群遗传多样性参数上有着显著的不同,综合各种群的多样性参数值,可以看出,不同种群的各多样性指数呈现出相同的变化规律,即低海拔的Pop1种群(PPL=65.33%,I=0.348 4,h=0.241 2)>Pop2种群>Pop3种群>高海拔的Pop4种群(PPL=53.77%,I=0.280 6,h=0.206 7),表现为低海拔的种群遗传多样性程度大于高海拔种群的遗传多样性程度。
2.2 种群间遗传结构从表 4的AMOVA分析结果可看出,光皮桦种群有32.74%的遗传变异发生在种群间,显著性检测表明这种分化较显著,67.26%的遗传变异发生在种群内,这与表 4中基于POPGENE 32估算所得的Nei’s遗传分化系数Gst为34.86%和Shannon多样性指数为35.07%相一致。综合以上3种方法的估算结果可看出,约1/3的总变异存在于光皮桦种群之间,而多数变异存在于种群内,这与我国濒危树种马褂木(Liriodendron chinense)(李建民等,2002)、银杉(Cathaya argyrophylla)(汪小全等,1997)等相似,而与广布性的树种相异。由表 4还可知,由Gst估算的基因流(Nm)为0.934 3,说明种群间的基因流较小。根据Wright(1951)的观点,若每代迁入个体数Nm<1,则遗传漂变可以导致种群间明显的遗传分化。因此,低水平的基因流和遗传漂变可能是导致光皮桦不同海拔种群间产生较大遗传分化的原因。
|
|
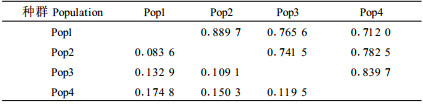
基于Nei’s遗传相似度(种群间RAPD片段共享性)和遗传距离可进一步分析种群间的遗传分化程度。武夷山不同光皮桦种群中(表 5),以海拔580 m的Pop1种群和海拔1 250 m的Pop4种群间的遗传相似度最低(0.712 0),相应地其遗传距离最远(0.174 8);而海拔580 m的种群Pop1和海拔750 m的Pop2种群间遗传相似度最高(0.889 7),遗传距离最小(0.083 6),说明不同海拔种群间存在一定的遗传分化,且海拔隔离越大的种群,其种群间的遗传多样性差异越明显。
|
|
运用SPSS 11.0中的Pearson相关将表 3中RAPD分析所得的光皮桦种群内遗传多样性指数与种群所在生境的生态因子作相关性分析(表 6),结果表明:5个多样性指数与海拔、土壤碳氮比之间存在显著(P<0.05)或极显著(P<0.01)的负相关关系;部分遗传多样性指数与气候因子(年均温和年降水)、土壤全氮及有机质含量存在显著的相关性,其中各个多样性指数与年均温呈正相关,而与其他生态因子则均呈负相关关系;5个多样性指数与土壤pH值呈负相关关系,但并不显著。由此说明海拔或土壤养分等单一或者几个生态因子的组合对光皮桦种群遗传多样性起一定的决定作用,也即生态因子对光皮桦RAPD多态性具有选择压力,RAPD多态性的变化与生态因子变量有关。这同利用RAPD方法对大针茅(Stipa grandis)种群(赵念席等,2004)、野生二粒小麦(Triticum dicoccoides)种群(Fahima et al., 1999)等其他植物的研究结果相似。
|
|
将不同海拔光皮桦种群各样地的生态因子标准化后,利用Mantel检验分别计算它们与种群遗传距离的相关关系,结果见表 7。由表 7可知,种群间Nei’s无偏遗传距离与海拔差距之间存在显著相关关系,说明海拔高度对光皮桦种群间遗传分化也有一定的影响,刘登义等(2003)、李丹等(2001)也得出了相似的结论。从表 7还可看出,不同海拔光皮桦种群间的Nei’s无偏差遗传距离与土壤养分因子(全氮、碳氮比、有机质含量)之间也存在显著的相关性,表明全氮、碳氮比及有机质含量等生态因子对种群间的分化均有显著影响。以上结果说明光皮桦种群间遗传分化显著实际上是多因子综合作用的结果。
|
|
从分子标记检测结果看,武夷山光皮桦天然群落具有较高的遗传多样性(PPL=87.44%,I=0.489 9,h=0.344 2),但野外调查发现其天然林内幼苗难更新成活,林下几无实生苗,年龄结构多为衰退型,据此推测现存较高的遗传多样性可能只是一种暂时现象,如不及时制定有效的保育策略,遗传多样性将无法长久保存;在种群水平,不同海拔光皮桦种群间遗传变异水平呈规律性变化,表现为水热及土壤条件配合较好的、处在海拔580 m种群的遗传变异水平较高(PPL=64.82%,h=0.271 2,I=0.378 4),随着海拔升高到750 m和980 m时,遗传变异水平降低,而当海拔继续升高到1 250 m时,其遗传变异水平更低(PPL=53.17%,h=0.220 7,I=0.320 6)。由此可见,海拔与光皮桦遗传变异水平高低有密切关系,随着海拔的升高,光皮桦遗传变异水平呈由高到低的分布。在本文的研究尺度上,这种海拔变化梯度主要由微地形地貌的变化来决定。如果将研究区域扩大到整个武夷山脉乃至整个分布区,那么距离和海拔因子对光皮桦群落的影响可能会相应增大。这有待于在今后的研究中进一步验证。
相关分析进一步表明,光皮桦遗传多样性指数随海拔而表现出的分布规律与随着海拔而表现出的生态环境因子的变化是密切相关的。种群内的遗传多样性与海拔、气候因子(年均温和年降水)以及土壤养分(全氮、碳氮比、有机质含量)因子呈极显著或显著的相关关系,说明海拔、土壤养分以及气候因子等可能在维持光皮桦种群遗传多样性方面起着较为重要的作用。本研究中,光皮桦种群间遗传分化与海拔距离、生态因子分异之间也存在显著的相关关系,由此可见,海拔、生境的变化等因素不仅影响光皮桦的遗传多样性,而且还影响其种群间的遗传分化。
遗传结构分析表明,武夷山光皮桦种群间的遗传分化程度较高,约2/3的遗传变异存在于种群内,种群间仅占1/3左右。植物种群间遗传分化主要受基因突变、地理距离、基因流、遗传漂变、自然选择及其生物学特性等因素的影响(Volis et al., 2001;Schaal et al., 1998)。其中,基因突变的发生几率很低,常常不被认为是引起种群间分化的主要因素。试验中的4个采样点地理距离之间相差不大,并且采样严格控制在群落内,随机采样,避免了群落边缘和林窗效应,因此光皮桦种群间遗传分化也不会是地理距离造成的。其次,光皮桦属阳性树种,对光的要求较严,在天然群落中多居林冠上层,自然更新能力弱,加上生境的片段化,这样就造成了现存的光皮桦个体多为成年老树的现象,因此其生物学特性可能是影响了种群间的遗传分化的一个潜在原因。此外,Volis等(2001)认为,如果植物种群间的遗传分化是由于种群间的遗传漂变引起的,那么种群间的遗传分化将不会与生态因子之间存在显著的相关关系。而在本研究中,武夷山光皮桦种群间的遗传分化与生态因子之间存在显著的相关关系,说明种群间的遗传分化不是由遗传漂变引起的,但受不同海拔区域小生境的自然选择压力的影响。总之,导致武夷山光皮桦天然群体间遗传分化大的原因除了其生物学特性外,还可能与不同海拔小生境的自然选择作用及种群间缺乏有效的基因流有关。至于是否还有其他原因还值得进一步探讨。
虽然目前没有被国家列为重点保护植物,但光皮桦是我国特有的具有重要经济价值的物种,保护好其现有遗传资源或种质,对进一步利用其遗传资源,解决光皮桦生产中的实际问题具有重要的意义。武夷山不同海拔区域生境的差异对光皮桦群体遗传多样性和遗传分化的影响较大,因此在今后的野生种质遗传资源保护和育种实践中,应注重不同海拔高度光皮桦野生种群的科学保护,同时还要有针对性地对其不同海拔生境的野生种质资源进行引种栽培和遗传改良,培育出适宜于各种生态环境下栽培的新品种。
董建文, 陈东阳, 赵大洲, 等. 2000. 天然光皮桦生长特性研究. 吉林林学院学报, 16(2): 76-78, 86. |
葛颂. 1988. 同功酶与林木群体遗传变异研究. 南京林业大学学报, 12(1): 68-77. |
李丹, 彭少麟. 2001. 三个不同海拔梯度马尾松种群的遗传多样性及其与生态因子的相关性. 生态学报, 21(3): 415-421. DOI:10.3321/j.issn:1000-0933.2001.03.011 |
李建民. 2000. 光皮桦天然林群落特征研究. 林业科学, 36(2): 122-124. DOI:10.3321/j.issn:1001-7488.2000.02.020 |
李建民, 周志春, 吴开云, 等. 2002. RAPD标记研究马褂木地理种群的遗传分化. 林业科学, 38(4): 61-66. DOI:10.3321/j.issn:1001-7488.2002.04.010 |
李志真, 谢一青, 黄儒珠, 等. 2006. 不同保存方法对光皮桦总DNA提取效果的影响. 分子植物育种, 4(1): 131-134. DOI:10.3969/j.issn.1672-416X.2006.01.022 |
刘登义, 沈浩, 杨月红, 等. 2003. 黄山花楸种群遗传多样性研究. 应用生态学报, 14(12): 2141-2144. DOI:10.3321/j.issn:1001-9332.2003.12.011 |
汪小全, 韩英, 邓峥嵘, 等. 1997. 松科系统发育的分子生物学证据. 植物分类学报, 35(2): 97-100. |
吴子诚, 王乐辉. 1996. 光皮桦选择群体特征和改良技术. 四川林业科技, 17(4): 17-28. |
谢芳, 李建民. 2000. 光皮桦育苗和造林实用技术. 林业科技开发, 14(6): 44-45. |
谢一青, 黄儒珠, 李志真, 等. 2006. 光皮桦RAPD分析体系优化设计方案比较. 福建林学院学报, 26(1): 73-77. DOI:10.3969/j.issn.1001-389X.2006.01.017 |
赵念席, 高玉葆, 王金龙, 等. 2004. 蒙古中东部草原大针茅种群遗传分化研究. 生态学报, 24(10): 2178-2185. DOI:10.3321/j.issn:1000-0933.2004.10.013 |
郑万钧. 1985. 中国树木志:第二卷. 北京: 中国林业出版社, 2113-2131.
|
周志春, 李建民, 陈炳星, 等. 2003. 几种亚热带速生乡土阔叶树种的制浆特性评价. 中国造纸, 22(2): 8-12. DOI:10.3969/j.issn.0254-508X.2003.02.003 |
Excoffier L. 1995. AMOVA 1.55 (Analysis of Molecular Variance). Genetics and Biometry Laboratory, University of Geneva, Suitzerland.
|
Fahima T, Sun G L, Beharav A, et al. 1999. RAPD polymorphism of wild emmer wheat populations, Triticum dicoccoides, in Israel. Theoretical and Applies Genetics, 98: 434-447. DOI:10.1007/s001220051089 |
Knapp E E, Rice K J. 1998. Comparison of isozymes and quantitative traits for evaluating patterns of genetic variation in purple needle grass (Nassella pulchra). Conservation Biology, 12: 1031-1041. DOI:10.1046/j.1523-1739.1998.97123.x |
Miller M P. 1997. AMOVA-PREP, a program for the preparation of AMOVA input files for use with WINAMOVA. Department of Biological Sciences, Northern Arizona University, Flagstaff, AZ.
|
Schaal B A, Hayworth D A, Olsen K M, et al. 1998. Phylogeographic studies in plants: problems and prospects. Molecular Ecology, 7: 465-474. DOI:10.1046/j.1365-294x.1998.00318.x |
Volis S, Yakubov B, Shulgina I, et al. 2001. Tests for adaptive RAPD variation in population genetic structure of wild barley, Hordeum spontaneum Koch. Biological Journal of Linnean Society, 74: 289-303. DOI:10.1111/j.1095-8312.2001.tb01393.x |
Wright S. 1951. The genetic structure of populations. Annals of Eugenics, 15: 323-354. |
Yeh F C, Yang R C, Boyle T. 1999. POPGENE version 1.32, Microsoft window base software for population genetic analysis: a quick user's guide. University of Alberta, Center for International Forestry Research, Alberta, Canada.
|
Zeng Jie, Zou Yuping, Bai Jiayu, et al. 2002. Preparation of total DNA from "Recalcitrant Plant Taxa". Acta Botanica Sinica, 44(6): 694-697. |
Zeng Jie, Zou Yuping, Bai Jiayu, et al. 2003. RAPD analysis of genetic variation in natural populations of Betula alnoides from Guangxi, China. Euphytica, 134(1): 33-41. DOI:10.1023/A:1026113506563 |
 2008, Vol. 44
2008, Vol. 44