文章信息
- 王艳敏, 魏志刚, 杨传平.
- Wang Yanmin, Wei Zhigang, Yang Chuanping.
- 白桦EST-SSR信息分析与标记的开发
- Data Mining for SSRs in ESTs and EST-SSR Marker Development in Betula platyphylla
- 林业科学, 2008, 44(2): 78-84.
- Scientia Silvae Sinicae, 2008, 44(2): 78-84.
-
文章历史
- 收稿日期:2006-11-21
-
作者相关文章
传统基因组来源的SSR开发需要基因组文库构建、探针杂交和克隆测序等繁杂的操作程序,这不仅费时、费力,而且开发成本很高,极大地制约了其发展和应用(Kota et al., 2001)。随着功能基因组学的迅速发展,GenBank(基因序列数据库)中积累了许多重要物种的大量ESTs(expressed-squence tags)序列,这些序列已经成为发展SSR标记(EST-SSR)的重要资源。与来自基因组中随机分布的SSR标记相比,基于EST开发的SSR标记位于基因的转录部分,反映了基因的编码部分,可以直接获得基因表达的信息,因而与功能基因紧密连锁,而且它在近缘物种间具有高度的可转移性和通用性。目前,EST-SSR的开发在农作物中应用广泛,在甘蔗(Saccharum officenarum)(Cordeiro et al., 2001)、小麦(Triticum aestivum)(Eujayl et al., 2002;Gupta et al., 2003;Gao et al., 2003;Yu et al., 2004)、黑麦(Secale cereale)(Hackauf et al., 2002)、大麦(Hordeum vulgare)(Thiel et al., 2003)、苜蓿(Medicago sativa)(Barrett et al., 2004)、葡萄(Vitis vinifera)(Decroocq et al., 2003)、水稻(Oryza sativa)(Cho et al., 2000)等中已有报道,而在树木中只有在杏(Prunus armeniaca)(Xu et al., 2004;Decroocq et al., 2003)、猕猴桃(Actinidia chinensis)(Fraser et al., 2004)、云杉(Picea asperata)(Rungis et al., 2004)和火炬松(Pinus taeda)(Liewlaksaneeyanawin et al., 2004)等物种中有过报道。
白桦(Betula platyphylla)是非常优良的短周期纸浆用材阔叶树种。目前,未见关于白桦EST-SSR方面的报道。本文从NCBI(美国国立生物技术信息中心)数据库中查找欧洲白桦(Betula pendula)的ESTs序列,并进行SSRs的查找和分析。在此基础上,根据SSRs两端的保守序列设计引物,从而开发了可以用于白桦的EST-SSR标记,并利用新开发的引物进行该树种的多态性研究。本研究是对现有白桦资源的进一步发掘和利用,建立这种标记对于加速白桦EST资源的开发利用、丰富其分子标记类型以及为绘制遗传图谱、实现目标性状的标记辅助选择具有重要的意义。
1 材料与方法 1.1 植物材料及基因组DNA的提取材料为东北林业大学校园内的白桦和白桦强化种子园内10株个体(欧洲白桦、日本白桦和来源于帽儿山、小北湖、凉水、清源、桓仁、乌伊岭、辉南、新疆的白桦)。每份材料取其新鲜叶片提取基因组DNA(杨传平等,2005)。
1.2 白桦EST-SSR序列的查找从GenBank/dbEST(http://www.ncbi.nlm.nih.gov/dbEST/index.html)下载欧洲白桦EST序列,共获得了2 548条欧洲白桦ESTs序列。
应用SSRIT(simple sequence repeat identification tool)软件(http://www.gramene.org/db/searches/ssrtool)在线搜索EST-SSR。搜索的标准为:二核苷酸、三核苷酸、四核苷酸重复序列的重复次数大于或等于5。
1.3 EST-SSR引物的设计用Primer3(http://frodo.wi.mit.edu/cgi-bin/primer3/primer3)设计EST-SSR引物,由上海生工生物技术公司合成。设计引物时设置的主要参数为:GC含量20%~80%,退火温度(tm)57~63 ℃,引物长约20 bp(18~27 bp),预期扩增产物长度120~350 bp。
用SSR Primer(http://hornbill.cspp.latrobe.edu.au/cgi-binpub/ssrprimer/indexssr.pl)设计EST-SSR引物,由上海生工生物技术公司合成。设计引物时设置的主要参数为:GC含量20%~70%,退火温度(tm)55~60 ℃,引物长度18~23 bp,预期扩增产物长度100~300 bp。
1.4 PCR扩增及电泳检测PCR反应体积为20 μL,内含DNA 100 ng,Mg2+浓度为3 mmol·L-1,Taq酶0.5 U,dNTP浓度为0.5 mmol·L-1,引物浓度为0.4 μmol·L-1。反应程序为:94 ℃预变性5 min;94 ℃变性30 s,复性30 s(退火温度视不同引物确定),72 ℃延伸30 s,35个循环;72 ℃延伸15 min;4 ℃保存。PCR产物在6%变性聚丙烯酰胺凝胶中电泳,银染,检测多态性。
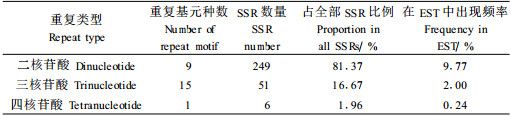
2 结果与分析 2.1 欧洲白桦EST-SSR分布、频率及特点在搜索的2 548个ESTs中,共发现了分布于260个ESTs中的306个SSRs,含有SSR的EST占全部EST的10.2%,SSR的出现频率是12.01%,这表明欧洲白桦EST中的SSR数量很丰富。在260条含有SSR的EST中,只有1个SSR的EST有216条,含有2个SSR的有43条,含有4个SSR的EST有1条。
由以上分析可以看出,欧洲白桦的EST-SSR主要有3种重复类型,即二核苷酸重复、三核苷酸重复、四核苷酸重复,其出现频率各不相同(表 1)。其中二核苷酸重复最为常见,共有249个,在全部EST中的出现频率为9.77%,所占SSR比例达到81.37%;其次为三核苷酸重复,出现频率为2.00%,所占SSR比例为16.67%;四核苷酸重复的出现频率为0.24%,所占SSR比例为1.96%。由此可见,在欧洲白桦EST-SSR中,二核苷酸重复占主导地位。
|
|
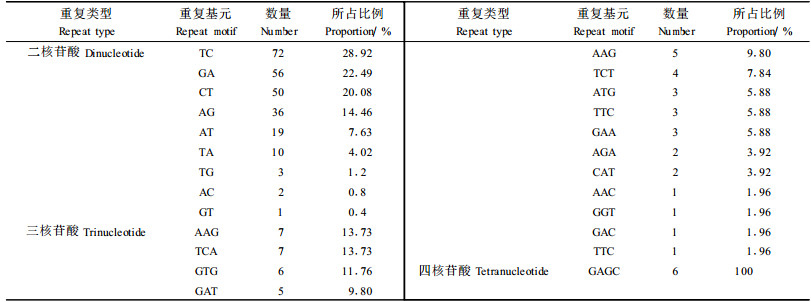
在搜索出的欧洲白桦EST-SSR中,共观察到25种核苷酸重复(表 2)。其中二核苷酸重复中,TC、GA、CT出现的次数较多,分别占二核苷酸重复的28.92%、22.49%、20.08%;在三核苷酸重复基元中,TCA、AAG出现较多,占三核苷酸重复的13.45%;四核苷酸中6个重复碱基均为GAGC。
|
|
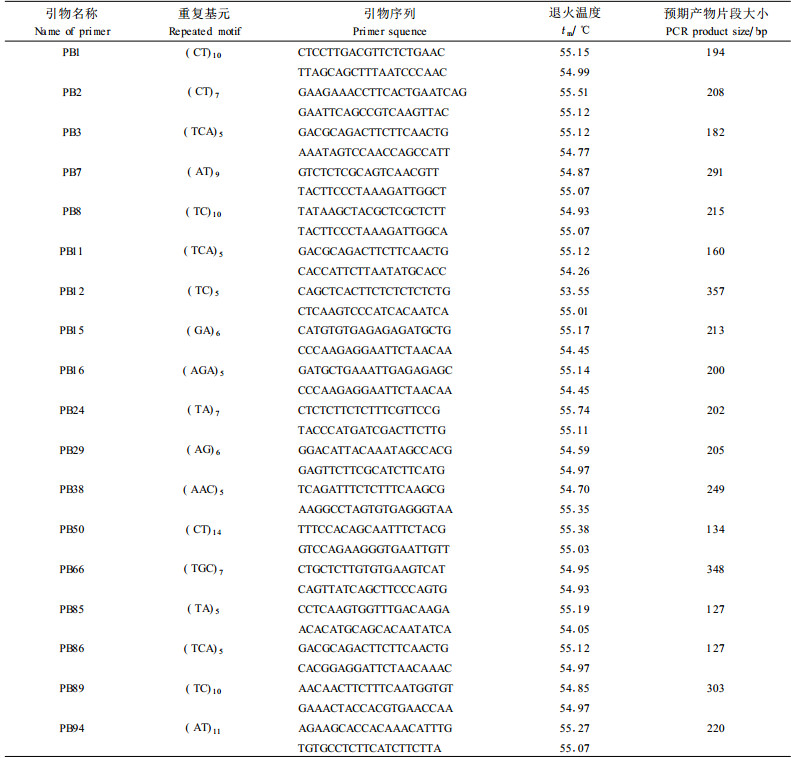
采用2种软件设计白桦的SSR引物,其中应用Primer3设计出176对引物,用SSR Primer共设计出100对引物。以东北林业大学校园内白桦对照为模板对用2种软件设计的引物进行扩增。其中应用Primer3设计出176对引物共有104对引物产生扩增产物,占合成引物的59.09%;而用SSR Primer设计出的100对引物中有37对引物产生了扩增产物,占合成引物的37%。
2.3 白桦EST-SSR的多态性分析为了进一步研究所设计引物的多态性,对能扩增出条带的141对引物用10株个体进行测试,结果用SSR Primer设计出的37对引物中共有18对引物具有多态性,占产生扩增产物引物总数的48.65%(图 1,表 3);而用Primer3设计的产生扩增产物的104对引物中仅有27对引物具有多态性,仅占产生扩增产物引物总数的25.96%(图 2,表 4)。由此可见,应用2种不同的设计软件所设计出的引物有所不同。

|
图 1 10个样品在SSR Primer设计的PB8号引物中的扩增效果 Figure 1 The amplification result of ten samples in PB8 designed by SSR Primer 1.欧洲白桦B. pendula; 2.日本白桦B.japonica; 3-10.不同种源白桦B.platyphylla in different provenances (3.帽儿山Maoershan; 4.小北湖Xiaobeihu; 5.凉水Liangshui; 6.清源Qingyuan; 7.桓仁Huanren; 8.乌伊岭Wuyiling; 9.辉南Huinan; 10.新疆Xinjiang).下同。The same below. |
|
|

|
图 2 10个样品在Primer3软件设计的P107引物中的扩增效果 Figure 2 The amplification result of ten samples in P107 designed by Primer3 |
|
|
随着EST和cDNA大规模测序的开展,EST-SSR引物的开发展现了广阔的前景。但到目前为止,相关研究还比较少,有待进一步加强EST-SSR的开发与应用研究。本次试验共对2 548个欧洲白桦EST进行了搜索,发现了307个SSRs,出现频率是12.05%。这个比率远高于其他物种EST数据库中筛选的SSR比率,如甘蔗的2.9%(Cordeiro et al., 2001)、水稻的4.7%(Kantety et al., 2002)、普通小麦的5.4%(Gupta et al., 2003)、白菜(Brassica pekinensis)的10.34%(忻雅等,2006),这种差异可能是物种间的真实SSR信息差异或搜寻SSR时所用长度最低标准不同造成的(Nicot et al., 2004)。当4种不同的碱基随机组合时, 若EST-SSR数目足够大且无偏倚性, 将可能分别产生2、4、10、33、102和350种单、二、三、四、五、六核苷酸重复(Rota et al., 2005)。在一些物种中三核苷酸重复占主要类型,如甘蔗EST中三核苷酸重复占90%以上(Cordeiro et al., 2001)、普通小麦EST中三核苷酸SSR占67.12%(Gupta et al., 2003)。而二核苷酸重复序列在多数植物基因组中分布几率较高(Cardle et al., 2000)。本研究中欧洲白桦EST中二核苷酸重复是主导类型, 所占SSR比例达到81.12%。这可能与白桦EST-SSR自身的特点有关。对欧洲白桦EST-SSR的分析结果显示, 大多数二核苷酸重复均有出现,但大多数三核苷酸重复和四核苷酸重复未出现,表现出明显的偏倚性。但本研究分析欧洲白桦EST序列数量不足5 000,所出现碱基偏倚性是白桦EST-SSR的真实反映还是由于分析EST数量较少或者二者兼而有之所造成, 还需要进一步研究。
GA在大多数植物二核苷酸重复中是主导核苷酸重复。Thiel等(2003)在大麦中研究得知二核苷酸重复中GA出现频率最高为52%,Nicot等(2004)在小麦的研究中所得到的结果为二核苷酸重复中GA出现频率为45%, 也与Kantety等(2002)和Morgante等(2002)对多种不同植物EST-SSR综合分析后所报道的结果相符。但在猕猴桃(Fraser et al., 2004)中频率最高的二核苷酸重复却为CT(45.3%), 其次才是GA(25.3%), 在番茄(Lycopersicon esculentum)(Areshchenkova et al., 2002)中也不是GA而是AT频率高。本研究中观察到TC、GA、CT数量较多,分别占二核苷酸重复的28.92%、22.49%、20.08%,TC在二核苷酸重复中占主导类型,其次才是GA(22.49%)。欧洲白桦三核苷酸重复中TCA、AAG数量较多(7个),占三核苷酸重复的13.45%。在白菜和拟南芥(Arabidopsis thaliana)中GAA较多,而在水稻、大麦、高粱(Sorghum vulgare)、玉米(Zea mays)中CCG则是主要三核苷酸重复(Gao et al., 2003), Nicot等(2004)报道小麦三核苷酸SSR中占优势的类型是CCG,Kantety等(2002)报道却是AAC。这种不同物种EST-SSR主导核苷酸重复的差异及同一物种中不同报道结果的差异, 可能是由于各报道中所用EST来源和EST数目不同所致。
本试验中,采用2种软件设计引物,应用Primer3软件设计出176对引物,共有104对引物产生扩增产物,占合成引物的59.09%,其中仅有27对引物具有多态性,仅占筛选引物总数的25.96%;而用SSR Primer软件设计出的100对引物中有37对引物产生了扩增产物,占合成引物的37%,其中有18对引物具有多态性,占筛选引物总数的48.65%。由此可见,利用2种不同的设计软件设计SSR引物其结果有一定的差异,建议利用EST序列开发SSR引物时尽量采用不同的软件进行设计,以丰富SSR的数量。在所有引物中,共有96对引物能够成功扩增但是没有多态性,这可能主要与EST建库植株与试验材料的亲缘关系以及引物设计的特异性有关,并不是所有的EST都有多态性,有些很保守的片断没有多态性是正常的。根据EST序列设计的引物对基因组DNA进行PCR扩增时,扩增产物通常会比目标EST片段长,这在以往的报道中普遍存在,如火炬松的EST作图和豌豆(Pisum sativum)(Konovalov et al., 2005)的EST-CAPS作图,这主要是由于内含子的存在。引物设计时尽量利用EST序列3′端的300 bp,将有助于去除内含子(Temesgen et al., 2001)。有135对引物不能进行有效扩增,可能是由于模板中含有抑制反应的杂质或模板含量低、引物和Mg2+浓度过低、退火温度过高、酶失活、反应条件不合适。除了进一步优化反应体系之外,还有可能是因为引物的序列落在2个外显子上或两引物间有一长的内含子,扩增不出产物。目前为止,可以搜索到10 304个欧洲白桦EST序列,随着EST序列的大量增长还可以进一步开发出更高效的可应用于白桦分子标记的SSR引物。
忻雅, 崔海瑞, 卢美贞, 等. 2006. 白菜EST-SSR信息分析与标记的建立. 园艺学报, 33(3): 549-554. DOI:10.3321/j.issn:0513-353X.2006.03.018 |
杨传平, 潘华, 魏志刚. 2005. 白桦ISSR-PCR反应体系的优化. 东北林业大学学报, 33(6): 1-3. DOI:10.3969/j.issn.1000-5382.2005.06.001 |
Areshchenkova T, GanalM W. 2002. Comparative analysis of polymorphism and chromosomal location of tomato microsatellite markers isolated from different sources. Theor Appl Genet, 104: 229-235. DOI:10.1007/s00122-001-0775-2 |
Barrett B, Griffiths A, Schreiber M, et al. 2004. A microsatellite map of white clover. Theor Appl Genet, 109: 596-608. |
Cardle L, Ramsay L, Milbourne D, et al. 2000. Computational and experimental characterization of physically clustered simple sequence repeats in plants. Genetics, 156: 847-854. |
Cho Y G, Ishii T, Temnykh S, et al. 2000. Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice (Oryza sativa L). Theor Appl Genet, 100: 713-722. DOI:10.1007/s001220051343 |
Cordeiro G M, Casu R, McIntyre C L, et al. 2001. Microsatellite markers from sugarcane (Saccharum spp.) ESTs cross transferable to erianthus and sorghum. Plant Sci, 160: 1115-1123. DOI:10.1016/S0168-9452(01)00365-X |
Decroocq V, Fave M G, Hagen L, et al. 2003. Development and transferability of apricot and grape EST microsatellite markers across taxa. Theor Appl Genet, 106: 912-922. DOI:10.1007/s00122-002-1158-z |
Eujayl I, Sorrells M E, Baum M, et al. 2002. Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat. Theor Appl Genet, 104: 399-407. DOI:10.1007/s001220100738 |
Fraser L G, Harvey C F, Crowhurst R N, et al. 2004. EST-derived microsatellites from Actinidia species and their potential for mapping. Theor Appl Genet, 108: 1010-1016. DOI:10.1007/s00122-003-1517-4 |
Gao L F, Tang J F, Li H W, et al. 2003. Analysis of microsatellites in major crops assessed by computational and experimental approaches. Mol Breed, 12: 245-261. DOI:10.1023/A:1026346121217 |
Gupta P K, Rustgi S, Sharma S, et al. 2003. Transferable EST-SSRs markers for the study of polymorphism and genetic diversity in bread wheat. Mol Gen Genomics, 270: 315-323. DOI:10.1007/s00438-003-0921-4 |
Hackauf B, Wehling P. 2002. Identification of microsatellite polymorphisms in an expressed portion of the rye genome. Plant Breed, 121: 17-25. DOI:10.1046/j.1439-0523.2002.00649.x |
Han Z G, Guo W Z, Song X L, et al. 2004. Genetic mapping of EST-derived microsatellites from the diploid Gossypium arboreum in allotetraploid cotton. Mol Gen Genomics, 272: 308-327. DOI:10.1007/s00438-004-1059-8 |
Kantety R V, RotaM L, Matthews D E, et al. 2002. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat. Plant Mol Biol, 48: 501-510. DOI:10.1023/A:1014875206165 |
Konovalov F, Toshchakova E, Gostimsky S. 2005. A CAPS marker set for mapping in linkage group Ⅲ of pea (Pisum sativum L.). Cell Mol Biol Lett, 10(1): 163-171. |
Kota R, Varshney R K, Thiel T, et al. 2001. Generation and comparison of EST-derived SSRs and SNPs in barley (Hordeum vulgare L.). Hereditas, 135: 145-151. |
Liewlaksaneeyanawin C, Ritland C E, El-Kassaby Y A, et al. 2004. Single-copy, species-transferable microsatellite markers developed from loblolly pine ESTs. Theor Appl Genet, 109: 361-369. |
Morgante M, Hanafey M, Powell W. 2002. Microsatellites are preferentially associated with nonrepeatitive DNA in plant genomes. Nat Genet, 30: 194-200. DOI:10.1038/ng822 |
Nicot N, Chiquet V, Gandon B, et al. 2004. Study of simple sequence repeat (SSRs) markers from wheat expressed sequence tags (ESTs). Theor Appl Genet, 109: 800-805. DOI:10.1007/s00122-004-1685-x |
Rota L R, Kantety R V, Yu J K. 2005. Nonrandom distribution and frequencies of genomic and EST-derived microsatellite markers in rice, wheat, and barley. BMC Genomics, 6: 23. DOI:10.1186/1471-2164-6-23 |
Rungis D, Berube Y, Zhang J, et al. 2004. Robust simple sequence repeat markers for spruce (Picea spp.) from expressed sequence tags. Theor Appl Genet, 109: 1283-1294. DOI:10.1007/s00122-004-1742-5 |
Temesgen B, Brown G R, Harry D E, et al. 2001. Genetic mapping of expressed sequence tag polymorphism (ESTP) markers in loblolly pine (Pinus taeda L.). Theor Appl Genet, 102(5): 664-675. DOI:10.1007/s001220051695 |
Thiel T, Michalek W, Varshney R K, et al. 2003. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L). Theor Appl Genet, 106: 411-422. DOI:10.1007/s00122-002-1031-0 |
Xu Yong, Ma Rongcai, Xie Hua, et al. 2004. Development of SSR markers for the phylogenetic analysis of almond trees from China and the Mediterranean region. Genome, 47(6): 1091-1104. DOI:10.1139/g04-058 |
Yu J K, Dake T M, Singh S, et al. 2004. Development and mapping of EST-derived simple sequence repeat markers for hexaploid wheat. Genome, 47: 805-818. DOI:10.1139/g04-057 |
 2008, Vol. 44
2008, Vol. 44