文章信息
- 李因刚, 周志春, 金国庆.
- Li Yingang, Zhou Zhichun, Jin Guoqing.
- 三尖杉种源遗传多样性
- Genetic Diversity for Different Provenances of Cephalotaxus fortunei
- 林业科学, 2008, 44(2): 64-69.
- Scientia Silvae Sinicae, 2008, 44(2): 64-69.
-
文章历史
- 收稿日期:2007-01-19
-
作者相关文章
三尖杉(Cephalotaxus fortunei)为三尖杉科(Cephalotaxaceae)中分布最广的中国特有种,是古老的孑遗植物,分布于浙江、安徽、福建、江西、湖南、湖北、河南、陕西、甘肃、四川、云南、贵州、广东和广西等省区,被国际松杉类植物专家组(CSG)列为第5级(即稀有的第2类)受胁植物(贺新强等,1996),其种子、枝、叶、根含有三尖杉酯碱和高三尖杉酯碱,均可入药(胡玉熹,1999)。临床上三尖杉酯碱和高三尖杉酯碱配合其他中药治疗各种类型白血病疗效较好,同时三尖杉酯碱还可用于治疗黑素瘤、肺癌、乳腺癌、脑肿瘤等。三尖杉为二倍体植物(顾志建等,1998),W. Dallimore和A. B. Jackson根据叶的长短建立了2个变种:长叶三尖杉(C. fortunei var. longifolia)与短叶三尖杉(C. fortunei var. brevifolia)(胡玉熹,1999)。三尖杉分布区东南部的种源种子较长,高生长量较大,分枝稀少,多为长叶类型;中西部的种源种子较短,高生长量较小,分枝较多,多为短叶类型,间有长叶类型分布(焦月玲等,2006)。自从三尖杉中提取出三尖杉双酯碱后,其重要经济价值逐渐被人们认识,而这却给三尖杉野生资源带来了毁灭性的破坏,生境严重片断化。现有的三尖杉野生资源主要存在于一些自然保护区的天然林分中,少有大面积分布,且多为砍伐后的萌生条。
ISSR标记技术检测的是2个SSR之间的一段短DNA序列上的多态性(Zietkiewicz et al., 1994),具有DNA用量少、操作简单、实验成本低等优点(Wolfe et al., l998;Harris,1999)。ISSR引物的退火温度比RAPD引物高,其产物的多态性比RAPD丰富,而且比RAPD技术更为稳定可靠,重复性更好(Tsumura et al., 1996)。近年来,ISSR分子标记已被广泛应用于物种鉴定(Huang,2000)、遗传多样性研究等(Martins et al.,2003;Panda et al., 2003)。目前对三尖杉的研究主要集中在资源分布(郭文杰等,1998)、种子变异(司马永康等,2002)和育苗技术(吕玉华等,2000)、生物碱的抗病活性(王定志等,1992)等方面。在三尖杉的分子生物学研究方面,陈少瑜等(2003a)对三尖杉属植物做了同工酶分析,其研究表明粗榧(Cephalotaxus drupacea)和高山三尖杉(C. fortunei var. alpina)的酯酶(EST)、苹果酸脱氢酶(MDH)等5种同工酶的酶谱具有较高的相似性;王艇等(1999)利用RAPD分子标记对三尖杉科植物做了系统学分析,证明在三尖杉科内建立篦子三尖杉组(Sect.Pectinatae)和三尖杉组(Sect.Cephalotaxus)的合理性。三尖杉的遗传多样性研究未见报道。本文在三尖杉种源苗期试验的基础上(焦月玲等,2006),利用ISSR分子标记进一步研究三尖杉不同种源的遗传多样性和遗传分化,旨在为科学合理地保护和利用现有的三尖杉天然资源提供理论依据。
1 材料与方法 1.1 材料试验叶样取自福建省明溪县绿色生态研究所圃地的三尖杉种源试验2年生留床苗。取样种源来自10个省的16个代表性种源,包括安徽黄山,江西崇义、于都,福建柘荣、武夷山、明溪,贵州从江、黎平,广东平远共9个长叶类型三尖杉种源,河南洛阳、湖北恩施、江西井冈山、湖南绥宁、贵州锦屏、四川峨眉、云南石屏共7个短叶类型三尖杉种源。三尖杉种源试验的采种林分为当地起源的天然林。由于三尖杉多为天然林分中的少见种,要求至少选择10株以上的优良母树采种,母树间距50 m以上,每种源提供种子5 kg。参试种源的基本情况和苗期遗传测定等详见焦月玲等(2006)。2005年9月下旬,每个种源随机抽取12个单株,分别采集其顶端新发枝条用于DNA提取。
1.2 基因组DNA提取每个单株取0.5 g新鲜嫩叶,采用改良CTAB法(Doyle et al., 1987)提取基因组DNA。提取的DNA溶于1×TE缓冲液中,用1.0%的琼脂糖凝胶电泳检测其质量,并在紫外分光光度计(日本岛津UV-2401PC)下检测其浓度,最后稀释至20 ng·μL-1,用于ISSR分析。
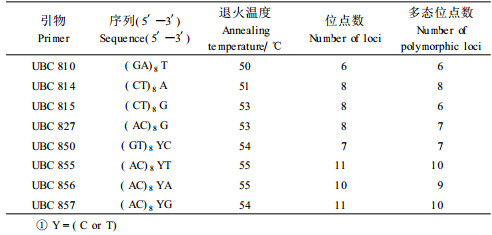
1.3 PCR扩增PCR扩增反应在PTC-100TM基因扩增仪上进行。ISSR扩增反应程序:94 ℃预变性5 min;94 ℃变性30 s,50~55 ℃退火(退火温度随引物而定,见表 1)45 s,72 ℃延伸90 s,40个循环;72 ℃延伸7 min,最后于4 ℃下保存。PCR反应经过比较和优化确定为20 μL体系:Taq酶0.5 U,dNTP 0.25 mmol·L-1,引物0.4 μmol·L-1,DNA模板80 ng,MgCl22.5 mmol·L-1,2%甲酰胺。扩增产物在3 V·cm-1电压条件下经1.5%琼脂糖凝胶电泳分离,对照标准分子量200 bp DNA Ladder (华美生物工程公司),经溴化乙锭染色,并利用复日FR-200紫外与可见光分析成像系统拍照记录结果。图 1给出了引物UBC856的ISSR扩增产物在部分三尖杉种源植株样品中的分离情况。
|
|

|
图 1 引物UBC856的ISSR扩增产物在部分三尖杉样品中的分离 Figure 1 Segregation of ISSR bands amplified by UBC856 in some samples of C. fortunei |
所用引物为加拿大哥伦比亚大学UBC公司公布的第9套ISSR引物。每个种源随机选择1个DNA模板进行扩增筛选,从60个ISSR引物中筛选出8个能产生多态、清晰且可重复条带的引物,用于全部16个种源样本分析。各引物名称、退火温度、检测的位点数和多态性位点数见表 1。
1.5 数据统计与分析ISSR为显性标记,同一引物扩增产物中电泳迁移率一致的条带被认为具有同源性。电泳图谱中的每一条带视为1个标记,并代表 1个引物的结合位点。按凝胶同一位置上DNA条带的有无进行统计,有带的记为“1”,无带的记为“0”,仅记录清晰且长度在200~1 800 bp范围的扩增带,建立0/1数据矩阵。
得到的ISSR数据矩阵用于以下分析:1)将ISSR标记视为表征性状,应用POPGENE软件(Version 1.3.1)计算多态位点百分率(PPL)和Shannon信息多样性指数(I);2)将ISSR标记视作同一基因座位上的2个等位基因(Williams et al., 1990;钱韦等,2001),基于Hardy-Weinberg平衡,删除基因频率小于3/N(N为样本数,N=192)的条带后(Lynch et al., 1994),分别计算Nei’s基因多样度指数(HE)、种源总基因多样度(HT)、种源内基因多样度(HS)、基因分化系数(GST)和Nei’s遗传距离(D);3)基于POPGENE软件对种源间Nei's(1978)无偏遗传距离(unbiased genetic distance)的估算,对16个种源进行UPGMA聚类分析;4)用AMOVA-PREP软件计算个体间欧氏距离所得的输出文件(距离文件、组文件、群体文件)作为输入文件,用WINAMOVA1.04软件进行分子方差分析,并以种源为单位,采用Mantel检验分析16个种源间遗传距离和地理距离的相关性。
2 结果与分析 2.1 种源及物种水平的遗传多样性利用8条ISSR引物对16个三尖杉种源共192个个体进行PCR扩增,每条引物扩增出的位点数目为6~11条不等,条带片断大小在300~1 800 bp间,共检测到69个位点,其中63个位点是多态的,多态位点百分率(PPL)为91.30%。16个种源的多态位点百分率(PPL)差别较大,在44.83%~89.66%之间,平均为72.52%,其中以湖南绥宁种源最高(89.66%),福建柘荣和贵州黎平种源次之(84.48%),四川峨眉种源最低(44.83%),最高值为最低值的2倍。不同类型种源的多态位点百分率均值也有一定差异,如长叶三尖杉种源均值为74.90%,短叶三尖杉为69.45%。长叶类型三尖杉种源间的多态位点百分率差异较小,最高种源(福建柘荣和贵州黎平,PPL=84.48%)为最低种源(广东平远,PPL=67.24%)的1.26倍;短叶类型三尖杉种源间的多态位点百分率差异较大,最高种源(湖南绥宁,PPL=89.66%)为最低种源(四川峨眉,PPL=44.83%)的2倍。种源的Nei’s基因多样性(HE)在0.148 6~0.343 1之间,Shannon信息多样性指数(I)在0.222 0~0.501 5之间,两者在种源间的大小变化趋势与多态位点百分率基本一致。此外,种源平均水平的各遗传多样性参数(PPL=72.52%,HE=0.252 8, I=0.375 3)低于物种水平(PPL=91.30%,HE=0.337 7, I=0.529 5)。
|
|
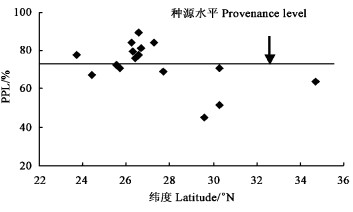
与产地经纬度的相关分析表明,三尖杉种源多态位点百分率不存在明显的地理变异模式。然而从不同纬度种源多态位点百分率的离散分布图(图 2)中可以清楚地看出,多态性百分率高的种源多集中在26°~28°N间。此范围内贵州黎平、江西于都等7个种源的多态位点百分率都高于种源水平(PPL=72.52%),均值为81.77%,为其余9个种源均值(PPL=65.33%)的1.25倍。
|
图 2 不同纬度种源多态位点百分率(PPL)离散图 Figure 2 Discrete figure of percentage of polymorphic loci (PPL) of provenances from different latitudes |
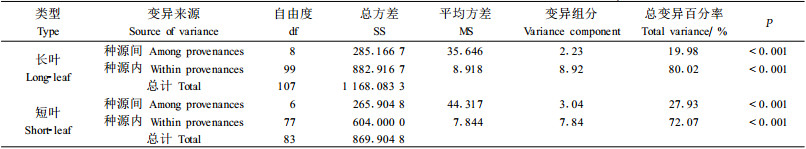
POPGENE的分析结果(表 3)表明,三尖杉种内总基因多样性为0.337 7,种源内基因多样性为0.252 8,种源间的基因多样性为0.084 8。基因分化系数达到0.251 0,基因流较小,仅为0.746 0。三尖杉乃是呈星散分布的广布性树种,为林分中的少见种,种群一般较小,现多龟缩于一些自然保护区和保护小区,生境严重片断化,结果导致种源间较大的遗传分化,25.10%的遗传变异存在于地理种源间,而种源内的变异占到了总变异的74.90%。
|
|
AMOVA分析结果(表 4)亦显示,三尖杉种源间和种源内都存在显著的遗传变异(P<0.001),其中种源间变异占25.90%,种源内变异占74.10%,与利用POPGENE软件分析获得的结论基本一致。三尖杉不同类型种源的AMOVA分析(表 5)表明,长叶类型三尖杉的种源间变异较小,仅占总变异的19.98%,而短叶类型三尖杉的种源间变异则较大,占总变异的27.93%。较之于长叶类型种源,短叶类型种源间的地理距离较远,这种距离隔离影响了种源间的基因流,结果致使短叶类型三尖杉种源间产生较大的遗传分化。
|
|
|
|
通过估算16个三尖杉种源间遗传距离,并据此使用UPMGA聚类法得出树形图(图 3)。从图中可以看出,江西于都、贵州从江、安徽黄山、贵州锦屏、福建明溪、福建柘荣、贵州黎平和湖南绥宁之间的遗传距离较近,首先聚在一起;江西井冈山、云南石屏和湖北恩施, 江西崇义、福建武夷山和四川峨嵋也各自聚为一类;而广东平远与其他种源的遗传距离均较大,最后才与其他种源聚类。福建明溪、福建柘荣、贵州黎平、湖南绥宁、江西于都、贵州从江、贵州锦屏7个相互间遗传距离较小的种源分布在三尖杉自然分布区的东部和偏中东部地区,包括福建、江西南部、湖南南部、贵州东南部等地,来自这一分布区的种源遗传多样性较高,且多为长叶类型三尖杉。江西井冈山、崇义和福建武夷山虽属于东部种源,但与福建明溪、柘荣等上述7个分布区东部和偏中东部种源的遗传距离较远而未能归在一类,可能与这3个种源的原产地种群特性有关。对三尖杉各种源间遗传距离和地理距离的Mantel检验表明,种源间遗传距离与地理距离间不存在显著相关性(r=0.253 5,P=0.320 6),如相距较远的江西于都和贵州从江种源最先聚在一起,但其地理距离却远大于贵州从江和黎平两者间的地理距离(132 km),另外江西于都种源也没有和邻近的江西井冈山种源聚在一起。

|
图 3 种源Nei’s遗传距离的UPGMA聚类图 Figure 3 UPGMA dendrogram for provenances of C. fortunei based on Nei's genetic distance |
地理分布范围是决定植物种遗传多样性的主要因素之一,宽分布区的种趋向于具有更高的遗传多样性水平(Hamrick et al., 1990)。三尖杉的分布区广阔,加之三尖杉是单性、雌雄异株的植物,保证了其严格的异花授粉,因而在物种水平上,三尖杉遗传多样性(HE=0.337 7)高于Nybom(2004)所统计的多种植物种群水平的遗传多样性(基于RAPD和ISSR等显性标记)平均值(HE=0.22),也高于分布区较窄的同属植物篦子三尖杉(Cephalotaxus oliveri)(陈少瑜等,2003b)。本文研究发现三尖杉16个种源间的遗传多样性差异很大,不仅表现在不同分布区的种源间,而且也表现在不同类型种源间。三尖杉东部和偏中东部(25°36′—27°15′N)的种源其基因多样性都高于种源平均值(HE=0.252 8),这一区域的种源多为长叶类型,生长较快,推测是三尖杉可能的分布中心。比较表明,参试的9个长叶类型种源的多态位点百分率均值为74.90%,7个短叶类型种源的遗传多样性均值为69.45%,前者为后者的1.08倍。长叶和短叶类型三尖杉种源间差异可能是造成分布中心和边缘分布区种源间遗传多样性差异的主要原因。
三尖杉种源间的遗传分化系数(GST=0.251 0)明显高于远交物种和多年生物种的平均值(GST=0.22和GST=0.19)(Nybom,2004),25.38%的遗传变异存在于地理种源间,74.62%的遗传变异存在于地理种源内。三尖杉为稀有渐危种,虽然分布广泛,但呈星散不连续分布,为林分中的少见种,种群较小,加之受到了严重的人为干扰,现主要分布在一些自然保护区中,其生境严重片断化,其结果因小种群效应及种群间缺乏有效的基因流(Nm=0.735 1<1),导致三尖杉种源间产生严重的遗传分化。此外,短叶类型三尖杉种源间的遗传分化明显大于长叶类型的三尖杉种源。通过聚类分析发现,三尖杉东部和偏中东部的7个参试种源(包括江西于都、贵州从江、安徽黄山、贵州锦屏、福建明溪、福建柘荣、贵州黎平和湖南绥宁)遗传距离较近而聚在一起,该区域的种源遗传多样性较高,应实行优先原地保护。除通过国家和省级自然保护区原地保护外,对于数量多、种群大、林相好的其他三尖杉天然林应加强自然保护小区的建设。此外,还应加强三尖杉种质资源的收集和评价,实现优良种质的异地保存,为其药用林培育提供丰富的育种材料。
陈少瑜, 司马永康, 方波, 等. 2003a. 三尖杉属植物的同工酶分析. 四川林业科技, 4(2): 42-44. |
陈少瑜, 司马永康, 方波, 等. 2003b. 篦子三尖杉的遗传多样性及濒危原因. 西北林学院学报, 18(2): 29-32. |
顾志建, 周其兴, 岳中枢. 1998. 三尖杉的核形态研究. 植物分类学报, 36(1): 47-52. |
郭文杰, 鲁雪华, 林勇. 1998. 三尖杉的资源利用与开发. 亚热带植物通讯, 27(1): 23-26. |
贺新强, 林金星, 胡玉熹, 等. 1996. 中国松杉类植物濒危等级划分的比较. 生物多样性, 4(1): 45-51. DOI:10.3321/j.issn:1005-0094.1996.01.010 |
胡玉熹. 1999. 三尖杉生物学. 北京: 科学出版社.
|
焦月玲, 周志春, 金国庆, 等. 2006. 三尖杉苗木生长和形态种源差异. 林业科学研究, 9(4): 452-456. DOI:10.3321/j.issn:1001-1498.2006.04.008 |
吕玉华, 沙珩. 2000. 三尖杉的育苗技术. 云南林业科技, (2): 12-14. DOI:10.3969/j.issn.1672-8246.2000.02.003 |
钱韦, 葛颂. 2001. 居群遗传结构中显性标记数据分析方法初探. 遗传学报, 28(3): 244-255. |
司马永康, 蒋宏, 方波, 等. 2002. 珍贵药用植物三尖杉种子大小变异. 云南林业科技, (4): 30-32. DOI:10.3969/j.issn.1672-8246.2002.04.006 |
王定志, 马广恩, 徐任生. 1992. 三尖杉属植物中生物碱的研究. 药学学报, 27(3): 178-184. |
王艇, 苏应娟, 黄超, 等. 1999. 三尖杉科植物RAPD分析及其系统学意义. 武汉植物学研究, 17(3): 227-230. DOI:10.3969/j.issn.2095-0837.1999.03.009 |
Doyle J J, Doyle J L. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf material. Phytochem Bull, 19: 11-15. |
Hamrick J L, Godt M J W. 1990. Allozyme diversity in plant species//Brown A H D, Clegg M T, Kahler A L, et al. Plant Population Genetics. Sunderland, MA, Sinauer, 43-63.
|
Harris J. 1999. RAPDs in systematics—a useful methodology?//Hollingsworth P M, Bateman R M, Gornall R J. Molecular Systematics and Plant Evolution. London, Taylor and Francis, 221-228.
|
Huang J C, Sun M. 2000. Genetic diversity and relationships of sweetpotato and its wild relatives in Ipomoea series Batatas (Convolvulaceae) as revealed by inter-simple sequence repeat (ISSR) and restriction analysis of chloroplast DNA. Theoretical and Applied Genetics, 100(4): 1050-1060. |
Lynch M, Milligan B G. 1994. Analysis of population genetic structure with RAPD markers. Molecular Ecology, 3: 9l-99. |
Martins M, Tenreiro R, Oliveira M M. 2003. Genetic relatedness of Portuguese almond cultivars assessed by RAPD and ISSR markers. Plant Cell Reports, 22: 71-78. DOI:10.1007/s00299-003-0659-9 |
Nei M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, (89): 583-590. |
Nybom H. 2004. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants. Molecular Ecology, 13: 1143-1155. DOI:10.1111/mec.2004.13.issue-5 |
Panda S, Martin J, Aguinagalde I. 2003. Chloroplast and nuclear DNA studies in a few members of the Brassica oleracea L. group using PCR-RFLP and ISSR-PCR markers:a population genetic analysis. Theoretical and Applied Genetics, 106: 1122-1128. DOI:10.1007/s00122-002-1134-7 |
Tsumura Y, Ohba K, Strauss S H. 1996. Diversity and inheritance of inter-simple sequence repeat polymorphism in Douglas-fir (Pseudotsuga menziesii) and sugi (Cryptomeria japonica). Theoretical and Applied Genetics, 92: 40-45. DOI:10.1007/BF00222949 |
Williams J G K, Kubelik A R, Livak K J, et al. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research, 18: 6531-6535. DOI:10.1093/nar/18.22.6531 |
Wolfe A D, Liston A. 1998. Contributions of PCR-based methods to plant systematics and evolutionary biology//Soltis D E, Soltis P S, Doyle J J. Molecular Systematics of Plants. Norwell, MA, Kluwer Academic Publishers, 43-86.
|
Zietkiewicz E, Afalski A, Labuda D. 1994. Genome finger-printing by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification. Genomics, 20: 176-183. DOI:10.1006/geno.1994.1151 |
 2008, Vol. 44
2008, Vol. 44