
陈晓亚,中国科学院上海生命科学研究院植物生理生态研究所、上海市辰山植物科学研究中心研究员,中国科学院院士,发展中国家科学院院士。主要从事植物次生代谢、植物-昆虫相互作用、表皮毛和棉纤维发育研究。2008年获得何梁何利科学技术进步奖,2014年获第三届国际棉花基因组(ICGI)组织奖。
2. 南京大学生命科学学院,南京 210023
3. 大连大学生命科学与技术学院,大连 116622
青蒿素(artemisinin)是目前治疗恶性疟疾(malaria)最有效的药物成分。1977年《科学通报》首次报道青蒿素[1],1979年,青蒿素的化学结构得以解析[2]。青蒿素结构中特有的过氧桥键(peroxidebridge),被认为与抑制疟原虫的活性直接相关。在自然界中,青蒿素的来源是菊科(Asteraceae)蒿属(Ar-temisia)的1年生草本植物黄花蒿(A.annuaL.)。黄花蒿广泛分布于欧亚大陆的寒温带、温带和亚热带地区,以及北美部分地区。该植物在中药文献里常称为"青蒿",青蒿素的中文名即源自于此。而在植物分类学中,青蒿这一名称被用于蒿属另一个种A.carvifoliaBach.-Ham.[3],该种含有艾蒿碱(abrotanine)等成分,但未发现含青蒿素。本文根据植物分类学文献使用黄花蒿一词。研究表明,在黄花蒿中,青蒿素含量约占其干重的0.01%~0.8%,主要富集在花和叶片中。
1 黄花蒿的化学成分除青蒿素外,黄花蒿还含有多种次生代谢成分(天然产物)。其中大量的萜类成分是黄花蒿特殊气味的主要来源。关于黄花蒿成分分析已经有大量的研究报道。1999年,Tellez等比较了黄花蒿有腺体和无腺体品种的萜类成分及其含量,发现在两种不同的品种之间,代谢产物存在着成分与含量的显著区别。有腺毛(glandulartrichome)品种中挥发油含量占鲜重的0.24%,而无腺毛品种只有0.06%。前者成分以α-蒎烯(α-pinene)、松香芹酮(pinocarvone)和蒿酮(arte-misiaketone)为主,而后者则以大根香叶烯D(germacrene-D)和β-石竹烯(β-caryophyllene)为主。青蒿素仅见于黄花蒿的有腺体品种[4]。
2 青蒿素的生物合成青蒿素是一种倍半萜内酯(sesquiterpenelactone)。高等植物中萜类合成代谢的底物是源于细胞质中甲羟戊酸(mev-alonicacid,MVA)途径或质体中磷酸甲基赤藓糖(methy-lerythritolphosphate,MEP)途径的五碳(C5)前体异戊烯基二磷酸(isopentenyldiphosphate,IPP)和二甲基丙烯基二磷酸(dimethylallyldiphosphate,DMAPP),在细胞质中经C15的法尼基二磷酸(farnesyldiphosphate,FPP)由倍半萜合酶和三萜合酶进一步形成各种倍半萜、三萜等产物;在质体中经C10的牻牛儿基二磷酸(geranyldiphosphate,GPP)由单萜合酶合成单萜、经C20的牻牛儿基牻牛儿基二磷酸(geranylgeranyldiphos-phate,GGPP)由二萜合酶催化生成二萜以及类胡萝卜素等产物。黄花蒿富含单萜和倍半萜,多个单萜、倍半萜合酶已经被克隆。如贾军伟等从黄花蒿中,通过同源基因克隆的策略获得了2个单萜合酶基因(QH1和QH5)并鉴定为催化由GPP合成3R-芳樟醇(3R-linalool)[5];蔡煜等从黄花蒿中克隆鉴定了一个倍半萜合酶,β-石竹烯合酶[6];卢山等克隆鉴定了一个单萜合酶,β-蒎烯合酶,并发现其表达受昼夜节律的调控[7]。
在过去20年里,随着分子生物学技术的发展,青蒿素的代谢途径已经得到了较为详细的研究。不仅其生物合成反应中大部分步骤已经阐明,而且绝大多数参与青蒿素合成的酶也得到克隆和鉴定(图 1)。FPP合酶(FPPsynthase,FPPS)催化IPP和DMAPP形成FPP。随后,不同的倍半萜合酶以FPP为底物,催化形成不同的倍半萜基本骨架,其中大多数是环状化合物,其中紫穗槐-4,11-二烯(amorpha-4,11-diene)是青蒿素合成的关键中间体。黄花蒿中编码FPPS的基因于1996年由Matsushita等克隆[8]。随后,Chang等和Mercke等2个实验室同时报道了黄花蒿的紫穗槐-4,11-二烯合酶(amor-pha-4,11-dienesynthase,ADS)编码基因[9, 10]。ADS催化青蒿素途径的第一步反应,是青蒿素生物合成的关键酶。在形成萜类的基本骨架之后,各种修饰酶继续对这些结构进行氧化、羟化、甲基化等修饰。Teoh等发现一个细胞色素P450单加氧酶,CYP71AV1,即紫穗槐二烯C-12氧化酶,催化紫穗槐-4,11-二烯的羟化和脱氢,依次产生青蒿醇(artemisinical-cohol)、青蒿醛(artemisinicaldehyde)和青蒿酸(artemisinicac-id)[11]。与CYP71AV1相互作用的细胞色素还原酶(cyto-chromeP450reductase,CPR)的编码基因也得到了克隆鉴定。后来Teoh等发现一个醛脱氢酶ALDH1可能也参与了从青蒿醛向青蒿酸的代谢过程[12]。2013年Paddon等[13]在构建青蒿酸工程酵母的过程中发现催化紫穗槐二烯形成青蒿酸的全过程还需要另外一个细胞色素CYB5与CYP71AV1共同催化紫穗槐二烯形成青蒿醇,而催化青蒿醇形成青蒿醛则需要一个醇脱氢酶ADH1。Bharel等[14]认为青蒿酸可能是经过青蒿素B(arteannuinB)和二氢青蒿素B(dihydroarteannuinB)形成青蒿素。此外,Zhang等[15]发现青蒿醛也可能通过一个还原酶(arte-misinicaldehydeΔ11(13)doublebondreductase,DBR2)的作用形成二氢青蒿醛(dihydroartemisinicaldehyde),并在ALDH1的作用下成为二氢青蒿酸(dihydroartemisinicacid),最终经一个过氧化物中间体形成青蒿素[12]。
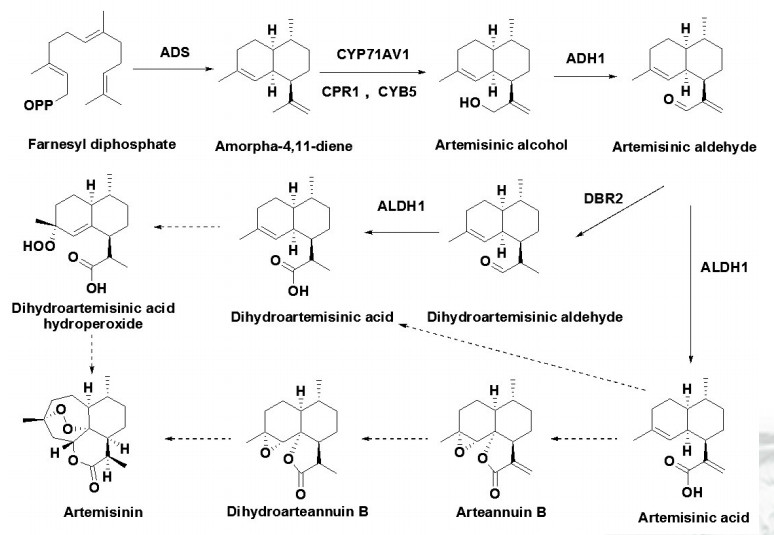 | 酶(黑体)包括紫穗槐-4,11 -二烯合酶(ADS)、紫穗槐二烯 C-12氧化酶(CYP71 AV1)、P450还原酶(CPR1)、细胞色素b5(CYB5)、醇脱氢酶(ADH1)、醛脱氢酶(ALDH1)和青蒿醛Δ11(13)双键还原酶(DBR2)。虚线表示推测的代谢过程图 1 青蒿素的生物合成途径 |
植物中特定的天然产物往往仅分布于独特的物种,同时其合成与积累具有组织、器官以及发育阶段的特异性,因此又被称为植物特异代谢物(plantspecializedmetabolites)。青蒿素是植物特殊代谢物的典型范例,处处体现着这一特性。早期Bertea等对黄花蒿腺毛中的基因表达进行了分析,发现多达47%的表达基因参与各种代谢过程,包括MVA、MEP途径以及下游的萜类合成与修饰过程[16]。Kim等[17]发现在参与青蒿素合成的酶基因中,ADS的启动子主要表达于花药和幼嫩叶片的腺毛中,而在代谢途径中位于其下游的CYP71AV1和DBR2基因也在腺毛中具有较高的表达水平[11, 15, 18]。
黄花蒿的腺毛由10个细胞组成,包括一对基细胞、一对柄细胞和3对分泌细胞。2009年Olsson等[19]利用微切割方法分离了每一对细胞,进而研究了各自的基因表达特异性,结果表明FPPS基因在所有的腺毛细胞及其基部的叶肉细胞中都表达,但是ADS、CYP71AV1和DBR2的编码基因只在最外侧的一对分泌细胞中表达。这些结果显示,青蒿素可能是在叶肉细胞和腺毛中合成其前体FPP,并在腺毛的顶部才最终生成的。
3 青蒿素的代谢调控青蒿素生物合成途径步骤较多,且代谢过程位于特定的腺毛细胞中,分离鉴定调控因子对于深入理解植物倍半萜及青蒿素生物合成具有重要意义。
转录因子的鉴定是认识植物代谢调控进而开展天然产物代谢工程的一个重要手段。在类黄酮的代谢工程研究中,转录因子的鉴定和运用取得了很好的进展。近5年来,在青蒿素生物合成的研究中也已经发现了多种转录因子的参与。WRKY、AP2/ERF、bHLH和bZIP等家族的转录因子都有报道[15,21~24]。其中,于宗霞等[24]发现黄花蒿的AaERF1和AaERF2两个转录因子都能够结合在ADS和CYP71AV1基因启动子区域的CBF2和RAA元件上。转基因工作显示,过表达这2个转录因子中的任何一个都能够促进黄花蒿中ADS和CYP71AV1的基因表达,同时增加植株中青蒿酸和青蒿素的含量;而抑制这2个转录因子的表达则出现相反的效果。AaERF1和AaERF2的基因表达都受到茉莉酸处理的诱导,显示出其调控作用与植物激素信号系统之间可能存在着联系[24]。张芳源等[25]发现一个bZIP家族的转录因子AabZIP1还能够与ADS和CYP71AV1上游的ABRE元件相结合,表明ABA可能也参与了青蒿素的代谢调控。
此外,Pandey等[26]的研究表明,青蒿素的合成受到UV-B的强烈诱导,而其合成途径中的DBR2基因的表达受到UV-B诱导的DNA甲基化的调控[27]。这一发现将表观遗传学的研究工作引进了青蒿素的代谢调控领域。而洪高洁等[28]发现在黄花蒿中过表达拟南芥蓝光受体CRY1也可以增加青蒿素的积累。
4 青蒿素代谢工程与合成生物学疟疾多发生在欠发达国家或地区,而青蒿素是首推的抗疟良药。因此,确保青蒿素的高效生产和稳定供应始终是一个重要的话题。目前栽培黄花蒿仍然是获得青蒿素最有效的手段。尽管黄花蒿生长环境比较宽泛,但植株中较低的青蒿素含量以及栽培条件等诸多因素的影响使得青蒿素的产量难以满足制药市场的需求。通过过量表达关键酶基因以及抑制代谢旁路等手段,可以在一定程度上提高转基因黄花蒿中的青蒿素含量[29, 30]。
除了增加通往青蒿素生物合成的代谢流,提高青蒿素的贮存能力也是解决产量问题的一个有效途径。2010年,Gra-ham等[31]利用转录组深度测序的方法,分析了可能与青蒿素含量相关的数量基因位点(quantitativetraitlocus,QTL)。根据基于青蒿素含量以及叶面积与腺毛密度等因素的分析,他们获得了包括DXR2和MAX3在内的一系列与青蒿素合成相关的位点。这一结果为通过遗传育种的手段获得青蒿素高产品系提供了依据。今年,通过表达一个β-葡萄糖苷酶,Singh等[32]也促进了转基因黄花蒿的腺毛密度,并提高了青蒿素含量。
细胞工程和基因工程等手段可用于提高植物中天然产物的合成与积累。国内外不同实验室先后在黄花蒿发根培养条件优化以及促进其中青蒿素含量的提高等方面开展了一系列的研究工作[33~41],这些研究对于以细胞或发根为材料,通过生物反应器大量培养以获得青蒿素提供了较为全面的数据,也揭示了在青蒿素生物合成过程中可能存在的一些调控因素。
随着参与青蒿素生物合成的各种酶基因不断得到克隆,通过转基因手段在植物体内促进其生物合成、或在微生物中重建其代谢途径的工作取得了较大的进展。1996年Vergauwe等[42]建立了黄花蒿的根癌农杆菌高效转化体系,不仅使黄花蒿的遗传转化成为可能,也将其在植物中的干重含量提高到0.17%。1998年陈大华等[36]也建立了发根农杆菌的转化体系,并开始尝试将一些参与萜类代谢的基因转入黄花蒿的发根。景福远等[43]通过在黄花蒿中过表达其自身的CYP71AV1,将植株中的青蒿素含量提高了约2.4倍。随着越来越多的基因得到鉴定,在微生物中合成青蒿素的可能性也日渐提升。Keasling实验室首先通过在大肠杆菌中表达ADS和来自酵母的MVA途径,合成了紫穗槐-4,11-二烯[44];随即他们又在酵母中表达ADS、CYP71AV1及CPR,实现了青蒿酸的微生物合成[45, 46]。而在加入DBR2后,酵母得以进一步产生二氢青蒿酸[15]。以此为底物,进行青蒿素的半化学合成工作已经显现出诱人的前景[13, 47],这也是合成生物学在天然产物合成领域的第一个成功案例。由于参与植物特异代谢的酶活性较低,运用蛋白工程方法对其进行改造能够进一步提高合成生物学体系的效率。李建戌等[48]通过改造ADS催化反应中影响反应产物选择性的可塑性氨基酸残基,将ADS的活性提高了约70%,这是青蒿素代谢工程的又一种新策略。
5 展望青蒿素的发现和应用表明,新的药物成分一经发现,将对人类健康产生重大的促进作用。植物的天然产物是一个巨大的药物资源宝库。对已知产物的功能分析和寻找尚未了解的新的天然产物将是一个长期但具有重大意义的研究工作。虽然中国植物资源丰富,但是与制药产业的需求量相比仍远远不敷供应。加强对植物天然产物的代谢途径及其调控方式的研究工作将有助于阐明这些成分的生物合成过程,并为利用代谢工程的方法促进其合成与积累、或通过合成生物学的手段异源合成这些产物提供支持。
| [1] | 青蒿结构研究协作组.一种新型的倍半萜内酯——青蒿素[J].科学通报,1977, 22(3): 142. |
| [2] | 刘静明,倪慕云,樊菊芬,等.青蒿素(Arteannuin)的结构和反应[J].化学学报, 1979, 37(2): 129-143. |
| [3] | 林有润.中国蒿属志——中国蒿属植物的系统分类、分布和主要经济用途[J].植物研究, 1988, 8: 1-61. |
| [4] | Tellez M R, Canel C, Rimando A M, et al. Differential accumulation of isoprenoids in glanded and glandless Artemisia annua L.[J]. Phytochem-istry, 1999, 52: 1035-1040. |
| [5] | Jia J W, Crock J, Lu S, et al. (3R)-Linalool synthase from Artemisia an-nua L.: cDNA isolation, characterization, and wound induction[J]. Archives of Biochemistry and Biophysics, 1999, 372: 143-149. |
| [6] | Cai Y, Jia J W, Crock J, et al. A cDNA clone for β-Caryophyllene syn-thase from Artemisia annua L.[J]. Phytochemistry, 2002, 61: 523-529. |
| [7] | Lu S, Xu R, Jia J W, et al. Cloning and functional characterization of a β-pinene synthase from Artemisia annua L. that shows a circadian pat-tern of expression[J]. Plant Physiology, 2002, 130: 477-486. |
| [8] | Matsushita Y, Kang W, Charlwood B V. Cloning and analysis of a cDNA encoding farnesyl diphosphate synthase from Artemisia annua[J]. Gene, 1996, 172: 207-209. |
| [9] | Chang Y J, Song S H, Park S H, et al. Amorpha-4,11-diene synthase of Artemisia annua: cDNA isolation and bacterial expression of a ter-pene synthase involved in artemisinin biosynthesis[J]. Archives of Bio-chemistry and Biophysics, 2000, 383: 178-184. |
| [10] | Mercke P, Bengtsson M, Bouwmeester H J, et al. Molecular cloning, expression, and characterization of amorpha-4,11-diene synthase, a key enzyme of artemisinin biosynthesis in Artemisia annua L.[J]. Ar-chives of Biochemistry and Biophysics, 2000, 381: 173-180. |
| [11] | Teoh K H, Polichuk D R, Reed D W, et al. Artemisia annua L. (As-teraceae) trichome-specific cDNAs reveal CYP71AV1, a cytochrome P450 with a key role in the biosynthesis of the antimalarial sesquiter-pene lactone artemisinin[J]. FEBS Letters, 2006, 580: 1411-1416. |
| [12] | Teoh K H, Polichuk D R, Reed D W, et al. Molecular cloning of an al-dehyde dehydrogenase implicated in artemisinin biosynthesis in Arte-misia annua[J]. Botany, 2009, 87: 635-642. |
| [13] | Paddon C J, Westfall P J, Pitera D J, et al. High-level semi-synthetic production of the potent antimalarial artemisinin[J]. Nature, 2013, 496: 528-532. |
| [14] | Bharel S, Gulati A, Abdin M Z, et al. Enzymatic synthesis of artemis-inin from natural and synthetic precursors[J]. Journal of Natural Prod-ucts, 1998, 61, 633-636. |
| [15] | Zhang Y, Teoh K H, Reed D W, et al. The molecular cloning of arte-misinic aldehyde D11(13) reductase and its role in glandular tri-chome-dependent biosynthesis of artemisinin in Artemisia annua[J]. Jouranl of Biological Chemistry, 2008, 283: 21501-21508. |
| [16] | Bertea C M, Voster A, Verstappen F W, et al. Isoprenoid biosynthesis in Artemisia annua: Cloning and heterologous expression of a germacrene A synthase from a glandular trichome cDNA library[J]. Archives of Biochemistry and Biophysics, 2006, 448: 3-12. |
| [17] | Kim S H, Chang Y J, Kim S U. Tissue specificity and developmental pattern of amorpha-4,11-diene synthase (ADS) proved by ADS pro-moter-driven GUS expression in the heterologous plant, Arabidopsis thaliana[J]. Planta Medica, 2008, 74: 188-193. |
| [18] | Wang H, Han J, Kanagarajan S, et al. Trichome-specific expression of the amorpha-4,11-diene 12-hydroxylase (cyp71av1) gene, encoding a key enzyme of artemisinin biosynthesis in Artemisia annua, as reported by a promoter-GUS fusion[J]. Plant Molecular Biology, 2013, 81: 119-138. |
| [19] | Olsson M E, Olofsson L M, Lindahl A L, et al. Localization of enzymes of artemisinin biosynthesis to the apical cells of glandular secretory trichomes of Artemisia annua L.[J]. Phytochemistry, 2009, 70: 1123-1128. |
| [20] | Ji Y, Xiao J, Shen Y, et al. Cloning and characterization of AabHLH1, a bHLH transcription factor that positively regulates artemisinin bio-synthesis in Artemisia annua[J]. Plant and Cell Physiology 2014, 55: 1592-1604. |
| [21] | Lu X, Jiang W, Zhang L, et al. Characterization of a novel ERF tran-scription factor in Artemisia annua and its induction kinetics after hor-mones and stress treatments[J]. Molecular Biology Reports, 2012, 39: 9521-9527. |
| [22] | Lu X, Zhang L, Zhang F, et al. AaORA, a trichome-specific AP2/ ERF transcription factor of Artemisia annua, is a positive regulator in the artemisinin biosynthetic pathway and in disease resistance to Botrytis cinerea[J]. New Phytologist, 2013, 198: 1191-1202. |
| [23] | Ma D, Pu G, Lei C, et al. Isolation and characterization of AaWRKY1, an Artemisia annua transcription factor that regulates the amorpha-4,11-diene synthase gene, a key gene of artemisinin biosyn-thesis[J]. Plant and Cell Physiology, 2009, 50: 2146-2161. |
| [24] | Yu Z X, Li J X, Yang C Q, et al. The jasmonate-responsive AP2/ERF transcription factors AaERF1 and AaERF2 positively regulate artemis-inin biosynthesis in Artemisia annua L.[J] Molecular Plant, 2012, 5: 353-365. |
| [25] | Zhang F, Fu X, Lü Z, et al. A basic leucine zipper transcription fac-tor, AabZIP1, connects abscisic acid signaling with artemisinin biosyn-thesis in Artemisia annua[J]. Molecular Plant, 2015, 8: 163-175. |
| [26] | Pandey N, Pandey-Rai S. Short term UV-B radiation-mediated tran-scriptional responses and altered secondary metabolism of in vitro propagated plantlets of Artemisia annua L.[J]. Plant Cell Tissue and Organ Culture, 2014, 116: 371-385. |
| [27] | Pandey N, Pandey-Rai S. Deciphering UV-B-induced variation in DNA methylation pattern and its influence on regulation of DBR2 ex-pression in Artemisia annua L.[J]. Planta, 2015, 242: 869-879. |
| [28] | Hong G J, Hu W L, Li J X, et al. Increased accumulation of artemis-inin and anthocyanins in Artemisia annua expressing the arabidopsis blue light receptor CRY1[J]. Plant Molecular Biology Reporter, 2009, 27(3): 334-341. |
| [29] | Shen Q, Chen Y F, Wang T, et al. Overexpression of the cytochrome P450 monooxygenase (cyp71av1) and cytochrome P450 reductase (cpr)genes increased artemisinin content in Artemisia annua (Asteraceae)[J]. Genetics and Molecular Research, 2012, 11: 3298-3309. |
| [30] | Zhang L, Jing F, Li F, et al. Development of transgenic Artemisia an-nua (Chinese wormwood) plants with an enhanced content of artemis-inin, an effective anti-malarial drug, by hairpin-RNA-mediated gene silencing[J]. Biotechnology and Applied Biochemistry, 2009, 52: 199-207. |
| [31] | Graham I A, Besser K, Blumer S, et al. The genetic map of Artemisia annua L. identifies loci affecting yield of the antimalarial drug artemis-inin[J]. Science, 2010, 327: 328-331. |
| [32] | Singh N D, Kumar S, Daniell H. Expression of β-glucosidase increas-es trichome density and artemisinin content in transgenic Artemisia an-nua plants[J]. Plant Biotechnology Journal, 2015: doi: 10.1111/ pbi.12476. |
| [33] | 罗桂芬,胡虹,段金玉.黄花蒿的组织培养[J].植物生理学通讯, 1995, 31: 207. |
| [34] | 蔡国琴,李国珍,叶和春,等. Ri质粒转化的青蒿发根培养及青蒿素的生物合成[J].生物工程学报, 1995, 11: 315-320. |
| [35] | 刘春朝,王玉春,欧阳藩,等.青蒿毛状根分支生长的动力学模型[J].应用与环境生物学报, 1997, 3: 385-388. |
| [36] | 陈大华,孟玉玲,叶和春,等.青蒿转杜松烯合成酶基因发根系的培养[J].植物学报, 1998, 40: 711-714. |
| [37] | 刘本叶,叶和春,李国凤,等.青蒿发根生长及青蒿素生物合成动态的研究[J].生物工程学报, 1998, 14: 401-404. |
| [38] | Wyslouzil B E, Waterbury R G, Weathers P J. The growth of single roots of Artemisia annua in nutrient mist reactors[J]. Biotechnology and Bioengineering, 2000, 70: 143-150. |
| [39] | Arsenault P R, Vail D R, Wobbe K K, et al. Effect of sugars on arte-misinin production in Artemisia annua L.: Transcription and metabo-lite measurements[J]. Molecules, 2010, 15: 2302-2318. |
| [40] | Weathers P J, Arsenault P R, Covello P S, et al. Artemisinin produc-tion in Artemisia annua: Studies in planta and results of a novel deliv-ery method for treating malaria and other neglected diseases[J]. Phyto-chem Reviews, 2011, 10: 173-183. |
| [41] | Nguyen K T, Towler M J, Weathers P J. The effect of roots and media constituents on trichomes and artemisinin production in Artemisia an-nua L.[J]. Plant Cell Reports, 2013, 32: 207-218. |
| [42] | Vergauwe A, Cammaert R, Vandenberghe D, et al. Agrobacterium tu-mefaciens-mediated transformation of Artemisia annua L. and regener-ation of transgenic plants[J]. Plant Cell Reports, 1996, 15: 929-933. |
| [43] | 景福远,张凌,李美芽,等.过量表达cyp71av1和cpr基因提高青蒿中青蒿素的含量[J].中国农业科技导报, 2008, 10: 64-70. |
| [44] | Martin V J, Pitera D J, Withers S T, et al. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids[J]. Nature Bio-technology, 2003, 21: 796-802. |
| [45] | Ro D K, Paradise E M, Ouellet M, et al. Production of the antimalari-al drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440: 940-943. |
| [46] | Lindahl A L, Olsson M E, Mercke P, et al. Production of the artemis-inin precursor amorpha-4,11-diene by engineered Saccharomyces cere-visiae[J]. Biotechnology Letters, 2006, 28: 571-580. |
| [47] | Paddon C J, Keasling J D. Semi-synthetic artemisinin: A model for the use of synthetic biology in pharmaceutical development[J]. Nature Reviews Microbiology, 2014, 12: 355-367. |
| [48] | Li J X, Fang X, Zhao Q, et al. Rational engineering Plasticity residues of sesquiterpene synthases from Artemisia annua: Product specificity and catalytic efficiency[J]. Biochemical Journal, 2013, 451(3): 417-426. |

