支气管哮喘(bronchial asthma)简称哮喘,目前全球有3亿左右的患者,并且患病人数逐年增加。哮喘是一种多因素异质性呼吸系统疾病,受基因与环境的共同作用[1]。大气污染是哮喘的危险因素之一。Hsu等[2]的研究表明,母亲怀孕(16~25)周期间增加PM2.5的暴露水平和儿童早期哮喘的发展呈显著相关。随着基因表达调控研究的深入,人们发现存在DNA序列以外的调控机制,这种调控机制被称为表观遗传学。Ivana等[3]的研究发现,长期暴露于污染空气、烟草烟雾等环境的人群与其表观遗传标志的改变有密切关联,同时增加了哮喘的发生风险。Kuo等[4]和Lee等[5]的研究表明,表观遗传学中组蛋白乙酰化与哮喘发病机制密切相关。
本实验通过建立PM2.5刺激大鼠哮喘加重模型,探究组蛋白去乙酰化酶、组蛋白不同位点乙酰化水平的改变,进而探究PM2.5加重哮喘的表观遗传学机制。
1 材料方法 1.1 主要仪器与试剂 1.1.1 仪器酶标分析仪(BioRad公司,美国),DYCZ-24DN型电泳仪(北京六一仪器厂),荧光定量PCR仪Line Gene 9640(杭州博日科技有限公司,BIOER),高分辨率倒置显微镜(OLYMPUS)
1.1.2 试剂EpiQuikTM总组蛋白提取试剂盒、EpiQuikTM总组蛋白H3K9、H3K18乙酰化(H3K9ac、H3K18ac)定量检测试剂盒(Epigentek, 美国),组蛋白H3K9ac、H3K18ac抗体(GeneTex, 美国),HDAC1、HDAC2抗体(Santa Cruz Biotechnology,美国),反转录试剂盒Primer Script RT Master Mix(Takara公司,日本),2×SYBR Green qPCR Master Mix(徐州溥博生物科技有限公司)。
1.2 动物分组及建立哮喘模型选择体重为(170~210) g的健康SPF级雄性SD大鼠45只,随机分为5组,每组9只,分别为生理盐水对照组(1.5 mg/kg)、哮喘组(1.5 mg/kg OVA)、PM2.5低剂量刺激哮喘组(1.5 mg/kg PM2.5+OVA)、PM2.5中剂量刺激哮喘组(6 mg/kg PM2.5+OVA)、PM2.5高剂量刺激哮喘组(24 mg/kg PM2.5+OVA)。实验第1、8、15天致敏。哮喘组、PM2.5低剂量刺激哮喘组、PM2.5中剂量刺激哮喘组、PM2.5高剂量刺激哮喘组于实验第1天腹腔注射生理盐水混悬液1 mL(含OVA100 mg、氢氧化铝100 mg)致敏,第8天和第15天经腹腔注射生理盐水混悬液1 mL(含OVA 10 mg、氢氧化铝100 mg)致敏;生理盐水对照组以生理盐水代替致敏剂注射。哮喘组、PM2.5刺激哮喘组第22天开始给予3%OVA激发液100 mL高频雾化吸入,每天1次,每次30 min,连续2周。生理盐水对照组以生理盐水代替雾化液。PM2.5低、中、高剂量刺激哮喘组于雾化开始前2 h进行PM2.5气管滴注染毒,每3 d染毒1次。动物于第36天处死、收集标本,具体实验细节参考本课题组发表文章[6]。
1.3 指标检测 1.3.1 肺组织HDAC1、HDAC2mRNA的测定取肺组织约50 mg加入1 mLTRizol提取总RNA,检测其纯度并定量,保证纯度在1.8~2.2之间。对定量后的RNA进行反转录。配制20 μL PCR反应液,包括:2×SYBR Green qPCR Master Mix 10 μL,Primer(10 μmol/L)0.8 μL,ddH2O(灭菌)6.4 μL,cDNA 2 μL。采用两步法PCR扩增标准程序:预变性,95℃ 30 s循环1次;PCR反应,95℃ 5 s,60℃ 30 s,循环40次;各基因引物序列如表 1所示。
| 基因 | 碱基序列 |
| β-actin-F | GGAGATTACTGCCCTGGCTCCTA |
| β-actin-R | GACTCATCGTACTCCTGCTTGCTG |
| HDAC1-F | GTGAGGACTGTCCGGTATTTGATG |
| HDAC1-R | GATGTCCGTCTGCTGCTTATTGA |
| HDAC2-F | TGGGCTGCTTCAACCTAACTGTC |
| HDAC2-R | ACTGCAGTCTCATACGTCCAACATC |
1.3.2 Western blot检测HDAC1、HDAC2水平
取肺组织约30 mg,加入增强型RIPA裂解液及PMSF抽提蛋白,选用BCA蛋白定量试剂盒测定浓度。将各组蛋白调至同一浓度,加入相应的蛋白示踪上样缓冲液。取约60 μg总蛋白进行十二烷基硫酸钠聚丙烯酰胺凝胶垂直电泳(SDS-PAGE)。电泳后湿转至硝酸纤维素NC膜上,用质量分数为5%的脱脂奶粉室温封闭2 h后加入HDAC1、HDAC2抗体孵育(工作浓度1 :500),4℃过夜。PBS漂洗10 min,3次,加入相应二抗37℃孵育2 h,PBS漂洗10 min,3次。避光条件下采用化学发光显色液进行条带曝光。
1.3.3 总组蛋白提取用EpiQuickTM总组蛋白提取试剂盒(Epigentek,美国)对总组蛋白进行提取。取肺组织约200 mg,剪碎,加入1×预裂解缓冲液1 mL,组织匀浆,匀浆液转移入2 mL离心管中,于4℃、10 000 rpm离心5 min,弃去上清。用3倍体积的裂解缓冲液重悬肺组织匀浆,冰上孵育30 min。于4℃、12 000 rpm离心5 min,将上清液转入另一个新离心管中。按说明书配置DTT平衡缓冲液后,立即向上清液中加入0.3倍体积的DTT平衡缓冲液,即为提取的总组蛋白。用微量BCA蛋白定量试剂盒进行组蛋白定量。保存于-80℃。
1.3.4 组蛋白H3K9ac、H3K18ac的检测用EpiQuickTM总组蛋白H3K9、H3K18乙酰化定量检测试剂盒(Epigentek, 美国)对H3K9ac、H3K18ac水平进行定量检测。将试剂盒中标准对照品进行梯度稀释,获取0、100、50、25、12.5、6.25、3.125 ng/μL的标品。根据蛋白定量结果,向待测样品各孔中加入150 ng待测组蛋白;按试剂盒说明书进行操作,最终经酶标仪450 nm处测定吸光度。根据标准对照品各蛋白浓度及吸光度绘制标准曲线;根据如下公式计算各样品的H3K9ac、H3K18ac水平:
Amount(ng/mg protein)=OD(sample-blank)/(protein(μg)×slope)×1 000
1.3.5 石蜡标本切片的免疫组化染色免疫组织化学染色采用SABC法。石蜡切片常规脱蜡至水。3% H2O2去离子水室温孵育10 min,消除内源性过氧化物酶活性。对抗原进行热修复。滴加一抗5% BSA封闭液37℃孵育30 min,甩干。滴加一抗(组蛋白H3K9ac、H3K18ac抗体,工作浓度1 :500),37℃孵育2 h,PBS冲洗。滴加生物素标记的羊抗小鼠/兔IgG(S-P试剂盒C试剂),湿盒内37℃孵育30 min,PBS冲洗。滴加过氧化物酶标记的链霉素亲和素(S-P试剂盒D试剂),37℃孵育30 min,PBS冲洗。DAB显色,镜下控制反应时间,自来水充分冲洗。苏木素复染,脱水,透明。中性树胶封片。
1.4 统计学分析采用SPSS 22.0软件进行统计分析。计量资料以x±s表示,组间比较采用方差分析,若各组方差齐则采用LSD法进行两两比较。检验水准α=0.05。
2 结果 2.1 大鼠肺组织HDAC1、HDAC2mRNA水平各组大鼠肺组织HDAC1mRNA表达量差异无统计学意义(F=1.07,P > 0.05;表 2)。HDAC2 mRNA表达量差异有统计学意义(F=4.65,P < 0.05);进一步多重比较显示,PM2.5中、高剂量刺激哮喘组与哮喘组间有显著差异(P < 0.05)。
| 分组 | HDAC1 | HDAC2 |
| 生理盐水对照组 | 1.19±0.11 | 1.09±0.25 |
| 哮喘组 | 0.84±0.24 | 1.04±0.13 |
| PM2.5低剂量刺激哮喘组 | 0.77±0.19 | 0.75±0.33 |
| PM2.5中剂量刺激哮喘组 | 0.76±0.08 | 0.66±0.21* |
| PM2.5高剂量刺激哮喘组 | 0.93±0.21 | 0.58±0.15* |
| 注:*表示与哮喘组比较,P < 0.05 | ||
2.2 大鼠肺组织HDAC1、HDAC2蛋白表达水平
Western blot结果显示(图 1,表 3),各组大鼠肺组织HDAC1蛋白表达差异有统计学意义(F=4.37,P < 0.05);多重比较显示,PM2.5中剂量刺激哮喘组与哮喘组间有显著差异(P < 0.05);HDAC2蛋白表达差异有统计学意义(F=3.21,P < 0.05);多重比较显示,PM2.5高剂量刺激哮喘组与哮喘组间有显著差异(P < 0.05)。

|
| 注:A:生理盐水对照组;B:哮喘组;C:PM2.5低剂量刺激哮喘组;D:PM2.5中剂量刺激哮喘组;E:PM2.5高剂量刺激哮喘组 图 1 PM2.5染毒对哮喘大鼠HDAC1、HDAC2蛋白表达的影响 |
| 分组 | HDAC1/β-actin | HDAC2/β-actin |
| 生理盐水对照组 | 1.598±0.350 | 1.203±0.329 |
| 哮喘组 | 1.243±0.287 | 1.003±0.272 |
| PM2.5低剂量刺激哮喘组 | 0.995±0.329 | 0.885±0.131 |
| PM2.5中剂量刺激哮喘组 | 0.720±0.298* | 0.850±0.276 |
| PM2.5高剂量刺激哮喘组 | 0.893±0.362 | 0.625±0.062* |
| 注:*表示与哮喘组比较,P < 0.05 | ||
2.3 大鼠肺组织组蛋白H3K9、H3K18乙酰化水平
各组大鼠肺组织H3K9乙酰化水平差异有统计学意义(F=5.51,P=0.01;表 4);多重比较显示,PM2.5低、中、高剂量刺激哮喘组分别与哮喘组间有显著差异(P < 0.05)。H3K18乙酰化水平差异有统计学意义(F=3.54,P=0.02);多重比较显示,PM2.5高剂量刺激哮喘组与哮喘组、PM2.5低、中剂量刺激哮喘组间有显著差异(P < 0.05)。
| ng/(mg ·pro) | ||
| 分组 | H3K9ac | H3K18ac |
| 生理盐水对照组 | 1 259.95±122.39 | 1 836.24±163.96 |
| 哮喘组 | 1 271.35±75.01 | 1 845.96±158.79 |
| PM2.5低剂量刺激哮喘组 | 1 431.72±115.97a | 1 882.15±138.32 |
| PM2.5中剂量刺激哮喘组 | 1 522.86±171.88a | 1 878.49±101.73 |
| PM2.5高剂量刺激哮喘组 | 1 492.86±73.58a | 2 133.45±192.97abc |
| 注:a表示与哮喘组比较,P < 0.05;b表示与PM2.5低剂量刺激哮喘组比较,P < 0.05;c表示与PM2.5中剂量刺激哮喘组比较,P < 0.05 | ||
2.4 大鼠支气管上皮细胞乙酰化H3K9、H3K18蛋白表达
免疫组织化学显示,H3K9ac、H3K18ac蛋白表达主要集中在支气管上皮细胞。生理盐水对照组大鼠各级支气管上皮细胞H3K9ac、H3K18ac蛋白呈阴性或弱阳性(图 2A、图 3A),哮喘组各级支气管上皮细胞H3K9ac、H3K18ac蛋白呈棕黄色强阳性表达(图 2B、图 3B),PM2.5刺激哮喘组各级支气管上皮细胞H3K9ac、H3K18ac蛋白的表达明显增强(图 2CDE、图 3CDE),各组相对吸光度见表 5。
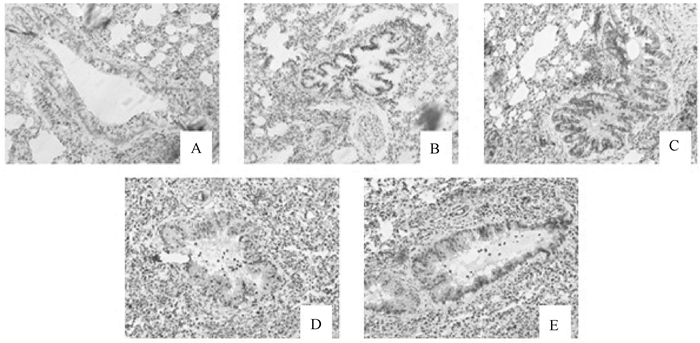
|
| 注:A生理盐水对照组;B哮喘组;C PM2.5低剂量刺激哮喘组;D PM2.5中剂量刺激哮喘组;E PM2.5高剂量刺激哮喘组 图 2 PM2.5染毒对哮喘大鼠支气管上皮细胞H3K9ac蛋白表达的影响(免疫组织化学×200) |
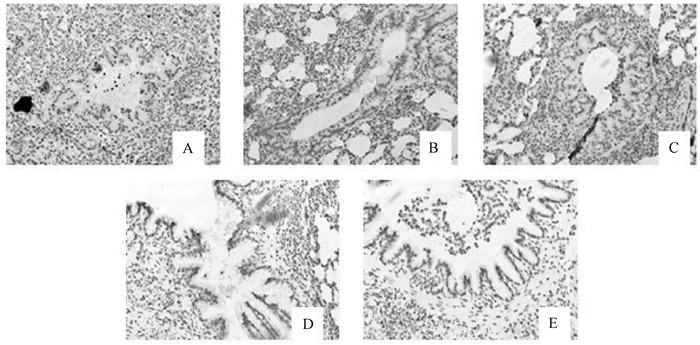
|
| 注:A生理盐水对照组;B哮喘组;C PM2.5低剂量刺激哮喘组;D PM2.5中剂量刺激哮喘组;E PM2.5高剂量刺激哮喘组 图 3 PM2.5染毒对哮喘大鼠支气管上皮细胞H3K18ac蛋白表达的影响(免疫组织化学×200) |
| 分组 | H3K9ac | H3K18ac |
| 生理盐水对照组 | 0.075 2±0.006 3 | 0.100 7±0.002 3 |
| 哮喘组 | 0.104 0±0.002 1a | 0.136 6±0.005 8a |
| PM2.5低剂量刺激哮喘组 | 0.106 1±0.005 5 | 0.153 8±0.001 1b |
| PM2.5中剂量刺激哮喘组 | 0.113 7±0.008 9b | 0.148 9±0.000 8b |
| PM2.5高剂量刺激哮喘组 | 0.115 0±0.003 1bc | 0.273 9±0.015 1bcd |
| 注:a表示与生理盐水对照组比较差异有统计学意义;b表示与哮喘组比较差异有统计学意义;c表示与PM2.5低剂量组比较差异有统计学意义;d表示与PM2.5中剂量组比较差异有统计学意义 | ||
3 讨论
本研究成功建立PM2.5诱导大鼠哮喘加重模型,主要表现为哮喘组和PM2.5刺激哮喘组大鼠行为的改变,摄食减少、狂躁不安、激发期间出现喘息及点头、端坐呼吸等;支气管肺泡灌洗液(BALF)中细胞总数明显升高,嗜酸粒细胞百分比增高;肺病理切片炎性细胞浸润、肺泡结构破坏及融合、腔内粘液量增加等典型哮喘特征[6]。
表观遗传调控机制对细胞的分化、增殖和炎症基因的表达起关键作用[7]。组蛋白尾端的多重翻译后修饰包括乙酰化、甲基化、磷酸化、泛素化,其中组蛋白的乙酰化修饰和甲基化修饰对染色质重构和基因表达起核心作用[8]。在基因转录活跃区,核心组蛋白的乙酰化程度高;在基因转录非活跃区则相反[9]。组蛋白去乙酰化酶(HDACs)使组蛋白去乙酰化,DNA与组蛋白八聚体紧密结合,DNA上与各种转录因子和协同转录因子结合的位点不易识别,抑制基因表达。
组蛋白去乙酰化酶是炎症基因诱导和细胞增殖的重要驱动因素[10]。本研究中,哮喘组HDAC2 mRNA表达量低于生理盐水对照组,PM2.5刺激哮喘组低于哮喘组,随PM2.5染毒剂量的增加而降低。其中PM2.5中、高剂量刺激哮喘组HDAC2mRNA转录水平明显低于哮喘组,差异有统计学意义。提示哮喘大鼠肺组织中HDAC2转录水平降低,PM2.5可进一步降低HDAC2mRNA的表达。Western blot结果显示哮喘组HDAC1、HDAC2蛋白表达量低于生理盐水对照组。PM2.5中剂量刺激哮喘组HDAC1蛋白表达量明显低于哮喘组、PM2.5高剂量刺激哮喘组HDAC2蛋白表达量明显低于哮喘组,差异有统计学意义。提示哮喘大鼠肺组织中HDAC1、HDAC2蛋白表达量降低,PM2.5可进一步降低哮喘大鼠肺组织HDAC1、HDAC2蛋白的表达。Ito K研究证明[11],在哮喘患者体内HDAC活性降低,同时HDAC1和HDAC2蛋白表达量降低。Gilmour等[12]人体外研究证实PM10与组蛋白乙酰化转移酶的活性增加有关。最新研究表明,HDAC3基因敲除小鼠PM2.5染毒后其肺组织中炎症相关信号高表达,促进炎细胞因子释放[13]。人支气管上皮细胞系暴露于柴油车尾气颗粒物(DEP)引起组蛋白去乙酰酶1(HDAC1)的降解[14]。崔彩云的研究证明,吸烟可以使哮喘气道HDAC2蛋白表达进一步降低[15]。以上实验与本研究结果一致。
组蛋白分为5类,包括H1、H2A、H2B、H3、H4,其中与哮喘相关研究较多的为组蛋白H3、H4。本研究中,PM2.5刺激哮喘组较哮喘组H3K9乙酰化水平升高,差异有统计学意义。PM2.5高剂量刺激哮喘组较哮喘组、PM2.5低、中剂量刺激哮喘组H3K18乙酰化水平升高,差异有统计学意义。以上结果提示哮喘大鼠肺组织中H3K9、H3K18乙酰化增强,PM2.5暴露可进一步增强二者乙酰化水平。
总组蛋白乙酰化定量检测从整体水平说明PM2.5对大鼠肺组织组蛋白的影响,为进一步探究其表达部位及水平,我们采用免疫组织化学法检测。其结果显示,H3K9ac、H3K18ac蛋白表达主要集中在支气管上皮细胞,哮喘组表达量高于生理盐水对照组,差异有统计学意义;PM2.5刺激哮喘组高于哮喘组,差异有统计学意义,且表达量随PM2.5染毒剂量的增加而增加。
苏新明等[16]研究表明,哮喘小鼠肺组织组蛋白H3的乙酰化增强,其中H3K9、H3K18等多位点乙酰化水平较正常对照组明显增高;Stefanowicz等人[17]发现,与健康受试者相比哮喘患者气道上皮细胞组蛋白H3K18乙酰化水平表达增强。同时,另有研究显示,空气污染中PM可导致组蛋白乙酰化水平升高[18];染色质组蛋白乙酰化在PM10介导的炎症反应中发挥作用[19]。Rui等[20]研究发现,大鼠暴露于交通相关空气污染后H3K9乙酰化水平改变。
综上所述,PM2.5可能通过降低HDAC2基因和HDAC1、HDAC2蛋白的表达,使组蛋白H3K9、H3K18乙酰化水平升高,从而加重大鼠哮喘。
| [1] | Bønnelykke K, Ober C. Leveraging gene-environment interactions and endotypes for asthma gene discovery[J]. J Allergy Clin Immunol, 2016, 137(3): 667–679. doi: 10.1016/j.jaci.2016.01.006 |
| [2] | Hsu HH, Chiu YH, Coull BA, et al. Prenatal particulate air pollution and asthma onset in urban children. identifying sensitive windows and sex differences[J]. Am J Respir Crit Care Med, 2015, 192(9): 1052–1059. doi: 10.1164/rccm.201504-0658OC |
| [3] | Yang IV, Schwartz DA. Epigenetic mechanisms and the development of asthma[J]. J Allergy Clin Immunol, 2012, 130(6): 1243–1255. doi: 10.1016/j.jaci.2012.07.052 |
| [4] | Kuo CH, Hsieh CC, Lee MS, et al. Epigenetic regulation in allergic diseases and related studies[J]. Asia Pac Allergy, 2014, 4(1): 14–18. |
| [5] | Lee KY, Ito K, Hayashi R, et al. NF-κB and activator protein 1 response elements and the role of histone modifications in IL-1β-induced TGF-β1 gene transcription[J]. J Immunol, 2006, 176(1): 603–615. doi: 10.4049/jimmunol.176.1.603 |
| [6] | 郑鑫, 张佳琪, 张志红, 等. iNOS基因甲基化对交通相关PM2.5诱导大鼠哮喘加重中NO的调控作用[J]. 环境与健康杂志, 2016, 33(11): 941–945, 1035. Zheng X, Zhang JQ, Zhang ZH, et al. iNOS gene methylation regulates NO levels in asthma exacerbation rats induced by traffic-related PM2.5[J]. J Environ Health, 2016, 33(11): 941–945, 1035. (in Chinese). |
| [7] | El Biaze M, Boniface S, Koscher V, et al. T cell activation, from atopy to asthma:more a paradox than a paradigm[J]. Allergy, 2003, 58(9): 844–853. doi: 10.1034/j.1398-9995.2003.00282.x |
| [8] | Marmorstein R. Protein modules that manipulate histone tails for chromatin regulation[J]. Nat Rev Mol Cell Biol, 2001, 2(6): 422–432. doi: 10.1038/35073047 |
| [9] | Hake SB, Xiao A, Allis CD. Linking the epigenetic 'language' of covalent histone modifications to cancer[J]. Br J Cancer, 2004, 90(4): 761–769. doi: 10.1038/sj.bjc.6601575 |
| [10] | Adcock IM, Ford P, Ito K, et al. Epigenetics and airways disease[J]. Respir Res, 2006, 7: 21. doi: 10.1186/1465-9921-7-21 |
| [11] | Ito K, Caramori G, Lim S, et al. Expression and activity of histone deacetylases in human asthmatic airways[J]. Am J Respir Crit Care Med, 2002, 166(3): 392–396. doi: 10.1164/rccm.2110060 |
| [12] | Gilmour PS, Rahman I, Donaldson K, et al. Histone acetylation regulates epithelial IL-8 release mediated by oxidative stress from environmental particles[J]. Am J Physiol Lung Cell Mol Physiol, 2003, 284(3): L533–L540. doi: 10.1152/ajplung.00277.2002 |
| [13] | Schiltz RL, Mizzen CA, Vassilev A, et al. Overlapping but distinct patterns of histone acetylation by the human coactivators p300 and PCAF within nucleosomal substrates[J]. J Biol Chem, 1999, 274(3): 1189–1192. doi: 10.1074/jbc.274.3.1189 |
| [14] | Cao DS, Bromberg PA, Samet JM. COX-2 expression induced by diesel particles involves chromatin modification and degradation of HDAC1[J]. Am J Respir Cell Mol Biol, 2007, 37(2): 232–239. doi: 10.1165/rcmb.2006-0449OC |
| [15] | 崔彩云, 杜永成, 胡晓芸, 等. 吸烟对致敏大鼠气道组蛋白去乙酰化酶2及血清中白介素8表达的影响[J]. 国际呼吸杂志, 2011, 31(5): 321–325. Cui CY, Du YC, Hu XY, et al. Effect of smoking on expressional levels of histone deacetylase 2 protein and interleukin-8 in sensitized rats airway and serum[J]. Int J Respir, 2011, 31(5): 321–325. (in Chinese). |
| [16] | 苏新明, 任媛, 赵萱, 等. 慢性支气管哮喘小鼠肺组织中组蛋白H3乙酰化修饰增强[J]. 中国组织化学与细胞化学杂志, 2016, 25(4): 315–321. Su XM, Ren Y, Zhao X, et al. Enhancement of histone H3 acetylation modification in the lung tissue of mice with chronic bronchial asthma[J]. Chin J Histochem Cytochem, 2016, 25(4): 315–321. (in Chinese). |
| [17] | Stefanowicz D, Lee JY, Lee K, et al. Elevated H3K18 acetylation in airway epithelial cells of asthmatic subjects[J]. Respir Res, 2015, 16(1): 95. doi: 10.1186/s12931-015-0254-y |
| [18] | Gilmour PS, Rahman I, Donaldson K, et al. Histone acetylation regulates epithelial IL-8 release mediated by oxidative stress from environmental particles[J]. Am J Physiol Lung Cell Mol Physiol, 2003, 284(3): L533–L540. doi: 10.1152/ajplung.00277.2002 |
| [19] | Donaldson K, Stone V, Borm PJA, et al. Oxidative stress and calcium signaling in the adverse effects of environmental particles (PM10)[J]. Free Rad Biol Med, 2003, 34(11): 1369–1382. doi: 10.1016/S0891-5849(03)00150-3 |
| [20] | Ding R, Jin YT, Liu XN, et al. H3K9 acetylation change patterns in rats after exposure to traffic-related air pollution[J]. Environ Toxicol Pharmacol, 2016, 42: 170–175. doi: 10.1016/j.etap.2016.01.016 |



