2. 中国科学院生态环境研究中心, 水污染控制实验室, 北京 100085;
3. 广西大学, 生命科学与技术学院, 南宁 530005;
4. 广西大学, 亚热带农业生物资源保护与利用国家重点实验室, 南宁 530005
2. Department of Water Pollution Control Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085;
3. College of Life Science and Technology, Guangxi University, Nanning 530005;
4. State Key Laboratory of Conservation and Utilization of Subtropical Agricultural Biological Resources, Guangxi University, Nanning 530005
抗生素因具有杀菌消炎和促进畜禽生长的功效, 从而被大量的应用于人类医疗和畜禽养殖(Bolan et al., 2004).抗生素的大量使用, 导致抗生素耐药菌(antibiotic resistant bacteria, ARB)和抗生素抗性基因(antibiotic resistance genes, ARGS)在环境中广泛传播.目前, 几乎每一种致病细菌都对至少一种抗生素产生了抗性(Munir et al., 2011).全球因感染耐药菌死亡的人数在逐年增加, 到2050年每年因耐药感染死亡的人数将达到1000万, 全球GDP损失累计达100万亿美元(Gustavo Arango-Argoty et al., 2018).而在我国每年生产大约21万t抗生素, 其中约45.4%的抗生素被用于人体医疗, 54.6%的抗生素被应用于畜禽养殖, 但多达30%~90%的抗生素无法被有效利用(Zhang et al., 2018b), 通过尿液和粪便排出, 这就导致污水处理厂和畜禽养殖场成为环境中抗生素抗性基因(ARGs)重要的源, 尤其是剩余污泥和畜禽粪便(Zhang et al., 2015a).
作为有机废弃物的重要处理技术, 厌氧消化技术可实现资源回收、降低环境中的危害(刘新媛等, 2020), 因此是剩余污泥和畜禽粪便处理处置的重要方式.其对ARGs消减的研究是当前的研究热点.然而厌氧消化对污泥、猪粪和鸡粪源ARGs的变化尚无普适性的结论.研究表明厌氧消化能在一定程度上消减污泥中的ARGs(Huang et al., 2016), 但也存在部分ARGs丰度上升的现象(Xu et al., 2019).污泥中温厌氧消化能够消减sulⅠ和sulⅡ(Ma et al., 2011), 而在另外的研究中sulⅠ和sulⅡ却分别富集了25.8%和48.2%(Zhang et al., 2015b).不同的研究对猪粪厌氧消化中ARGs的变化也存在相反的结果, 而污泥和猪粪以及鸡粪源的ARGs在厌氧消化中的差异则更为显著, 近期的研究表明在猪粪的厌氧消化中sulⅠ是先富集后消减(Wu et al., 2020).对于ermB, 在污泥中温厌氧消化中具有富集作用, 而在猪粪厌氧消化中ermB得到大量的消减(Zhang et al., 2017), 在鸡粪中则是先富集后消减(Zhang et al., 2019).这些结果的差异同反应器构型、操作条件、底物组成有关, 而且已经有较多的研究关注反应器构型、操作条件(如SRT和温度等对厌氧消化中ARGs变化趋势的影响), 而关于底物的影响研究尚不足, 因此有必要针对底物类型差异对厌氧消化中ARGs的动态变化进行研究.
厌氧消化中ARGs存在两种形式, 胞内DNA(Intracellular DNA)中的ARGs(Intracellular ARGs, iARGs)和胞外DNA中的ARGs(Extracellular ARGs, eARGs), 而ARGs在厌氧消化中的传播主要通过基因垂直转移(Vertical gene transfer, VGT)和基因水平转移(Horizontal gene transfer, HGT)(Zhang et al., 2013), iARGs主要通过结合或转导来转移, 而eARGs则通过自然转化而传播(Zhang et al., 2018c).eDNA可以来源于死细胞的裂解和活细胞的分泌, 并且可以通过吸附在土壤胶体、沙粒、粘土矿物和腐殖质上而免受核酸酶的攻击(Pietramellara et al., 2008).先前的研究表明, 存在于微生物中的胞外DNA构成了一个动态的基因库, 可通过移动基因原件在环境中扩散(Lu et al., 2010).从而通过生态循坏威胁人类健康.董培艳等研究发现eARGs和iARGs普遍存在于各种典型的环境样品中, 且丰度较高, 并通过模拟实验发现, eARGs具有较高的转化活性, 且吸附在底泥颗粒上的eARGs的转化效率高于游离的eARGs(Dong et al., 2019).然而目前针对污泥和畜禽粪便厌氧消化过程中的ARGs在iDNA和eDNA中的贡献, 以及在不同底物的厌氧消化过程中的变化和传播的研究不足.有必要针对不同底物类型中iDNA和eDNA中的ARGs在厌氧消化过程中的变化进行研究.
综上所述, 本文选取了猪粪、鸡粪和剩余污泥3种不同环境样品进行厌氧消化批次实验, 采用绝对荧光定量PCR技术对其中13种ARGs进行定量检测, 以16S rRNA基因扩增子测序测微生物群落的变化.研究结果为厌氧消化过程中胞内外抗生素抗性基因在不同底物类型中的动态变化提供了理论依据与参考.
2 材料与方法(Materials and methods) 2.1 样品采集本实验使用的剩余污泥来自北京某污水处理厂剩余污泥, 鸡粪和猪粪分别来自北京郊区集约化养猪和养鸡场.接种泥分别收集其各自厌氧消化反应器中.猪粪、鸡粪和城市污泥的总固体(Total solids, TS)和挥发性固体(Volatile solids, VS)分别为31.0%和22.9%、43.6%和35.8%、14.5%和9.1%.3种接种泥的TS和VS分别为7.6%和5.1%、6.5%和3.8%、5.9%和2.7%.猪粪、鸡粪和污泥分别通过常规高压灭菌锅(121 ℃, 30 min)微生物灭菌方法进行热处理以杀死这些底物中的微生物, 然后采用R2A培养基培养灭菌底物为阴性对照验证灭菌效率.
2.2 实验设计本次实验采用碧普全自动甲烷潜力测试系统(Automatic Methane Potential Test System Ⅱ, AMPTS Ⅱ).有效运行体积为0.4 L, 设置150 r·min-1的速度间隙搅拌, 期间1 min搅拌和1 min静置交替.底物类型为猪粪(PM)、鸡粪(CM)、城市污泥(SS)、高压灭菌处理的猪粪(TPM)、高压灭菌处理的鸡粪(TCM)和高压灭菌处理的城市污泥(TSS))和其各自的接种泥按照3∶1(TS)的比例充分混合, 然后将0.4 L的混合物转移到每个瓶中. 每种处理重复3次. 最终的TS约为8%, 装罐后氮吹3~5 min, 以在密封后除去氧气. 然后将瓶子放在水浴中温度控制在37 ℃, 通过装有3 mol·L-1 NaOH溶液的气体洗涤瓶除去产气中的二氧化碳.开启系统并自动测量甲烷的产生.取0、6、16、24、55 d厌氧消化的样品进行后续分析.其具体实验设计流程如图 1所示.
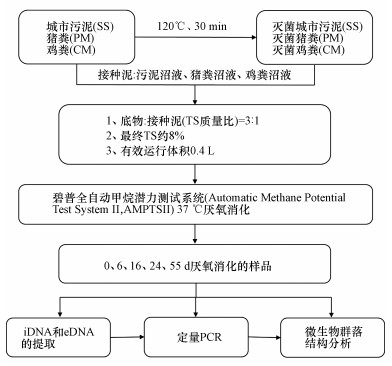 |
| 图 1 实验设计流程图 Fig. 1 Flow chart of experimental design |
吸取样品1 mL, 加入4 mL PBS缓冲溶液, 加入20 μL proteinase K (蛋白酶K 20 mg·mL-1)在37 ℃水浴30 min, 期间每10 min混匀摇晃1次.加入0.2 g PVPP, 样品在250 r·min-1 25 ℃振荡10 min, 之后10000 g 10 min 4 ℃离心, 将离心后的沉积物重悬于4 mL PBS缓冲溶液, 振荡、离心分离操作重复3次.将离心沉淀物放入FastDNATM Spin Kit for Soil试剂盒(MpBio, 美国)提供的Lysing Matrix E tube中进行胞内DNA的提取.将统一收集的上清液在0.22 μm滤膜上过滤, 收集滤过液置于冰上, 向滤液中加入相同体积的异丙醇和1/10体积的乙酸钠, 置于-20 ℃过夜放置, 之后在9600 g 4 ℃离心30 min弃上清液, 沉淀物用TIANamp Micro DNA Kit提取胞外DNA.具体流程见图 2.
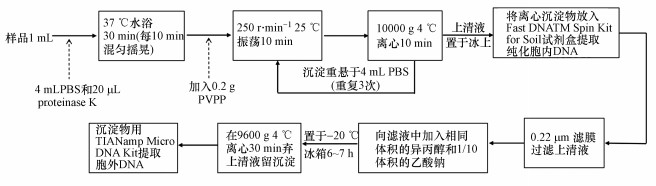 |
| 图 2 DNA提取流程图 Fig. 2 Flow chart of DNA extraction |
本研究采用SYBR Premix Ex TaqTM绝对荧光定量PCR技术对目标ARGs及16sRNA进行定量.本实验测试胞内外目标ARGs包括tetM、tetG、tetX、sulⅠ、sulⅡ、ermB、ermF 、mefA 、ereA、blaCTX-M、blaTEM 、intI1、mcr-1等13种抗生素抗性基因.首先进行质粒拷贝数的计算, 然后将克隆质粒进行10倍梯度稀释进行qPCR反应构建标准曲线, 标准曲线的R2均大于0.998, 扩增效率为82.1%~100.3%.本文中qPCR方法与之前的研究相同(Zhang et al., 2018a), 其引物和退火温度如表 1所示.
| 表 1 本研究中使用的引物及其相应的抗性类型和机制 Table 1 The primers used in this study and their corresponding resistance types and mechanisms |
本研究采用的细菌通用引物515FmodF(5′-GTGYCAGCMGCCGCGGTAA-3′)和806RmodR(5′-GGACTACNVGGGTWTCTAAT-3′)开展微生物群落结构分析(Sampson et al., 2016, Walters et al., 2016).PCR建库测序使用Illumina MiSeq平台并委托上海美吉生物医药科技有限公司完成.其大致流程为:合并成对的末端读数(PEAR:-x, 0.1), 并根据唯一的条形码分配给每个样品; 对合并的读段进行质量控制(PRINSEQ), 并过滤嵌合体(USEARCH)以得到纯净序列; 干净的序列经过归一化处理.处理后的数据使用核糖体数据库项目(RDP)分类器进行分类学分类, 去除低于0.01%的分类单元(Wang et al., 2007), 并通过RDP相关模块计算多样性指数.
2.6 数据分析甲烷产气量以及ARGs使用origin2018分析; 主成分分析(PCA)和Procrustes分析是使用Canoco 5.0(Microcomputer Power, USA)进行的; 使用Paleontological STatistics软件(PAST 3.07)进行了Mantel测试, 并使用Heml 1.0构建了热图(Deng et al., 2014)
3 结果与讨论(Results and discussion) 3.1 不同底物产甲烷效果的对比研究图 3为不同底物厌氧消化累计甲烷产气量趋势图.对于猪粪组, 在36 d之前灭菌猪粪(TPM)的累计产气量要高于猪粪(PM), 且TPM最高甲烷产气量增加了25.8%.而鸡粪组则在48 d前灭菌鸡粪(TCM)累计产气量高于鸡粪(CM), 和猪粪组具有相似的特点.然而到了一定时间后甲烷产气量趋于接近, 猪粪和鸡粪均有类似的现象.高压蒸汽灭菌对于不同底物厌氧消化结束时产甲烷量没有明显的影响, 特别是污泥, 整个过程都未出现明显的变化.在CM和TCM中, 在厌氧消化15 d前出现产气抑制现象, 这种现象与之前的研究一致(Zhang et al., 2019).这与鸡粪中的蛋白质组分较其他畜禽粪便要高, 且在厌氧消化的水解阶段, 易被转化为氨氮有关(Yin et al., 2018).氨氮浓度超过临界值后, 对厌氧菌的正常生理代谢产生不利的影响, 导致消化效率下降, 出现氨抑制.并且未处理的鸡粪中丙酸含量在AD第24 d高达2.38 g·L-1, 在厌氧消化结束时乙酸含量明显降低至0.88 g·L-1.高压蒸汽灭菌的鸡粪和鸡粪具有相同的特点, 在厌氧消化结束时丙酸含量均显著下降.综上所述, 高压蒸汽灭菌处理底物虽然提高了猪粪和鸡粪厌氧消化前中后期的产气速率, 但未显著改变最终的甲烷累计产气量.而预处理污泥厌氧消化产气效果低于未预处理污泥的厌氧消化, 其影响因素主要有高压蒸汽灭菌(即微生物灭菌的方式)此方法较为温和, 目的是为了研究底物中微生物的影响, 未明显改变污泥中的组成.另一方面污泥预处理在厌氧消化前期的甲烷产气效果要高于未处理组, 这符合预处理组要高于未处理组, 而在大概8 d以后污泥预处理后的产气效果低于未处理污泥, 虽然在厌氧消化的中后期预处理组未高于处理组的甲烷产气量, 但也没有显著的差异.
 |
| 图 3 厌氧消化甲烷产气量 Fig. 3 Dynamic changes of accumulative methane production |
不同底物类型中eDNA和iDNA的浓度如图 4所示, iDNA含量为8.3~163.9 μg·g-1, eDNA含量为0.1~1.8 μg·g-1.在厌氧消化结束后, TCM的eDNA与总DNA的比例最高(8.3%), 其次为TPM(7.1%), 在PM、CM、SS和TSS中eDNA与总DNA的比例分别为5.0%、3.2%、3.3%和2.9%.猪粪和鸡粪的高压蒸汽灭菌处理组在厌氧消化结束时的eDNA占总DNA的比例均大于未处理组, 将底物进行高压蒸汽灭菌处理对消减eDNA的量没有明显的效果.对于剩余污泥未有显著的改变(图 4).对不同底物厌氧消化的样品中DNA和16S rRNA进行相关性分析, 观察到DNA浓度与16S rRNA基因的丰度之间存在显著相关性(R2=0.66, p < 0.05), 这表明eDNA主要来自死细胞的裂解(Pietramellara et al., 2008).且iDNA和eDNA在厌氧消化结束后均比开始时降低, 结果表明厌氧消化能够消减不同底物中iDNA, 同时eDNA在3种不同环境底物中均增加.
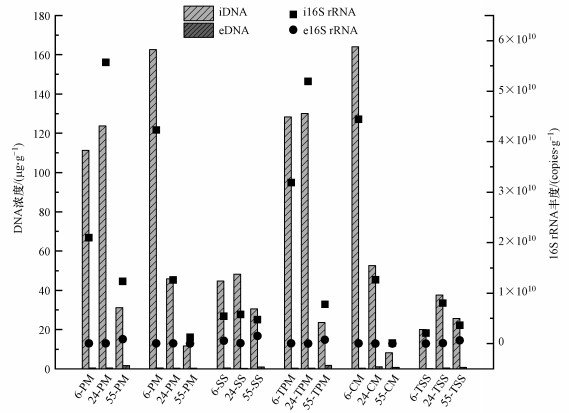 |
| 图 4 不同样品中eDNA和iDNA的浓度以及不同样品中eDNA和iDNA中16S rRNA基因的丰度 (猪粪: PM鸡粪: CM污泥: SS灭菌猪粪: TPM灭菌鸡粪: TCM灭菌污泥: TSS) Fig. 4 The concentration of eDNA and iDNA in different samples and abundance of 16S rRNA gene in eDNA and iDNA in different samples |
将ARGs的绝对丰度进行取log值后从图 5a中可以看出, iARGs的绝对丰度要大于eARGs, 而PM中的iARGs较高于eARGs 1~2 logs(copies·g-1, 以干重计, 下同), CM和SS在本研究中也具有相似的特带你.而对于不同底物类型中胞内外ARGs的变化, iARGs从9.8 logs(copies·g-1)降低至9.4 logs(copies·g-1), eARGs则从6.5 logs(copies·g-1)增加到7.7 logs(copies·g-1).在PM中blaCTX-M和blaTEM在厌氧消化初期胞外DNA中未检测到, 而在AD结束后分别增加了4.6 logs(copies·g-1)和1.9 logs(copies·g-1), CM有相同现象.mcr-1则为相反的结果, 经过AD结束后被去除, 在SS中mcr-1则是经过厌氧消化后中存在于胞外DNA.这与厌氧消化过程中ARGs主要以基因的垂直转移和基因水平转移密切相关.通过对比不同底物高压蒸汽灭菌后经过厌氧消化iARGs和eARGs没有显著的变化, 说明底物微生物对ARGs的转归的影响不大, 而具体的尚没有确定的结论.而对于不同底物以及处理组经过厌氧消化过程中iARGs和eARGs与总ARGs的占比见表 2.
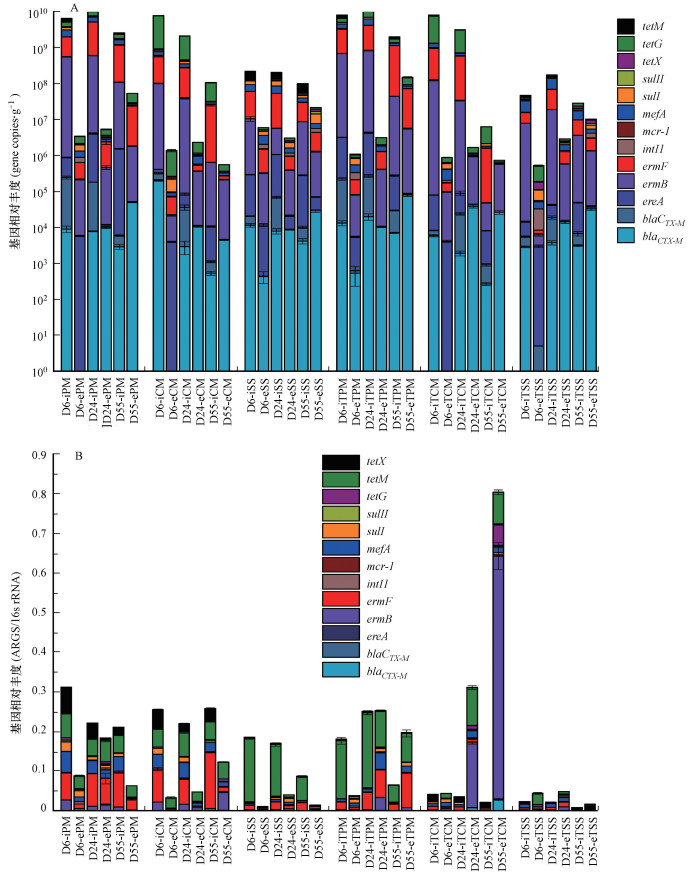 |
| 图 5 样品胞内外DNA中抗生素抗性基因绝对含量(a)和相对丰度 (ARG/16S rRNA gene)(b) Fig. 5 Absolute abundance (a) and relative abundance (ARG/16S rRNA gene) (b) of antibiotic resistance genes in the intracellular and extracellular DNA of the sample |
| 表 2 不同底物类型厌氧消化过程中iARGs和eARGs与总ARGs的占比 Table 2 The proportion of iARGs and eARGs to total ARGs in the anaerobic digestion process of different types of substrate |
从表中可以得出剩余污泥中的eARG在总ARGs中的比例最高(5.47%), 这可能归因于颗粒物的吸附, 从而降低了核酸酶的敏感性.猪粪和鸡粪的eARG占总ARGs的比例分别为0.29%和0.04%.采用微生物灭菌底物处理组中的eARG占总ARGs的比例没有显著的变化.因此高压蒸汽灭菌未改变底物微生物胞内外特征.
PM和SS高压灭菌后ARGs的相对丰度均降低, CM则呈相反的结果.SS和TSS在厌氧消化的每个阶段中细胞外检测到的ARGs均大于细胞内检测到的ARGs, 是因为污泥组中的胞外DNA浓度较高.而污泥中胞外DNA浓度较高的原因为污泥主要由微生物组成, 污泥厌氧消化过程中, 破胞是一个关键环节, 破胞使胞内DNA释放到胞外, 导致胞外DNA浓度过高, 从而使eARGs增加.由此可知厌氧消化使污泥组中的胞外ARGs增加, 增加了环境中ARGs传播的风险.PM和CM中微生物多属于肠道微生物居多, 在经过厌氧消化的每个阶段微生物细胞内的ARGs的相对丰度均大于细胞外的ARGs的相对丰度.从相对丰度图中可以看出在猪粪组中主要的ARGs为ermF、tetM、tetX、mefA, 从tetX主要存在于iDNA, 在eDNA存在较少, 且随着AD的进行, ARGs丰度逐渐降低.ermF在PM中胞内相对丰度呈增加趋势, 而在胞外相对丰度降低, 且在TPM中的变化趋势则相反.在CM的胞内中tetM的含量最高且随着AD进行丰度逐渐降低, 而在胞外检测到的ARGs总体较低.在TCM中, tetM、ermF在细胞内依然是主要的ARGs.对于胞内的ARGs在AD结束时, 其总的相对丰度有所降低.在胞外的ARGs检测中, 厌氧消化初期丰度较低, 而在中期和末期丰度显著增加.正面反映了ARGs在胞内外的转移.而转移的机制尚没有确切的结论.在SS中, 胞内的ARGs相对丰度均较低.胞外的ARGs在AD中后期ermB、tetM、tetG、mefA和blaCTX-M的相对丰度都有所增加, ermB的相对丰度从24 d的0.16增加到AD结束时的0.61, 大约增加了5倍.intI1作为抗生素抗性基因的载体, 在3种不同底物环境中的iDNA和eDNA的相对丰度如表 3所示, intI1在3种环境中主要存在于eDNA中, 丰度最高的存在于第55 d的污泥组的eDNA中, 在其他研究中在来自医院污水处理, 工业污水处理和河水沉积物中均存在eDNA中intI1的相对丰度均高于iDNA中的相对丰度的现象(Dong et al., 2019).与染色体上的16S rRNA基因相比, eARGs的相对丰度比iARGs更高, 可能是因为其降解速度较慢, 且带有ARGs的质粒eDNA的持久性更高(Mao et al., 2014).综上可知, 在环境样品中大量的eARGs可能会促进抗生素耐药性的扩散, 从而通过各种途径对人类健康造成危害.并且同一种抗生素抗性基因在同种底物的细胞内外的变化差异较大, 而在不同底物之间的变化更是具有差异性.
| 表 3 3种典型环境样品中intI1在iDNA和eDNA中的相对丰度 Table 3 The relative abundance of intI1 in iDNA and eDNA in three typical environmental samples |
通过分析3种不同环境样品以及高压灭菌处理后的样品的iDNA和eDNA中门水平的群落分析(图 6).iDNA不仅包含了活的微生物群落中的DNA而且还有死细胞内的DNA, 而胞外DNA则是由胞内释放到胞外的DNA.经过测序结果表明eDNA中的微生物α多样性比iDNA中的多(表 4), 是由于胞内DNA释放到胞外后, 一部分的胞外DNA降解了, 而另外一部分DNA由于难降解和具有持久性的特征随着厌氧消化进行了累计.堆积柱状图反映了3个不同底物中各个反应体系门水平微生物的组成图, 共检测到厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、螺旋菌门(Spirochaetes)和放线菌门(Actinobacteria)在内的5个优势微生物门, 在剩余污泥组中Thermotogae也是优势微生物门之一.在反应初期(第6 d)PM中Firmsxicutes(33%)、Bacteroidetes(34%)、Spirochaetes(19.9%)为iDNA中3个优势门, 而反应初期的eDNA的优势门主要为Firmicutes(44.3%)、Bacteroidetes(15.6%)、Proteobacteria(26.9%).经过高压灭菌后的猪粪在厌氧消化初期, iDNA中的优势门和PM的相同, 但丰度不同, 分别为32.49%、45.7%和9.9%, 而胞外DNA中则与PM有显著的差异, 主要门为Proteobacteria, 其丰度为62.6%.说明厌氧消化和高压蒸汽灭菌预处理共同造成变形菌门大量死亡后释放DNA到环境中.在厌氧消化结束后, PM的iDNA中Firmicutes(42%)、Bacteroidetes(45%), 两个优势门在结束时均富集, eDNA的主要优势门与iDNA的相同, 但其比例分别为51%和23%, 同样也是出现了富集的现象, 而另一个优势门Proteobacteria(11.8%)则呈现下降的趋势, 说明厌氧消化能够消减变形菌门.对于CM和TCM的分析, 厌氧消化的前期和后期iDNA和eDNA中的优势门的变化较小.在AD初期, CM中iDNA中的微生物优势门分别为Firmicutes(24%)、Bacteroidetes(47%); eDNA中优势门主要为Firmicutes(73%).TCM中的iDNA优势门为Firmicutes(40%)、Bacteroidetes(31%), 在AD结束后, CM中的iDNA中的微生物优势门和AD前期相同, 但丰度却分别为54%和39%, eDNA中Proteobacteria由初期的4.4%增加到60%, 而在初期的优势门也分别降低至4.1%和3.6%, 变化显著; 在TCM中, eDNA中的优势门为Proteobacteria(58%), 这种门水平的变化与CM的变化非常相似.SS中iDNA主要的优势微生物门有Firmicutes(23.3%)、Bacteroidetes(28.9%)和Thermotogae(10.3%), 而eDNA中Thermotogae仅仅为0.7%, 且在厌氧消化结束后iDNA中降低, eDNA中增加.其他优势菌群门在厌氧消化前后的iDNA和eDNA均没有显著的变化.不同底物类型经过厌氧消化后菌群的变化趋势各不相同, 同一个优势门在不同底物中的消减和富集没有普遍性的规律.通过属水平热图分析(图 8), PM中主要的优势属为Petrimonas、unclassified_Bacteroidales、Sphaerochaeta、Acholeplasma、Bacteroides、Acinetobacter、Anaerococcus.CM中主要的优势属为Petrimonas、unclassified_Clostridiales、unclassified_Bacteroidales、Bacteroides. SS中的主要优势属unclassified_Bacteria、Petrimonas、Tissierella、Anaerobaculum、Defluviitoga.从优势菌属可以看出中温厌氧消化中优势产甲烷菌以及嗜温菌是相似的.底物中的大多数优势属不能很好地适应厌氧消化的微环境, 且随着AD的进行而消减, 通过属水平胞内外样品的主成分分析(PCA)(图 7), 可知高压蒸汽灭菌在AD过程中对微生物群落动力学没有很大的影响, 属水平群落在胞内外中的分区明显.针对在厌氧消化过程中胞内外优势属的变化不同, 具体如下:PM中胞内的优势属Sphaerochaeta和Bacteroides在厌氧消化结束后分别降低了90%和87.8%, 在CM中也呈现消减状态, 分别降低了74.15%和96.59%.Petrimonas在CM厌氧消化过程中则增加了89.5%, 而其他属没有明显的变化.SS中unclassified_Bacteria、Petrimonas均在消化结束后分别增加了29.66%和37.98%, Defluviitoga、Macellibacteroides和Pirellula分别降低了90.34%、97.90%和95.78%.在胞外DNA中, PM中主要优势属Petrimonas和unclassified_Clostridiales, 其在厌氧消化结束后均增加, 而Acholeplasma、Acinetobacter、unclassified_Erysipelotrichaceae、Anaerococcus均降低.在CM中Globicatella、Ruminococcus、Acetanaerobacterium和Erysipelothrix分别降低了99.90%、98.66%、99.8%和99.83%.胞外SS中主要以unclassified_Bacteria、Petrimonas和Tissierella, 且在厌氧消化结束后均降低.结果表明3种底物在经过高压蒸汽灭菌预处理后优势属的变化不明显, 因此底物中微生物的变化对后期的贡献有限.高压蒸汽灭菌处理底物对厌氧消化中iDNA和eDNA的菌群未造成较大的改变, 在不同底物中, PM和CM中的优势属较为相似, 以嗜温性和厌氧菌属为主.而在污泥厌氧消化结束后一些球菌属和厌氧发酵产氢细菌属均降低(98.66%~99.90%).同时厌氧消化能够消减eDNA中的优势门丰度.
 |
| 图 6 3种不同底物厌氧消化门水平微生物群落在细胞内外的丰度动态变化 Fig. 6 The abundance dynamic changes of three different substrate types in the phylum community in iDNA and eDNA |
| 表 4 3种环境样品中iDNA和eDNA物种多样性指数 Table 4 iDNA and eDNA of species diversity indexes in three environmental samples |
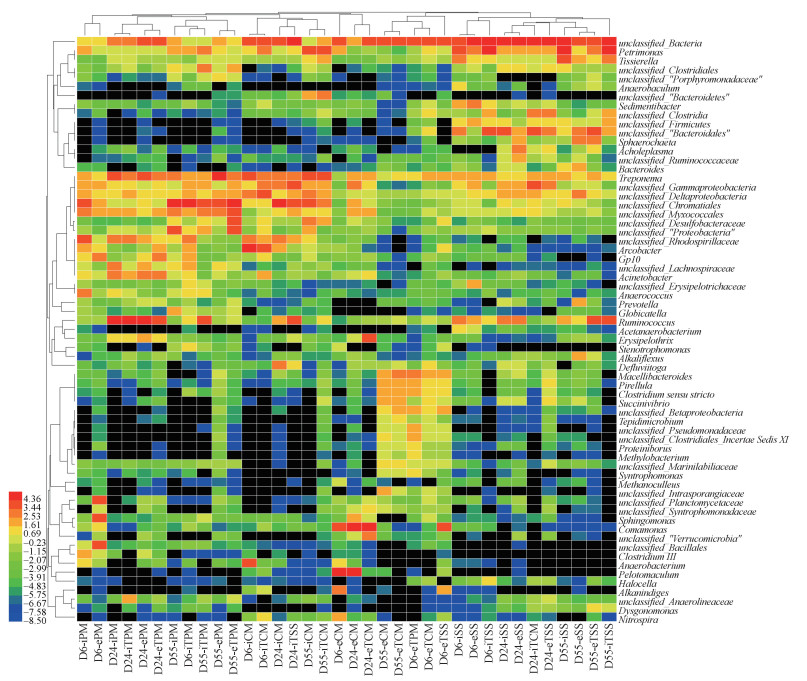 |
| 图 8 热图显示属水平上微生物群落的动态变化(log2)(每个样本仅收集前10个属) Fig. 8 Heatmap showing the dynamic changes of the microbial community(MC)at the genera level(Only the top 10 genera in each sample were collected) |
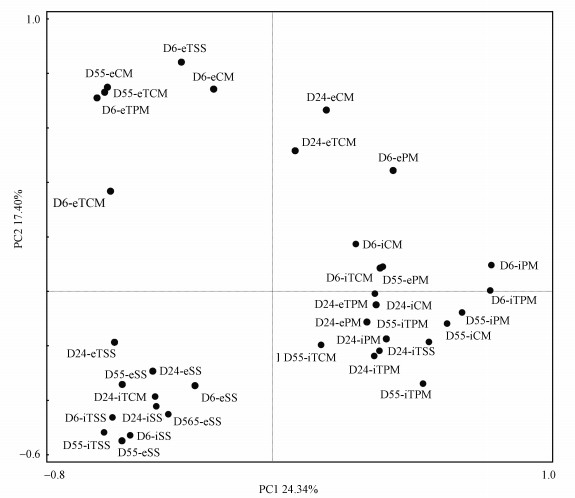 |
| 图 7 属水平微生物群落变化的主成分分析 Fig. 7 Principal component analysis (PCA) of the microbial community changes based on the genera data |
微生物群落可能直接或间接地影响ARGs丰度的变化, Mantel test和Procrustes分析表明微生物群落对于ARGs变化的重要性.基于Bray-Curtis和Mantel test得出A(胞内DNA中微生物群落和ARGs的相关性)和B(胞外DNA中微生物群落和ARGs的相关性)的p值分别为0.0004和0.0008, R值分别为0.362和0.3654, 表明微生物群落与ARGs之间呈弱相关.且在Procrustes分析中3种不同底物的胞内外DNA中微生物群落对ARGs的影响的结果, 进一步证明了两者之间的联系, 其A和B的解释变量分别为46.2%和17.7%, 胞内微生物群落与胞内ARGs之间呈显著相关性, 而胞外微生物群落与胞外ARGs相关性则不显著.造成胞内外微生物群落与其ARGs之间相关性差异的原因可能受多种因素的影响, 其一, 微生物群落是影响ARGs的一个重要因素; 其二, DNA释放到胞外后可能一部分经过厌氧消化降解或者DNA片段断裂未能被检测出等, 但其中具体的影响因素, 还需要进一步的研究.结果表明, 厌氧消化过程中大部分ARGs主要存在于活的微生物内, 因此, 胞内大多数微生物群落结构随着优势微生物的丰度增加或降低而改变, 微生物中的ARGs也随之降低, 这与之前的研究一致.因胞外ARGs与胞外微生物相关性弱, 胞外ARGs的来源尚不明晰, 需要更深一步的研究.
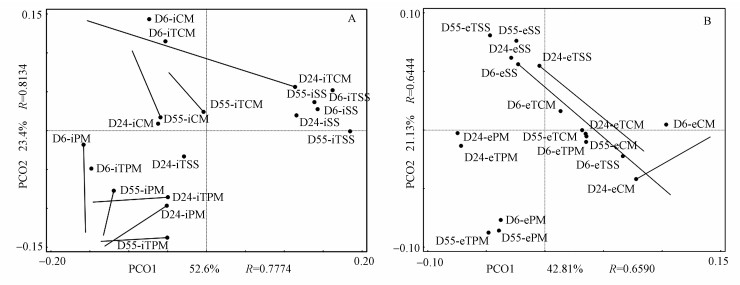 |
| 图 9 普鲁克分析3种不同底物和高压蒸汽灭菌后底物厌氧消化过程中微生物群落与ARGs变化之间的相关性(A和B分别表示胞内DNA中微生物群落和ARGs和胞外DNA中微生物群落和ARGs的相关性) Fig. 9 Prucker analyzed the correlation between the microbial community and the changes in ARGs during the anaerobic digestion of three different substrates and substrates after autoclaving(A and B respectively indicate the correlation between microbial community and ARGs in intracellular DNA and the correlation between microbial community and ARGs in extracellular DNA) |
1) 底物类型对厌氧消化过程中胞内外ARGs具有显著的影响.ermB在PM的胞内外均是消减的, 在CM的胞内呈消减的趋势, 而在胞外则是富集; ermB在SS厌氧消化结束时则是大量富集的.blaCTX-M和blaTEM在PM、CM和SS的胞外ARGs均是富集, 且在SS中富集的更明显.
2) 高压蒸汽灭菌处理底物, 未能显著增加猪粪和鸡粪厌氧消化的总累积甲烷产气量, 但增加了厌氧消化过程中的甲烷产气速率.结合预处理和未处理样品中DNA浓度和ARGs丰度的变化, 高压蒸汽灭菌处理底物没有显著改变DNA的浓度以及ARGs的归趋, 因此底物中的微生物对厌氧消化过程中的ARGs影响有限.
3) 3种底物中iDNA浓度均大于eDNA浓度, qPCR分析得知, PM和CM中eARGs占总ARGs的比例分别为0.29%和0.04%, SS中eARGs占总ARGs的5.47%, SS中eARGs相对较高, 因此剩余污泥中的eARGs的归趋成为目前研究的热点.
4) Mantel test和Procrustes分析表明胞内微生物群落与iARGs呈显著相关性, 而胞外的微生物群落与eARGs相关性则不显著.这可能是因为eARGs受多种因素的影响, 例如胞外微生物群落和一些化学影响等.具体的机制需要进一步的研究.
Arango-Argoty G, Garner E, Pruden A, et al. 2018. DeepARG: a deep learning approach for predicting antibiotic resistance genes from metagenomic data[J]. Microbiome, 6(1): 23. DOI:10.1186/s40168-018-0401-z |
Aminov R I, Chee-Sanford J C, Garrigues N, et al. 2002. Development, validation, and application of PCR primers for detection of tetracycline efflux genes of gram-negative bacteria[J]. Applied Environmental Microbiology, 68: 1786-1793. DOI:10.1128/AEM.68.4.1786-1793.2002 |
Aminov R I, Garrigues-Jeanjean N, Mackie R I. 2001. Molecular ecology of tetracycline resistance: development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Applied Environmental Microbiology, 67: 22-32. DOI:10.1128/AEM.67.1.22-32.2001 |
Birkett C I, Ludlam H A, Woodford N, et al. 2007. Real-time TaqMan PCR for rapid detection and typing of genes encoding CTX-M extended-spectrum beta-lactamases[J]. Journal of Medical Microbiology, 56: 52-55. DOI:10.1099/jmm.0.46909-0 |
Bolan N, Adriano D, Mahimairaja S. 2004. Distribution and bioavailability of trace elements in livestock and poultry manure by-products[J]. Critical Reviews in Environmental Science and Technology, 34: 291-338. DOI:10.1080/10643380490434128 |
Chen J, Yu Z, Michel F C J R, et al. 2007. Development and application of real-time PCR assays for quantification of erm genes conferring resistance to macrolides-lincosamides-streptogramin B in livestock manure and manure management systems[J]. Applied Environmental Microbiology, 73: 4407-4416. DOI:10.1128/AEM.02799-06 |
Colom K, Pã Rez J, Alonso R, et al. 2003. Simple and reliable multiplex PCR assay for detection of blaTEM, blaSHV and blaOXA-1 genes in Enterobacteriaceae[J]. FEMS Microbiology Letters, 223: 147-151. DOI:10.1016/S0378-1097(03)00306-9 |
Deng W, Wang Y, Liu Z, et al. 2014. HemI: a toolkit for illustrating heatmaps[J]. PLoS One, 9: e111988. DOI:10.1371/journal.pone.0111988 |
Dong P, Wang H, Fang T, et al. 2019. Assessment of extracellular antibiotic resistance genes (eARGs) in typical environmental samples and the transforming ability of eARG[J]. Environmental International, 125: 90-96. DOI:10.1016/j.envint.2019.01.050 |
Huang H, Chen Y, Zheng X, et al. 2016. Distribution of tetracycline resistance genes in anaerobic treatment of waste sludge: The role of pH in regulating tetracycline resistant bacteria and horizontal gene transfer[J]. Bioresource Technology, 218: 1284-1289. DOI:10.1016/j.biortech.2016.07.097 |
Lu N, Zilles J L, Nguyen T H. 2010. Adsorption of extracellular chromosomal DNA and its effects on natural transformation of Azotobacter vinelandii[J]. Applied Environmental Microbiology, 76: 4179-4184. DOI:10.1128/AEM.00193-10 |
刘新媛, 胡文甲, 欧阳帆, 等. 2020. 消化污泥序批式驯化中产气性能和微生物演替[J]. 环境工程: 1-8. |
孟晓山, 张玉秀, 隋倩雯, 等. 2018. 氨氮浓度对猪粪厌氧消化及产甲烷菌群结构的影响[J]. 环境工程学报, 12(8): 212-222. |
Ma Y, Wilson C A, Novak J T, et al. 2011. Effect of various sludge digestion conditions on sulfonamide, macrolide, and tetracycline resistance genes and class I integrons[J]. Environmental Science and Technology, 45: 7855-7861. DOI:10.1021/es200827t |
Mao D, Luo Y, Mathieu J, et al. 2014. Persistence of extracellular DNA in river sediment facilitates antibiotic resistance gene propagation[J]. Environmental Science and Technology, 48: 71-78. DOI:10.1021/es404280v |
Munir M, Xagoraraki I. 2011. Levels of antibiotic resistance genes in manure, biosolids, and fertilized soil[J]. J Environmental Quality, 40: 248-255. DOI:10.2134/jeq2010.0209 |
Ng L K, Martin I, Alfa M, et al. 2001. Multiplex PCR for the detection of tetracycline resistant genes[J]. Mol Cell Probes, 15: 209-215. DOI:10.1006/mcpr.2001.0363 |
Pei R, Kim S C, Carlson K H, et al. 2006. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research, 40: 2427-2435. DOI:10.1016/j.watres.2006.04.017 |
Pietramellara G, Ascher J, Borgogni F, et al. 2008. Extracellular DNA in soil and sediment: Fate and ecological relevance[J]. Biology and Fertility of Soils, 45: 219-235. |
Sampson T R, Debelius J W, Thron T, et al. 2016. Gut microbiota regulate motor deficits and neuroinflammation in a model of parkinson's disease[J]. Cell, 167: 1469-1480. DOI:10.1016/j.cell.2016.11.018 |
Stokes H W, Nesbo C L, Holley M, et al. 2006. Class 1 integrons potentially predating the association with tn402-like transposition genes are present in a sediment microbial community[J]. Journal of Bacteriology, 188: 5722-5730. DOI:10.1128/JB.01950-05 |
Sutcliffe J, Grebe T, Tait-Kamradt A, et al. 1996. Detection of erythromycin-resistant determinants by PCR[J]. Antimicrobial Agents and Chemotherapy, 40: 2562-2566. DOI:10.1128/AAC.40.11.2562 |
Suzuki M T, Taylor L T, Delong E F. 2000. Quantitative analysis of small-subunit rRNA genes in mixed microbial populations via 5'-nuclease assays[J]. Applied and Environmental Microbiology, 66: 4605-4614. DOI:10.1128/AEM.66.11.4605-4614.2000 |
Walters W, Hyde E R, Berg-Lyons D, et al. 2016. Improved bacterial 16S rRNA Gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys[J]. Msystems, 1(1). |
Wang Q, Garrity G M, Tiedje J M, et al. 2007. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied Environmental Microbiology, 73: 5261-5267. DOI:10.1128/AEM.00062-07 |
Wu X, Tian Z, Lv Z, et al. 2020. Effects of copper salts on performance, antibiotic resistance genes, and microbial community during thermophilic anaerobic digestion of swine manure[J]. Bioresource Technology, 300: 122728. DOI:10.1016/j.biortech.2019.122728 |
Xu R, Yang Z H, Zheng Y, et al. 2019. Metagenomic analysis reveals the effects of long-term antibiotic pressure on sludge anaerobic digestion and antimicrobial resistance risk[J]. Bioresource Technology, 282: 179-188. DOI:10.1016/j.biortech.2019.02.120 |
Yin F, Dong H, Zhang W, et al. 2018. Antibiotic degradation and microbial community structures during acidification and methanogenesis of swine manure containing chlortetracycline or oxytetracycline[J]. Bioresource Technology, 250: 247-255. DOI:10.1016/j.biortech.2017.11.015 |
于雯超, 郑利兵, 魏源送, 等. 2020. 磁混凝对市政污水中抗生素抗性基因和重金属抗性基因的削减效能[J]. 环境科学, 41(2): 815-822. |
Zhang J, Lu T, Shen P, et al. 2019. The role of substrate types and substrate microbial community on the fate of antibiotic resistance genes during anaerobic digestion[J]. Chemosphere, 229: 461-470. DOI:10.1016/j.chemosphere.2019.05.036 |
Zhang J, Wang Z, Wang Y, et al. 2017. Effects of graphene oxide on the performance, microbial community dynamics and antibiotic resistance genes reduction during anaerobic digestion of swine manure[J]. Bioresource Technology, 245: 850-859. DOI:10.1016/j.biortech.2017.08.217 |
Zhang J, Yang M, Zhong H, et al. 2018a. Deciphering the factors influencing the discrepant fate of antibiotic resistance genes in sludge and water phases during municipal wastewater treatment[J]. Bioresource Technology, 265: 310-319. DOI:10.1016/j.biortech.2018.06.021 |
Zhang M, Liu Y S, Zhao J L, et al. 2018b. Occurrence, fate and mass loadings of antibiotics in two swine wastewater treatment systems[J]. Science of the Total Environmental, 639: 1421-1431. DOI:10.1016/j.scitotenv.2018.05.230 |
Zhang Q Q, Ying G G, Pan C G, et al. 2015a. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science and Technology, 49: 6772-6782. DOI:10.1021/acs.est.5b00729 |
Zhang T, Yang Y, Pruden A. 2015b. Effect of temperature on removal of antibiotic resistance genes by anaerobic digestion of activated sludge revealed by metagenomic approach[J]. Applied Microbiology Biotechnology, 99: 7771-7779. DOI:10.1007/s00253-015-6688-9 |
Zhang Y, Niu Z, Zhang Y, et al. 2018c. Occurrence of intracellular and extracellular antibiotic resistance genes in coastal areas of Bohai Bay (China) and the factors affecting them[J]. Environmental Pollution, 236: 126-136. DOI:10.1016/j.envpol.2018.01.033 |
Zhang Y, Snow D D, Parker D, et al. 2013. Intracellular and extracellular antimicrobial resistance genes in the sludge of livestock waste management structures[J]. Environmental Science and Technology, 47: 10206-10213. DOI:10.1021/es401964s |
 2021, Vol. 41
2021, Vol. 41


