2. 河南大学黄河中下游数字地理技术教育部重点实验室, 开封 475004;
3. 河南大学环境与规划国家级实验教学示范中心, 开封 475004;
4. 河南大学民生学院, 开封 475004
2. Key Laboratory of Geospatial Technology for the Middle and Lower Yellow River Regions(Henan University), Ministry of Education, Kaifeng 475004;
3. National Demonstration Center for Environment and Planning, Henan University, Kaifeng 475004;
4. Minsheng College, Henan University, Kaifeng 475004
土壤动物是土壤系统中最活跃的组成部分, 也是一个重要的生物多样性储存库, 对土壤功能维持和生态资源的可持续利用起着重要作用(Bardgett et al., 2014; 邵元虎等, 2015).蚯蚓是最重要的土壤动物类群之一, 其活动能够影响土壤结构和功能, 并参与土壤有机质分解和营养元素的地球化学循环(张卫信等, 2007; Blouin et al., 2013).事实上, 蚯蚓的很多生态功能都是由其肠道微生物来协助完成的.蚯蚓肠道不仅影响土壤碳、氮元素的生物地球化学循环(Drake et al., 2007; Zhang et al., 2013), 其肠道排出的蚓粪还有助于土壤中除草剂的降解(Liu et al., 2011).此外, 蚯蚓肠道微生物可以为宿主提供必要的营养资源, 研究表明, 肠道微生物组是宿主最大的共生体系, 并能调节宿主的生理免疫和代谢, 从而影响宿主对环境的适应能力(Thakuria et al., 2010; Zhu et al., 2018; Wang et al., 2019).如Liebeke等(2015)发现蚯蚓肠道微生物能够分泌“破防御素”来降低多酚类化合物的毒性.鉴于蚯蚓肠道微生物组在减缓外界污染物对蚯蚓毒害等方面发挥重要作用, 因此, 探讨蚯蚓肠道微生物对污染物的响应, 将有助于全面了解蚯蚓肠道微生物组的组成和功能.蚯蚓肠道是一个厌氧和多糖的微环境, 并位于土壤地表以下, 通常容易被人们所忽视.目前关于肠道微生物菌群的研究多集中于老鼠(Blacher et al., 2019; Lahiri et al., 2019)、白蚁(Bourguignon et al., 2018)和蜜蜂(Raymann et al., 2017)等常见动物, 但关于土壤动物, 特别是“生态系统工程师”蚯蚓肠道微生物的报道还较少.
自20世纪以来, 抗生素的问世为医疗进步和人类健康做出了巨大贡献, 并随后广泛应用于畜牧养殖等产业中.然而近几十年来, 随着现代化集约产业的快速发展, 农业生产中越来越多的抗生素被大量滥用, 并经动物或污染水体排泄, 最终导致土壤环境中出现大量抗生素的残留和富集(王敏等, 2010; Zhu et al., 2013).这些抗生素的长期存在不仅会导致大量耐药菌的产生, 影响抗生素本身的应用效果, 而且这些耐药菌携带了大量抗生素抗性基因, 可通过食物链进行水平转移, 从而对全球生态系统稳定造成巨大损害(苏建强等, 2013).目前, 抗生素滥用导致的抗生素抗性基因在全球范围内的传播已严重威胁到人类的健康和生命安全(Zhu et al., 2017; Chen et al., 2019).此外, 土壤动物是土壤健康和毒性的“晴雨表”, 并经常作为土壤污染物影响的指示性生物(罗洁文等, 2016).当大量抗生素进入农田土壤系统, 必然会对土壤中的非靶向动物产生影响(Ding et al., 2019).已有研究证实, 抗生素暴露能引起动物生长和繁殖等生理代谢的紊乱(Gorokhova et al., 2015; Zhu et al., 2018).但有关抗生素对土壤动物肠道微生物的影响却极少引起人们的关注.蚯蚓肠道微生物群落是土壤生态系统功能和生物多样性的重要组成部分.因此, 研究土壤中抗生素污染对蚯蚓肠道微生物群落结构的变化对于全面理解抗生素的生物危害过程具有重要的意义.
鉴于此, 本研究以常见蚯蚓—赤子爱胜蚓(Eisenia fetida)为研究对象, 通过高通量测序技术研究土壤中常见抗生素—磺胺甲恶唑对蚯蚓肠道内微生物群落结构组成及多样性的影响.这将有助于提升人们对土壤动物肠道内微生物群落的认识, 也有助于了解抗生素对土壤生态系统的影响.
2 材料与方法(Materials and methods) 2.1 试验材料试验土壤采自本地无污染农田, 将采集的土样自然风干后, 捡出枯枝落叶和砂石等杂物, 用玛瑙研钵研磨后分别过10和100目筛, 备用.供试蚯蚓为赤子爱胜蚓(Eisenia fetida), 购自本地专业养殖场.实验前, 选择相似长度和体重, 且具有明显环带的性成熟蚯蚓, 放于采集土壤中进行驯化.本次试验选用磺胺甲恶唑作为常见的抗生素.其他试剂若无特殊说明, 均为分析级纯度, 购自国药控股化学试剂有限公司.实验所用水经Milli-Q系统净化(18.2 MΩ cm, 美国).
2.2 试验设计称取1000 g过10目筛的风干土样添加到聚乙烯塑料保鲜盒(长×宽×高, 20 cm×18 cm×12 cm)中, 将适量的磺胺甲恶唑充分融入溶液中, 然后将溶液充分淹没土壤并混匀, 放在实验室避光低温处老化14 d, 从而得到一系列磺胺甲恶唑浓度(分别为空白对照、0.1、1和10 mg·kg-1)的抗生素污染土壤样品.同时每个保鲜盒中放入10条长度相似的蚯蚓, 每条蚯蚓的质量在0.2~0.3 g左右.每个浓度梯度实验设置3个平行.最后将所有保鲜盒放入培养箱内, 培养条件设置为:每天持续光照/黑暗循环各12 h, 相对湿度为75%, 温度控制在20~22 ℃.实验期间, 定期向土壤中加入超纯水, 保证土壤含水率保持在30%左右.
2.3 试验分析方法 2.3.1 样品DNA提取试验28 d后, 采集土壤和蚯蚓肠道样品.用无菌镊子将蚯蚓从土壤样品取出, 并将其放于装满无水乙醇的离心管中杀死封存, 在无菌超纯水中漂洗5次, 从而去除蚯蚓表层微生物影响, 然后在无菌操作台上使用无菌镊子和无菌剪刀将蚯蚓解剖, 取出其肠道内含物.称取大约0.5 g土样和肠道样品, 使用Fast DNA® Spin Kit (MP Biomedical, 美国)规格的土壤提取试剂盒提取土样和肠道样品总DNA.最后将DNA样品储存于-20 ℃冰箱, 备用.
2.3.2 16S rRNA基因扩增和生物信息分析以DNA总长为模板, 扩增细菌16S rRNA基因的可变V4~V5区.该区扩增引物包含上游通用引物515F(5′-GTGCCAGCMGCCGCGG-3′)及下游通用引物907R (5′-CCGTCAATTCMTTTRAGTTT-3′), 其中, 下游引物含有6个核苷酸片段的barcode, 用来区别各个样品DNA信息.本研究采用50 μL的PCR扩增反应体系:TaKaRa Ex Taq酶25 μL, 无菌水22 μL, 10 mmol·L-1上、下游引物各1 μL, DNA样品1 μL.同时设置该扩增体系的温度时间条件:94 ℃预变性5 min;然后30个循环扩增, 每个循环含94 ℃变性30 s, 58 ℃退火30 s, 72 ℃延伸30 s;最终72 ℃延伸5 min(Wang et al., 2019).PCR产物经纯化回收后, 送测序公司(诺禾致源, 中国)进行高通量测序.之后用QIIME 1服务器对测序所得原始序列进行下游分析(Caporaso et al., 2010).用Uclust聚类法(97%的相似度水平)将高质量序列进一步聚类为OTU单元(Edgar, 2010).通过不同测序深度下细菌群落多样性的比较, 从而得到Chao1、Shannon和PD Whole Tree指数等样品的α多样性数据.通过计算相同测序深度下的数据得到微生物的β多样性等数据, 主要由基于Bray-Curtis距离的主坐标轴分析方法(PCoA)来表示.
2.3.3 样品理化分析土壤pH值采用pH仪进行测定.土壤TC和TN使用C/N分析仪(Vario MAX C/N, 德国), 采用燃烧法进行测定.土壤重金属消解采用微波消解仪(CEM Microwave Technology Ltd., 英国), 使用硝酸(Merck Millipore, 65%, 德国)-氢氟酸(ThermoFisher Scientific, 49%, 美国)体系消解土壤.土壤中Cd、Cr、Cu、Ni、Pb和Zn含量由电感耦合等离子体质谱(ICP-MS, Agilent 7500 ce, Agilent Technologies, 美国)测定.每批消解样品都有3个空白对照, 以及两个样品技术平行, 样品间平行重复的相对偏差均在8%以内.此外, 本研究使用标准物质黄棕壤GBW07403进行校准验证, 样品回收率均在92.1%~106.7%之间, 符合标准.
2.4 数据处理试验数据分析如均值、百分比、变异系数、标准差、标准误等均采用Excel 2010(Microsoft, 美国).各个样品间显著性分析采用软件SPSS V18.0完成(IBM, 美国), 其中, 单因素方差分析(one-way ANOVA)用来比较数据在0.05水平上的显著性, 显著性检验的方法采用t检验和Tukey HSD检验.同时, 考虑到数据的分布特征和来源, 在进行ANOVA分析前要对数据进行正态分布检验.此外, 本文中相关性分析也使用SPSS软件来完成, 采用Pearson相关系数.微生物数据分析主要基于R (version 3.4.3)软件, 如PCoA分析和Adonis显著性检验.其他数据绘图均由Origin Pro8.5 (OriginLab, 美国)完成.
3 结果(Results) 3.1 磺胺甲恶唑对蚯蚓生长存活率的影响本次试验土壤的理化性质如表 1所示.由表 1可知, 试验土壤均呈中性, pH均值在6.8左右, 且在试验处理间变化不大.土壤总氮和总碳的含量均值分别为2.6和28.3 g·kg-1, 土壤中各个重金属的含量也都在安全范围内.同时, 随着土壤中抗生素浓度的增加, 蚯蚓的存活率在所有处理间均未发生显著改变(ANOVA, p>0.05), 其中, 死亡数最多的是SMZ-1处理组, 但也仅死亡3条左右.此外, 蚯蚓的体重在0.42~0.54 g之间, 且在4个处理间均未发生显著变化(p>0.05).
| 表 1 土壤理化性质和蚯蚓毒理结果 Table 1 Descriptive statistics of soil physicochemical properties and earthworm toxicology |
通过对土壤和蚯蚓肠道样品进行高通量测序, 共得到950276条高质量序列, 其中, 每个样品平均序列数为39595, 总体序列数值为29913~59083条.通过97%的相似度归类, 共得到18093个OTUs, 其中, 土壤和蚯蚓肠道样品之间大约有33.3%的共享OTUs.同时, 还发现土壤样品和蚯蚓肠道菌群组成之间存在显著差异(Adonis test, p=0.001).基于Bray-Curtis距离算法的主坐标分析表明, 蚯蚓肠道细菌群落样品聚集在右侧, 并沿着第一坐标轴(PC1)与聚集在左侧的土壤细菌群落显著分离, 该坐标轴解释了47.9%的变异量(R=0.99, p < 0.01, 图 1a).此外, 通过计算样品间的细菌α多样性, 还发现土壤微生物多样性显著高于蚯蚓肠道内微生物多样性.如Chao1和Shannon指数均表明蚯蚓肠道细菌多样性显著低于土壤细菌多样性(p < 0.001, 图 1b).
 |
| 图 1 土壤和蚯蚓肠道微生物多样性分析(a.主坐标分析, b.箱线图) Fig. 1 Analysis of microbial diversity in soil and earthworm gut(a.Principal coordinates (PCoA) analysis, b. Boxplot) |
从细菌群落组成成分来看, 放线菌门(Actinobacteria, 占总序列的39.2%)、变形菌门(Proteobacteria, 26.9%)和厚壁菌门(Firmicutes, 18.7%)是肠道内最主要的3类细菌群落, 然而土壤中的优势菌群主要是变形菌门(Proteobacteria, 48.7%)、厚壁菌门(Firmicutes, 14.3%)、拟杆菌门(Bacteroidetes, 10.1%)和放线菌门(Actinobacteria, 9.1%)(图 2a).其中, 肠道内放线菌门的相对丰度是土壤中该菌相对丰度的4.3倍(t-test, p < 0.01), 同时, 肠道内厚壁菌门的相对丰度也显著高于土壤中该菌的相对丰度(p=0.007), 但土壤中变形菌门的相对丰度显著高于肠道内的相对丰度(p=0.001), 且拟杆菌门的相对丰度比肠道内该菌的相对丰度高13.1倍(p < 0.01).此外, 在科水平上土壤和肠道内存在显著差异的主要细菌群落组成如图 2b所示.如蚯蚓肠道内芽胞杆菌科(Bacillaceae, 11.8%)的相对丰度最高, 显著高于土壤中该菌的相对丰度(8.6%)(p < 0.01), 肠道内微杆菌科(Microbacteriaceae)和分支杆菌科(Mycobacteriaceae)的相对丰度分别为9.7%和5.4%, 且均显著高于土壤中的相对丰度(分别为0.7%和0.3%)(p < 0.05).土壤中相对丰度最高的细菌为气单胞菌科(Aeromonadaceae, 14.0%), 其丰度是肠道内该菌的4.3倍(p < 0.01), 黄杆菌科(Flavobacteriaceae)的相对丰度为6.5%, 明显高于肠道内的相对丰度(0.1%)(p < 0.01).
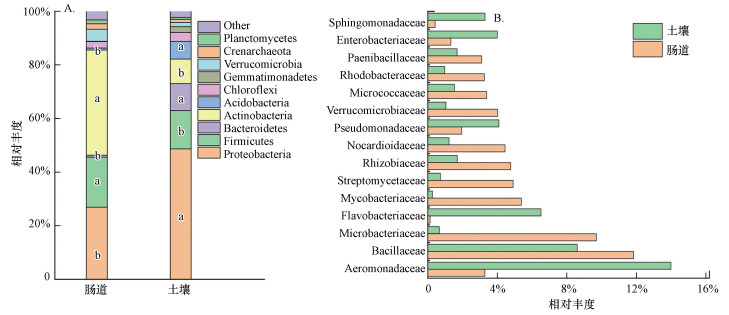 |
| 图 2 土壤和蚯蚓肠道微生物群落组成之间的差异(A.门水平, 相对丰度小于1%的归为“Other”, 不同小写字母表示细菌的相对丰度在肠道和土壤间存在显著差异, B.科水平, 仅给出相对丰度排名前15且在肠道和土壤之间均存在显著差异的细菌) Fig. 2 Differences of microbial community composition in soil and earthworm gut (A.phylum level, B.family level) |
土壤中磺胺甲恶唑的添加能改变蚯蚓肠道细菌群落的多样性, 如随着土壤中磺胺甲恶唑浓度的不断增加, 蚯蚓肠道细菌的PD Whole Tree指数不断降低(图 3), 并在抗生素浓度为10 mg·kg-1处理组中达到最低值(165.4).同时, 当土壤中磺胺甲恶唑浓度达到1 mg·kg-1左右时, 与对照相比, 该抗生素能够显著降低蚯蚓肠道细菌的Chao1指数(p < 0.05).此外, 通过对肠道内微生物群落结构的PCoA分析, 发现随着土壤中磺胺甲恶唑的添加, 土壤和蚯蚓肠道细菌群落均沿着第二坐标轴逐步分开, 并解释了其中6.9%的变异量(图 1a).本研究通过Adonis test统计分析发现, 磺胺甲恶唑的添加能显著改变蚯蚓肠道微生物群落结构(p=0.005).
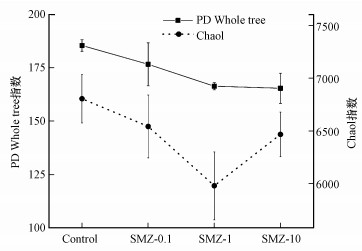 |
| 图 3 不同处理中蚯蚓肠道微生物多样性指数 Fig. 3 Diversity indexes of earthworm gut microbiome in different treatments |
土壤中磺胺甲恶唑的添加还能引起蚯蚓肠道内细菌组成的改变(图 4和图 5).图 4结果显示, 蚯蚓肠道内放线菌门、变形菌门和厚壁菌门等优势菌群受抗生素影响较小, 其丰度仅发生微弱变化.此外, 与空白对照相比, 抗生素的添加能够显著降低蚯蚓肠道内分支杆菌属(Mycobacterium)和Luteolibacter的相对丰度(p < 0.05), 特别是在磺胺甲恶唑浓度为10 mg·kg-1处理组中分支杆菌属的相对丰度(2.1%)仅为对照组的0.2倍(图 5).处理组中低浓度磺胺甲恶唑(0.1 mg·kg-1)也能使肠道内副球菌属(Paracoccus)的相对丰度显著降低, 降低幅度高达65.5%(p < 0.05).相比之下, 对照处理组蚯蚓肠道内拜纳蒙纳斯属(Balneimonas)的相对丰度仅为0.96%, 而高浓度抗生素的添加能显著增加其肠道内的相对丰度, 增加到2.4%(p < 0.05).在磺胺甲恶唑浓度为10 mg·kg-1处理组中蚯蚓肠道内f(Verrucomicrobiaceae)的相对丰度是其对照处理组中的3.2倍(p < 0.05).
 |
| 图 4 磺胺甲恶唑对蚯蚓肠道门水平细菌群落组成的影响 Fig. 4 Effects of SMZ-exposure on earthworm gut bacterial composition at the phylum level |
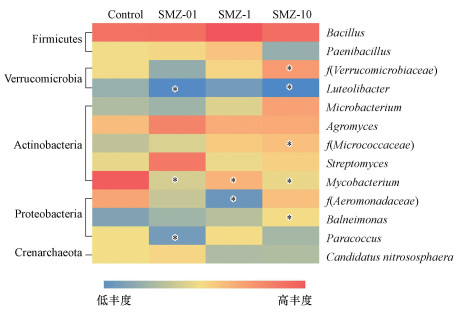 |
| 图 5 磺胺甲恶唑对蚯蚓肠道微生物群落组成的影响(热图仅显示属水平上相对丰度排名前13的细菌变化, 星号表示抗生素处理组与对照处理组之间在0.05水平上存在显著差异) Fig. 5 Effects of SMZ-exposure on earthworm gut microbial community composition |
本次试验结果显示, 蚯蚓肠道微生物群落与周围土壤环境微生物群落显著不同(p < 0.01), 这可能是由于蚯蚓肠道和土壤所处生境不同.众所周知, 蚯蚓肠道是一个中性pH且含高糖分的独特缺氧环境, 而周围土壤处于好氧条件, 且由复杂的矿物质组成(Drake et al., 2007).比如, 酸杆菌门(Acidobacteria)具有嗜酸性, 且大量存在于贫瘠环境或有机营养不足的环境中, 而从土壤到肠道环境酸杆菌门的相对丰度显著减少, 可能反映了相对于土壤的有机质含量, 蚯蚓肠道内有较高含量的有机物质(Drake et al., 2007; Pass et al., 2015), 这一发现也在一定程度上反映了肠道和土壤环境之间的区别.肠道内放线菌门和壁厚菌门的大量存在也证明了蚯蚓肠道内的厌氧环境可以增加兼性或者专性厌氧微生物的丰度(Wang et al., 2019).本研究还发现蚯蚓肠道内链霉菌科(Streptomycetaceae)和芽胞杆菌科的丰度显著增加, 这些细菌涉及到简单或复杂有机物的降解或磷酸的矿化等过程(Wan et al., 2004; Singh et al., 2016).如肠道内链霉菌科丰度的增加可能被认为是其利用葡萄糖异构酶参与到肠道内聚合底物(如纤维素、木聚糖和木糖等)的同化(Killham et al., 2007).此外, 蚯蚓肠道微生物多样性显著低于周围土壤, 这与之前的研究发现一致.赤子爱胜蚓生活在土壤表层(0~10 cm), 主要以土壤中腐殖有机物为食物来源, 蚯蚓肠道会对瞬时土壤选择地吸收同化, 从而降低土壤微生物多样性.同时, 蚯蚓肠道和土壤间还存在大量共享的OTUs, 这表明周围的土壤也是蚯蚓肠道内部微生物主要的物质来源, 土壤微生物群落在肠道微生物的最初形成中发挥着重要作用(Drake et al., 2007).总的来说, 蚯蚓肠道微生物群落组成可通过筛选土壤细菌, 并由独特的肠道微环境共同塑造而成.反过来, 肠道微生物群落通过分解矿化营养元素来协助宿主蚯蚓健康生长.
本次试验中磺胺甲恶唑的添加对土壤中蚯蚓无明显毒性作用, 这与之前研究结果类似.Pino等(2015)研究发现, 当土壤中磺胺甲恶唑的浓度高达4000 mg·kg-1时, 赤子爱胜蚓的生存未受到较大影响.磺胺甲恶唑属于磺胺类抗生素, 该抗生素在土壤中能够迅速降解, 半衰期仅为9.0~58.7 d左右(Lin et al., 2011).同时, 磺胺甲恶唑的降解产物也可能对蚯蚓无毒害作用.当然, 抗生素的应用效果还受土壤类型和微生物活动等因子的共同影响.
本研究结果也表明, 随着污染物磺胺甲恶唑浓度的不断增加, 蚯蚓肠道微生物群落多样性显著降低.首先, 磺胺甲恶唑属于磺胺类抗生素, 它是一类广谱性抗生素, 可以通过抑制革兰氏阳性菌和许多革兰氏阴性菌的增殖来对抗大多数细菌的增长, 并广泛应用于保护动物和人类的健康.已有研究证实, 磺胺甲恶唑能够对土壤微生物群落施加选择性压力, 敏感菌的生长会受到抑制甚至死亡, 少部分耐受菌可以存活并大量增加积累, 从而降低土壤细菌多样性(Sarmah et al., 2006; 李彦文等, 2009).而蚯蚓主要以富含有机质的土壤为食, 进入肠道的土壤微生物群落组成较为单一, 势必会影响肠道微生物多样性的来源.其次, 磺胺甲恶唑抗生素本身吸附在土壤颗粒表面, 会经蚯蚓摄食活动进入肠道内, 该抗生素可直接作用于肠道内细菌群落的结构, 从而降低肠道内自身微生物群落多样性.以上两点可能是该抗生素引起蚯蚓肠道细菌群落多样性降低的主要原因, 类似的结果也在抗生素暴露后的跳虫(Zhu et al., 2019)和老鼠(Yin et al., 2015)肠道内观察到.此外, 磺胺甲恶唑的添加还能改变蚯蚓肠道内的细菌组成成分.拟杆菌门广泛存在于部分动物肠道内, 且具有一定的降解复杂环境有机化合物的能力, 如复杂的多糖、蛋白质等生物聚合物, 以及宿主来源的碳水化合物(Thomas et al., 2011; Naas et al., 2014).暴露于磺胺甲恶唑的蚯蚓肠道内拟杆菌门丰度的增加表明, 该菌可能参与了降解磺胺甲恶唑或进一步降解其代谢产物等过程.尽管抗生素暴露能改变一些细菌的相对丰度, 但蚯蚓肠道内优势核心细菌的相对丰度变化不大, 这可能与肠道独特的环境保护某些细菌免受伤害有关, 也与肠道内微生物菌群不断形成对抗生素的耐药性有一定关联(Agamennone et al., 2015; Yin et al., 2015; Zhu et al., 2018).后续研究将不断探索其他类型的抗生素污染, 以及抗生素不同时间段暴露对蚯蚓肠道微生物群落变化演替的影响, 从而全面掌握新型污染物—抗生素对非靶向土壤动物微生物组的影响.
5 结论(Conclusions)1) 赤子爱胜蚓肠道和周围土壤的微生物群落之间存在显著差异, 且肠道细菌的多样性显著低于土壤.此外, 蚯蚓肠道在门水平上主要以放线菌门(39.2%)、变形菌门(26.9%)和厚壁菌门(18.7%)为主.这些数据对于深入认识土壤动物肠道微生物组等方面的知识具有重要意义.
2) 污染物磺胺甲恶唑的添加对蚯蚓生长、死亡等指标无显著影响, 但能显著降低肠道细菌群落多样性, 并能显著改变蚯蚓肠道细菌群落的组成结构, 这可为开展基于土壤动物微生物组的生态毒理学评价提供科学依据.
Agamennone V, Jakupović D, Weedon J T, et al. 2015. The microbiome of Folsomia candida:an assessment of bacterial diversity in a Wolbachiacontaining animal[J]. FEMS Microbiology Ecology, 91(11): 298-303. |
Bardgett R D, van der Putten W H. 2014. Belowground biodiversity and ecosystem functioning[J]. Nature, 515: 505-511. DOI:10.1038/nature13855 |
Blacher E, Bashiardes S, Shapiro H, et al. 2019. Potential roles of gut microbiome and metabolites in modulating ALS in mice[J]. Nature, 572(7770): 474-480. DOI:10.1038/s41586-019-1443-5 |
Blouin M, Hodson M E, Delgado E A, et al. 2013. A review of earthworm impact on soil function and ecosystem services[J]. European Journal of Soil Science, 64: 161-182. DOI:10.1111/ejss.12025 |
Bourguignon T, Lo N, Dietrich C, et al. 2018. Rampant host switching shaped the termite gut microbiome[J]. Current Biology, 28(4): 649-654. DOI:10.1016/j.cub.2018.01.035 |
Caporaso J G, Kuczynski J, Stombaugh J, et al. 2010. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 7: 335-336. DOI:10.1038/nmeth.f.303 |
Chen Q L, Cui H L, Su J Q, et al. 2019. Antibiotic resistomes in plant microbiomes[J]. Trends in Plant Science, 24(6): 530-541. DOI:10.1016/j.tplants.2019.02.010 |
Ding J, Zhu D, Hong B, et al. 2019. Long-term application of organic fertilization causes the accumulation of antibiotic resistome in earthworm gut microbiota[J]. Environment International, 124: 145-152. DOI:10.1016/j.envint.2019.01.017 |
Drake H L, Horn M A. 2007. As the worm turns:the earthworm gut as a transient habitat for soil microbial biomes[J]. Annual Review of Microbiology, 61: 169-189. DOI:10.1146/annurev.micro.61.080706.093139 |
Edgar R C. 2010. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
Gorokhova E, Rivetti C, Furuhagen S, et al. 2015. Bacteria-mediated effects of antibiotics on Daphnia nutrition[J]. Environmental Science & Technology, 49(9): 5779-5787. |
Killham K, Prosser J I. 2007. The Prokaryotes.Soil Microbiology, Ecology and Biochemistry(3rd edtion, vol.5)[M]. Pittsburgh: Academic Press, 19e143.
|
Lahiri S, Kim H, Garcia-Perez I, et al. 2019. The gut microbiota influences skeletal muscle mass and function in mice[J]. Science Translational Medicine, 11(502): eaan5662. DOI:10.1126/scitranslmed.aan5662 |
李彦文, 莫测辉, 赵娜, 等. 2009. 菜地土壤中磺胺类和四环素类抗生素污染特征研究[J]. 环境科学, 30(6): 1762-1766. |
Liebeke M, Strittmatter N, Fearn S, et al. 2015. Unique metabolites protect earthworms against plant polyphenols[J]. Nature Communications, 6: 7869. DOI:10.1038/ncomms8869 |
Lin K, Gan J. 2011. Sorption and degradation of wastewater-associated non-steroidal anti-inflammatory drugs and antibiotics in soils[J]. Chemosphere, 83: 240-246. DOI:10.1016/j.chemosphere.2010.12.083 |
Liu Y J, Zaprasis A, Liu S J, et al. 2011. The earthworm Aporrectodea caliginosa stimulates abundance and activity of phenoxalkanoic acid herbicide degraders[J]. ISME Journal, 5(3): 473-485. DOI:10.1038/ismej.2010.140 |
罗洁文, 黄玫英, 殷丹阳, 等. 2016. 蚯蚓在土壤污染风险评价中的应用研究进展[J]. 江苏农业科学, 44(8): 24-29. |
Naas A E, Mackenzie A K, Mravec J, et al. 2014. Do rumen bacteroidetes utilize an alternative mechanism for cellulose degradation?[J]. mBio, 5: e01401. |
Pass D A, Morgan A J, Read D S, et al. 2015. The effect of anthropogenic arsenic contamination on the earthworm microbiome[J]. Environmental Microbiology, 17: 1884-1896. DOI:10.1111/1462-2920.12712 |
Pino M R, Val J, Mainar A M, et al. 2015. Acute toxicological effects on the earthworm Eisenia fetida of 18 common pharmaceuticals in artificial soil[J]. Science of the Total Environment, 518-519: 225237. |
Raymann K, Shaffer Z, Moran N. 2017. A.Antibiotic exposure perturbs the gut microbiota and elevates mortality in honeybees[J]. PLoS Biology, 5(3): e2001861. |
Sarmah A K, Meyer M T, Boxall A B A. 2006. A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics (VAs) in the environment[J]. Chemosphere, 65: 725-759. DOI:10.1016/j.chemosphere.2006.03.026 |
邵元虎, 张卫信, 刘胜杰, 等. 2015. 土壤动物多样性及其生态功能[J]. 生态学报, 35(20): 6614-6625. |
Shin N R, Whon T W, Bae J W. 2015. Proteobacteria:microbial signature of dysbiosis in gut microbiota[J]. Trends in Biotechnology, 33: 496-503. DOI:10.1016/j.tibtech.2015.06.011 |
Singh A, Tiwari R, Sharma A, et al. 2016. Taxonomic and functional diversity of the culturable microbiomes of epigeic earthworms and their prospects in agriculture[J]. Journal of Basic Microbiology, 56: 1009-1020. DOI:10.1002/jobm.201500779 |
苏建强, 黄福义, 朱永官. 2013. 环境抗生素抗性基因研究进展[J]. 生物多样性, 21(4): 481-487. |
Thakuria D, Schmidt O, Finan D, et al. 2010. Gut wall bacteria of earthworms:a natural selection process[J]. ISME Journal, 4: 357-366. DOI:10.1038/ismej.2009.124 |
Thomas F, Hehemann J H, Rebuffet E, et al. 2011. Environmental and gut bacteroidetes:the food connection[J]. Frontiers in Microbiology, 2: 93. |
Wan J H C, Wong M H. 2004. Effects of earthworm activity and P-solubilizing bacteria on P availability in soil[J]. Journal of Plant Nutrition and Soil Science, 167: 209e213. |
Wang H T, Zhu D, Li G, et al. 2019. Effects of arsenic on gut microbiota and its biotransformation genes in earthworm Metaphire sieboldi[J]. Environmental Science & Technology, 53: 3841-3849. |
王敏, 唐景春. 2010. 土壤中的抗生素污染及其生态毒性研究进展[J]. 农业环境科学学报, 29(增刊): 261-266. |
Yin J, Zhang X X, Wu B, et al. 2015. Metagenomic insights into tetracycline effects on microbial community and antibiotic resistance of mouse gut[J]. Ecotoxicology, 24(10): 2125-2132. DOI:10.1007/s10646-015-1540-7 |
张卫信, 陈迪马, 赵灿灿. 2007. 蚯蚓在生态系统中的作用[J]. 生物多样性, 15(2): 142-153. |
Zhang W X, Hendrix P F, Dame L E, et al. 2013. Earthworms facilitate carbon sequestration through unequal amplification of carbon stabilization compared with mineralization[J]. Nature Communications, 4: 2576. DOI:10.1038/ncomms3576 |
Zhu D, An X L, Chen Q L, et al. 2018. Antibiotics disturb the microbiome and increase the incidence of resistance genes in the gut of a common soil collembolan[J]. Environmental Science & Technology, 52(5): 3081-3090. |
Zhu Y G, Johnson T A, Su J Q, et al. 2013. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 110: 3435-3440. DOI:10.1073/pnas.1222743110 |
Zhu Y G, Michael G, Pascal S, et al. 2017. Microbial mass movements[J]. Science, 357(6356): 1099-1100. DOI:10.1126/science.aao3007 |
 2020, Vol. 40
2020, Vol. 40


