2. 广东省环境污染控制与修复技术重点实验室, 广州 510275
2. Guangdong Provincial Key Laboratory of Environmental Pollution Control and Remediation Technology, Guangzhou 510275
已被广泛应用70多年的磺胺类抗生素作为最早系统投入使用的抗生素类药物之一, 目前仍被大量应用于水产养殖、畜牧业及作为人类药物治疗细菌和某些微生物引起的多种感染(Zhang et al., 2009; Dmitrirnko et al., 2014).近年来, 随着在地下水、沉积物、土壤甚至食物中均检测出了不同残留程度的磺胺类抗生素(Šinigoj-Gačnik et al., 2000; Jiang et al., 2011; Yang et al., 2011; Baran et al., 2011; Wang et al., 2014; Cecinato et al., 2017), 包括磺胺二甲基嘧啶(Sulfamethazine, SM2)在内的磺胺类抗生素对环境和人类健康造成的危害也正逐步引起人们的高度关注.相关毒理学研究表明, SM2对哺乳动物、水生动物、藻类、微生物、人类等都有直接致敏、致畸、致癌、致突变、激素失衡等毒害作用(Sanderson et al., 2004; Moore et al., 2010; Yi et al., 2010; Tao et al., 2014); 且土壤与水环境中长期残留的磺胺类抗生素能够诱发环境中产生耐药细菌, 对人类和动物健康也将构成极大的潜在威胁(Rysz et al., 2004; Hsu et al., 2014).因此, 如何去除环境中广泛存在的SM2等磺胺类抗生素已成为当前急需解决的环境热点问题.
目前, SM2的去除方法主要有物理化学法和生物法(Weiwei et al., 2009; Klavarioti et al., 2009; Na et al., 2015; Shi et al., 2018).与能耗高、处理费用昂贵、易形成对环境有害的高毒性中间体(Michael et al., 2004)的物理化学法相比较, 通过生物(主要为微生物)改变抗生素的结构, 使大分子化合物降解为小分子化合物从而降低毒性的生物处理法方法(付泊明等, 2017)具有能耗及处理费用低、反应条件温和、无二次污染等明显优势.近年来, 生物处理法在利用活性污泥、生物膜、好氧颗粒污泥等降解SM2的污水处理领域及用微生物对SM2污染土壤进行修复的领域均取得了很大的进展并展现出良好的应用前景.
但生物处理法当前面临的瓶颈之一是高效降解SM2功能微生物的筛选研究相对滞后.Ma Jesús等(2011)最早开始探究白腐菌杂色云芝(Trametes versicolor)对磺胺二甲基嘧啶的降解效能, 发现完全降解9 mg·L-1的SM2需要20 h.赵方等(2012)从长期受SM2污染的土壤中筛选出一株具有SM2降解功能的细菌, 根据其生理生化特性, 鉴定为克雷伯氏菌属(Klebsiella Trevisan SX5), 但菌株SX5对磺胺二甲基嘧啶的降解周期较长, 在60 d可使20 mg·L-1的SM2降解至10 mg·L-1, 同时降解过程符合一级降解动力学方程.之后陆续有嗜热-地芽孢杆菌(Thermophile-Geobacillus sp. S-07)(Pan et al., 2017)、无色杆菌(Achromobacter sp. S-3)(Huang et al., 2012)、不动杆菌(Acinetobacter sp)(Wang et al., 2018)、微杆菌属(Microbacterium sp.)(Topp et al., 2013)、黄孢原毛平革菌(Phanerochaete Chrysosporium)(郭晓丹娜等, 2017)等被报道.这些能降解SM2的微生物的降解效率普遍不高, 部分还需要外加碳源进行共代谢, 同时降解SM2的中间产物、降解途径及降解机理等仍不明确.
鉴于此, 本研究利用微生物纯培养技术分离纯化出一株能以SM2为唯一碳源的高效降解菌, 系统考察SM2浓度、温度、pH、初始接菌量对菌株生长及SM2降解效率的影响, 确定菌株的最佳降解条件, 并通过分析降解中间产物推测可能存在的生物降解途径.以期提供高效的降解SM2的功能微生物, 并为受SM2污染的环境修复提供技术支撑.
2 材料与方法(Materials and methods) 2.1 主要试剂磺胺二甲基嘧啶(SM2, 分析纯, 98%)由德国CNW Technologies GmbH公司生产.称取1.0 g磺胺二甲基嘧啶, 加入到100 mL 0.1 mol·L-1的NaOH溶液中, 配制成10000 mg·L-1的SM2储备液.根据不同的培养需要, 实验中分别选择筛选培养基、富集培养基.筛选培养基成分:Na2HPO4·12H2O 15580 mg·L-1, KH2PO4 675 mg·L-1, MgSO4·H2O 112 mg·L-1, NH4Cl 20.4 mg·L-1, FeSO4·7H2O 1 mg·L-1, MnSO4·H2O 1 mg·L-1, CuCl·2H2O 0.25 mg·L-1, 钼酸钠0.25 mg·L-1, CaCl2 0.015 mg·L-1, SM2 5~50 mg·L-1.富集培养基成分:蛋白胨10000 mg·L-1, 牛肉膏3000 mg·L-1, NaCl 5000 mg·L-1, 琼脂15000 mg·L-1, SM2 50 mg·L-1.
2.2 实验方法 2.2.1 SM2降解菌的富集与分离筛选以广州市猎德污水处理厂A2/O工艺的好氧池活性污泥作为种泥, 在好氧条件下以SM2为唯一碳源和能源进行SM2降解菌的富集培养.具体操作为:在250 mL锥形瓶内加入100 mL筛选培养基(SM2浓度为10 mg·L-1), 以10%的接种量加入泥水混合物, 混匀后取样进行HPLC分析.将锥形瓶置于30 ℃、150 r·min-1的摇床中振荡培养(为防止SM2光解, 摇床已做避光处理).每天取样至锥形瓶中, SM2被降解完全后倒掉上清液, 加入新鲜筛选培养基并逐步提高SM2浓度至50 mg·L-1.降解完全后进行菌种的分离筛选.
在超净工作台上取1 mL上述富集液进行10-1~ 10-8的逐级梯度稀释, 分别吸取0.5 mL各倍数的稀释液在固体筛选培养基(SM2浓度为50 mg·L-1)上培养, 挑取单菌落进行多次划线分离, 得到纯菌落后接种于筛选培养基上(SM2浓度为10 mg·L-1)验证其降解效果, 并用紫外分光光度计在600 nm下的吸光度(OD600)表示生物量的变化.
2.2.2 菌株的保存将筛选到的SM2降解菌接种到斜面培养基中, 在30 ℃生化培养箱中培养至斜面上长出菌落后放入4 ℃冰箱冷藏.每隔1~2月, 将斜面菌株接到液体培养基中活化, 活化后再划线到固体平板上, 挑取单菌落接种到斜面培养基上.每次实验前24 h, 将菌株划线接种到富集固体培养基上, 保证实验使用菌量足够.
2.2.3 菌株的生理生化鉴定形态观察:将菌株在平板上划线, 待长出单菌落后, 观察其菌落大小、形状、色泽、边缘是否隆起、质地、颜色、透明程度、是否易挑起等特征, 并用光学显微镜观察菌株的形态大小.
生理生化试验:革兰氏染色试验、甲基红试验、过氧化氢酶试验、苯丙氨脱氢酶试验、葡萄糖酸盐试验, 具体方法见参考文献(杨基先等, 2002).
2.2.4 菌株16S rDNA基因序列分析采用天根细菌基因组DNA提取试剂盒提取细菌DNA, 进行细菌的16S rDNA V3+V4区域的PCR扩增, 引物为:27f: AGAGTTTGATCMTGGCTCAG, 1492R: TACGG YTACCTTGTTACGACTT.将样品送至北京六合华大基因科技有限公司广州分公司进行纯化测序, 所测得序列在NBCI上进行比对, 确定微生物所属菌种, 并挑选相关序列, 采用MEGA 6.0软件构建系统发育树.
2.2.5 HPLC分析SM2浓度测定采用高效液相色谱法, 使用高效液相色谱仪(HPLC-1260 InfinityⅡ Agilent Technologies, USA).每次取样1 mL, 过0.22 μm尼龙有机滤膜于2 mL棕色样品瓶, 并将样品置于4 ℃环境下保存待测.高效液相色谱仪检测器为SPD-20AV, 检测波长270 nm, 色谱柱采用SHIMADZU VP-ODS C18, 流动相为A/B=75/25(V/V), 其中, A为3 mL冰醋酸加入到750 mL超纯水, B为乙腈, 流速1.0 mL·min-1, 柱温40 ℃.
2.2.6 菌株降解SM2的条件优化从富集平板培养基上挑取生长24 h以内的菌落到筛选培养基, 培养24 h后按照适宜的接种量将其接种到新鲜的筛选培养基中, 观察在不同温度(10、20、30、40 ℃)、pH(4、6、8、10)、SM2浓度(5、10、20、50、70、100 mg·L-1)、接种量(OD600分别为0.02、0.1、0.3、0.5、1.0)下菌株J2对SM2的降解情况.每个批次做3组平行实验.
2.2.7 SM2降解中间产物降解中间产物的定性分析采用三重四极杆液相色谱-质谱联用仪(LC-MS/MS), 参数设置如下:色谱柱为Thermo Hypersil GOLD C18柱(100 mm×2.1 mm, Particle Sz. 1.9 μm); 进样量3 μL; 柱温40 ℃; 流动相:A相为0.1%甲酸水, B相为甲醇; 洗脱方式为梯度洗脱.
3 结果(Results) 3.1 菌株的分离筛选和鉴定用经过富集的活性污泥进行多次稀释涂布、平板划线等操作后, 最终筛选出4株能在以SM2为唯一碳源的固体筛选培养基上生长且具有不同菌落形态的菌株, 分别命名为J1、J2、J3、J4.将4株细菌分别接种到以20 mg·L-1 SM2为唯一碳源的筛选培养基上, 30 ℃、150 r·min-1下振荡培养72 h后, 观察细菌的生长情况及对SM2的降解效果.其中, 菌株J2和J3在以SM2为唯一碳源和能源的条件下生长得相对较好, 且在72 h内对SM2的降解效率也较高, 分别达到94.21%和80.84%, 而另外的菌株J1和J4在72 h内的降解效率仅分别为44.91%和9.01%.因此, 挑选生长量较大、降解效能最好的J2菌株作为实验菌株进行进一步研究.
菌株J2的形态如图 1所示, 菌落呈圆形, 表面略有光泽, 挑起有丝状, 略有粘连, 白色, 不透明, 培养较长时间后表面转为浅红色.其生理生化特征为:革兰氏染色、运动性、过氧化氢酶及葡萄糖酸盐试验均呈阳性, 甲基红及苯丙氨酸脱氢酶试验则显示阴性.菌株的16S rRNA基因序列分析(Genbank登记号: MK424313), 菌株系统发育树见图 2.菌株J2与蜡样芽胞杆菌(Bacillus cereus)同源性为99%, 因此, 菌株被鉴定为蜡样芽胞杆菌的(Bacillus cereus).
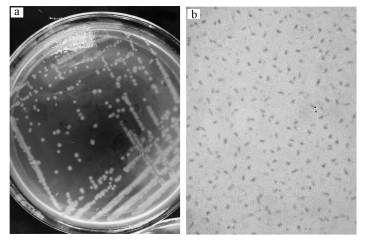 |
| 图 1 细菌J2的形态特征 (a.在平板培养基上的菌落形态; b.在光学显微镜下的形态) Fig. 1 Colonial morphology of strain J2 (a.the image of strain J2 in the salt medium; b.the image of strain J2 under light microscope) |
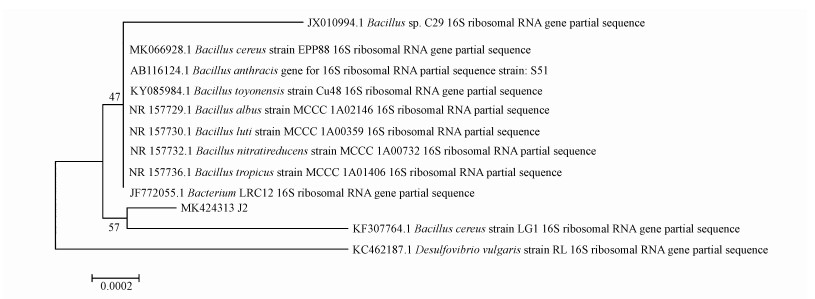 |
| 图 2 菌株J2的系统发育树 Fig. 2 Phylogenetic tree of strain J2 |
在SM2初始浓度分别为5、10、20、50、70、100 mg·L-1的条件下对菌株J2进行振荡培养, 设置培养温度为30 ℃, 初始pH为8.0, 初始OD600为0.1.不同SM2初始浓度下菌株J2对SM2的降解结果如图 3a所示.由图可知, 在SM2初始浓度分别为5、10、20 mg·L-1的情况下, J2能快速降解SM2, 没有观察到延滞, 并分别在4、10、12 h内降解率达到100%.而在SM2初始浓度为50和70 mg·L-1的情况下分别出现了12和36 h的延滞, SM2初始浓度为50 mg·L-1时在36 h内对SM2的降解率达到100%, SM2初始浓度为70 mg·L-1时在72 h内降解率达到99.51%.当SM2初始浓度为100 mg·L-1时, 48 h内没有观察到明显的SM2降解, 72 h后SM2的降解率仅为26.24%, 菌株的OD600也由最初的0.092增长到0.136, 120 h后对SM2的降解率才超过90%(120 h的数据未显示在图 3a中).初始SM2浓度升高会使J2菌株降解SM2的延滞时间增加, 浓度越高降解速率越慢.这可能是由于抗生素对细菌生长具有抑制作用, 且浓度越高, 该抑制作用对磺胺类化合物的降解产生的影响越大(Mao et al., 2018).尽管J2菌株对SM2的耐受浓度可达到100 mg·L-1, 但在实际应用中为了确保降解效果的同时处理更高浓度的SM2, 选取50 mg·L-1作为菌株J2降解SM2的最佳浓度.
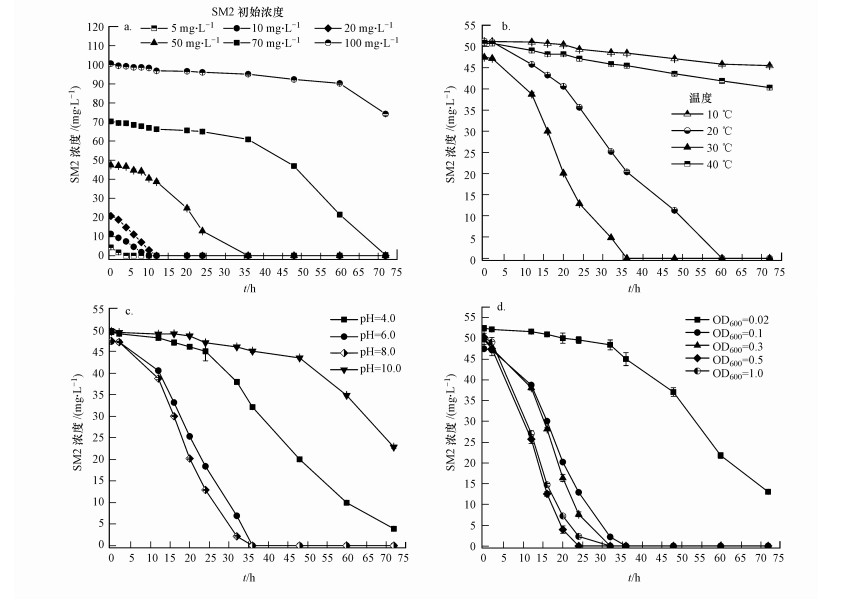 |
| 图 3 SM2初始浓度(a)、温度(b)、pH (c)和初始菌体浓度(OD600)(d)对J2菌株降解SM2的影响 Fig. 3 The effect of SM2 concentration(a), temperature(b), pH(c) and OD600 (d) cues on the removal rate of SM2 by J2 |
分别在10、20、30、40 ℃ 4个温度条件下对菌株J2进行振荡培养, 设置SM2初始浓度为50 mg·L-1, 初始pH为8.0, 初始OD600为0.1.不同温度下菌株J2对SM2的降解效果见图 3b.可以看出, 温度对J2菌株降解SM2具有显著的影响.10 ℃时, 细菌的生长和SM2的降解均受到了明显的抑制, 仅能降解11.08%的SM2;20 ℃时的降解速率明显加快, 72 h即可将SM2降解完全; 在30 ℃时SM2的降解速率达到最大, 36 h即可达到100%的降解率; 然而, 继续提高培养温度到40 ℃, 菌株的生菌浊度反而下降, 对SM2的降解率也大幅下降, 72 h时的降解率仅为20.75%.菌株降解性能下降可能是由于温度过高或过低影响了细菌体内酶的活性(任南琪等, 2005).
3.2.3 不同pH条件下的降解实验设置了4个不同初始pH(4、6、8、10)的培养环境, 初始SM2浓度为50 mg·L-1, 培养温度为30 ℃, 初始OD600为0.1.不同pH下菌株J2的SM2降解结果如图 3c所示.由图 3c可知, 改变培养基的初始pH值对SM2的降解有显著影响, 当pH为6.0~8.0时, 菌株降解SM2的效果最好且没有明显差异, 在36 h时内降解达到100%.而当pH分别为4.0和10.0时, 菌株对SM2的降解效果下降明显, 36 h的降解率率仅分别为35.12%和9.24%, 将反应时间延长到72 h, 降解率分别为92.03%和54.02%.在pH为10.0的反应后期, 菌株降解SM2的速率明显增大, 可能是由于反应过程中产生了酸性产物(Yang et al., 2015), 使体系pH略有降低, 促进了微生物对SM2的降解.
3.2.4 不同初始菌体浓度(OD600)下的降解本实验以菌悬液在600 nm下的吸收波长(OD600)来控制初始接菌量, 分别设置5个OD600值(0.02、0.1、0.3、0.5、1.0)对菌株J2进行振荡培养, 初始SM2浓度为50 mg·L-1, 初始pH为8.0, 培养温度为30 ℃.不同初始菌体浓度(OD600)下菌株J2对SM2的降解效果如图 3d所示.由图 3d可知, 随着菌体浓度的增加, 菌株J2对SM2的降解率逐渐增大.初始OD600为0.1~1.0的体系中, 菌株J2对SM2的降解率达到100%的用时分别为36、32、24、32 h, 总体上差异不明显; 而初始OD600为0.02时, 72 h内的降解率仅为75.16%, 这是因为体系内初始微生物数量过少, 需要较长的适应期(赵锐, 2012).
3.3 菌株J2的降解途径根据三重四极杆液相色谱-质谱联用仪(LC-MS/MS)测出SM2的标准色谱图和质谱图见图 4a, SM2的出峰时间为4.31 min, 荷质比(m/z)为278.92752.图 4b~4f为检测到的中间产物的一级和二级质谱图.根据一级质谱和二级质谱的m/z推算, 共有5种中间产物, 分别为产物1:N-(4, 6-二甲基嘧啶-2基)-1, 4-二苯胺(m/z=214.95);产物2:苯胺(m/z=93.01);产物3:2-氨基-4, 6-二甲基嘧啶(m/z=123.91);产物4:N-(3, 5-二甲基嘧啶)-苯磺酰胺(m/z=262.60);产物5:苯亚砜(m/z=141.74).这5种中间产物的结构式分别标注在图 4中.
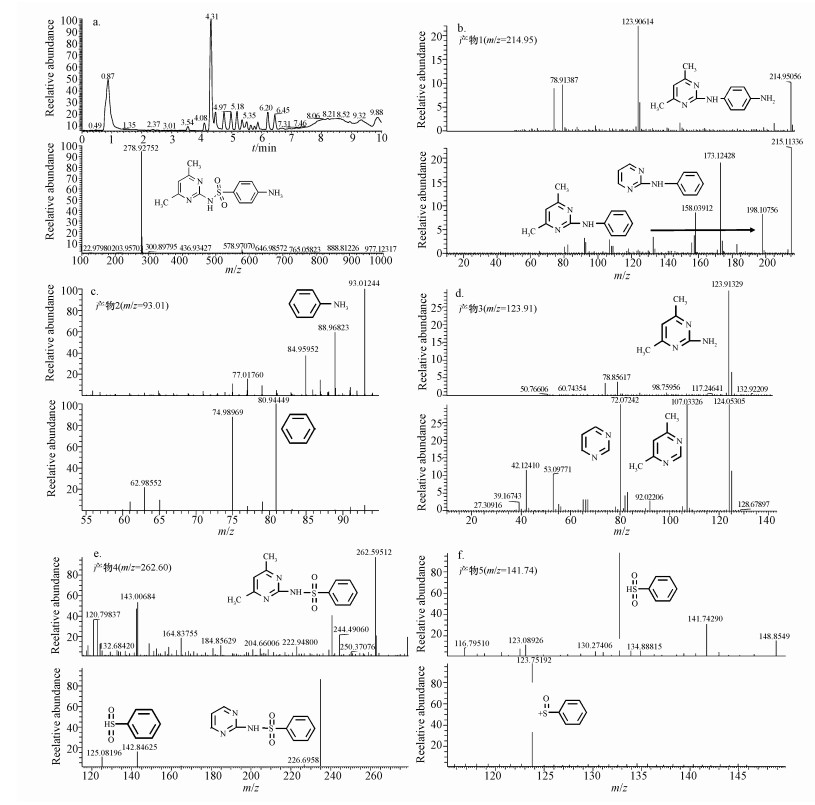 |
| 图 4 SM2的标准色谱图和标准质谱图(a)及J2菌株降解SM2中间产物的质谱图(b~f) Fig. 4 Standard chromatogram spectrogram of SM2 and standard mass spectrogram of SM2 (a) and the mass spectrum of SM2 intermediate products(b~f) |
根据产物的种类组成推测菌株J2降解SM2的两种路径如图 5所示, 路径Ⅰ:与大多数磺胺类物质(Boreen et al., 2008; Batchu et al., 2014)类似, 磺胺二甲基嘧啶分子首先在酶促反应作用下(Ma et al., 2011)脱除SO2, 生成嘧啶环和苯胺环自由基, 这两种自由基再经过环间耦合生成产物1(N-(4, 6-二甲基嘧啶-2基)-1, 4-二苯胺(m/z=214.95)), 在活性氧物种如·OH作用下(Werner et al., 2007), 产物1分子中的C—N键断开生成产物2(苯胺(m/z=93.01)和产物3(2-氨基-4, 6-二甲基嘧啶(m/z=123.91)).Ma等(2011)在研究白腐真菌对磺胺二甲基嘧啶的降解机理时得到了类似的结果.同样, 在物理化学法和光降解法处理磺胺二甲基嘧啶的过程中, 也是首先脱除分子中的SO2(Katherine et al., 2010; Periša et al., 2013).路径Ⅱ:在一种漆酶的作用下(Ma et al., 2011) N4键断裂, 产生产物4(N-(3, 5-二甲基嘧啶)-苯磺酰胺(m/z=262.60)), 之后N—S键断裂, 进一步形成产物3和产物5(苯亚砜(m/z=141.74)).
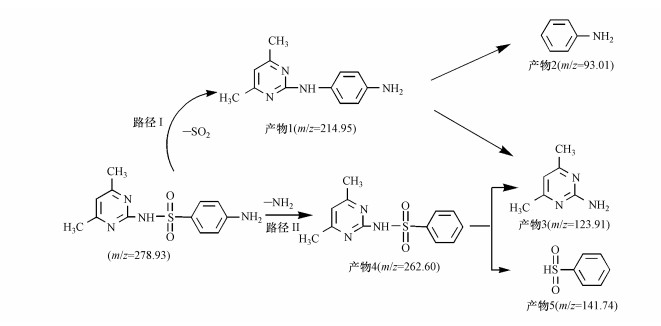 |
| 图 5 J2菌株降解SM2可能的代谢途径 Fig. 5 Proposed metabolic pathway of the biodegradation of SM2 by J2 |
将现有报道中具有SM2降解能力的几株纯菌株与J2进行了对比, 结果汇总如表 1所示.由表 1可知, 目前报道的以SM2为唯一碳源的研究相当有限, 菌株J2的最佳降解浓度为50 mg·L-1, 远远超过最佳浓度范围在0.05~2 mg·L-1之间的其他菌株.菌株J2显示出对SM2具有极高的耐受性, 这种对SM2抑制作用的耐受力是保证高效、稳定的SM2去除率的前提(Essam et al., 2010).
| 表 1 SM2降解效果比较 Table 1 Comparison of SM2 degradation effect |
从表 1还可以看出, 大多数已分离的以SM2为唯一碳源的纯种菌株对SM2的降解效果也不是十分理想, 降解率从低到高分别仅为18.53%、23%、33%、42%, 且降解速度也较慢, 分别在168、120、120和96 h内才能达到这个降解率.而菌株J2却具有更为优异的降解效果, 36 h内即可将50 mg·L-1的SM2全部降解.
Na等(2015)利用实验室SBR反应器驯化活性污泥降解SM2, 在24 h内初始浓度为1 mg·L-1的SM2仅能降解56%;Bing等(2010)采用两套A/O工艺反应器对11种抗生素进行去除研究, 结果发现, 在48 h内反应器可以降解39.1%的SM2;梅子凡(2017)利用多级A/O工艺处理养猪废水中的抗生素, 发现系统对1 mg·L-1的SM2的降解率仅为65.29%.可见, 本研究分离出的能以SM2为唯一碳源的菌株J2具有高效降解SM2的能力, 同样在磺胺类抗生素废水处理领域展现出良好的应用潜力.
4 结论(Conclusions)1) 本研究从城市污水处理厂活性污泥中筛选出一株能以SM2为唯一碳源和能源的菌株J2, 初步鉴定为蜡样芽胞杆菌(Bacillus cereus)(Genbank登记号:MK424313).
2) 菌株J2有效降解SM2的最佳条件为:SM2浓度50 mg·L-1, 温度30 ℃, pH=8.0, 初始OD600=0.1, 在此条件下, 36 h内菌株J2对SM2的降解率即可达到100%.同时, 该菌株可以利用SM2为唯一碳源, 且具有较高的SM2耐受性及优异的SM2降解效率.
3) 分析出菌株J2降解SM2的中间产物有5种, 并推断出菌株J2降解SM2的途径有两条, 路径Ⅰ:磺胺二甲基嘧啶分子首先在酶促反应作用下脱除SO2, 生成嘧啶环和苯胺环自由基, 两种自由基再经过环间耦合生成N-(4, 6-二甲基嘧啶-2基)-1, 4-二苯胺, 该分子中的C—N键在活性氧物种作用下断开生成苯胺和2-氨基-4, 6-二甲基嘧啶; 路径Ⅱ:N4键断裂, 产生N-(3, 5-二甲基嘧啶)-苯磺酰胺, 之后N-(3, 5-二甲基嘧啶)-苯磺酰胺的N—S键断裂, 进一步形成2-氨基-4, 6-二甲基嘧啶和苯亚砜.
Baran W, Adamek E, Ziemiańska J, et al. 2011. Effects of the presence of sulfonamides in the environment and their influence on human health[J]. Journal of Hazardous Materials, 196(1): 1–15.
|
Bing L, Tong Z. 2010. Biodegradation and adsorption of antibiotics in the activated sludge process[J]. Environmental Science & Technology, 44(9): 3468–3473.
|
Cecinato A, Romagnoli P, Perilli M, et al. 2017. Pharmaceutical substances in ambient particulates:A preliminary assessment[J]. Chemosphere, 183: 62–68.
DOI:10.1016/j.chemosphere.2017.05.100
|
Dmitrienko S G, Kochuk E V, Apyari V V, et al. 2014. Recent advances in sample preparation techniques and methods of sulfonamides detection-A review[J]. Analytica Chimica Acta, 850: 6–25.
DOI:10.1016/j.aca.2014.08.023
|
Essam T, Amin M A, Tayeb O E, et al. 2010. Kinetics and metabolic versatility of highly tolerant phenol degrading Alcaligenes strain TW1[J]. Journal of Hazardous Materials, 173(1): 783–788.
|
付泊明, 陈立伟, 蔡天明, 等. 2017. 诺氟沙星降解菌NOR-36的分离筛选及降解特性研究[J]. 环境科学学报, 2017, 37(2): 576–584.
|
郭晓丹娜, 郭夏丽. 2017. 白腐真菌共培养对磺胺二甲基嘧啶降解的影响[J]. 生物技术通报, 2017, 33(5): 197–202.
|
Hsu J T, Chen C Y, Young C W, et al. 2014. Prevalence of sulfonamide-resistant bacteria, resistance genes and integron-associated horizontal gene transfer in natural water bodies and soils adjacent to a swine feedlot in northern Taiwan[J]. Journal of Hazardous Materials, 277(4): 34–43.
|
Jiang L, Hu X, Yin D, et al. 2011. Occurrence, distribution and seasonal variation of antibiotics in the Huangpu River, Shanghai, China[J]. Chemosphere, 82(6): 822–828.
DOI:10.1016/j.chemosphere.2010.11.028
|
Klavarioti M, Mantzavinos D, Kassinos D. 2009. Removal of residual pharmaceuticals from aqueous systems by advanced oxidation processes[J]. Environment International, 35(2): 402–417.
DOI:10.1016/j.envint.2008.07.009
|
Ma Jesús G, Carlos E R, Teresa V, et al. 2011. Biodegradation of sulfamethazine by Trametes versicolor:Removal from sewage sludge and identification of intermediate products by UPLC-QqTOF-MS[J]. Science of the Total Environment, 409(24): 5505–5512.
DOI:10.1016/j.scitotenv.2011.08.022
|
Huang M, Tian S, Chen D, et al. 2012. Removal of sulfamethazine antibiotics by aerobic sludge and an isolated Achromobacter sp.S-3[J]. Journal of Environmental Sciences, 24(9): 1594–1599.
DOI:10.1016/S1001-0742(11)60973-X
|
Mao F, Liu X, Wu K, et al. 2018. Biodegradation of sulfonamides by Shewanella oneidensis MR-1 and Shewanella sp.strain MR-4[J]. Biodegradation, 29(11): 1–12.
|
Michael C D, Ching-Hua H. 2004. Transformation of the antibacterial agent sulfamethoxazole in reactions with chlorine:kinetics, mechanisms, and pathways[J]. Environmental Science & Technology, 38(21): 5607–5615.
|
Moore J E, Moore P J A, Millar B C, et al. 2010. The presence of antibiotic resistant bacteria along the River Lagan[J]. Agricultural Water Management, 98(1): 217–221.
DOI:10.1016/j.agwat.2010.09.001
|
梅子凡.2017.多级A/O工艺处理养猪废水中抗生素的试验研究[D].重庆: 重庆大学
http://cdmd.cnki.com.cn/Article/CDMD-10611-1017838656.htm |
Na Y, Junfeng W, Shiju Z, et al. 2015. Removal of concentrated sulfamethazine by acclimatized aerobic sludge and possible metabolic products[J]. Peerj.
DOI:10.7717/peerj.1359
|
Pan L, Tang X, Li C, et al. 2017. Biodegradation of sulfamethazine by an isolated thermophile-Geobacillus sp.S-07[J]. World Journal of Microbiology & Biotechnology, 33(5): 85.
|
Rysz M, Alvarez P J J. 2004. Amplification and attenuation of tetracycline resistance in soil bacteria:aquifer column experiments[J]. Water Research, 38(17): 3705–3712.
DOI:10.1016/j.watres.2004.06.015
|
任南琪, 李建政. 2005. 污染控制微生物生态学[M]. 哈尔滨: 哈尔滨工业大学出版社.
|
Sanderson H, Brain R A, Johnson D J, et al. 2004. Toxicity classification and evaluation of four pharmaceuticals classes:antibiotics, antineoplastics, cardiovascular, and sex hormones[J]. Toxicology, 203(1): 27–40.
|
Shi B J, Wang Y, Geng Y K, et al. 2018. Application of membrane bioreactor for sulfamethazine-contained wastewater treatment[J]. Chemosphere, 193: 840–846.
DOI:10.1016/j.chemosphere.2017.11.051
|
šinigoj-Gačnik K, Doganoc D Z. 2000. Contamination of farm animals and fishes from slovenia with heavy metals and sulfonamides[J]. Bulletin of Environmental Contamination & Toxicology, 64(2): 235–241.
|
Tao L, Shilin Y, Yanqiu C, et al. 2014. Integrated biomarker responses in zebrafish exposed to sulfonamides[J]. Environmental Toxicology & Pharmacology, 38(2): 444–452.
|
Topp E, Chapman R, Devers-Lamrani M, et al. 2013. Accelerated biodegradation of veterinary antibiotics in agricultural soil following long-term exposure, and isolation of a sulfamethazine-degrading sp[J]. Journal of Environmental Quality, 42(1): 173–178.
DOI:10.2134/jeq2012.0162
|
Wang D, Qian S, Zhao W T, et al. 2014. Pharmaceutical and personal care products in the surface water of China:A review[J]. Chinese Science Bulletin, 59(59).
DOI:10.1360/972013-370
|
Wang J, Zhou A, Zhang Y, et al. 2016. Research on the adsorption and migration of sulfa antibiotics in underground environment[J]. Environmental Earth Sciences, 75(18).
DOI:10.1007/s12665-016-6056-9
|
Wang S, Hu Y, Wang J. 2018. Biodegradation of typical pharmaceutical compounds by a novel strain Acinetobacter sp[J]. Journal of Environmental Management, 217: 240–246.
DOI:10.1016/j.jenvman.2018.03.096
|
Weiwei B, Zhimin Q, Xun P, et al. 2009. Removal of veterinary antibiotics from sequencing batch reactor (SBR) pretreated swine wastewater by Fenton's reagent[J]. Water Research, 43(17): 4392–4402.
DOI:10.1016/j.watres.2009.06.057
|
王强锋, 朱彭玲, 夏中梅, 等. 2018. 三种农用抗生素降解真菌的筛选及其降解性能[J]. 农业资源与环境学报, 2018, 35(6): 533–539.
|
Yang J F, Ying G G, Zhao J L, et al. 2011. Spatial and seasonal distribution of selected antibiotics in surface waters of the Pearl Rivers, China[J]. Journal of Environmental Science and Health, Part B, 46(3): 272–280.
DOI:10.1080/03601234.2011.540540
|
Yi L, Daqing M, Michal R, et al. 2010. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 44(19): 7220–7225.
|
杨基先, 马放, 任南琪. 2002. 污染控制微生物学实验[M]. 哈尔滨: 哈尔滨工业大学出版社.
|
Zhang Y, Marrs C F, Simon C, et al. 2009. Wastewater treatment contributes to selective increase of antibiotic resistance among Acinetobacter spp[J]. Science of the Total Environment, 407(12): 3702–3706.
DOI:10.1016/j.scitotenv.2009.02.013
|
赵方.2012.磺胺二甲基嘧啶的微波与微生物降解研究[D].郑州: 郑州大学
http://cdmd.cnki.com.cn/Article/CDMD-10459-1012354213.htm |
赵锐.2012.青岛近海两种生态环境可培养细菌多样性研究及3株海洋新菌的分类鉴定[D].青岛: 中国海洋大学
http://cdmd.cnki.com.cn/Article/CDMD-10423-1012506638.htm |
 2019, Vol. 39
2019, Vol. 39


