2. 工业聚集区污染控制与生态修复教育部重点实验室, 广州 510006;
3. 广东省环境风险防控与应急处置工程技术研究中心, 广州 510006;
4. 广东省环境纳米材料工程技术研究中心, 广州 510006
2. The Key Lab of Pollution Control and Ecosystem Restoration in Industry Clusters, Ministry of Education, Guangzhou 510006;
3. Guangdong Provincial Engineering and Technology Research Center for Environmental Risk Prevention and Emergency Disposal, South China University of Technology, Guangzhou Higher Education Mega Centre, Guangzhou 510006;
4. Guangdong Engineering and Technology Research Center for Environmental Nanomaterials, Guangzhou 510006
抗生素的发现是人类医学的一个伟大进步, 然而抗生素在降低细菌感染类疾病的发病率和死亡率的同时, 也导致了携带抗生素抗性基因(antibiotic resistance genes, ARGs)的菌株的出现, 细菌获得抗药性后, 本来对其有效的抗生素失去功效.抗生素抗药性给人类健康造成了极大的威胁, 由此引发的生态风险更是引人关注(Martínez, 2008; Lee et al., 2013; Blair et al., 2015; Qiao et al., 2018).ARGs产生的选择压力主要来自于临床上大量抗生素的使用, 另一个很重要的来源就是农业畜牧业的抗生素使用, 最后一方面则是自然选择过程.虽然细菌在自然条件下就抗生素对细菌产生选择压力, 能产生ARGs, 但工业化抗生素的大规模生产和使用大大加速了细菌获得ARGs这一过程.目前医用和兽用抗生素的大量使用造成抗生素进入到自然环境中, 在各种环境介质中, 包括地表水、地下水、沉积物和土壤中都发现了抗生素痕迹.特别是在污水处理厂进出水、医院废水、养殖废水和制药废水中能够检测到持久性存在的低浓度抗生素(Boxall et al., 2005a; 庄榆佳等, 2017; 邹高龙等, 2018; 孙秋根等, 2018).这些残留于环境的抗生素对细菌产生选择压力, 使被抗生素污染的环境介质中抗性菌及抗性基因的水平远远高于环境背景值(Bougnom et al., 2017; 张宁等, 2018).显然, 非常有必要更好地了解抗生素对细菌的影响, 以减缓耐药性的出现.
更深入地理解促进抗生素抗性发展的分子机制和澄清导致细胞中ARG产生的抗生素的分子反应至关重要.细菌获得抗生素抗药性的途径主要有3种, ①通过自身基因突变, 这是群体中的药物特异性选择并且天然存在的抗性变异, ②通过水平基因转移(horizontal gene transfer, HGT), 分别对应ARGs在微生物中的产生和传播过程, ③超突变菌株中增加的诱变(Joseph et al., 2015; Olsen, 2015; Mei-Ting et al., 2015; Mathieu et al., 2016).目前, 越来越多的证据表明抗生素的亚MIC可以刺激ARGs的产生并增加细菌突变率(Kohanski et al., 2010b; Jutkina et al., 2018).鉴于抗生素的大量使用, 它们最终被释放到不同的环境, 如土壤、沉积物、地表水和地下水中(Boxall et al., 2005b; Juan et al., 2012; Zhu et al., 2017; 陈卫平等, 2017).因此, 细菌经常在其栖息地持久性接触低剂量抗生素.这就值得强调研究抗生素亚MIC对细菌影响的重要性.
在抗生素胁迫条件下, 细菌会启动许多胁迫响应基因.蛋白质是生物体中重要的功能分子, 外源污染物和微生物细胞之间的相互作用机制可能涉及一系列特定功能蛋白质的改变.此外, 与污染物的转化或解毒相关的代谢过程通常受整套蛋白质及其网络的调节, 而不是单一的有效酶.相对和绝对定量同位素标记(isobaric tags for relative and absolute quantification, iTRAQ)技术是一种多肽体外标记技术, iTRAQ结合串联质谱分析技术已成为蛋白定性和定量研究的重要工具之一, 并且在各个领域得到广泛应用, 特别是近年来在环境分子机理领域中得到越来越多青睐.因此, 在本研究中选择了这种方法.
目前, 用亚抑菌浓度的四环素持续诱导野生型大肠杆菌(敏感菌株)产生抗性并对比蛋白差异的研究未见报道.因此, 本研究以野生型大肠杆菌为基础材料, 四环素为模型抗生素, 通过亚抑菌浓度四环素对其进行持续性诱导后产生抗性, 分析对比抗性菌株和敏感菌株的蛋白差异, 此外, 进一步分析差异蛋白质表达以检测与ARGs突变相关的蛋白质和基因, 然后在低剂量四环素胁迫下鉴定细胞生长和代谢的潜在机制, 以阐明潜在相关蛋白在该过程中的作用.同时, 旨在探索并提供一种对ARGs的产生分子机制更深入的见解.
2 材料与方法(Materials and methods) 2.1 供试大肠杆菌, 抗生素和培养条件所有实验均用野生型大肠杆菌ATCC25922进行, 在Luria-Bertani(LB)培养基(10 g胰蛋白胨, 5 g酵母提取物和10 g氯化钠在1 L去离子水中)或Mueller-Hinton Broth(MHB)培养基中, 37 ℃培养.上述培养基原料均购于北京索莱宝公司, 四环素购于美国Sigma公司.
将大肠杆菌接种到LB培养基中, 置于37 ℃条件下并以250 r·min-1摇动过夜.将培养物以高速振荡温育, 是为了适当的通气, 防止细菌在烧瓶中形成反应性氧化物质(ROS).接下来, 将制备的样品应用于亚抑制浓度测定, 并且结合实验和机理研究进行以下实验.所有实验一式3份进行.
2.2 最低抑菌浓度(MIC)分析本实验使用美国临床和实验室标准研究所的微量肉汤稀释法测定MIC.简而言之, 将来自每种处理条件的等分细胞稀释到MHB培养基中, 使得600 nm处的光密度(OD600)为0.05并分配到含有四环素(从128 μg·mg-1到0.25 μg·mg-1)的96孔板中.将96孔板在37 ℃下孵育24 h, 之后使用酶标仪测量OD600.计算每种药物浓度的OD600值, 并将MIC确定为OD600抑制率为90%生长的浓度.
2.3 供试菌株的诱导主要使用抗生素四环素诱导野生型大肠杆菌ATCC25922敏感菌株.课题组早期的实验观察到该大肠杆菌对四环素的MIC值为2 μg·mL-1.最初, 将大肠杆菌的过夜培养物以1:10000稀释到含有亚抑制性抗生素1 μg·mL-1(相当于1/2倍MIC)的新鲜LB培养基中, 然后置于平底烧瓶中在37 ℃和250 r·min-1培养24 h.此后, 每天从上一天的培养液中转接到新鲜的含有亚抑菌浓度的LB培养基中, 重复上述步骤, 连续培养20 d.每隔2 d测1次MIC值.
2.4 抗性基因的定量检测本实验采用实时荧光定量PCR法(Real-time fluorescence quantitative PCR, qPCR)测定敏感菌株经诱导后产生的ARGs, 使用qPCR测定来评估可能受四环素抗生素影响的接合过程相关的基因的转录.研究选取了8种靶基因, 包括四环素抗性基因(tetA、tetB、tetE和tetZ), β-内酰胺类抗性基因(blaTEM和blaFOX), 多重耐药性基因(acrA、marA和marR)和16S rRNA.本研究中使用的引物如表 1所示.选取原始的大肠杆菌(培养至对数期)为对照组, 四环素处理20 d的大肠杆菌(培养至对数期)为实验组.
| 表 1 目的基因引物序列 Table 1 Target gene primer sequences |
RNA的提取, 首先菌体离心, 加1 mL的Trizol, 用Tissue Ruptor匀浆器处理1 min, 室温放置10 min.然后加200 μL的氯仿, 充分混匀, 15000 r·min-1离心5 min.取上清液, 至1.5 mL的Eppendorf管中, 加600 μL的氯仿, 混匀, 15000 r·min-1离心5 min.取上清液, 至1.5 mL的Eppendorf管中, 加500 μL异丙醇, 混匀, 15000 r·min-1离心10 min.弃上清液, 用75%的乙醇1 mL冲洗, 高速离心5 min.弃上清液, 于空气中干燥3 min, 将干燥后的RNA溶解于水中, 取1 μL总RNA测OD260并定量, 得到RNA提取物, 待用.
实时荧光定量PCR反应体系由16 μL的反应体系组成, 反应在ABI ViiA 7 PCR(Applied Biosystems)仪上对384孔板中进行, 每个样品设3次平行实验.将反应循环在94 ℃保持2 min, 然后进行40个循环:在反应体系中94 ℃反应10 s, 60 ℃反应10 s和72 ℃反应40 s.每经过一个循环, 收集1次荧光强度信号, 这样就可以通过荧光强度变化监测产物量的变化, 结合相应的软件分析, 可以得到荧光扩增曲线, 计算待测样品初始模板的量.反应中的Ct值数据的采集采用校正的阈值设定, 实时荧光定量PCR的方法以16S rRNA作为内参基因, 进行定量.
2.5 iTRAQ标记和LC-MS / MS蛋白质组学分析将细胞样品在液氮中研磨成粉末, 然后在含有8 mL用Tris-HCl饱和的苯酚的50 mL离心管中进行超声处理.随后, 在6000 g离心20 min后, 将匀浆用8 mL提取缓冲液(0.7 mol·L-1的蔗糖, 0.1 mol·L-1的KCl, 50 mmol·L-1的EDTA, 0.5 mol·L-1的Tris, pH 7.5, 2%b-巯基乙醇和1 mmol·L-1的PMSF)涡旋.在4 ℃下, 收集上清液酚相, 再次用萃取缓冲液再萃取两次.然后, 通过在甲醇中加入0.1 mol·L-1乙酸铵, 然后在-20 ℃下进一步温育过夜, 进行酚相中蛋白质的沉淀.在4 ℃下以6000 g离心20 min后, 分别用甲醇和丙酮冲洗收集的蛋白质两次.最后, 将蛋白质样品溶解在裂解缓冲液(7 mol·L-1尿素, 2 mol·L-1硫脲, 20 mmol·L-1 Tris碱)中, 并使用牛血清白蛋白作为标准, 通过Bradford方法估计蛋白质浓度.
用FASP方法进行蛋白质消化.简而言之, 每个样品中的200 μg蛋白质, 首先与制造商提供的4 μL还原剂在60 ℃下反应1 h, 然后与2 μL半胱氨酸封闭试剂在室温下孵育10 min, 最后转移至10 kD超滤管.在12000 r·min-1离心20 min后, 加入100 μL的0.25 mol·L-1 TEAB并在相同条件下进一步进行离心, 以收集捕获在超滤膜上的蛋白质, 该过程重复3次.随后, 将从3次重复混合的收集的蛋白质样品转移到新管中, 并加入50 μL的0.5 mol·L-1 TEAB和胰蛋白酶(胰蛋白酶:蛋白质的质量比= 1:50), 然后在37 ℃下过夜反应.然后, 补充胰蛋白酶(胰蛋白酶:蛋白质= 1:100)并进一步培养4 h, 通过离心收集滤液后, 加入50 μL 0.5 mol·L-1TEAB, 通过再离心收获的滤液与从前一个获得的滤液合并.最后一次离心.
然后将提取的蛋白质(每个样品中100 μg)还原, 烷基化, 水解, 并用同量异位标签标记, 用于相对和绝对定量(iTRAQ)试剂.将标记的肽合并, 并使用旋转真空浓缩器干燥, 重悬于缓冲液(含有2%乙腈(ACN)的5 mmol·L-1甲酸铵; pH=10)中, 并通过高pH反相液相色谱(RPLC-MS)分离.A相和B相分别是在水中的20 mmol·L-1甲酸铵(pH=10)和在100%ACN(pH=10)中的20 mmol·L-1甲酸铵.第一维高pH-RP液相分离先用95%的A相和5%的B相平衡柱子30 min, 然后将标记后抽干的混合多肽用200 μL的A相复溶, 取100 μL进样后以0.2 mL·min-1的速率进行梯度洗脱:B相从5%到37%, 最后在5 min内使B相的比例上升至95%并保持5 min, 共85 min.整个洗脱过程在214 nm吸光度下进行监测, 从线性梯度开始根据峰型收取24个组分, 每管每50 s接1次, 梯度为85 min, 反复循环接样.使用连接至Q Exactive hybrid quadrupole-Orbitrap质谱仪(Thermo Scientific Q Exactive)的Nano Aquity UPLC系统(Thermo Dionex Ultimate 3000 RSLCnano)进行所有收集的分级的LC-MS / MS分析.第二维反相液质联用RPLC-MS的肽段用样品溶解液(0.1%甲酸、5%乙腈)溶解, 充分振荡涡旋, 13500 r·min-1, 4 ℃离心20 min, 将上清液转移到上样管中, 进行质谱鉴定.进行3次生物组重复.
2.6 蛋白质鉴定和定量在分析蛋白质的基础上, 超过1.3倍的选择被定义为蛋白质的差异上调表达, 并且少于1.3倍的选择被定义为蛋白质的差异性下调表达, 即平均值≥1.3是上调蛋白, AVG≤0.77是下调蛋白.
使用Gene Ontology(GO)分析注释鉴定的蛋白质的功能, 并使用Kyoto Encyclopedia of Genes and Genomes(KEGG)数据库分析代谢组学途径.
3 结果与讨论(Results and discussion) 3.1 四环素诱导的实验结果 3.1.1 MIC的变化结果诱导前供试野生型大肠杆菌的MIC为2 μg·mL-1, 判定为敏感(MIC小于16 μg·mL-1判定为敏感).图 2所示, 经过6 d诱导后MIC为8 μg·mL-1, 并在接下来6 d的诱导都保持MIC为8 μg·mL-1.在第14 d, MIC为16 μg·mL-1, 处于敏感和抗性判断的临界值, 第20 d时, MIC为32 μg·mL-1, 为对四环素的抗性菌株.在低浓度四环素环境下诱导的过程中可以发现当MIC上升到8 μg·mL-1时, 较难上升, 该MIC值保持了8 d后继续上升到16 μg·mL-1, 并在此基础上再经6 d后达到32 μg·mL-1.这说明野生型大肠杆菌在受到低浓度四环素胁迫时, 开始时耐受性提高的较快, 到抗性临界点附近时保持了较长时间的耐受值, 但随着时间再次继续延长也会产生抗性.
 |
| 图 1 MIC的增长趋势 Fig. 1 Trend of MIC within 15 days |
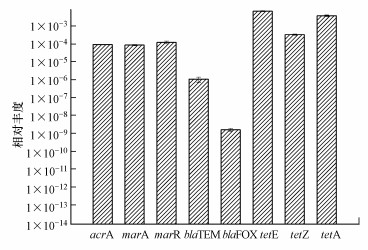 |
| 图 2 ARGs的相对丰度 Fig. 2 Abundance of ARGs |
在该研究中, 在用四环素处理后的所有样品中观察到tetA、tetE、tetZ、blaTEM、blaFOX、acrA、marA和marR, 而在野生型大肠杆菌中没有检测到它们.其中, arcA是一种双组分系统转录因子, 含有传感器结构域和DNA结合结构域(Kohanski et al., 2010).相关研究已经证实, 双组分系统是细胞死亡的共同机制中的重要元素, 并且有研究表明acrA外排泵在大肠杆菌多重耐药性性(Mar)突变体的抗生素抗性表型中起主要作用(Okusu et al., 1996).大肠杆菌的Mar突变体对多种抗生素具有抗性, 其增加的主动外排是导致某些药物抗性的原因, 而marA是参与Mar表型的基因的转录激活因子, marR是操纵子的抑制因子, 主要参与抗生素抗性和氧化应激基因的激活.还有2个外排泵抗性基因tetA, tetE和一个核糖体保护抗性基因tetZ被检测到(Stone et al., 2016).另外β-内酰胺抗性基因blaTEM属于A类β-内酰胺酶家族, blaFOX属于C型β-内酰胺酶家族.值得注意的是, 没有在处理后的样品中检索到tetB.得到的ARGs的表达如图 2所示.在这8种基因中, tetE的丰度最高, 为6.0×10-3 copies/16S rRNA, 其次是tetA, 超过3.0×10-3 copies/16S rRNA, 随后依次是, tetE(3.3×10-4), marR(1.2×10-4), acrA(9.2×10-5), marA(8.6×10-5), blaTEM(1×10-6), 而blaFOX相对丰度最低, 仅有1.58×10-9 copies/16S rRNA.发现其中的外排泵类基因(tetA、tetE)的相对丰度高于核糖体保护蛋白类基因(tetE).多重耐药性基因丰度也高于两个β-内酰胺类抗性基因, 这不仅说明野生型大肠杆菌在四环素胁迫下能够诱导产生四环素类抗性基因, 还能诱导产生多重耐药性基因和β-内酰胺类抗性基因.
3.2 iTRAQ鉴定的蛋白信息本次研究, 通过ProteinPilot软件鉴定了1857种独特蛋白质, 其置信水平为> 95%(未使用的截止值>1.3).对大肠杆菌数据库的分析表明, 实验组(第20 d菌株)与对照处理(野生型大肠杆菌)相比, 抗生素处理后部分鉴定的蛋白质受到显著调节.为了检测不同应激物后大肠杆菌中的蛋白质变化, 比较了在大肠杆菌四环素胁迫后上调或下调的蛋白质的表达量.发现共同表达564种差异蛋白, 21种蛋白质的对照处理上调(p≤0.05, fold change>1.3), 543种蛋白质下调(p≤0.05, fold change<0.77).
3.3 差异蛋白的GO富集分析GO是1个国际标准化的基因本体功能分类体系工具, 用来全面描述生物体中基因和蛋白质的属性.GO共有3个大类, 分别描述构成的细胞组件(cell component)、分子功能(molecular function)以及参与的生物过程(biological processes)
根据UniProt和Gene Ontology数据库中列出的分子功能, 主要涉及10大功能蛋白类别包括离子结合(39%), 氧化还原酶活性(15%), 离子结合(8%), DNA结合(7%), 裂解酶活性(6%), 连接酶活性(6%), 异构酶活性(5%), 跨膜转运蛋白活性(5%), 激酶活性(5%), ATP酶活性(4%).与野生型大肠杆菌对比, 处理过的大肠杆菌关于离子结合蛋白整体是下调的, 差异蛋白主要集中于对各种离子或分子的结合能力, 如对金属离子、NAD、铁硫簇及血红素等的结合.其次是关于氧化还原酶活性蛋白整体下调, 这说明, 四环素胁迫下会抑制细胞体内的氧化还原反应.
根据数据库中列出的生物过程, 本研究中主要包括了生物合成过程(18%), 细胞氮化合物代谢过程(15%), 小分子代谢过程(15%), 对压力的反应(11%), 分解代谢过程(8%), 碳水化合物代谢过程(8%), 细胞氨基酸代谢过程(7%), 辅因子代谢过程(6%), 前体代谢产物和能量的产生(6%), 翻译(6%).在生物过程中, 生物合成, 代谢过程、能量生成中有较多差异蛋白富集, 说明四环素作用改变了细胞合成、代谢和能量产生过程.关于细胞氮化合物, 碳水化合物, 细胞氨基酸, 辅因子代谢过程的等蛋白的显著下调, 细胞减少代谢和能量的消耗, 可能类似于1种节能的过程, 同样减少了细胞内自由基, 从而保护细菌免受四环素诱导的产生细胞毒性.
在细胞组件这个大类, 主要包括了细胞溶质(43%), 细胞质(27%), 质膜(11%), 含蛋白质的复合物(10%), 细胞内(6%), 核糖体(3%).菌体在四环素作用下, 其细胞质和细胞表面或膜成分可能受到影响或被破坏, 会影响细胞表面或膜结构, 促进的渗透胁迫耐受性和固有的与四环素结合的能力.
3.4 差异蛋白的生物学通路富集分析本研究通过KEGG途径进一步分析受四环素影响的途径和网络(图 4).图中很直观地可以发现, 主要分为4个大类细胞过程, 环境信息处理和遗传信息处理, 其中代谢途径的蛋白的表达变化是占比最大的, 这表明代谢在抗生素反应中的重要作用.
 |
| 图 3 差异表达蛋白的功能分类 Fig. 3 Functional classifications of differentially expressed proteins |
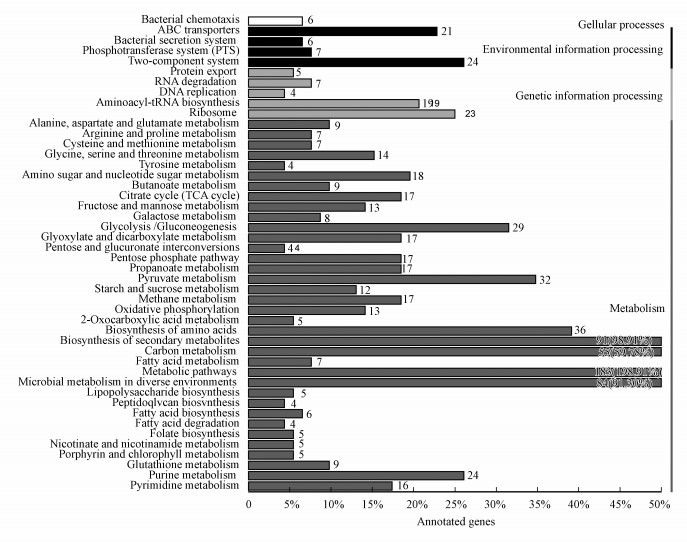 |
| 图 4 KEGG分类 Fig. 4 KEGG classification |
细菌趋药性(Bacterial chemotaxis)对于细菌通过向最高浓度的食物分子游动或逃离毒物来寻找食物(例如葡萄糖)是十分重要的(Wadhams et al., 2004).其中周质二肽转运蛋白(DppA)是可以经受渗透压休克的转运系统的二肽结合蛋白, 同样是肽趋化性所必需的蛋白(Pulvermacher et al., 2009).研究显示原核细胞都具有趋化记忆能力(Monica et al., 2014).在原核生物中, 该机制涉及甲基化受体的称为甲基接受趋化蛋白(MCP)的.这导致它们脱敏并且允许原核生物“记住”并适应化学梯度.本研究中接受甲基的趋化蛋白I (Tsr)是趋化信号转导器, 响应环境中引诱剂和驱避剂浓度的变化, 从外部向细胞内部转导信号, 并通过甲基化水平的变化促进感觉适应(Gegner et al., 1992).细菌趋药性6个蛋白的下调, 一定程度上说明通过四环素改变大肠杆菌的内源趋化能力可以降低或抑制感染.
3.4.2 转运蛋白微生物ABC转运蛋白(ABC transporters)在理解毒力和抗药性方面具有重要意义, 四环素的暴露不可避免地诱导细菌细胞中的渗透压的改变, 进而导致细胞损伤.ABC转运蛋白通常由多个亚基组成, 其中1个或2个是跨膜蛋白, 其中1个或2个是膜相关的ATP酶(Dawson et al., 2006).ATP酶亚基利用三磷酸腺苷(ATP)结合和水解的能量来激发跨膜的各种底物的转运, 用于摄取或输出底物(Hung et al., 1998).本研究中21个相关蛋白的表达与敏感菌株对比发现其表达量下调, 因为ABC转运蛋白摄取与搬运吸收大量的营养素, 生物合成前体, 微量金属和维生素, 而输出则运输脂类, 药物以及各种初级和次级代谢产物, 消耗ATP, 这表明该菌株在四环素胁迫期间的节能存活模式, 这与前文中GO分析得到的结果一致.另外, 有研究表明ABC转运蛋白也参与了多重耐药性.
3.4.3 碳水化合物代谢碳水化合物代谢(Carbon metabolism)是许多必需代谢途径的核心(Machado et al., 2015).比较处理前后菌株之间的蛋白质组学谱, 显示处理后的具有下调的55种蛋白质丰度.参与TCA循环的酶, 比如富马酸水合酶II类(FumC)和乌头酸水合酶(AcnB和AcnA)等, 糖酵解降解碳水化合物的烯醇化酶(Eno)等和丝氨酸羟甲基转移酶(GlyA)等, 这些碳基团的主要来源的蛋白在处理后的菌体中的丰度较低, 说明在四环素压力期间, 菌体减少了细胞内自由基, 从而保护细菌免受四环素诱导的细胞毒性.这种自我保护机制, 表明该菌株的节能存活模式有利于解决四环素毒性.这种能量守恒和存活可以被视为针对环境胁迫(包括抗生素的暴露)的应变的固有“平衡”生理状态.
3.4.4 脂肪酸代谢脂肪酸代谢(Fatty acid metabolism)由产生能量的分解代谢过程和产生生物学重要分子(甘油三酯、磷脂、第二信使、局部激素和酮体)的合成代谢过程组成(Douglas et al., 2003).脂肪酸在代谢中的1个作用是能量产生, 以三磷酸腺苷(ATP)的形式捕获.同时脂肪酸是磷脂的组成部分, 磷脂构成细胞的大部分质膜或细胞膜.本研究中发现7个脂肪酸代谢下调, 同样是代谢能量节约的1种, 进一步辅证了这种节能存活模式.
3.4.5 柠檬酸循环(TCA循环)几种分解代谢途径会聚在柠檬酸循环Citrate cycle (TCA cycle)上, 大多数这些反应在TCA循环中添加中间体, 因此TCA循环被称为补充反应.该循环消耗乙酸盐(以乙酰-CoA的形式)和水, 将NAD +还原为NADH, 并产生二氧化碳作为废物副产物, 由柠檬酸循环产生的NADH被送入氧化磷酸化(电子传输), 而处理后的菌体中17种关于TCA循环的蛋白下调, 说明这个途径的过程被抑制.这样, 营养物的氧化以产生ATP形式的可用化学能减少.相当于处以一种“蓄势”状态, 以增加对四环素压力耐受性.
3.4.6 嘌呤和嘧啶代谢在处理后的细胞蛋白组中观察到四环素对嘌呤代谢(Purine metabolism)和嘧啶代谢(pyrimidine metabolism)蛋白还是有一定影响, 其中, 腺苷酸琥珀酸合成酶(PurA)下调, 其在嘌呤核苷酸生物合成的从头途径中发挥重要作用, 催化IMP生物合成AMP的第一步(Sun et al., 2012).嘌呤代谢和嘧啶代谢大部分蛋白下调, 除了DNA聚合酶Ⅲ亚基α(DnaE), 一种复杂的多链酶, 负责细菌中的大多数复制合成.这些蛋白被四环素胁迫改变, 归因于其在四环素暴露后, 细胞为了维持稳定生理状态的, 对环境做出反应应力较强的细胞防御和修复系统生理状态(包括嘌呤和嘧啶代谢)的诱导.
4 结论(Conclusions)1) 野生型大肠杆菌经低浓度四环素诱导, 20 d后可由敏感转至耐药, 表明环境中的低浓度四环素的刺激对大肠杆菌耐药性的形成影响较大.
2) 大肠杆菌用四环素处理后, 检测到四环素抗性基因tetA、tetE和tetZ, β-内酰胺类抗性基因blaTEM和blaFOX, 多重耐药性基因acrA、marA和marR, 其中四环素外排泵类抗性基因(tetA和tetE)高于其他ARGs.
3) 大肠杆菌在四环素胁迫下具有特定的节能存活模式, 有利于忍耐四环素毒性, 是处于四环素暴露下的“平衡”生理状态.
4) 大肠杆菌可以通过诱导细胞防御和修复系统来应对环境压力, 这使得该菌株能够在相对稳定的生理反应的情况下暴露于四环素下.
5) 四环素胁迫下, 大肠杆菌表现出对离子传输的严格调节(下调), 并改变诱导的渗透胁迫.
6) 环境中长期存在的低剂量药物等可使细菌产生ARGs, 本研究提供了低剂量四环素胁迫下, 鉴定细胞生长和代谢的潜在分子机制, 并阐明潜在相关蛋白在该过程中的作用, 研究结果为进一步解析抗生素胁迫的抗性机制提供了参考.
Blair J M, Webber M A, Baylay A J, et al. 2015. Molecular mechanisms of antibiotic resistance[J]. Nature Reviews Microbiology, 13(1): 42–51.
DOI:10.1038/nrmicro3380
|
Bougnom B P, Piddock L J V. 2017. Wastewater for urban agriculture:A significant factor in dissemination of antibiotic resistance[J]. Environmental Science & Technology, 51(11): 5863–5864.
|
Boxall A, Long C. 2005a. Veterinary medicines and the environment[J]. Environmental Toxicology & Chemistry, 24(4): 759.
|
陈卫平, 彭程伟, 杨阳, 等. 2017. 北京市地下水中典型抗生素分布特征与潜在风险[J]. 环境科学, 2017, 38(12): 5074–5080.
|
Dawson R J, Locher K P. 2006. Structure of a bacterial multidrug ABC transporter[J]. Nature, 443(7108): 180–185.
DOI:10.1038/nature05155
|
Douglas E, Haller R G, Walton M E. 2003. Energy contribution of octanoate to intact rat brain metabolism measured by 13C nuclear magnetic resonance spectroscopy[J]. Journal of Neuroscience the Official Journal of the Society for Neuroscience, 23(13): 5928–5935.
DOI:10.1523/JNEUROSCI.23-13-05928.2003
|
Gegner J A, Graham D R, Roth A F, et al. 1992. Assembly of an MCP receptor, CheW, and kinase CheA complex in the bacterial chemotaxis signal transduction pathway[J]. Cell, 70(6): 975–982.
DOI:10.1016/0092-8674(92)90247-A
|
Guo M T, Yuan Q B, Yang J. 2015. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater[J]. Environmental Science & Technology, 49(9): 5771–5778.
|
Hung L W, Wang I X, Nikaido K, et al. 1998. Crystal structure of the ATP-binding subunit of an ABC transporter[J]. Nature, 396(6712): 703.
DOI:10.1038/25393
|
Joseph N, Pascal S. 2015. The soil resistome:a critical review on antibiotic resistance origins, ecology and dissemination potential in telluric bacteria[J]. Environmental Microbiology, 17(4): 913–930.
DOI:10.1111/emi.2015.17.issue-4
|
Juan L, Thanh W, Bing S, et al. 2012. Plasmid-mediated quinolone resistance genes and antibiotic residues in wastewater and soil adjacent to swine feedlots:potential transfer to agricultural lands[J]. Environmental Health Perspectives, 120(8): 1144–1149.
DOI:10.1289/ehp.1104776
|
Jutkina J, Marathe N P, Flach C F, et al. 2018. Antibiotics and common antibacterial biocides stimulate horizontal transfer of resistance at low concentrations[J]. Science of the Total Environment, 616(11/12): 172–178.
|
Kohanski M A, Depristo M A, Collins J J. 2010a. Sublethal antibiotic treatment leads to multidrug resistance via radical-induced mutagenesis[J]. Molecular Cell, 37(3): 311–320.
DOI:10.1016/j.molcel.2010.01.003
|
Lee K Y, Jeon S Y, Hong J W, et al. 2013. Human Health Risk Assessment (HHRA) for environmental development and transfer of antibiotic resistance[J]. Environmental Health Perspectives, 121(9): 993–1001.
DOI:10.1289/ehp.1206316
|
Machado D, Herrgård M J, Rocha I. 2015. Modeling the contribution of allosteric regulation for flux control in the central carbon metabolism of E. coli[J]. Frontiers in Bioengineering & Biotechnology, 3(1): 2–11.
|
Martínez J L. 2008. Antibiotics and antibiotic resistance genes in natural environments[J]. Science, 321(5887): 365–367.
DOI:10.1126/science.1159483
|
Mathieu A, Fleurier S, Frã Noy A, et al. 2016. Discovery and function of a general core hormetic stress response in E. coli induced by sublethal concentrations of antibiotics[J]. Cell Reports, 17(1): 46–57.
DOI:10.1016/j.celrep.2016.09.001
|
Monica S, Haicen Y, Michael E, et al. 2014. Cellular memory in eukaryotic chemotaxis[J]. Proc Natl Acad Sci U S A, 111(40): 14448–14453.
DOI:10.1073/pnas.1412197111
|
Okusu H, Ma D H. 1996. AcrAB efflux pump plays a major role in the antibiotic resistance phenotype of Escherichia coli multiple-antibiotic-resistance (Mar) mutants[J]. Journal of Bacteriology, 178(1): 306–308.
DOI:10.1128/jb.178.1.306-308.1996
|
Olsen I. 2015. Biofilm-specific antibiotic tolerance and resistance[J]. European Journal of Clinical Microbiology & Infectious Diseases, 34(5): 877–886.
|
Pulvermacher S C, Stauffer L T, Stauffer G V. 2009. Role of the Escherichia coli Hfq protein in GcvB regulation of oppA and dppA mRNAs[J]. Microbiology, 155(Pt 1): 115–123.
|
Qiao M, Ying G G, Singer A C, et al. 2018. Review of antibiotic resistance in China and its environment[J]. Environment International, 110: 160–172.
DOI:10.1016/j.envint.2017.10.016
|
Stone L K, Baym M, Lieberman T D, et al. 2016. Compounds that select against the tetracycline-resistance efflux pump[J]. Nature Chemical Biology, 12(11): 902–904.
DOI:10.1038/nchembio.2176
|
孙秋根, 王智源, 董建玮, 等. 2018. 太湖流域河网4种典型抗生素的时空分布和风险评价[J]. 环境科学学报, 2018, 38(11): 4400–4410.
|
Sun Y, Fukamachi T, Saito H, et al. 2012. Respiration and the F Fo-ATPase enhance survival under acidic conditions in Escherichia coli[J]. Plos One, 7(12): 52577.
DOI:10.1371/journal.pone.0052577
|
Wadhams G H, Armitage J P. 2004. Making sense of it all:bacterial chemotaxis[J]. Nat Rev Mol Cell Biol, 5(12): 1024–1037.
DOI:10.1038/nrm1524
|
张宁, 李淼, 刘翔. 2018. 土壤中抗生素抗性基因的分布及迁移转化[J]. 中国环境科学, 2018, 38(7): 2609–2617.
DOI:10.3969/j.issn.1000-6923.2018.07.029 |
Zhu Y G, Zhao Y, Li B, et al. 2017. Continental-scale pollution of estuaries with antibiotic resistance genes[J]. Nature Microbiology, 2: 16270.
DOI:10.1038/nmicrobiol.2016.270
|
庄榆佳, 赵忆, 苏建强, 等. 2017. 抗生素抗性基因在养殖废水中的分布与去除[J]. 环境化学, 2017, 36(11): 2311–2318.
DOI:10.7524/j.issn.0254-6108.2017041801 |
邹高龙, 刘志文, 董洁平, 等. 2018. 环丙沙星在污水处理过程中的迁移转化以及对污水生物处理的影响[J]. 环境科学学报, 2018, 39(2): 308–317.
|
 2019, Vol. 39
2019, Vol. 39


