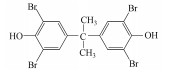
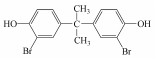
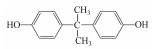
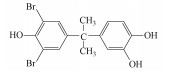
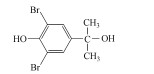
在过去的几十年中, 四溴双酚A(Tetrabromobisphenol A, TBBPA)作为典型的反应型或添加型阻燃剂被大量生产并广泛应用于纸张、纺织品、塑料制品, 特别是电子产品中(Lyche et al., 2015).其中, 2013年TBBPA全球需求量超过2×105 t(Li et al., 2015).大量使用的TBBPA经过一系列的迁移释放到环境中(Lilienthal et al., 2008), 在多种环境介质, 如大气、土壤、沉积物和地表水中均能检测到TBBPA的存在(肖潇等, 2012; Liu et al., 2016).另外, 在人体血液中也有检测到TBBPA(Barghi et al., 2017).值得注意的是, TBBPA具有内分泌干扰毒性、神经毒性、免疫毒性和生殖毒性(Pullen et al., 2003; Kuiper et al., 2007; Guyot et al., 2014; Wojtowicz et al., 2014), 对人类和有机体具有潜在危害, 甚至可能产生致癌效应(Abdallah, 2016).
由于TBBPA具有疏水性和持久性等特点, 环境中TBBPA的去除比较困难.近年来, 关于环境中TBBPA的去除研究越来越多, 常用的处理方法有化学氧化法(Bao et al., 2015; Ji et al., 2016)、化学还原法(Li et al., 2016)、吸附法(Shen et al., 2018)和微生物降解法(Ren et al., 2016).其中, 微生物降解可将复杂的有机物转化为简单的有机物, 操作简便且一般不会对环境造成二次污染, 被认为是去除环境中污染物的主要途径(Stiborova et al., 2015).Xu等(2018)研究了在好氧条件下红球菌(Rhodococcusjostii RHA1)降解14C标记的TBBPA的效果, 结果表明, TBBPA在5 d内首先快速转化为甲基醚-TBBPA, 然后转化为疏水性更高但毒性更低的二甲醚-TBBPA, TBBPA的半衰期为0.32 d, 其中0.6%的TBBPA完全矿化.已有研究表明, TBBPA可以被多种微生物好氧降解, 如赤红球菌(Rhodococcus ruber)(Chang et al., 2012)、短小芽孢杆菌(Bacillus pumilus)(Chang et al., 2012)、链球菌(Streptococcus sp.)(Peng et al., 2014)、假单胞菌(Pseudomonas sp.)(Peng et al., 2014; Gu et al., 2016; Ma et al., 2017)、红球菌(Rhodococcus jostii)(钱艳园等, 2012; Xu et al., 2018)、苍白杆菌(Ochrobactrum sp.)(An et al., 2011)和节杆菌(Arthrobacter sp.)(Ren et al., 2016)等.尽管目前有很多研究致力于TBBPA的微生物降解, 并提出了TBBPA的模拟降解途径, 但系统分析TBBPA微生物降解过程中涉及的功能酶及其具体作用机制的研究却鲜有报道.
本研究在电子废物拆解区域的沉积物中, 以TBBPA为唯一碳源, 筛选得到一株可耐受并具有一定TBBPA降解活性的菌株, 研究该菌株对TBBPA的降解特性, 并模拟其降解途径.此外, 结合Kyoto Encyclopedia of Genes and Gnomes(KEGG)等数据库对降解菌株降解TBBPA的代谢途径、功能酶和编码基因等进行对比分析, 进而探究其降解机理.以期为好氧微生物降解TBBPA的机理和功能机制等研究提供参考.
2 材料与方法(Materials and methods) 2.1 样品采集采集台州地区电子废物拆解区表层沉积物样品, 采样深度为0~5 cm, 将采集的样品(原始样品一般约为1 kg)去除石块等杂质后, 置于4 ℃冰箱中低温保存, 备用.
2.2 培养基与储备液的配制无机盐培养基(MSM)(g·L-1):KH2PO4 1.5, K2HPO4 2.3, (NH4)2SO4 1, NaNO3 0.5, MgCl2·6H2O 0.008, MnSO4·H2O 0.002, FeSO4·7H2O 0.002, CaCl2 0.002, ZnSO4·7H2O 0.002, Na2MoO4·2H2O 0.001, H3BO3 0.0004, pH=7.0.
LB培养基:蛋白胨10 g, 酵母粉5 g, 氯化钠10 g, 去离子水定容至1000 mL, pH=6.2.
TBBPA储备液:称取0.1 g TBBPA, 加入0.1% NaOH使其溶解, 转移到100 mL棕色容量瓶中, 定容, 配制成浓度为1000 mg·L-1的储备液, pH=12.0.
2.3 降解菌株的筛选和鉴定将5 g沉积物样品加入到含有1 mg·L-1 TBBPA的MSM中, 置于恒温振荡箱中培养10 d.除非另有说明, 本实验均在30 ℃、150 r·min-1条件下避光进行.培养液10 d转移一次, 对应MSM中TBBPA的浓度从1 mg·L-1逐渐增加5、10、20 mg·L-1, 驯化时间为40 d, 取最后一个培养周期的菌液用于降解菌株的分离纯化.
对该菌株进行16S rDNA序列测定(上海睿迪生物科技有限公司), 测序结果在NCBI数据库中进行BLASTn比对, 选取从NCBI GenBank中获得的同源性较高的序列, 通过MEGA 5.1软件将其进行比对, 采用邻位相连法(Neighbor-joining)构建降解菌株的系统发育树.
2.4 TBBPA的好氧降解挑取适量纯化菌株转接入LB培养基中, 放入恒温振荡箱中培养24 h后, 将此菌液经8000 r·min-1离心8 min, 弃上清液, 加入无菌生理盐水洗涤2次后配成一定浓度的菌悬液(OD600=1.0).将10%的菌悬液接种到添加一定浓度TBBPA的MSM中, 置于恒温振荡箱中培养.反应一定时间后取样, 用高效液相色谱(HPLC)定量分析TBBPA浓度.每组设置3个平行, 以未接种菌悬液的体系作为对照组.取样前用1%的NaOH调节使体系的pH为9, 在150 r·min-1的恒温振荡箱中振荡30 min使溶液中的TBBPA完全溶解并均匀分布.
2.5 降解菌株功能酶的注释根据菌株TP-1的16S rDNA测序结果进行同源性分析, 选择与其同源性最高的同种属菌株的全基因组序列进行KEGG功能注释, 基于注释结果将相关功能酶与检测到的TBBPA代谢中间产物进行匹配, 构建该菌株代谢TBBPA的代谢通路图, 进而分析菌株TP-1对TBBPA的降解机制.然后, 在NCBI数据库中, 将降解菌株功能酶的蛋白序列进行BLASTp比对, 下载相似度较高的14条序列, 通过MEGA 5.1软件构建系统发育树, 分析功能酶蛋白的同源性, 进一步分析该菌株降解TBBPA的功能酶特性.此外, 采用SyntTax数据库分析该菌株降解TBBPA的功能酶蛋白的编码基因, 分析其基因簇同线性, 得到可靠的基因同线性关系和功能酶的分类.
2.6 TBBPA分析方法 2.6.1 定量分析本研究采用HPLC(Shimadzu LC-20AT)测定TBBPA浓度.色谱条件:色谱柱为C18反相柱(5 μm × 4.6 mm × 250 mm), 流动相为甲醇:水=80:20(V:V), 流速为1 mL·min-1, 进样量为10 μL, 检测波长为220 nm, 测定时间为10 min.
2.6.2 定性分析样品预处理:于反应24 h后整瓶取样, 加入10 mL二氯甲烷振荡2 min, 待分层后, 将下层液体转移至圆底烧瓶中.重复上述操作3次, 将收集的液体旋转蒸发至干(温度为42 ℃), 用甲醇定容.经0.22 μm滤膜过滤后保存至棕色样品瓶中, 用高效液相色谱-质谱联用仪(HPLC-MS)(TSQ QUANTUM ULTRA)对TBBPA降解产物进行定性分析.
HPLC-MS质谱条件:电喷雾电离, 喷雾电压3500 V, 雾化温度0 ℃, 毛细管温度270 ℃, 质谱扫描范围(m/z)100~600.
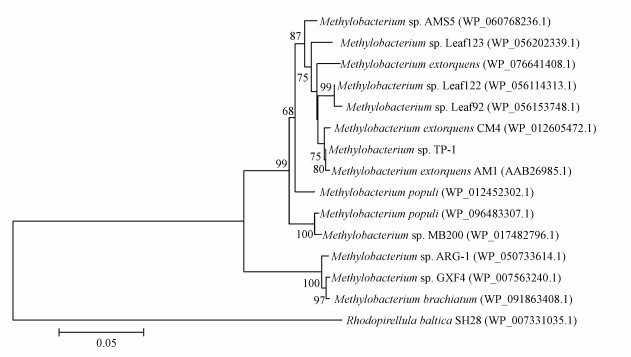
3 结果与讨论(Results and discussion) 3.1 降解菌株的鉴定通过一系列的驯化和分离过程, 从沉积物中筛选得到一株TBBPA降解菌株, 命名为TP-1.将菌株TP-1的16S rDNA序列提交到NCBI数据库中进行BLASTn比对, 选取相似度最高的15条已命名纯培养菌株的16S rRNA基因序列构建系统发育树, 分析其同源进化关系(图 1).菌株TP-1与甲基杆菌属菌株Methylobacterium extorquens、Methylobacterium zatmani和Methylobacterium pseudosasae等聚类于同一支, 且节点值较高, 因此, 认定该菌株属于甲基杆菌属(Methylobacterium sp.).甲基杆菌属在降解环境中的有机污染物和生物修复过程中起到重要作用, 如Srivastva等(2017)的研究表明, 菌株Methylobacterium sp. PV1可以降解各种碳氢化合物, 包括支链烯烃.此外, 已有研究证实甲基杆菌属菌株Methylobacterium rhodesianum可以脱卤降解卤代化合物二氯甲烷(刘洪霞等, 2013; Yu et al., 2017).
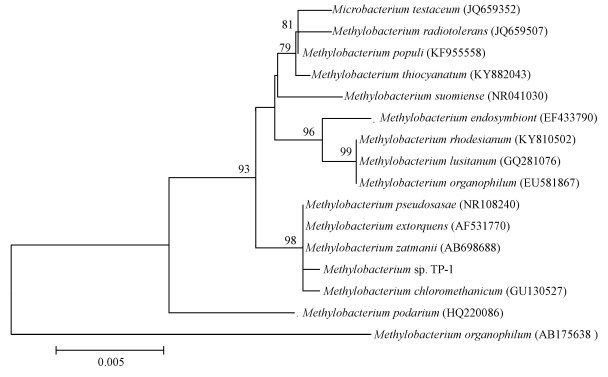 |
| 图 1 菌株TP-1 16S rDNA系统发育树 Fig. 1 Phylogenetic tree of strain TP-1 based on 16S rDNA sequence analysis |
底物初始浓度是影响微生物降解的重要因素.实验设置不同的TBBPA初始浓度, 考查其对降解率的影响(图 2).结果表明, 60 h后, 菌株TP-1对初始浓度为1、5、10、15和20 mg·L-1的TBBPA的降解率分别为12.19%、54.32%、74.87%、49.33%和30.12%.与其他体系相比, TBBPA初始浓度为10 mg·L-1时, 菌株TP-1表现出最高的降解率(74.87%), 因此, 在之后的微生物降解实验中TBBPA的初始浓度固定为10 mg·L-1.此外, 菌株TP-1对初始浓度为1 mg·L-1的TBBPA的降解率明显低于其他浓度下的降解率, 过低的底物浓度可能无法为菌株的生长提供足够的碳源(Peng et al., 2014);而在TBBPA浓度为20 mg·L-1的MSM中, TBBPA降解率降低至30.12%, 表明该菌株对于TBBPA的利用效率有限, 且高浓度的TBBPA可能对菌株TP-1具有一定的毒性作用(Debenest et al., 2010; Zhang et al., 2014).
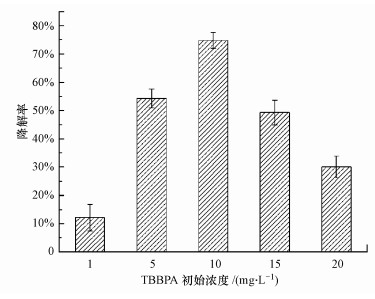 |
| 图 2 TBBPA初始浓度对菌株TP-1降解率的影响 Fig. 2 Effects of initial TBBPA concentration on degradation efficiency by TP-1 |
pH值对TBBPA在水溶液中的溶解度和微生物降解特性均有影响.本文针对不同pH值条件下菌株TP-1降解TBBPA的特性进行研究(图 3), 发现pH=7体系中, TBBPA在36 h内迅速降解, 60 h后降解率达74.87%;而pH=5体系中, 菌株TP-1对TBBPA的降解率较低, 为63.92%;pH=9体系中, 降解率降低至58.27%.由此表明, 在碱性和酸性条件下, 菌株TP-1对TBBPA降解率明显降低.此外, 反应前24 h内, pH为9时降解率降低可能还与TBBPA的溶解度有关, 此时溶液中的TBBPA基本处于溶解状态, 而悬浮态的有机物更容易吸附于微生物表面进而被微生物利用.此前的研究表明, pH会对蛋白质的功能产生影响, 过低或过高的pH都会降低酶的活性, 不利于降解酶功能的表达(史延华等, 2015).
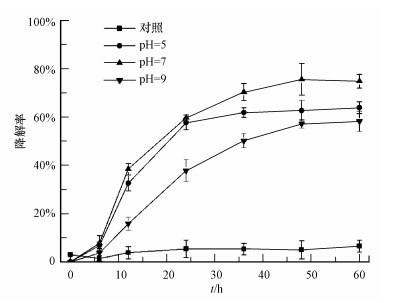 |
| 图 3 初始pH对TBBPA降解效果的影响 Fig. 3 Effects of initial pH on TBBPA degradation efficiency |
为进一步了解TBBPA在环境介质中的归趋和可能造成的生态危害, 本研究采用HPLC-MS检测了菌株TP-1降解TBBPA的中间产物, 并据此推测了降解途径和机理.根据检测结果, 菌株TP-1降解TBBPA可能的中间产物主要有6种(表 1).
| 表 1 基于HPLC-MS推测的中间产物 Table 1 The proposed intermediates based on HPLC-MS |
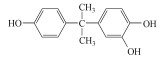
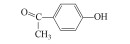
基于HPLC-MS检测到的中间产物, 提出菌株TP-1降解TBBPA的模拟途径(图 4), 其中, 双酚A(编号C)是TBBPA降解的典型脱溴产物(Peng et al., 2014), 同分异构体(编号E)是TBBPA通过β断裂产生的中间产物, 而4-对羟基苯乙酮(编号G)为还原脱溴后进一步被菌株TP-1氧化的产物(An et al., 2011).因此, 基于上述3种中间产物, 推测TBBPA的降解方式为β断裂、还原脱溴和氧化.首先, TBBPA被氧化失去一个电子(An et al., 2011; Feng et al., 2013), 虽然未检测到TBBPA的氧化形式, 但在质谱中发现其断裂产物E.两个自由基中间体由β断裂反应在TBBPA的一个苯环和异丙基之间裂解产生, 然后转化为同分异构体E.同时, 也检测到TBBPA的脱溴产物, 包括二溴双酚A(B)和双酚A(C).此外, 双酚A(C)可进一步被氧化为产物G(An et al., 2011).
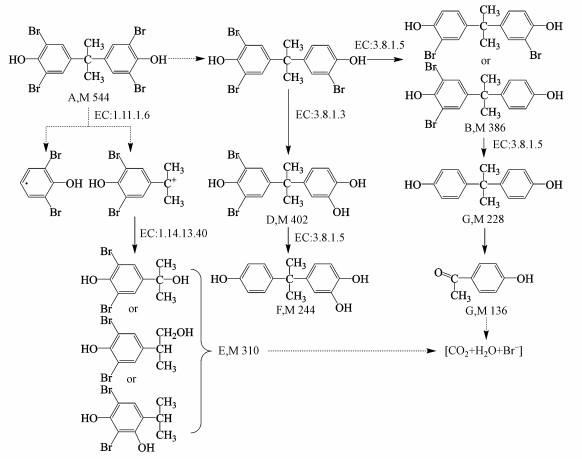 |
| 图 4 功能酶注释的TBBPA模拟降解途径(图中虚线箭头表示未检测到的产物) Fig. 4 The proposed degradation pathway of TBBPA by enzymes annotation |
微生物降解有机物的过程中涉及到多种酶催化反应(Abbasian et al., 2015).KEGG pathway数据库有机物参考代谢通路分析和KEGG注释结果表明, 菌株TP-1含有多种降解有机物的功能酶, 包括过氧化氢酶、脱卤酶和单加氧酶等.结合菌株TP-1降解TBBPA的模拟途径推测其参与TBBPA降解的功能酶包括过氧化氢酶(EC:1.11.1.6), 可以催化酚类底物的氧化反应(Lu et al., 2017);卤代烷脱卤酶(EC:3.8.1.5), 可对TBBPA进行还原脱溴作用(Pang et al., 2014);卤乙酸脱卤酶(EC:3.8.1.3), 通过水分子直接攻击底物, 取代卤素原子(Kurihara et al., 2008);单加氧酶(EC:1.14.13.40), 以氧气作为氧化剂氧化有机物(Patel et al., 1982).菌株TP-1功能酶注释的TBBPA降解途径如图 4所示, TBBPA在过氧化氢酶的作用下, 其中一个苯环和异丙基之间裂解产生自由基中间体(Feng et al., 2017), 然后在单加氧酶的作用下转化为产物E.此外, TBBPA被还原为三溴双酚A(未检测到), 然后在卤代烷脱卤酶的催化作用下C—Br键断裂, 进一步脱溴还原为二溴双酚A(B)和双酚A(C), 而卤乙酸脱卤酶通过水解脱卤作用将其转化为产物D, 在卤代烷脱卤酶的作用下生成产物F.由此表明, 菌株TP-1可以通过脱卤酶和氧化还原酶等多种功能酶的作用, 对TBBPA进行还原脱溴和氧化矿化.
3.5 降解功能酶分析已有研究表明, 同源性高的蛋白一般会具有相似的生化功能, 因此, 可以通过同源性较高的蛋白质功能来推测未知蛋白在分解代谢有机物过程中的具体作用, 这对于解释未知蛋白质的功能具有重要意义(Hegyi et al., 1999).菌株TP-1过氧化氢酶的蛋白序列系统发育树见图 5.比对分析结果表明, 过氧化氢酶序列与菌株Methylobacterium extorquens AM1(AAB26985.1)的同源性最高, 相似度为80%, 推测其在功能上也具有相似性.且甲基杆菌属菌株Methylobacterium jeotgali和Methylobacterium indicum中均含有同源性较高的过氧化氢酶(Aslam et al., 2007; Chaudhry et al., 2016).在自然环境中, 氧化还原酶被认为是可以高效去除废水中微量污染物的有前景的工具(Feng et al., 2013), 其中包括过氧化物酶, 它能够催化酚类底物的氧化偶联反应(Lu et al., 2017).因为有机碳氢化合物在细胞内的代谢是一个氧化过程, 而氧的激活和结合是加氧酶和过氧化物酶催化的酶促反应(Peixoto et al., 2011; Abbasian et al., 2015).此外, 根据Feng等(2017)的研究, 过氧化物酶可以有效降解TBBPA.
 |
| 图 5 过氧化氢酶蛋白序列系统发育树 Fig. 5 Phylogenetic tree based on the based on catalase amino acid sequence |
此外, 若某一微生物种属内有多个连续蛋白编码基因广泛存在, 则其更多的来源于遗传, 说明该编码基因在该种属的菌株中是保守的(Gaur et al., 2015; 刘莉莉等, 2017).将降解菌株TP-1的过氧化氢酶编码基因序列与SyntTax数据库中同种属菌株序列进行比对, 分析功能酶的基因簇同线性, 结果如图 6所示.菌株TP-1的过氧化氢酶与Methylobacterium extorquens AM1、Methylobacterium extorquens PA1、Methylobacterium extorquens CM4、Methylobacterium extorquens PSBB040、Methylobacterium extorquens DM4、Methylobacterium zatmanii PSBB041和Methylobacterium extorquens AMS5的基因簇的同线性较好, 并且在这些菌株中均有编码过氧化氢酶(Catalase)基因的存在, 表明过氧化氢酶在甲基杆菌属中普遍存在, 即来源于遗传而非偶然获得(Gaur et al., 2015), 它们可以通过氧化还原作用降解TBBPA.
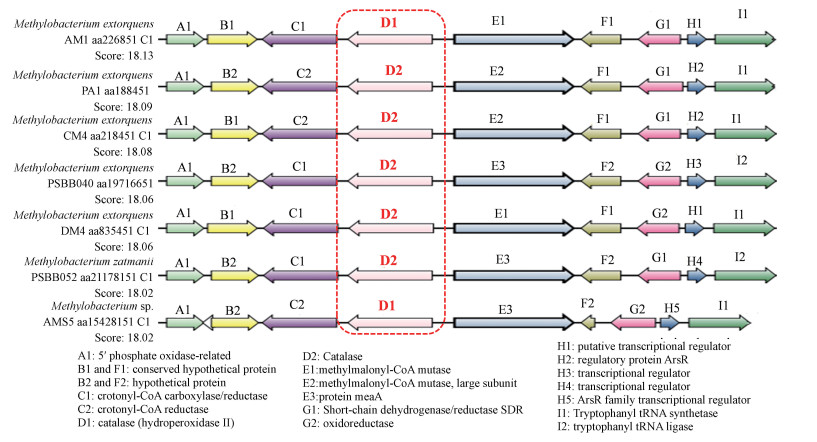 |
| 图 6 过氧化氢酶基因在不同甲基杆菌属间的比较 Fig. 6 Comparison of the catalase gene in different Methylobacterium sp. |
对菌株TP-1降解TBBPA的其他功能酶卤代烷脱卤酶(EC:3.8.1.5)、卤乙酸脱卤酶(EC: 3.8.1.3)和单加氧酶(EC: 1.14.13.40)的同源性和编码基因分析表明, 这3种酶在其他甲基杆菌属菌株中均具有较高的同源性酶, 且其基因簇与甲基杆菌属菌株的相应基因簇均具有较高的同线性, 推测这些功能酶在甲基杆菌属菌株中是保守的, 即其他甲基杆菌也有类似的TBBPA降解活性.
4 结论(Conclusions)1) 筛选得到一株TBBPA降解菌株TP-1, 通过16S rDNA序列分析鉴定该菌株属于甲基杆菌属(Methylobacterium sp.).
2) 在pH=7的条件下反应60 h, 菌株TP-1对初始浓度为10 mg·L-1的TBBPA的降解率为74.87%.菌株TP-1降解TBBPA的产物为二溴双酚A、双酚A、4-对羟基苯乙酮和低溴化芳香族化合物, 其降解方式主要为β断裂、还原脱溴和氧化.
3) 基于KEGG pathway数据库参考代谢通路和KEGG注释结果发现, 菌株TP-1含有过氧化氢酶和脱卤酶等多种功能酶, 结合TBBPA的模拟降解途径推测过氧化氢酶、卤代烷脱卤酶、卤乙酸脱卤酶和单加氧酶为菌株TP-1降解TBBPA的关键酶.
4) 对菌株TP-1降解TBBPA的功能酶过氧化氢酶、卤代烷脱卤酶、卤乙酸脱卤酶和单加氧酶的同源性和编码基因分析表明, 这4种酶与甲基杆菌属菌株的相应功能酶均具有较高的同源性, 且其基因簇在甲基杆菌属菌株内均具有较高的同线性, 推测其他甲基杆菌也有类似的TBBPA降解活性.
Abbasian F, Lockington R, Mallavarapu M, et al. 2015. A comprehensive review of aliphatic hydrocarbon biodegradation by bacteria[J]. Applied Biochemistry and Biotechnology, 176(3): 670–699.
DOI:10.1007/s12010-015-1603-5
|
Abdallah M A E. 2016. Environmental occurrence, analysis and human exposure to the flame retardant tetrabromobisphenol-A (TBBPA)-A review[J]. Environment International, 94: 235–250.
DOI:10.1016/j.envint.2016.05.026
|
An T, Zu L, Li G, et al. 2011. One-step process for debromination and aerobic mineralization of tetrabromobisphenol-A by a novel Ochrobactrum sp.T isolated from an e-waste recycling site[J]. Bioresource Technology, 102(19): 9148–9154.
DOI:10.1016/j.biortech.2011.06.080
|
Aslam Z, Lee C S, Kim K H, et al.2007.Methylobacterium jeotgali sp.nov., a non-pigmented, facultatively methylotrophic bacterium isolated from jeotgal, a traditional Korean fermented seafood[J].International Journal of Systematic and Evolutionary Microbiology, 57(3): 566-571
|
Bao Y, Niu J. 2015. Photochemical transformation of tetrabromobisphenol A under simulated sunlight irradiation:Kinetics, mechanism and influencing factors[J]. Chemosphere, 134: 550–556.
DOI:10.1016/j.chemosphere.2014.12.016
|
Barghi M, Shin E S, Kim J C, et al. 2017. Human exposure to HBCD and TBBPA via indoor dust in Korea:Estimation of external exposure and body burden[J]. Science of the Total Environment, 593.
|
Chang B V, Yuan S Y, Ren Y L. 2012. Aerobic degradation of tetrabromobisphenol-A by microbes in river sediment[J]. Chemosphere, 87(5): 535–541.
DOI:10.1016/j.chemosphere.2011.12.057
|
Chaudhry V, Baindara P, Pal V K, et al. 2016. Methylobacterium indicum sp.nov., a facultative methylotrophic bacterium isolated from rice seed[J]. Systematic and Applied Microbiology, 39(1): 25–32.
DOI:10.1016/j.syapm.2015.12.006
|
Debenest T, Gagné F, Petit A N, et al. 2010. Ecotoxicity of a brominated flame retardant (tetrabromobisphenol A) and its derivatives to aquatic organisms[J]. Comparative Biochemistry and Physiology Part C:Toxicology and Pharmacology, 152(4): 407–412.
DOI:10.1016/j.cbpc.2010.06.009
|
Feng Y, Colosi L M, Gao S, et al. 2013. Transformation and removal of tetrabromobisphenol A from water in the presence of natural organic matter via laccase-catalyzed reactions:reaction rates, products, and pathways[J]. Environmental Science and Technology, 47(2): 1001–1008.
DOI:10.1021/es302680c
|
Feng Y, Lu K, Gao S, et al. 2017. The fate and transformation of tetrabromobisphenol A in natural waters, mediated by oxidoreductase enzymes[J]. Environmental Science Processes and Impacts, 19(4): 596–604.
DOI:10.1039/C6EM00703A
|
Gaur R, Jeena G, Shah N, et al. 2015. High density linkage mapping of genomic and transcriptomic SNPs for synteny analysis and anchoring the genome sequence of chickpea[J]. Scientific Reports, 5: 1–11.
DOI:10.9734/JSRR
|
Gu C, Wang J, Liu S, et al. 2016. Biogenic Fenton-like reaction involvement in cometabolic degradation of tetrabromobisphenol A by Pseudomonas sp.fz[J]. Environmental Science and Technology, 50(18): 9981–9989.
DOI:10.1021/acs.est.6b02116
|
Guyot R, Chatonnet F, Gillet B, et al. 2014. Toxicogenomic analysis of the ability of brominated flame retardants TBBPA and BDE-209 to disrupt thyroid hormone signaling in neural cells[J]. Toxicology, 325: 125–132.
DOI:10.1016/j.tox.2014.08.007
|
Hegyi H, Gerstein M. 1999. The relationship between protein structure and function:a comprehensive survey with application to the yeast genome[J]. Journal of Molecular Biology, 288(1): 147–164.
DOI:10.1006/jmbi.1999.2661
|
Ji Y, Kong D, Lu J, et al. 2016. Cobalt catalyzed peroxymonosulfate oxidation of tetrabromobisphenol A:Kinetics, reaction pathways, and formation of brominated by-products[J]. Journal of Hazardous Materials, 313: 229–237.
DOI:10.1016/j.jhazmat.2016.04.033
|
Kuiper R V, Brandhof E J V D, Leonards P E G, et al. 2007. Toxicity of tetrabromobisphenol A (TBBPA) in zebrafish (Danio rerio) in a partial life-cycle test[J]. Archives of Toxicology, 81(1): 1–9.
DOI:10.1007/s00204-006-0117-x
|
Kurihara T, Esaki N. 2008. Bacterial hydrolytic dehalogenases and related enzymes:occurrences, reaction mechanisms, and applications[J]. The Chemical Record, 8(2): 67–74.
DOI:10.1002/(ISSN)1528-0691
|
Li F, Wang J, Jiang B, et al. 2015. Fate of tetrabromobisphenol A (TBBPA) and formation of ester- and ether-linked bound residues in an oxic sandy soil[J]. Environmental Science and Technology, 49(21): 12758–12765.
DOI:10.1021/acs.est.5b01900
|
Li Y, Li X, Xiao Y, et al. 2016. Catalytic debromination of tetrabromobisphenol A by Ni/nZVI bimetallic particles[J]. Chemical Engineering Journal, 284: 1242–1250.
DOI:10.1016/j.cej.2015.09.079
|
Lilienthal H, Verwer C M, Lt V D V, et al. 2008. Exposure to tetrabromobisphenol A (TBBPA) in Wistar rats:neurobehavioral effects in offspring from a one-generation reproduction study[J]. Toxicology, 246(1): 45–54.
DOI:10.1016/j.tox.2008.01.007
|
刘洪霞, 朱润晔, 欧阳杜娟, 等. 2013. 二氯甲烷降解菌Methylobacterium rhodesianum H13的分离鉴定及降解特性研究[J]. 环境科学, 2013, 34(9): 3613–3619.
|
Liu K, Li J, Yan S, et al. 2016. A review of status of tetrabromobisphenol A (TBBPA) in China[J]. Chemosphere, 148: 8–20.
DOI:10.1016/j.chemosphere.2016.01.023
|
刘莉莉, 郭倩倩, 陈鑫, 等. 2017. 城市污水处理厂四环素抗性菌的筛选及其抗性基因分析[J]. 环境科学学报, 2017, 37(11): 4039–4046.
|
Lu K, Hu J, Gao S, et al. 2017. Multi-walled carbon nanotubes facilitated the removal of tetrabromobisphenol a mediated by horseradish peroxidase[J]. Environmental Pollution, 231: 863–870.
DOI:10.1016/j.envpol.2017.08.093
|
Lyche J L, Rosseland C, Berge G, et al. 2015. Human health risk associated with brominated flame-retardants (BFRs)[J]. Environment International, 74: 170–180.
DOI:10.1016/j.envint.2014.09.006
|
Ma Y, Zhao Y, Wang Y, et al. 2017. Effects of Cu2+ and humic acids on degradation and fate of TBBPA in pure culture of Pseudomonas sp.strain CDT[J]. Journal of Environmental Sciences, 62: 60–67.
DOI:10.1016/j.jes.2017.07.012
|
Pang S Y, Jiang J, Gao Y, et al. 2014. Oxidation of flame retardant tetrabromobisphenol a by aqueous permanganate:reaction kinetics, brominated products, and pathways[J]. Environmental Science and Technology, 48(1): 615–623.
DOI:10.1021/es4041094
|
Patel R N, Hou C T, Laskin N, et al. 1982. Microbial oxidation of hydrocarbons:properties of a soluble methane monooxygenase from a facultative methane-utilizing organism, Methylobacterium sp.strain CRL-26[J]. Applied and Environmental Microbiology, 44(5): 1130–1137.
|
Peixoto R S, Vermelho A B, Rosado A S. 2011. Petroleum-degrading enzymes:bioremediation and new prospects[J]. Enzyme Research, 2011: 1–7.
|
Peng X, Qu X, Luo W, et al. 2014. Co-metabolic degradation of tetrabromobisphenol A by novel strains of Pseudomonas sp.and Streptococcus sp[J]. Bioresource Technology, 169: 271–276.
DOI:10.1016/j.biortech.2014.07.002
|
Pullen S, Boecker R G. 2003. The flame retardants tetrabromobisphenol A and tetrabromobisphenol A-bisallylether suppress the induction of interleukin-2 receptor alpha chain (CD25) in murine splenocytes[J]. Toxicology, 184(1): 11–22.
DOI:10.1016/S0300-483X(02)00442-0
|
钱艳园, 刘莉莉, 于晓娟, 等. 2012. 四溴双酚A好氧降解菌的筛选及其降解特性研究[J]. 环境科学, 2012, 33(11): 3962–3966.
|
Ren L, Jia Y, Ruth N, et al. 2016. Biotransformations of bisphenols mediated by a novel Arthrobacter sp.strain YC-RL1[J]. Applied Microbiology and Biotechnology, 100(4): 1967–1976.
DOI:10.1007/s00253-015-7076-1
|
Shen J, Huang G, An C, et al. 2018. Removal of tetrabromobisphenol A by adsorption on pinecone-derived activated charcoals:synchrotron FTIR, kinetics and surface functionality analyses[J]. Bioresource Technology, 247: 812–820.
DOI:10.1016/j.biortech.2017.09.177
|
史延华, 任磊, 贾阳, 等. 2015. 施氏假单胞菌YC-YH1的萘降解特性及产物分析[J]. 微生物学通报, 2015, 10(42): 1866–1876.
|
Srivastva N, Vishwakarma P, Bhardwaj Y, et al. 2017. Kinetic and molecular analyses reveal isoprene degradation potential of Methylobacterium sp[J]. Bioresource Technology, 242: 87–91.
DOI:10.1016/j.biortech.2017.02.002
|
Stiborova H, Vrkoslavova J, Lovecka P, et al. 2015. Aerobic biodegradation of selected polybrominated diphenyl ethers (PBDEs) in wastewater sewage sludge[J]. Chemosphere, 118: 315–321.
DOI:10.1016/j.chemosphere.2014.09.048
|
Wojtowicz A K, Szychowski K A, Kajta M. 2014. PPAR-γ agonist GW1929 but not antagonist GW9662 reduces TBBPA-induced neurotoxicity in primary neocortical cells[J]. Neurotoxicity Research, 25(3): 311–322.
DOI:10.1007/s12640-013-9434-z
|
肖潇, 陈德翼, 梅俊, 等. 2012. 贵屿某电子垃圾拆解点附近大气颗粒物中氯代/溴代二噁英、四溴双酚A污染水平研究[J]. 环境科学学报, 2012, 32(5): 1142–1148.
|
Xu S, Wang Y F, Yang L Y, et al. 2018. Transformation of tetrabromobisphenol A by Rhodococcus jostii RHA1:Effects of heavy metals[J]. Chemosphere, 196: 206–213.
DOI:10.1016/j.chemosphere.2017.12.173
|
Yu J, Liu Q, Liu L, et al. 2017. Cloning and characterization of dichloromethane dehalogenase from Methylobacterium rhodesianum for dichloromethane degradation[J]. Bioremediation Journal, 21(2): 71–80.
DOI:10.1080/10889868.2017.1282938
|
Zhang W, Chen L, An S, et al. 2014. Toxic effects of the joint exposure of decabromodiphenyl ether (BDE209) and tetrabromobisphenol A (TBBPA) on soil microorganism and enzyme activity[J]. Environmental Toxicology and Pharmacology, 38(2): 586–594.
DOI:10.1016/j.etap.2014.08.015
|
 2018, Vol. 38
2018, Vol. 38