2. 苏州科技大学环境科学与工程学院, 苏州 215009
2. School of Environmental Science and Engineering, Suzhou University of Science and Technology, Suzhou 215009
近年来, 驯化和富集培养活性污泥微生物进行脱氮除磷是污水处理工艺的研究热点(Ye et al., 2011; Limpiyakorn et al., 2011; Mußmann et al., 2011).随着分子生物技术的发展, 很多研究者通过实时荧光定量PCR技术对活性污泥细菌的种类和数量进行准确的定性和定量(赵大勇等, 2010).实时荧光定量PCR准确性的前提是活性污泥中的微生物尽可能的被抽提完全, 模板的质量较好, 纯度较高, 定量PCR时抑制效应尽可能降到最低(Wang et al., 2017; Bachoon et al., 2001).污水处理的活性污泥样品成分复杂(尹军等, 2012), Wilen等(2003)在研究活性污泥主要化学成分时发现, 腐殖酸是除蛋白质之外所占比重最大的高分子化合物;佟娟等(2009)在剩余污泥碱性发酵液中也发现了腐殖酸的存在, 并且李欢等(2009)研究了采用碱破解方法提取污泥中腐殖酸的方法.腐殖酸广泛地存在于天然水体和污泥中(王文东等, 2016), 其中, 大量的腐殖酸在DNA抽提时较难去除(Dong et al., 2006), 基因组中残留的腐殖酸会抑制酶的活性, 从而抑制PCR和实时荧光定量PCR(Margit et al., 2013; Davorka et al., 2005), 降低实时荧光定量PCR的准确性, 影响对活性污泥样品中微生物数量丰度或功能丰度的检测及对污水处理工艺中微生物所扮演角色的意义的准确解析(Didier et al., 2010; 王行等, 2016).
已有的研究表明, 稀释模板、去除腐殖酸都可以降低腐殖酸对定量PCR的抑制效应(Wang et al., 2017; Davorka et al., 2005).目前主要的腐殖酸去除方法有两大类, 一类是在基因组抽提前去除土壤中的腐殖酸, 另一类是在基因组抽提后去除基因组中残留的腐殖酸(Zipper et al., 2003).活性污泥本质上是人工驯化培养的含有功能微生物的土壤, 腐殖酸的含量较高(Lovley, 1996; Anesio, 2005; Wilen et al., 2003), 去除活性污泥腐殖酸和梯度稀释活性污泥基因组模板对定量PCR抑制效应的影响尚未见报道, 模板稀释到何种程度对于抑制效应有明显的改善尚不明确.
基于此, 本文通过研究不同方法去除活性污泥腐殖酸的效果, 以及外加内标质粒、梯度稀释模板后定量PCR抑制效应的变化, 以阐明腐殖酸的去除对活性污泥定量PCR准确性的影响.
2 材料与方法(Materials and methods) 2.1 实验材料取厌氧氨氧化中试反应启动成功的活性污泥, 置于-80 ℃条件下保存备用.
2.2 腐殖酸的去除取上述活性污泥, 使用购自于上海生工的土壤腐殖酸清除剂, 按照说明书进行抽提前活性污泥腐殖酸的去除;使用购自于上海生工的柱式腐殖酸清除剂, 按照说明书对抽提后的基因组DNA进行腐殖酸的去除.实验共4组, 分别为抽提前腐殖酸去除组(前)、抽提后腐殖酸去除组(后)、无腐殖酸去除的对照组(对照)、抽提前后腐殖酸共同处理组(前+后).
2.3 基因组DNA的抽提在抽提DNA前为了防止活性污泥的异质性带来的误差, 准确称取厌氧氨氧化中试反应启动成功的1.00 g(湿重)活性污泥和1.00 g(湿重)抽提前去除腐殖酸的活性污泥于带刻度的10 mL试管中, 用PBS缓冲液(pH=7.4, 浓度为0.20 mol·L-1)定容至10 mL后, 充分混匀成土壤匀液, 混匀后确保试管底部没有固体颗粒物残留(王行等, 2016).吸取5 mL匀液于5 mL的离心管中(合0.5 g鲜土), 4 ℃、12000 r·min-1离心2 min, 倒掉上清液.离心管中的活性污泥再用PBS洗涤2次后, 用美国MP公司生产的DNA提取试剂盒(FastDNA Spin kit for soil, MP-116560)对活性污泥进行基因组的提取.
2.4 DNA酶消化基因组DNA取50 μL上述4组抽提的基因组DNA, 分别加入DNA酶Ⅰ5 μL、10×Buffer 5 μL(普洛麦格公司), 37 ℃水浴30 min后,加终止液5 μL, 65 ℃水浴10 min, 即得到4组不含DNA的腐殖酸杂质(王行等, 2016).分别为DNA酶Ⅰ消化抽提前腐殖酸去除组(前+酶)、DNA酶Ⅰ消化抽提后腐殖酸去除组(后+酶)、DNA酶Ⅰ消化无腐殖酸去除的对照组(对照+酶)、DNA酶Ⅰ消化抽提前后腐殖酸共同处理组(前+后+酶).
2.5 DNA浓度的测定及PCR反应利用核酸蛋白测定仪NanoDrop2000检测基因组的A260/230、A260/280和浓度.采用2%的琼脂糖凝胶, 95 V电泳35 min, 检测基因组抽提的完整性及质量.以上述8种处理后的基因组为模板进行PCR反应, 引物序列见表 1.0.2 mL PCR管中配置20 μL体系如下:0.2 μL TaKaRa Taq (5 U·mL-1)、2 μL 10×PCR Buffer (Mg2+ Plus)、2 μL dNTP Mixture (各2.5 mmol·L-1)、2 μL模板(10 ng·μL-1)、上、下游引物各1 μL(1 nmol·L-1)、灭菌蒸馏水加至20 μL.将上述反应液离心、混匀、再离心, 在PCR仪中进行以下循环:95 ℃预变性5 min;98 ℃变性10 s, 63 ℃退火30 s, 72 ℃延伸5 s, 30个循环;72 ℃延伸5 min;反应体系温度最后降至4 ℃, 1.5%琼脂糖凝胶电泳分析.
| 表 1 实验所用引物 Table 1 The primers of the experiment |
按照TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.4.0的操作说明对PCR产物进行胶回收(总细菌V3~V4区产物长度190 bp).回收的PCR产物根据天根pGM-T克隆试剂盒的说明进行连接、转化、涂平板、挑取阳性单克隆菌落、摇菌、抽质粒(质粒快速提取试剂盒, 天根生化科技有限公司), 送上海生工生物公司进行测序鉴定, 最终得到总细菌V3~V4区的质粒标准品.用核酸蛋白测定仪测定质粒标准品的A260/230、A260/280和浓度.根据公式(1)计算质粒标准品的起始拷贝数G(copies·μL-1).
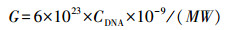
|
(1) |
式中, M为拷贝DNA分子的碱基对数(bp·copy-1);W为1个碱基对的分子质量(660 u·bp-1);CDNA为DNA浓度(ng·μL-1).
2.7 标准曲线的建立及总细菌16S rDNA V3~V4区的定量检测10倍梯度稀释10 ng·μL-1的总细菌质粒成5个浓度, 作为绝对定量PCR的模板, 建立总细菌的标准曲线.以10 ng·μL-1的4个处理组DNA及DNA酶Ⅰ消化的4个基因组DNA为模板, 进行定量PCR.根据标准曲线计算各组总细菌16S rDNA的绝对拷贝数.将浓度为10 ng·μL-1的质粒标准品作为内标质粒与2.4节中的前+酶组、对照+酶组混合后, 用灭菌的超纯水将上述混合液10倍梯度稀释成5个浓度, 作为绝对定量PCR的模板, 进行定量PCR.将10 ng·μL-1的2.2节中的前组和对照组DNA10倍梯度稀释成5个浓度, 作为绝对定量PCR的模板, 进行定量PCR.反应体系为20 μL, 反应组分为:Mix 12.5 μL, 上、下游引物各0.58 μL(1 nmol·L-1), 模板2 μL, 灭菌三蒸水9.5 μL.反应条件同常规PCR, 总延伸结束后, 加入熔解曲线的制备(95 ℃保持15 s, 60 ℃保持1 min, 95 ℃保持15 s)步骤.
2.8 绝对拷贝数及抑制效应的计算Ct值为每个反应管内的荧光信号到达设定的域值时所经历的循环数.将实时荧光定量PCR所得的Ct值作为标准曲线关系式中的Y值, 得到X值, 10X即为各组模板的绝对拷贝数, 单位为copies·g-1.腐殖酸的抑制效率I=(P-T)/P×100%, 其中, P为加入的总细菌内标质粒拷贝数(copies·g-1), T为内标质粒与腐殖酸混合组的总细菌拷贝数(copies·g-1).
2.9 数据分析所有的数据都来自于3个独立重复试验的平均值±标准差, 使用Dunnett′s检验比较各个处理组与对照组的差异, 在0.05的水平上进行数据显著性差异分析.
3 结果(Results) 3.1 基因组DNA的浓度及质量为了研究不同腐殖酸去除方法对活性污泥基因组抽提质量的影响, 表 2调查了不同去除方法和DNA酶Ⅰ消化后的模板浓度和质量, 抽提前腐殖酸去除组的A260/230值(1.34)、A260/280值(1.80)较对照组高, 与其它各组相比, 更接近于高质量基因组的标准(Krsek et al., 1999; Lyons et al., 2000).这可能与抽提后的去除法引入了一些残留的有机溶剂, 而抽提前的去除法引入的有机溶剂通过DNA提取过程得以消除有关(徐德峰等, 2013).表 2中抽提前腐殖酸去除组的DNA浓度(162.46 ng·μL-1)高于其它各组.以上结果表明, 抽提前腐殖酸去除的方法既保证了DNA的得率又提高了模板质量.
| 表 2 各基因组浓度和质量 Table 2 The concentration and quality of the genome |
对8种处理后的模板进行琼脂糖凝胶电泳, 由图 1可知, 未用DNA酶Ⅰ消化后的各组都有基因组主带, 明亮单一, 可以作为实时荧光定量PCR的模板;用DNA酶Ⅰ消化后的各组基因组被降解, 没有条带, 说明剩余杂质的主要成分为腐殖酸(王行等, 2016; Lakay et al., 2006).
 |
| 图 1 各基因组的琼脂糖凝胶电泳 (M:Maker;1:对照;2:对照+酶;3:前;4:前+酶;5:后;6:后+酶;7:前+后;8:前+后+酶) Fig. 1 The agarose gel electrophoresis of the genome |
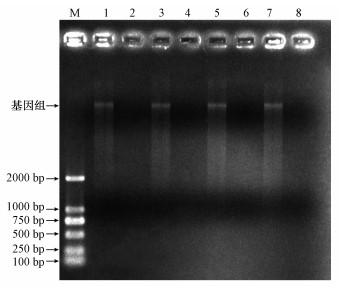
为了验证DNA酶处理后的基因组能否作为模板进行总细菌V3~V4区的PCR扩增, 以总细菌V3~V4区的引物进行PCR, 琼脂糖电泳结果如图 2所示.由图 2可知, 未经DNA酶Ⅰ处理的组1、3、5、7在190 bp处扩增出目的条带, 经过DNA酶Ⅰ处理的组2、4、6、8没有扩增出目的条带.与此同时, 以DNA酶Ⅰ消化过的4个组为模板, 进行实时荧光定量PCR, 所得的Ct值与阴性对照基本相同, 说明DNA酶Ⅰ已将各活性污泥基因组模板消化完全.
 |
| 图 2 总细菌16S rDNA V3~V4区的PCR琼脂糖凝胶电泳 (M:Maker;1:对照;2:对照+酶;3:前;4:前+酶;5:后;6:后+酶;7:前+后;8:前+后+酶) Fig. 2 The agarose gel electrophoresis of PCR of total bacteria 16S rDNA V3~V4 area |
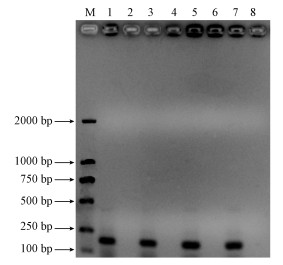
为了探究腐殖酸的去除是否影响活性污泥实时荧光定量PCR, 分别以10 ng·μL-1的抽提前腐殖酸去除组、抽提后腐殖酸去除组、抽提前后腐殖酸同时去除组、对照的基因组为模板, 进行实时荧光定量PCR, 结果见图 3.图 3中, 抽提前腐殖酸去除组的拷贝数(3.71×109 copies·g-1)最高, 抽提后腐殖酸去除组的拷贝数(3.64×109 copies·g-1)和抽提前后腐殖酸共同去除组的拷贝数(3.62×109 copies·g-1)次之, 对照组的拷贝数(2.68×109 copies·g-1)显著低于前3组(p < 0.05), 说明抽提前去除腐殖酸的扩增效率最高, 抑制效应最低.这与表 2的结果一致, 说明抽提前去除活性污泥腐殖酸的方法更为可取.
 |
| 图 3 腐殖酸去除后总细菌的绝对拷贝数 (标准曲线扩增效率90.73%, 标准曲线R2=0.988) Fig. 3 The absolute copies number of total bacteria after removal of humic acid |
为了深入研究抽提前活性污泥腐殖酸的去除及模板浓度对定量PCR扩增的影响, 以杂质腐殖酸10倍梯度稀释已知浓度的总细菌内标质粒, 定量的Ct值如图 4a所示.由图 4a可知, 随着目的基因拷贝数(GA)对数值的降低, 抽提前去除过腐殖酸的内标质粒组Ct值逐渐低于对照组, 梯度稀释模板1~100倍的Ct值差值增加, 梯度稀释模板100~10000倍的Ct值基本无变化, 说明梯度稀释去除腐殖酸的活性污泥基因组模板100倍以上(含100倍), 可提高定量PCR的扩增效率, 降低腐殖酸对活性污泥定量PCR的抑制效应.
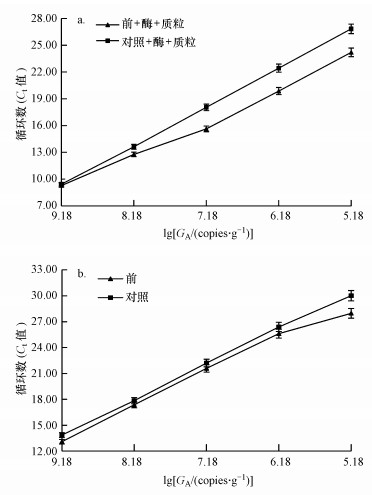 |
| 图 4 10倍梯度稀释的腐殖酸总细菌内标质粒(a)及抽提前去除组总细菌(b)的绝对拷贝数 (标准曲线扩增效率91.65%, 标准曲线R2=0.932) Fig. 4 The absolute copies number of standard plasmid of total bacteria(a) and total bacteria before genome extraction(b) by 10 times gradient dilute humic acid removal group |
为了研究梯度稀释抽提前去除腐殖酸的活性污泥总细菌定量PCR结果是否与上述结果一致, 以10倍梯度稀释抽提前去除腐殖酸的基因组为模板, 进行定量PCR(图 4b).图 4b中抽提前腐殖酸去除组1~1000倍梯度稀释模板的Ct值稍低于对照组;10000倍梯度稀释模板的Ct值较对照组有明显的降低.图 4说明, 去除腐殖酸与一定浓度的稀释模板都能降低腐殖酸对活性污泥总细菌定量PCR的抑制, 模板稀释虽然降低了腐殖酸的抑制效应, 但Ct值可能会超出可信的范围(Harms, 2003; Chang et al., 2008), 直接去除活性污泥腐殖酸比稀释活性污泥基因组模板更实用.
3.5 腐殖酸去除后的抑制效应为了进一步研究活性污泥腐殖酸的去除及活性污泥基因组模板浓度的变化对抑制效应的影响, 表 3对腐殖酸去除后的抑制效应进行了调查.表 3结果显示, 抽提前腐殖酸去除组和对照组的杂质中加入的内标质粒浓度为1.53×105 copies·g-1, 抽提前腐殖酸去除的杂质组抑制效应为6.16%,显著低于对照杂质组的72.68%(p < 0.05), 说明抽提前去除腐殖酸降低了抑制效应, 显著提高了活性污泥定量PCR的准确性.
| 表 3 腐殖酸去除后的抑制效应 Table 3 The inhibition efficiency after humic acid removal |
反应器不同运行阶段, 其含水率、pH、溶解氧、反应速率和效能不同(周健等, 2016), 活性污泥样品引入的干扰可能不同, 特别是通过影响酶活性来影响定量PCR扩增效率的杂质腐殖酸(王行等, 2016).Lindberg等(2007)发现, 湖泊沉积物中的内参基因受到腐殖酸的抑制明显.厌氧氨氧化活性污泥样品提取的DNA颜色有点发黄, 与图 3、图 4和表 3的结果一起说明活性污泥中存在腐殖酸(王行等, 2016).通常情况下, 通过DNA提取过程中的纯化步骤来减弱腐殖酸对PCR反应的抑制效应(王行等, 2016).本研究通过基因组抽提前腐殖酸去除试剂盒和梯度稀释模板100倍降低定量PCR的抑制效率, 显著提高了定量PCR的准确性.
4.2 影响活性污泥定量准确性的因素活性污泥的含水率和杂质不同, 可能会导致基因组抽提时DNA得率的差异较大, 影响定量PCR检测的准确性(Giles et al., 2012; Miller et al., 1999; Zhou et al., 1996).本研究通过抽提前3次PBS的重复清洗及3组重复抽提混合后使用, 减少样本的异质性和抽提随机性的误差, 提高定量结果的准确率.在定量PCR之前, 通过提高DNA得率的方法可以间接提高目标基因绝对定量的准确性, 然而得率的提高往往意味着DNA条带断裂的增加及样品纯度的降低(王行等, 2016).本研究通过基因组琼脂糖电泳图证明DNA抽提的完整性较好(图 1), 在腐殖酸有效去除的同时, 保证了较高的基因组得率162.46 ng·μL-1(表 2).此外, 当腐殖酸无法有效去除时, 也可通过在基因组抽提前加入特异性基因作为内参加标物计算腐殖酸的抑制效应,来评估定量PCR结果以实现准确定量(王行等, 2016).
4.3 抽提前去除腐殖酸的意义活性污泥中的细菌紧紧地粘附在泥样上, 反应器进水和出水时, 细菌随泥样一起停留在反应器中得以扩大培养.这种污泥微生物聚集体可能会导致细菌裂解不完全, 抽提的基因组低于污泥中的总细菌含量.降低细菌与污泥之间的粘附力, 使其在抽提时较好的分离, 对定量PCR准确性的提高意义重大(王行等, 2016).腐殖酸对系统内菌群种类、数量及系统的运行效能有较大影响, 可以促进污泥良好结构体的形成, 影响污泥表面性质, 抽提前去除活性污泥中的腐殖酸可能对细菌与污泥的分离具有积极意义(尹军等, 2012).抽提前去除也减少了抽提过程中或抽提后的纯化对DNA完整性的破坏.本研究中的图 1和表 2显示, 抽提前去除较抽提后去除腐殖酸, DNA的得率和质量更好.此外, 稀释基因组模板100倍以上, 基因组中腐殖酸的浓度得到了稀释, 对定量PCR的抑制效应降低, 但活性污泥抽提的基因组浓度有限, 较大倍比地稀释基因组, 定量PCR的Ct值可能会超出可信的范围, 降低定量PCR的可信度.图 4表明, 基因组抽提前去除活性污泥中的腐殖酸较稀释模板更能有效地降低腐殖酸对定量的抑制效应, 提高定量PCR的准确性.
5 结论(Conclusions)1) 基因组抽提前通过反复洗脱的方式对活性污泥中的腐殖酸进行去除, 基因组的得率(162.46 ng·μg-1)最多, A260/230值为1.34, A260/280值为1.80, 腐殖酸的残留最少, 基因组质量最高.
2) 腐殖酸对定量PCR的扩增具有抑制效应, 随着浓度的降低抑制效应先降低后保持不变.
3) 使用基因组抽提前腐殖酸去除试剂盒去除活性污泥腐殖酸, 稀释模板100倍(0.1 ng·μg-1), 定量PCR的抑制效率由72.68%降低到6.16%(p < 0.05), 显著提高了定量PCR的准确性.
Anesio A M. 2005. Effect of humic substance photo degradation on bacterial growth and respiration in lake water[J]. Applied and Environmental Microbiology, 71(10): 6267–6275.
DOI:10.1128/AEM.71.10.6267-6275.2005
|
Bachoon D S, Otero E, Hodson R E. 2001. Effects of humic substances on fluorometric DNA quantification and DNA hybridization[J]. Journal of Microbiological Methods, 47(1): 73–82.
DOI:10.1016/S0167-7012(01)00296-2
|
Chang P Y, Sarak F. 2008. Development and application of real-time PCR assays for quantifying total and aerolysin gene-containing aeromonas in source, intermediate, and finished drinking water[J]. Environmental Science & Technology, 42: 1191–1200.
|
Davorka S, Marija D G, Šimun A, et al. 2005. Taq polymerase reverses inhibition of quantitative real time polymerase chain reaction by humic acid[J]. Croatian Medical Journal, 46(4): 556–562.
|
Didier T, Claudia M C, Marielle D I, et al. 2010. Novel perspectives to purify genomic DNA from high humic acid content and contaminated soils[J]. Separation and Purification Technology, 75: 81–86.
DOI:10.1016/j.seppur.2010.07.014
|
Dong D, Yan A, Liu H, et al. 2006. Removal of humic substances from soil DNA using aluminium sulfate[J]. Journal of Microbiological Methods, 66(2): 217–222.
DOI:10.1016/j.mimet.2005.11.010
|
Giles M, Morley N, Baggs E M, et al. 2012. Soil nitrate reducing processes-drivers, mechanisms for spatial variation, and significance for nitrous oxide production[J]. Frontiers in Microbiology, 3: 407.
|
Han P, Gu J D. 2013. More refined diversity of anammox bacteria recovered and distribution in different ecosystems[J]. Applied Microbiology and Biotechnology, 97: 3653–3663.
DOI:10.1007/s00253-013-4756-6
|
Harms G. 2003. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant[J]. Environmental Science & Technology, 37: 343–351.
|
Krsek M, Wellington E M. 1999. Comparison of different methods for the isolation and purification of total community DNA from soil[J]. Journal of Microbiological Methods, 39: 1–16.
DOI:10.1016/S0167-7012(99)00093-7
|
Lakay F M, Botha A, Prior B A. 2006. Comparative analysis of environmental DNA extraction and purification methods from different humic acid-rich soils[J]. Journal of Applied Microbiology, 102(2007): 265–273.
|
李欢, 金宜英, 聂永丰. 2009. 污水污泥中腐殖酸的提取和利用[J]. 清华大学学报, 2009, 49(12): 1980–1983.
DOI:10.3321/j.issn:1000-0054.2009.12.017 |
Limpiyakorn T, Sonthiphand P, Rongsayamanont C. 2011. Abundance of amoA genes of ammonia-oxidizing archaea and bacteria in activated sludge of full-scale wastewater treatment plants[J]. Bioresource Technology, 102(4): 3694–3701.
DOI:10.1016/j.biortech.2010.11.085
|
Lindberg E, Albrechtsen H J, Jacobsen C S. 2007. Inhibition of real-time PCR in DNA extracts from aquifer sediment[J]. Geomicrobiology Journal, 24(3/4): 343–352.
|
Lovley D R. 1996. Humic substances as electron acceptors for microbial respiration[J]. Nature, 382(6590): 445–448.
DOI:10.1038/382445a0
|
Lyons S R, Griffen A L, Leys E J. 2000. Quantitative real-time PCR for porphyromonas gingivalis and total bacetria[J]. Journal of Clinical Microbiology, 38(6): 2362–2365.
|
Margit B, Andrea R, Alexandra N, et al. 2013. Effect of DNA polymerases on PCR-DGGE patterns[J]. International Biodeterioration & Biodegradation, 84: 244–249.
|
Miller D N, Bryant J E, Madsen E L, et al. 1999. Evaluation and optimization of DNA extraction and purification procedures for soil and sediment samples[J]. Applied and Environmental Microbiology, 65(11): 4715–4724.
|
Mußmann M, Brito I, Pitcher A. 2011. Thaumarchaeotes abundant in refinery nitrifying sludges express amoA but are not obligate autotrophic ammonia oxidizers[J]. Proceedings of the National Academy of Sciences of the United States of America, 108(40): 16771–16776.
DOI:10.1073/pnas.1106427108
|
Muyzer G, De W E, Uitterlinden A G. 1993. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 59: 695–700.
|
Tong J, Chen Y G. 2009. Recovery of nitrogen and phosphorus from alkaline fermentation liquid of waste activated sludge and application of the fermentation liquid to promote biological municipal wastewater treatment[J]. Water Research, 43(12): 2969–2976.
DOI:10.1016/j.watres.2009.04.015
|
王行, 曹萍麟, 张志剑. 2016. 内参基因加标法定量土壤微生物目标基因绝对拷贝数[J]. 微生物学通报, 2016, 43(2): 424–433.
|
Wang H, Qi J F, Xiao D R, et al. 2017. A re-evaluation of dilution for eliminating PCR inhibition in soil DNA samples[J]. Soil Biology & Biochemistry, 106: 109–118.
|
王文东, 张轲, 范庆海, 等. 2016. 紫外辐射对腐殖酸溶液理化性质及其混凝性能的影响[J]. 环境科学, 2016, 37(3): 994–999.
|
Wilen B M, Jin B, Lant P. 2003. The influence of key chemical constituents in activated sludge on surface and flocculating prope rties[J]. Water Research, 37(9): 2127–2139.
DOI:10.1016/S0043-1354(02)00629-2
|
徐德峰, 李彩虹, 孙力军, 等. 2013. 南美白对虾肠道微生物宏基因组提取方法的比较[J]. 生物技术通报, 2013(12): 119–122.
|
Ye L, Zhang T. 2011. Ammonia-oxidizing bacteria dominates over ammonia-oxidizing archaea in a saline nitrification reactor under low DO and high nitrogen loading[J]. Biotechnology and Bioengineering, 108(11): 2544–2552.
DOI:10.1002/bit.v108.11
|
尹军, 吴磊, 刘志生, 等. 2012. 腐殖活性污泥有机质成分分析[J]. 吉林大学学报, 2012, 42(3): 252–258.
|
赵大勇, 燕文明, 冯景伟. 2010. 实时荧光定量PCR技术对氨氧化细菌的研究[J]. 环境科学与技术, 2010, 33(12): 8–13.
|
周健, 黄勇, 刘忻, 等. 2016. 硫自养反硝化耦合厌氧氨氧化脱氮条件控制研究[J]. 环境科学, 2016, 37(3): 1061–1069.
|
Zhou J Z, Bruns M A, Tiedje J M. 1996. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology, 62(2): 316–322.
|
Zipper H, Buta C, Lammle K, et al. 2003. Mechanisms underlying the impact of humic acids on DNA quantification by SYBR Green I and consequences for the analysis of soils and aquatic sediments[J]. Nucleic Acids Research, 31(7): e39.
DOI:10.1093/nar/gng039
|
 2018, Vol. 38
2018, Vol. 38


