2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085;
3. 中国建筑上海设计研究院有限公司, 上海 200063
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085;
3. China Shanghai Architecture Design & Resetitute Co., Ltd, Shanghai 200063
氮循环过程是自然界的一个重要元素转化过程.生物固氮作用、硝化和反硝化是传统氮循环过程的主要组成,厌氧氨氧化(Anaerobic Ammonium Oxidation, anammox)的发现完善了人们对氮循环过程的认知.厌氧氨氧化细菌可以在厌氧/缺氧的环境下,将铵离子(NH4+)用亚硝酸根(NO2-)氧化为氮气(Strous et al., 1998).厌氧氨氧化对全球氮循环有着重要意义,它使人们对氮循环的限速步骤—氨氧化过程有了全新的认识,同时改变了全球氮循环通量的计算方法(Zhu et al., 2010).关于厌氧氨氧化的研究,最先产生于海洋生态系统(Dalsgaard et al., 2003),目前已计算出厌氧氨氧化反应在海洋生态系统中对氮气产生的贡献率为24%~67%(Dalsgaard et al., 2005),随后在陆地生态系统(Zhu et al., 2011)和淡水生态系统(Wang et al., 2012)中也陆续报道出厌氧氨氧化细菌在氮循环过程中发挥着不可忽视的作用.
以往对厌氧氨氧化细菌的研究大多是利用16S rRNA基因,但直接利用其功能基因能更具体地反映厌氧氨氧化细菌在样本中的群落特征.联氨合成酶(Hydrazine synthase, HZS)是厌氧氨氧化反应的重要酶(Strous et al., 2006),hzsB基因负责编译该酶的一个亚基(hydrazine synthase β-subunit),用它作为厌氧氨氧化的生物标记,能够更全面地展现厌氧氨氧化细菌的群落多样性(Zhu et al., 2011).高通量技术的应用是微生物学研究方法的一次革新,几十万乃至几百万条DNA分子序列能同时被测定,具有高效方便的优势,故本研究拟采用新一代高通量测序技术.
研究发现,厌氧氨氧化菌在海洋生态系统中广泛存在但生物多样性很低(Schmid et al., 2007),在陆地生态系统中生物多样性较高(Zhu et al., 2011),然而目前对于湿地中厌氧氨氧化细菌的群落特征,特别是在大尺度下其地理分布的认识还严重不足.我国地域广阔,从南到北有明显的温度梯度带,且湿地类型众多,这也为微生物群落和功能产生差异创造了可能性.本文通过Illumina Hiseq 2500平台对10个典型湿地沉积物样品的厌氧氨氧化hzsB功能基因进行高通量测序,研究并比较典型湿地沉积物中厌氧氨氧化细菌的丰度、物种组成、多样性,以及这些特性和关键理化因子之间的关系.
2 材料与方法(Materials and methods) 2.1 样品采集与保存于2014年10月采集中国10个不同纬度的典型湿地沉积物样品,样点按纬度增大的趋势依次为:珠江(ZJ)、洞庭湖(DTH)、嘉兴稻田(JX)、巢湖(CH)、兰州黄河(LZ)、天津永定新河(TJ)、北京香山山泉沉积物(XS)、北运河(BYH)、乌梁素海(WLSH)、盘锦淡水沼泽(PJ),采样点具体信息见表 1.
| 表 1 典型湿地沉积物采样点信息 Table 1 Table 1Information of sites in wetland sediments |
每个采样点随机取6个表层沉积物样品,去除其中的植物残体、石子后充分混匀;同时现场测定了上覆水的溶解氧(DO)和采样点气温.收集完毕后,密封并保存在聚乙烯无菌自封袋中,用冰盒储存带回实验室.一部分样品于4 ℃下保存用于基本理化指标的测定,剩下部分经冷冻干燥后保存在-80 ℃超低温冰箱直到DNA提取.
2.2 样品理化指标检测选择氨氮(NH4+-N)、硝氮(NO3--N)、亚硝氮(NO2--N)、pH值、盐度(Salinity)、总有机质(TOM)、含水率(MC)、总氮(TN)、总碳(TC)、总硫(TS)为基本理化指标进行检测.沉积物样品在采集并在4 ℃条件下运输到实验室后,依照《土壤农化分析》(鲍士旦,2000)进行相应测定,其中,氨氮、硝氮、亚硝氮测定时,先用2 mol·L-1的KCl溶液浸提沉积物样品1 h,将浸提液用0.45 μm微孔滤膜过滤后用连续流动分析仪(SAN plus,Skalar Analytical B.V.,Breda,the Netherlands)测定;pH值和盐度是在样品与超纯水质量比为1: 5的浸提液条件下进行测定;总有机质是以样品在550 ℃的烧失量计算(LOI550)(张文河等,2007);含水率以样品在105 ℃下的失水量计算;总氮、总碳、总硫利用元素分析仪同时进行测定.所有理化性质指标都选用3组平行样品进行测定,结果取3组平行样品的平均值(表 2).
| 表 2 典型湿地沉积物理化性质 Table 2 Chemical characteristics of wetland sediments |
样品经过冷冻干燥处理后,称取约0.33 g,用Fast DNA Spin Kit for Soil(MP Biomedicals,USA)提取试剂盒提取样品中的总DNA,并用Nano Drop 2000 UV-Vis Spectrophotometer(Thermo Fisher Scientific,USA)测定DNA浓度.采用厌氧氨氧化细菌功能基因hzsB的一对特征引物hzsB_396F和hzsB_742R(下文称引物1、引物2)对样品进行PCR扩增(Kartal et al., 2011;Harhangi et al., 2012).扩增的50 μL体系为:10×buffer 5 μL,dNTP(2.5 mmol·L-1) 4 μL,浓度为10 mmol·L-1的正、反向引物各1 μL,BSA 0.5 μL,Taq酶(2.5 U) 0.25 μL,稀释10倍的DNA模板2 μL,ddH2O补足至50 μL(Wang et al., 2012).扩增条件为:95 ℃预变性10 min;95 ℃变性60 s,59 ℃退火60 s,72 ℃延伸45 s,35个循环.PCR扩增产物用1%凝胶电泳验证后用Promega Agarose Gel DNA(Promega,Madison,WI)纯化试剂盒纯化,将纯化产物通过Hiseq 2500平台(Illumina,SanDiego,CA,USA)进行测序(诺禾致源生物信息科技有限公司,北京).
2.4 PCR定量分析采用实时荧光定量PCR方法对厌氧氨氧化细菌进行定量分析,其定量PCR采用SYBR Green法,选用引物1、引物2作为特征引物.扩增的20 μL体系包括:SYBRs Premix Ex Taq酶(Takara, 大连)10 μL,ROX50 0.4 μL,浓度为200 nmol·L-1的正、反向引物各0.2 μL,稀释10倍的DNA模板2 μL,BSA 0.4 μL,用ddH2O补足至20 μL.利用ABI7300 Real-Time PCR System扩增仪(Applied Biosystems,CA,USA)进行定量,扩增测序如下(Wang et al., 2012):95 ℃预变性3 min,95 ℃变性10 s,59 ℃退火30 s,72 ℃延伸30 s,共40个循环.将已知拷贝数质粒的DNA按照10倍进行梯度稀释,得到的6个标准样品同时进行定量扩增,得到标准曲线;实验设有阴性对照;每个样品取3次平行,要求扩增效率大于95%,可决系数R2大于0.98,溶解曲线为单一峰.
2.5 序列处理测序公司返回序列结果后,序列处理流程如下.①序列的质量控制:根据每个样品独立的条形码信息,所有序列被分配到每一个样本,然后去除条形码信息和引物序列.首先通过Uchime软件检测并筛除在PCR扩增过程中由于不完整的扩增得到的嵌合体序列,然后通过Qiime软件筛除长度不统一序列和低质量的序列,最后通过Mothur软件得到非冗余的核酸序列(Schloss et al., 2009).②序列的聚类:用Bioedit软件将非冗余的核酸序列翻译成氨基酸序列,然后通过Uclust软件将氨基酸序列按照90%的相似度进行聚类(Edgar et al., 2010),得到所有OTUs的序列分配结果.③在OTUs水平的生态学分析:将每个OTUs代表的氨基酸序列在Genbank数据库进行BLAST比对,比对后得到注释结果;通过编写的Perl脚本计算多样性等(庄林杰等,2017).
2.6 统计分析根据Bray-Curtis差异度指数进行群落的分类和排序,分类选择UPGMA算法,排序选用主坐标分析(Principle Coordinate Analysis, PCoA).用Origin9.0软件及Heatmap软件作图,用SPSS21.0软件(Statistical Product and Service Solutions)进行相关性分析(采用Person相关系数),用软件Canoco4.5做冗余分析(Redundancy Analysis, RDA).
3 结果(Results) 3.1 厌氧氨氧化细菌的高通量测序结果提取10个典型湿地表层沉积物样品DNA,对hzsB基因进行PCR扩增后,在Illumina 2500 Hiseq平台进行高通量测序.从每个样品的原始核酸序列中随机选取20000条,使用生物信息软件对序列进行质控、聚类、统计分析.OUTs的注释结果显示,质控后的序列均隶属于厌氧氨氧化细菌.各样点质控前后序列条数和在90%的相似度下序列聚类得到的OTUs数及覆盖率的大小如表 3所示.
| 表 3 Illumina高通量测序结果 Table 3 Summary of Illumina high-throughput sequencing results |
表 3的数据显示,原始序列在经过质控处理后,样品的序列数在18593~19562之间.样点的OTUs数在22~60之间,JX样品的OTUs最多(60),WLSH样品的OTUs最少(22),覆盖率均在99.90%之上.
以85%~97%相似度(1%递增)划分OTU得到各相似度下的OTUs数(图 1a),由图可知,相似度在85%~89%之间,划分的OTUs数量较少(8~55个),相似度在91%~97%之间,划分的OTUs数量较多(142~8586个),相似度为90%时最为合理.以500条序列为梯度,进行连续累加来计算OTUs的数量,得出OTUs随序列数增加的变化趋势(图 1b).从图中可以观察到,刚开始随着序列数的增加,OTUs数量迅速增加;而随着序列数的进一步增大,样本中OTUs数量的增加趋势逐渐趋于平缓;稀释曲线较高的平滑程度表明样品的序列数充足,高通量测序结果的覆盖度高,高覆盖率(均在99.90%之上)也说明了这一结果.
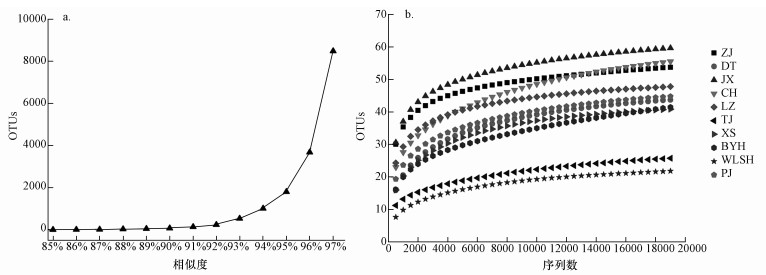 |
| 图 1 OTUs数随相似度变化的增长曲线(a)及典型湿地沉积物厌氧氨氧化细菌的稀释曲线(b) Fig. 1 OTUs growth curves as change of similarity(a) and the rarefaction curve of hzsB OTUs in wetland sediments(b) |
对10个典型湿地沉积物样品做实时PCR定量分析,得到厌氧氨氧化细菌hzsB基因的丰度,结果如图 2所示.由图 2可知,ZJ、JX、CH、BYH、PJ这五地的厌氧氨氧化细菌的hzsB基因丰度值在106 copies·g-1的水平,范围在1.24×106~4.20×106 copies·g-1之间,BYH的hzsB基因丰度值最大,为4.20×106 copies·g-1;DT、LZ、TJ、XS的厌氧氨氧化细菌的hzsB基因丰度值在105 copies·g-1的水平上,范围在2.14×105~5.04×105 copies·g-1之间;WLSH的hzsB基因丰度值最低,为5.42×104 copies·g-1.
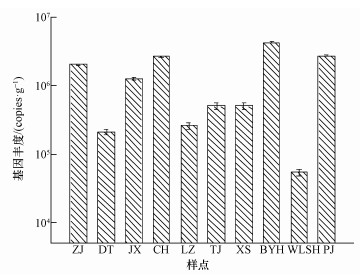 |
| 图 2 典型湿地沉积物厌氧氨氧化细菌的丰度 Fig. 2 Abundance of anammox bacteria in wetland sediments |
利用Uclust软件对10个典型湿地沉积物样品中厌氧氨氧化细菌质控后的序列进行聚类分析(90%相似度),删除序列贡献率很低的OTUs后得到77个OTUs,将每个OTUs的代表氨基酸序列在Genbank数据库进行BLAST比对,结果表明其均与厌氧氨氧化菌hzsB序列相似.
如图 3a所示,所有序列分为5个部分,54.09%的厌氧氨氧化序列与‘Ca. Brocadia’的同源性较高,30.97%的厌氧氨氧化序列与‘Ca. Jettenia’的同源性较高,6.95%和7.91%的厌氧氨氧化序列分别与‘Ca. Kuenenia’、‘Ca. Scalindua’的同源性较高,相似度均在92%~99%之间,以及微量和已知的hzsB基因序列相似度较低的序列,推测为未被探明的新型厌氧氨氧化细菌,将这一簇序列命名为Anammox-like cluster.由图 3b可知,‘Ca. Brocadia’中‘Candidatus Brocadia fulgida’和‘Candidatus Brocadia sinica’比例较大,分别占48.12%和37.6%,‘Candidatus Brocadia anammoxidans’和‘Candidatus Brocadia caroliniensis’分别占11.46%和2.82%.
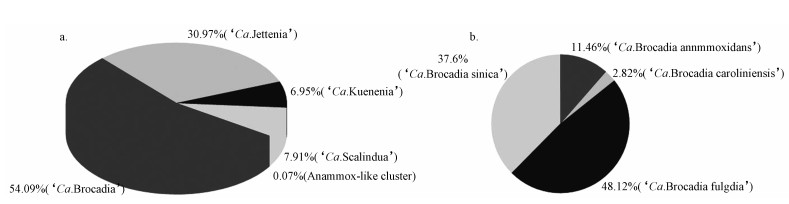 |
| 图 3 典型湿地沉积物厌氧氨氧化细菌的总体组成(a)及‘Ca. Brocadia’ (b)的组成 Fig. 3 Overall composition of anammox bacteria(a) and composition of 'Ca. Brocadia' (b) in wetland sediments |
统计隶属于每个种属中OTUs的序列条数,计算各样点中厌氧氨氧化细菌各种属的相对丰度,计算结果如图 4所示.由图可知,不同湿地沉积物的厌氧氨氧化菌落结构差别较大.
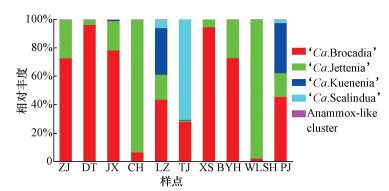 |
| 图 4 典型湿地沉积物厌氧氨氧化细菌相对丰度图 Fig. 4 Relative abundance of anammox bacteria in wetland sediments |
‘Ca. Brocadia’在ZJ、DT、JX、XS、BYH样点中的比例较大(> 50%),且在LZ、TJ、PJ样点中均有明显优势;‘Ca. Jettenia’在CH和WLSH两地的比例很大(93.30%、97.71%),在TJ样点中的比例最小(0.82%);‘Ca. Kuenenia’在LZ和PJ两地中有较为显著的占比(32.74%、35.24%),在其它样点只占很小比例,其中,隶属于‘Ca. Kuenenia’的OTUs在DT样点中没有检测到;‘Ca. Scalindua’在TJ样点的占比最大(70.54%),所有隶属于‘Ca. Scalindua’的序列基本由TJ样点贡献,除了在LZ和PJ样点有部分占比(5.95%、2.38%)外,在其它样点的比例很小;Anammox-like cluster在各地的比例都很小,在TJ、XS两地并没有检测到属于该菌群的序列.
各样品中厌氧氨氧化细菌的群落结构在OTUs水平下的展示如图 5所示,其中一个方块代表一个OTU,方块的颜色表示该OTU下序列丰度(颜色从蓝-白-红的渐变,代表序列丰度从低到高的变化),右边为各种属中OTUs的分配结果.结果表明,隶属于‘Ca. Brocadia’、‘Ca. Jettenia’、‘Ca. Kuenenia’、‘Ca. Scalindua’和Anammox-like cluster的OTUs数分别为41、27、2、4、3;各样点方块呈现不同的颜色排布规律,代表每个样点中单个OTU所分配得到的序列丰度不同,说明在OTUs水平下,各样点的厌氧氨氧化细菌群落差异较大.其中,隶属于‘Ca. Brocadia’的序列主要分布在OTU7和OTU1,隶属于‘Ca. Jettenia’的序列主要分布在OTU2和OTU4.
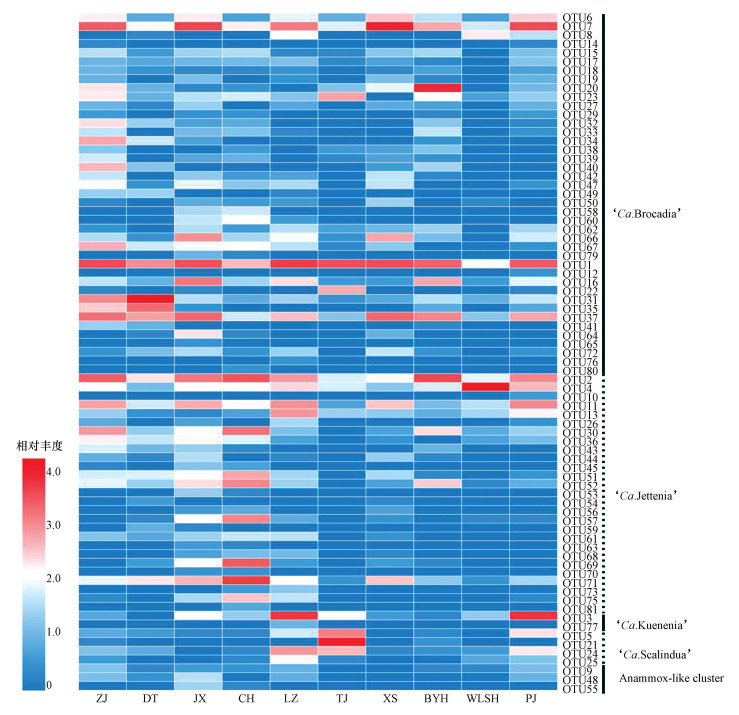 |
| 图 5 典型湿地沉积物厌氧氨氧化细菌群落分布热图 (相对丰度值用lg(x+1)处理) Fig. 5 The heatmap of anammox bacteria community distribution in wetland sediments |
α-多样性指标可以在一定程度上表示样点的群落特征.本文中计算的α-多样性指标有丰富度指数(Chao1指数),表示一个群落中物种的复杂程度,值越高说明群落内物种的数目越多;优势度指数(Simpson指数),表示一个群落中优势菌群的地位和贡献,值越高表明群落内物种数量差异越大,优势菌群的主导地位越突出;多样性指数(Shannon指数),是物种丰富度和均匀度的综合指标,也是对Chao1指数和Simpson指数综合的一个指数(庄林杰等,2017).为了保证每个样品的序列数一致,在计算各指标前对样品进行了重取样(Rousk et al., 2010).计算结果如表 4所示,在不同湿地沉积物样品中,Chao1指数最高的是JX(63.00),最低的是WLSH(25.00);Simpson指数最高的WSLH(0.94),最低的是ZJ(0.12);Shannon指数最高的是ZJ(2.53),最低的是WLSH(0.20).
| 表 4 典型湿地沉积物厌氧氨氧化细菌的α-多样性指数 Table 4 α-diversity index of anammox bacteria in wetland sediments |
比较各样点的多样性指数(Shannon指数),发现在纬度在由低到高的变化范围(由南到北),Shannon指数呈现减小的趋势.其中,最高的3个样点为ZJ(2.53)、JX(2.37)、CH(2.19),均是南方典型的湿地样点;最低的样点在WLSH(0.20),且TJ(1.26)、XS(1.47)、BYH(1.67)均属于北方典型湿地样点.在一定程度上可以反映南方典型湿地沉积物的厌氧氨氧化细菌多样性高于北方.
β-多样性可以用来比较各样点间群落的差异.基于Bray-Curtis差异度指数,计算各样点间的差异,进而对厌氧氨氧化细菌群落排序,选取解释度最大的两轴做PCoA图(图 6),它们对排序结果的解释度分别为28.04%和19.79%.图 6结果显示,在10个典型湿地沉积物中,LZ、PJ、XS三地样品距离很近,说明这3个点的厌氧氨氧化细菌的群落结构更相似;JX和ZJ两地样品距离较近,反映这两地的厌氧氨氧化细菌的群落结构比较类似;其它样点之间距离较远,尤其是TJ和WLSH两地,与其它各样点间的群落结构都有较大差异.
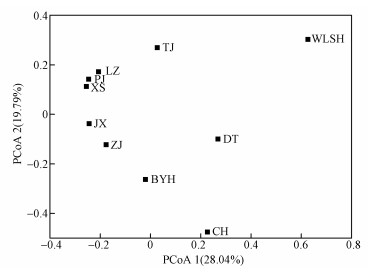 |
| 图 6 典型湿地沉积物厌氧氨氧化细菌的PCoA图 Fig. 6 PCoA in wetland sediments |
为了探究厌氧氨氧化群落结构是否受到关键环境因子的影响,用每个样点厌氧氨氧化细菌hzsB基因丰度和4类厌氧氨氧化菌群的相对丰度分别与土壤的理化性质做SPSS相关性分析(表 5)和冗余分析(RDA)(图 7).
| 表 5 典型湿地沉积物厌氧氨氧化细菌hzsB基因丰度与环境因子的相关性分析(n=10) Table 5 Correlation analysis between abundance of anammox bacteria and chemical characters of wetland sediments (n=10) |
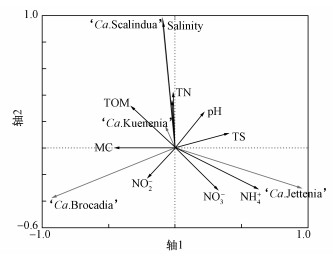 |
| 图 7 典型湿地沉积物厌氧氨氧化细菌的群落结构和环境因子的RDA图 Fig. 7 RDA between community structure of anammox bacteria and chemical characters of soil in wetland sediments |
SPSS结果显示,在所测样品中,厌氧氨氧化细菌的丰度与TOM(r=-0.658,p=0.039)、TN(r=-0.555,p=0.096)、TC(r=-0.608,p=0.062)、TS(r=-0.614,p=0.059)呈负相关,与其它所测土壤理化指标无相关性.表明在国内典型湿地沉积物中,部分理化指标含量(TOM、TN、TC、TS)与厌氧氨氧化细菌的整体丰度呈负相关.
运用Canoco 4.5软件对样点的10个沉积物理化因子和4类厌氧氨氧化菌群的相对丰度数据进行分析,考察湿地中厌氧氨氧化细菌群落结构和环境因子之间的关系,分析结果如图 7所示.由图可知,前2个轴的特征值分别为0.637、0.254,共解释了厌氧氨氧化菌群变异程度的89.1%,物种的两个排序轴之间相关系数和环境因子的两个排序轴之间的相关系数均为0,表明排序结果是可靠的.
RDA排序图显示:排序轴1与NH4+的正相关性最大,与MC的负相关性最大;排序轴2与盐度的正相关性最大,与NO2-的负相关性最大.分别代表物种信息和理化因子的虚线箭头和实线箭头之间的夹角和长短可以反映彼此间的相关性,分析结果表明,‘Ca. Jettenia’与NH4+呈显著正相关,‘Ca. Scalindua’与盐度呈显著正相关,‘Ca. Kuenenia’在所有的理化因子中与盐度的正相关性最显著.
4 讨论(Discussion)本文针对厌氧氨氧化细菌的功能基因hzsB进行扩增,结合新一代高通量技术,对国内10个具有代表性的典型湿地沉积物中厌氧氨氧化细菌的丰度和群落组成从不同角度进行分析:样点的α-多样性、样点间的β-多样性、基因丰度和群落组成及其与环境因子的关系.
本研究结果表明,样品中所检测到的厌氧氨氧化细菌最主要隶属于Brocadia,其次隶属于Jettenia,还有部分隶属于Kuenenia和Scalindua,而各个样点的主导菌属有差异.主要原因可能是:除了天津采样点位于盐碱地带而导致盐度偏高(1.89%)外,其它采样点的盐度大多较低(0.008%~0.076%),远小于标准海水盐度3.5%.‘Ca. Brocadia’主要存在于淡水环境中(Dale et al., 2009),这就解释了‘Ca. Brocadia’在整体上及在各个地点上处于主导地位的结果,本文RDA图中‘Ca. Brocadia’与盐度呈负相关也证明这一点.在海洋生态系统中,已有的厌氧氨氧化细菌严格隶属于Scalindua(Schmid et al., 2007);Sonthiphand等(2014)用16S rRNA基因序列确定了‘Ca. Scalindua’在海洋环境中的专一分布,研究表明,‘Ca. Scalindua’细胞在盐度小于1.5%的环境下会失去厌氧氨氧化活性(Awata et al., 2013),这些解释了‘Ca. Scalindua’在采样点TJ的相对丰度最高且在RDA图中‘Ca Scalindua.’与盐度呈显著正相关.Kartal等(2006)推断‘Ca. Kuenenia’适合生活在淡水环境中,但本研究中LZ和PJ两地的盐度相对于其他采样点较高,‘Ca. Kuenenia’在这两个样品的相对丰度较高,RDA图中‘Ca. Kuenenia’与盐度呈一定程度的正相关,这表明‘Ca. Kuenenia’对盐度可能具有更强的适应性.研究表明,隶属于Jettenia的16S rRNA序列来源于淡水生态系统(Kartal et al., 2006;Humbert et al., 2010;2012;Yoshinaga et al., 2011;Zhu et al., 2011;Wang et al., 2012;Hu et al., 2013;Long et al., 2013;Shen et al., 2013),RDA分析结果表明,该菌群与盐度呈负相关,说明‘Ca. Jettenia’适合在淡水环境中生存.
有研究表明,温度对厌氧氨氧化细菌群落结构、丰度及活性均有重要影响(Hou et al., 2013;Teixeira et al., 2012;Brin et al., 2015).本研究中南方的代表样点ZJ、JX、CH为多样性Shannon指数最高的3个点,北方的代表样点WLSH、TJ、XS为多样性Shannon指数最低的3个点,这可能是因为环境温度对厌氧氨氧化细菌的群落结构造成的潜在影响.各采样点的位置在纬度梯度下分布进而有温度差异,加上各地地理因子和环境因子差异等的综合因素,造成了各样点下厌氧氨氧化菌群落组成的不同.
本文通过定量PCR技术考察了厌氧氨氧化细菌在典型湿地沉积物中的分布,结果表明,5个样点的厌氧氨氧化细菌hzsB基因丰度值在106 copies·g-1的水平,4个样点在105 copies·g-1的水平,1个样点在104 copies·g-1的水平.以往研究表明,厌氧氨氧化细菌倾向于在高浓度或有稳定氮营养盐的沉积物中生存(Risgaard-Petersen et al., 2004; 2005;Rich et al., 2008; Nicholls et al., 2009; Dang et al., 2010; Rush et al., 2012; Teixeira et al., 2012; Kalvelage et al., 2013; Zhu et al., 2013).本文中各样点的氨氮和硝氮浓度有差异,且SPSS相关性分析中厌氧氨氧化细菌丰度与氨氮、硝氮没有显著的相关性,需要考虑更多的环境和气候因素来研究影响厌氧氨氧化细菌丰度变化的机制.在海洋生态系统中,硫化物被认为是影响厌氧氨氧化细菌分布和活性的一个重要因子(Wakeham et al., 2007;Jensen et al., 2008).此处得到TS与厌氧氨氧化细菌丰度呈负相关,可以推测TS对厌氧氨氧化菌的影响可以拓展到湿地生态系统.
厌氧氨氧化细菌生态位的差异一直是研究的热点(Ding et al., 2013; Oshiki et al., 2016).RDA结果表明,‘Ca. Jettenia’与NH4+呈显著正相关,‘Ca. Jettenia’相对于其它菌属可以在更高的NH4+浓度下生存(Ali et al., 2014).‘Ca. Brocadia sinica’倾向于生存在NH4+和NO2-高的环境中,因为与它们的亲和力低,而‘Ca. Kuenenia stuttgartiensis’对NH4+和NO2-的亲和力较高(Tsushima et al., 2007; Hu et al., 2010; Oshiki et al., 2011; Osaka et al., 2012),RDA图显示,‘Ca. Brocadia’与NH4+并无显著相关,可能是由于其它‘Ca. Brocadia sp.’的不同生理机能或者存在其它影响因子.
综上,本文运用高通量测序技术对厌氧氨氧化功能基因hzsB扩增,一定程度上揭示了不同地理分布的典型湿地沉积物中厌氧氨氧化细菌的丰度、群落组成、多样性,以及其与部分理化因子之间的关系.但厌氧氨氧化菌各种属和关键理化因子间的生物调节机制及它们生长动力学还有待深入研究.
Ali M, Oshiki M, Awata T, et al. 2014. Physiological characterization of anaerobic ammonium oxidizing bacterium "Candidatus Jettenia caeni"[J]. Environmental Microbiology, 17(6): 2172–2189.
|
Awata T, Oshiki M, Kindaichi T, et al. 2013. Physiological characterization of an anaerobic ammonium-oxidizing bacterium belonging to the "Candidatus scalindua" group[J]. Applied & Environmental Microbiology, 79(13): 4145–4148.
|
鲍士旦. 2000. 土壤农化分析(第3版)[M]. 北京: 中国农业出版.
|
Brin L D, Giblin A E, Rich J J. 2015. Effects of experimental warming and carbon addition on nitrate reduction and respiration in coastal sediments[J]. Biogeochemistry, 125(1): 81–95.
DOI:10.1007/s10533-015-0113-4
|
Dale O R, Tobias C R, Song B K. 2009. Biogeographical distribution of diverse anaerobic ammonium oxidizing (anammox) bacteria in Cape Fear River Estuary[J]. Environmental Microbiology, 11(5): 1194–1207.
DOI:10.1111/emi.2009.11.issue-5
|
Dalsgaard T, Canfield D E, Petersen J, et al. 2003. N2 production by the anammox reaction in the anoxic water column of Golfo Dulce, Costa Rica[J]. Nature, 422(6932): 606–608.
DOI:10.1038/nature01526
|
Dalsgaard T, Bo T, Canfield D E. 2005. Anaerobic ammonium oxidation (anammox) in the marine environment[J]. Research in Microbiology, 156(4): 457–464.
DOI:10.1016/j.resmic.2005.01.011
|
Dang H Y, Chen R P, Lin W, et al. 2010. Environmental factors shape sediment anammox bacterial communities in hypernutrified Jiaozhou Bay, China[J]. Applied & Environmental Microbiology, 76(21): 7036–7047.
|
Ding S, Zheng P, Lu H, et al. 2013. Ecological characteristics of anaerobic ammonia oxidizing bacteria[J]. Applied Microbiology & Biotechnology, 97(5): 1841–1849.
|
Edgar R C. 2010. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 26(19): 2460–2461.
DOI:10.1093/bioinformatics/btq461
|
Harhangi H R, Le R M, Van A T, et al. 2012. Hydrazine synthase, a unique phylomarker with which to study the presence and biodiversity of anammox bacteria[J]. Applied & Environmental Microbiology, 78(3): 752–758.
|
Hu B L, Zheng P, Tang C J, et al. 2010. Identification and quantification of anammox bacteria in eight nitrogen removal reactors[J]. Water Research, 44(17): 5014–5020.
DOI:10.1016/j.watres.2010.07.021
|
Hu B L, Shen L, Liu S, et al. 2013. Enrichment of an anammox bacterial community from a flooded paddy soil[J]. Environmental Microbiology Reports, 5(3): 483–489.
DOI:10.1111/1758-2229.12038
|
Hou L, Zheng Y, Liu M, et al. 2013. Anaerobic ammonium oxidation (anammox) bacterial diversity, abundance, and activity in marsh sediments of the Yangtze Estuary[J]. Journal of Geophysical Research, 118(3): 1237–1246.
|
Humbert S, Tarnawski S, Fromin N, et al. 2010. Molecular detection of anammox bacteria in terrestrial ecosystems: distribution and diversity[J]. Isme Journal Emultidisciplinary Journal of Microbial Ecology, 4(3): 450–454.
|
Humbert S, Zopfi J, Tarnawski S. 2012. Abundance of anammox bacteria in different wetland soils[J]. Environmental Microbiology Reports, 4(5): 484–490.
DOI:10.1111/emi4.2012.4.issue-5
|
Jensen, Marlene M, Kuypers M M M, et al. 2008. Rates and regulation of anaerobic ammonium oxidation and denitrification in the Black Sea[J]. Limnology & Oceanography, 53(1): 23–36.
|
Kartal B, Koleva M, Arsov R. 2006. Adaptation of a freshwater anammox population to high salinity wastewater[J]. Journal of Biotechnology, 126(4): 546–553.
DOI:10.1016/j.jbiotec.2006.05.012
|
Kartal B, Maalcke W J, de Almeida N M, et al. 2011. Molecular mechanism of anaerobic ammonium oxidation[J]. Nature, 479(7371): 127–130.
DOI:10.1038/nature10453
|
Kalvelage T, Lavik G, Lam P, et al. 2013. Nitrogen cycling driven by organic matter export in the South Pacific oxygen minimum zone[J]. Nature Geoscience, 6(3): 228–234.
DOI:10.1038/ngeo1739
|
Long A, Heitman J, Tobias C, et al. 2013. Co-occurring anammox, denitrification, and codenitrification in agricultural soils[J]. Applied & Environmental Microbiology, 79(1): 168–176.
|
Nicholls J C, Trimmer M. 2009. Widespread occurrence of the anammox reaction in estuarine sediments[J]. Aquatic Microbial Ecology, 55(2): 105–113.
|
Osaka T, Kimura Y, Otsubo Y, et al. 2012. Temperature dependence for anammox bacteria enriched from freshwater sediments[J]. Journal of Bioscience & Bioengineering, 114(4): 429–434.
|
Oshiki M, Shimokawa M, Fujii N, et al. 2011. Physiological characteristics of the anaerobic ammonium-oxidizing bacterium 'Candidatus Brocadia sinica'[J]. Microbiology, 157(6): 1706–1713.
DOI:10.1099/mic.0.048595-0
|
Oshiki M, Satoh H, Okabe S. 2016. Ecology and physiology of anaerobic ammonium oxidizing (anammox) bacteria[J]. Environmental Microbiology, 18(9): 2784–2796.
DOI:10.1111/1462-2920.13134
|
Rich J J, Dale O R, Song B, et al. 2008. Anaerobic ammonium oxidation (anammox) in Chesapeake Bay sediments[J]. Microbial Ecology, 55(2): 311–320.
DOI:10.1007/s00248-007-9277-3
|
Risgaard-Petersen N. 2004. Anaerobic ammonium oxidation in an estuarine sediment[J]. Aquatic Microbial Ecology, 36(3): 293–304.
|
Risgaard-Petersen N, Meyer R L, Revsbech N P. 2005. Denitrification and anaerobic ammonium oxidation in sediments: effects of microphytobenthos and NO3-[J]. Aquatic Microbial Ecology, 40: 67–76.
DOI:10.3354/ame040067
|
Rousk J, Baath E, Brookes P C, et al. 2010. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. Isme Journal Multidisciplinary Journal of Microbial Ecology, 4(10): 1340–1351.
|
Rush D, Wakeham S G, Hopmans E C, et al. 2012. Biomarker evidence for anammox in the oxygen minimum zone of the Eastern Tropical North Pacific[J]. Organic Geochemistry, 53(12): 80–87.
|
Schmid M C, Risgaard-Petersen N, Vossenberg J V D, et al. 2007. Anaerobic ammonium-oxidizing bacteria in marine environments: widespread occurrence but low diversity[J]. Environmental Microbiology, 9(6): 1476–1484.
DOI:10.1111/emi.2007.9.issue-6
|
Schloss P D, Westcott S L, Ryabin T, et al. 2009. Introducing mothur: Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied & Environmental Microbiology, 75(23): 7 537–7541.
|
Shen L, Liu S, Lou L, et al. 2013. Broad distribution of diverse anaerobic ammonium-oxidizing bacteria in Chinese agricultural soils[J]. Applied & Environmental Microbiology, 79(19): 6167–6172.
|
Sonthiphand P, Hall M W, Neufeld J D. 2014. Biogeography of anaerobic ammonia-oxidizing (anammox) bacteria[J]. Frontiers in Microbiology, 5(399): 399.
|
Strous M, Heijnen J J, Kuenen J G, et al. 1998. The sequencing batch reactor as a powerful tool for the study of slowly growing anaerobic ammonium-oxidizing microorganisms[J]. Applied Microbiology and Biotechnology, 50(5): 589–596.
DOI:10.1007/s002530051340
|
Strous M, Pelletier E, Mangenot S, et al. 2006. Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J]. Nature, 440(7085): 790–794.
DOI:10.1038/nature04647
|
Teixeira C, Magalhaes C, Joye S B, et al. 2012. Potential rates and environmental controls of anaerobic ammonium oxidation in estuarine sediments[J]. Aquatic Microbial Ecology, 66(1): 23–32.
DOI:10.3354/ame01548
|
Tsushima I, Ogasawara Y, Kindaichi T, et al. 2007. Development of high-rate anaerobic ammonium-oxidizing (anammox) biofilm reactors[J]. Water Research, 41(8): 1623–34.
DOI:10.1016/j.watres.2007.01.050
|
Wakeham, Stuart G, Amann, et al. 2007. Microbial ecology of the stratified water column of the Black Sea as revealed by a comprehensive biomarker study[J]. Organic Geochemistry, 38(12): 2070–2097.
DOI:10.1016/j.orggeochem.2007.08.003
|
Wang S, Zhu G, Peng Y, et al. 2012. Anammox bacterial abundance, activity, and contribution in riparian sediments of the Pearl River estuary[J]. Environmental Science & Technology, 46(16): 8834–8842.
|
Wang Y, Zhu G, Harhangi H R, et al. 2012. Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil[J]. Fems Microbiology Letters, 336(2): 79–88.
DOI:10.1111/fml.2012.336.issue-2
|
Yoshinaga I, Amano T, Yamagishi T, et al. 2011. Distribution and diversity of anaerobic ammonium oxidation (anammox) bacteria in the sediment of a eutrophic freshwater lake, Lake Kitaura, Japan[J]. Microbes & Environments, 26(3): 189–197.
|
张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理, 2007, 30(3): 455–459.
|
Zhu G, Jetten M S M, Kuschk P, et al. 2010. Potential roles of anaerobic ammonium and methane oxidation in the nitrogen cycle of wetland ecosystems[J]. Applied Microbiology & Biotechnology, 86(4): 1043–1055.
|
Zhu G, Wang S, Wang Y, et al. 2011. Anaerobic ammonia oxidation in a fertilized paddy soil[J]. Isme Journal, 5(12): 1905–1912.
DOI:10.1038/ismej.2011.63
|
Zhu G, Wang S, Wang W, et al. 2013. Hotspots of anaerobic ammonium oxidation at land-freshwater interfaces[J]. Nature Geoscience, 6(2): 103–107.
DOI:10.1038/ngeo1683
|
庄林杰, 夏超, 田晴, 等. 2017. 高通量测序技术研究典型湖泊岸边陆向深层土壤中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 2017, 37(1): 261–271.
|
 2017, Vol. 37
2017, Vol. 37


