大量不合理的抗生素使用导致全球范围内抗生素抗性菌和抗性基因的广泛传播, 引起了越来越多的关注(Christgen et al., 2015).由于抗生素并不能被机体完全吸收, 30%~90%的抗生素以原形的形式随粪便和尿液排出体外(Zhang et al., 2016), 导致在地下水、土壤、空气, 甚至饮用水中都能检测到抗生素、抗性菌和抗性基因的存在(Jiang et al., 2013; Huang et al., 2014; Li et al., 2016; Wang et al., 2015; 高俊红等, 2016).污水处理厂是抗生素抗性菌和抗性基因的重要储存库, 废水中的抗生素可能诱导抗生素抗性微生物富集, 甚至通过抗性基因水平转移使微生物获得多重抗生素抗性进而产生“超级细菌”(Pruden, 2014).
四环素类抗生素, 作为一种广谱抗生素广泛用于人和动物细菌感染治疗, 并且作为饲料添加剂用于促进动物生长(Burch et al., 2014), 导致环境中四环素类抗生素的广泛高浓度分布.据报道, 禽畜废水和城市污水中检测到四环素类抗生素的浓度分别高达4.1~32.67 μg · L-1(Kim et al., 2013)和10~1000 ng · L-1(Topal et al., 2016).同时, 四环素抗性菌在污水处理厂中频繁检出, 例如微杆菌属(Microbacterium)、无色杆菌属(Achromobacter)(Li et al., 2013)、节杆菌属(Arthrobacter)、副球菌属(Paracoccus)(Chen et al., 2016)、肠杆菌属(Enterobacter)(Huang et al., 2016)等.相应的四环素抗性基因也具有较高的检出率, Zhang等(2009)从上海、香港、加利福尼亚的3个污水处理厂活性污泥中检测到tetA、tetB、tetC、tetD、tetE、tetG、tetM、tetO、tetQ、tetX、tetS、tetL、tetK基因.Zhang等(2011)从我国不同地区的15个污水处理厂中检测到tetA、tetC、tetD、tetG、tetM、tetX、tetS等13种四环素抗性基因.Du等(2014)从浙江和上海的5个污水处理厂进出水中检测到tetA、tetB、tetC、tetE、tetG、tetM、tetO、tetQ、tetW、tetX四环素抗性基因的存在.目前, 关于污水处理厂中四环素抗性菌的研究多集中在抗生素抗性及抗性基因的表达(Feichtinger et al., 2016; Liu et al., 2016), 因此, 研究和分析污水处理厂活性污泥中四环素抗性菌中抗性基因的种类和遗传特性, 对于废水生物处理系统中抗生素抗性菌的环境行为和风险评估具有重要意义.
本研究采用四环素培养基从上海市某污水处理厂二沉池活性污泥中分离筛选出一株四环素抗性菌, 在菌株鉴定、生理生化特性分析的基础上, 结合KEGG蛋白功能注释, 抗性蛋白特性及基因簇分析, 考查该四环素抗性菌中抗性基因的种类及其遗传特性, 为评估污水处理系统中抗生素抗性菌的环境行为和风险提供参考数据.
2 材料与方法(Materials and methods) 2.1 污泥样品的采集与处理活性污泥样品取自上海市某污水处理厂二沉池活性污泥, 该污水处理厂主体工艺采用INKA曝气AO法处理工艺.取样设备经消毒处理, 样品采集后保存于冰盒运回实验室.采用固相萃取-高效液相色谱-串联质谱法(SPE-HPLC/MS/MS)检测污水中四环素类抗生素的浓度.采集的污水样品经过滤、调节pH、加内标后, 在24 h内完成固相萃取前处理过程, 处理后的样品采用高效液相色谱-串联质谱法(Aglient, USA)检测四环素类抗生素浓度.污水厂进水、A/O池出水、污水厂出水中四环素类抗生素浓度检测结果如表 1所示, 作为实验室筛选抗性菌的四环素浓度本底参考值.
| 表 1 城市污水处理厂四环素类抗生素浓度水平 Table 1 The concentrations of tetracycline antibiotics in municipal sewage treatment plant |
富集培养基:蛋白胨10 g, 酵母粉5 g, 氯化钠10 g, 加水定容到1000 mL, 使用前120 ℃高压灭菌20 min.
四环素储存液:四环素储存液使用无水乙醇配制, 经0.22 μm滤膜过滤后避光保存于-20 ℃冰箱, 保存时间不超过30 d.
选择培养基:富集培养基120 ℃灭菌20 min后, 加入四环素储存液, 4 ℃保存.
2.3 四环素抗性菌的分离纯化取沉降浓缩后的活性污泥样品, 按接种量40%加入到一系列四环素浓度梯度的选择培养基中, 四环素浓度梯度为1、5、10、50、100、500 μg · L-1, 置于恒温振荡培养箱中(30 ℃, 150 r · min-1)培养10 d.培养液用灭菌生理盐水进行梯度稀释后, 均匀涂布于对应四环素浓度的选择培养基平板上, 于30 ℃恒温生化培养箱中培养48 h.挑取单菌落, 在等浓度选择培养基平板上划线分离纯化.
2.4 抗性菌株的鉴定 2.4.1 生理生化鉴定菌株的形态和生理生化鉴定参照微生物学实验及《伯杰氏鉴定手册》.
2.4.2 分子生物学鉴定将选择培养基上分离得到的单菌落进行16S rDNA的PCR扩增与序列测定(上海睿迪生物科技有限公司).PCR引物为通用引物27F:AGAGTTTGATCCTGGCTCAG;1492R:GGTT ACCTTGTTA CGACTT.用Blast软件将测序结果与GenBank中已知菌株的16S rDNA序列进行同源性对比, 选取同源性较高的序列, 利用MEGA 5.1软件构建菌株的系统发育树.
2.5 菌株抗性蛋白特性分析依据菌株TC-1的16S rDNA鉴定结果, 选择与之同种的全基因组序列在KEGG数据库中进行蛋白功能注释.根据KEGG蛋白功能注释结果, 将菌株TC-1的抗性蛋白与NCBI数据库中已知的蛋白序列进行Blastp比对, 下载相似度最高的前13~14条蛋白序列.然后, 采用MEGA 5.1软件的邻位相连法(Neighbor-joining)构建系统发育树, 对这些抗性蛋白进行同源性分析, 研究这些抗性蛋白在节杆菌属细菌间的分类学关系.
2.6 抗生素抗性基因的基因簇分析根据菌株TC-1抗性蛋白同源性分析结果, 对其编码基因进行基因簇同线性分析.在菌株TC-1的KEGG蛋白注释结果中选择包括抗性蛋白序列在内的前后各5条蛋白序列, 共11条蛋白序列, 将其与SyntTax数据库中已知蛋白序列的同种属菌株进行比对, 选择与菌株TC-1抗性蛋白编码顺序一致性较高的基因簇图, 进行抗性基因的基因簇同线性分析.一般认为, 多个连续蛋白编码基因在某一微生物种属内广泛存在, 则其更多来源于遗传而非偶然获得的, 说明该编码基因在相应菌株中具有一定保守性(Gaur et al., 2015).
3 结果与讨论(Results and discussion) 3.1 抗性菌株的鉴定 3.1.1 抗性菌的生理生化特征自四环素浓度10 μg · L-1的选择培养基中分离得到一株四环素抗性菌, 命名为TC-1.菌株TC-1为革兰氏阳性菌, 菌落呈米黄色、圆形、微凸起, 边缘光滑整齐有光泽, 菌体在不同生长阶段呈杆状或球形, 具有明显的杆、球周期变化, 稳定期时几乎全部呈球状(图 1).菌株TC-1的生理生化鉴定结果见表 2.
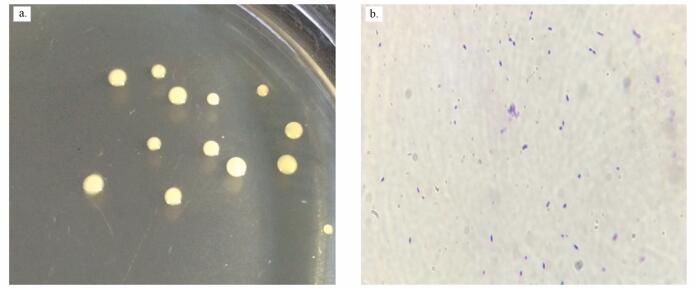 |
| 图 1 菌株TC-1形态观察(a.菌落, b.菌体形状) Fig. 1 Morphology of strain TC-1 (a. colony, b. cell shape) |
| 表 2 菌株TC-1的生理生化指标 Table 2 The physiological and biochemical indexes of strain TC-1 |
菌株TC-1的细菌DNA经PCR扩增后的16S rDNA大小为1411 bp, 将测序结果提交到NCBI数据库并进行BLASTN比对(序列号KY 678786), 发现菌株TC-1与节杆菌属的序列具有较高的同源性, 故选取与菌株TC-1同源性较高的菌株序列构建16S rDNA系统发育树(图 2).菌株TC-1与Arthrobacter uratoxydans strain DSM 20647(NR 026238)的同源性最高, 对比相似度达97%, 结合生理生化实验鉴定菌株TC-1属于微球菌科的节杆菌属.该菌属微生物广泛分布于自然环境, 尤其是土壤中, 属于条件致病菌(Fondi et al., 2012).
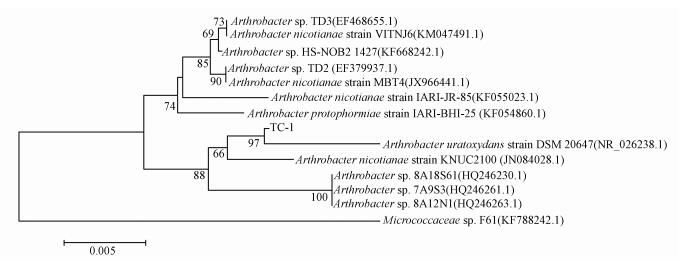 |
| 图 2 菌株TC-1的16S rDNA系统发育树 Fig. 2 Phylogenetic tree based the 16S rDNA sequence of strain TC-1 |
研究认为, 具有同源性的蛋白拥有相似甚至相同的生化功能, 因此对于一条给定的、未知功能的蛋白序列, 通常可根据其同源性蛋白的功能来推测它自身的功能(Hegyi et al., 1999), 此外, 同源性蛋白对于验证给定蛋白的功能注释具有至关重要的作用.通过NCBI数据库进行相似性检索发现, 菌株TC-1的16S rDNA序列与已知全基因序列的菌株Arthrobacter arilaitensis Re117(CBT 76982)有较高同源性.因此, 通过对菌株Arthrobacter arilaitensis Re117全基因序列的KEGG蛋白功能注释, 可近似分析菌株TC-1的蛋白序列信息.注释结果发现菌株TC-1中具有多种抗生素抗性蛋白, 如四环素外排蛋白(YP003916303)、氯霉素外排蛋白(YP003916969)、大环内酯类外排蛋白(YP003917752)和青霉素结合蛋白(YP003915290).城市污水处理厂接收生活污水和医院废水等, 污水成分复杂, 可能含有多种抗生素, 如四环素类、β-内酰胺类、大环内酯类、氯霉素类抗生素等, 因此污水处理厂中具有一种或多种抗生素抗性基因的微生物逐渐被富集(Rizzo et al., 2013).通过对菌株TC-1抗性蛋白编码基因的同源性分析, 可定性研究抗性蛋白在节杆菌属间的分类学关系, 并分析其抗性蛋白的功能特性.
四环素外排蛋白、氯霉素外排蛋白、大环内酯类外排蛋白属于细菌主动外排系统中的MFS药物转运蛋白.大多数主动外排泵系统是由质粒和染色体编码的, 根据氨基酸序列的同源性可以分为5个主要超家族, 包括小多耐药蛋白家族(Small multidrug resistance family, SMR)、多药及毒物外排家族(Multidrug and toxic compound extrusion family, MATE)、耐药结节分化超家族(Resistance-nodulation division family, RND)、主要易化子超家族(Major facilitator superfamily, MFS)、ATP结合盒超家族(ATP-binding cassette, ABC)(Poole, 2005).目前认为膜主动外排机制是由各种外排蛋白系统介导的把抗生素等药物从细菌细胞内泵出的主动排出过程.由于其底物的广泛性, 外排泵与细菌的多重耐药性密切相关.青霉素结合蛋白(Penicillin binding proteins, PBPs)是广泛存在于细菌表面的一种膜蛋白, 参与肽聚糖(Peptidoglycan, PG)合成的最后阶段, 是β-内酰胺类抗生素的主要作用靶位, 包括PBP1a、PBP1b、PBP1c、PBP2和PBP3(Sauvage et al., 2008).细菌有多种β-内酰胺类抗生素抗性机制, 例如产生β-内酰胺酶;通过降低对β-内酰胺类抗生素的亲和力产生新的PBPs;使用多重抗体从细胞中排出β-内酰胺类抗生素, 并通过孔蛋白突变减少β-内酰胺类抗生素进入(Zeng et al., 2013).在β-内酰胺类抗生素抗性机制中, PBPs的结构改变直接影响结合的抗生素的抗菌活性, 是重要的β-内酰胺类抗性机制(Sauvage et al., 2016).
将注释结果中发现的4种抗性蛋白序列与NCBI数据库中已知微生物蛋白序列进行Blastp相似性检索, 发现菌株TC-1的4种抗性蛋白序列与节杆菌属菌株的同源性较高, 相似度达99%以上, 进一步构建系统发育树同样表明其具有菌属内同源性(图 3).4种抗性蛋白序列的比对结果都是与节杆菌属菌株Arthrobacter sp. W1中蛋白序列的同源性最好, 相似度高达100%, 并且与多种节杆菌属菌株如Arthrobacter sp. YC-RL1、Arthrobacter sp. LS16、Arthrobacter sp. NIO1057等具有较高的同源性, 表明其抗性蛋白序列与菌株Arthrobacter sp. W1的对应功能酶具有很高的同源性, 功能上具有高度相似性.据此推测该菌株的四环素外排蛋白、氯霉素外排蛋白、大环内酯类外排蛋白和青霉素结合蛋白具有节杆菌属菌株中同源性蛋白的共性.鉴于此前的研究表明, 节杆菌属具有β-内酰胺类、大环内酯类、四环素类、喹诺酮类、磺胺类等20余种抗生素抗性, 是一种具有广谱抗生素抗性的条件致病菌(Kim et al., 2016; Tam et al., 2015), 节杆菌属菌株的存在和传播对于接收污水处理厂出水的环境可能产生严重的潜在致病威胁, 分析其抗性蛋白特性对节杆菌属抗生素抗性机理研究具有重要意义.
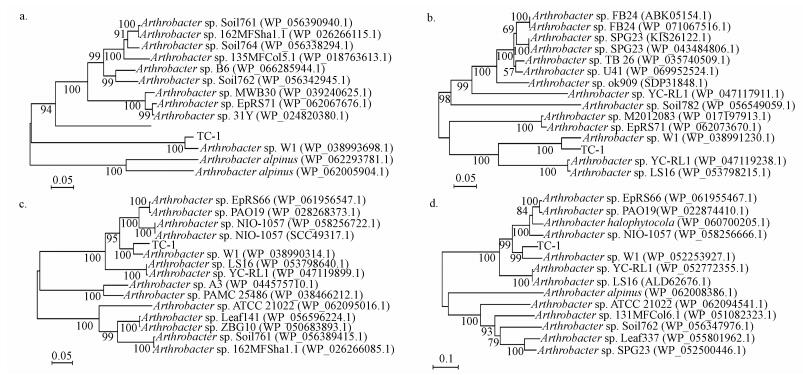 |
| 图 3 菌株TC-1抗性蛋白序列系统发育树(a.四环素外排蛋白, b.氯霉素外排蛋白, c.大环内酯类外排蛋白, d.青霉素结合蛋白) Fig. 3 Phylogenetic tree based the antibiotic resistance protein sequence of strain TC-1 (a. tetracycline efflux protein, b.chloramphenicol efflux protein, c. macrolide efflux protein, d. penicillin binding protein) |
将菌株TC-1抗性蛋白序列与SyntTax数据库中同种属菌株蛋白序列进行比对, 以分析抗性蛋白编码基因的基因簇同线性及抗性基因的保守性.结果表明, 其四环素外排蛋白、氯霉素外排蛋白、大环内酯类外排蛋白的编码基因与节杆菌属菌株Arthrobacter sp. QXT31、Arthrobacter sp. U41、Arthrobacter sp. LS16、Arthrobacter sp. YC RL1、Arthrobacter sp. ZXY2、Arthrobacter sp. A3和Arthrobacter sp. ERGS406的基因簇同线性较好, 同线性分数较高, 并且在菌株Arthrobacter sp. QXT31、Arthrobacter sp. U41、Arthrobacter sp. LS16、Arthrobacter sp. YC RL1、Arthrobacter sp. ZXY2、Arthrobacter sp. A3和Arthrobacter sp. ERGS406基因簇中发现可以编码MFS transporter的基因(图 4a、4b、4c).与四环素外排蛋白、氯霉素外排蛋白、大环内酯类外排蛋白的药物外排作用相吻合, 因此, 推测该菌株可通过外排泵作用将抗生素泵出体外, 使菌株获得了抗生素抗性.鉴于其抗性蛋白编码基因序列的高度同线性, 推测菌株TC-1可能与其他节杆属的菌株一样, 可通过外排泵作用将抗生素排出菌体外.对比节杆菌属抗生素抗性基因的基因簇分析表明, 四环素、氯霉素和大环内酯类抗生素抗性基因在节杆菌属中具有普遍存在性, 即来源于遗传而非单个菌株的偶然获得(Gaur et al., 2015).青霉素结合蛋白的基因簇同线性分析见图 4d, 菌株TC-1中的青霉素结合蛋白基因编码序列与多株节杆菌属菌株Arthrobacter sp. FB24、Arthrobacter sp. U41、Arthrobacter sp. QXT31、Arthrobacter sp. ATCC21022、Arthrobacter sp. YC RL1和Arthrobacter sp. ZXY2的基因簇同线性较好, 并且在Arthrobacter sp. FB24、Arthrobacter sp. U41、Arthrobacter sp. QXT31、Arthrobacter sp. ATCC21022、Arthrobacter sp. YC RL1和Arthrobacter sp. ZXY2菌株的基因簇中发现编码青霉素结合蛋白的基因, 鉴于其抗性基因序列的高度同线性, 推测菌株TC-1可能与其他节杆菌属菌株相同, 通过改变青霉素结合蛋白结构产生青霉素抗性.对比节杆菌属抗生素抗性基因的基因簇分析表明, 青霉素抗性基因在节杆菌属中具有普遍存在性, 即来源于遗传而非单个菌株的偶然获得(Gaur et al., 2015).
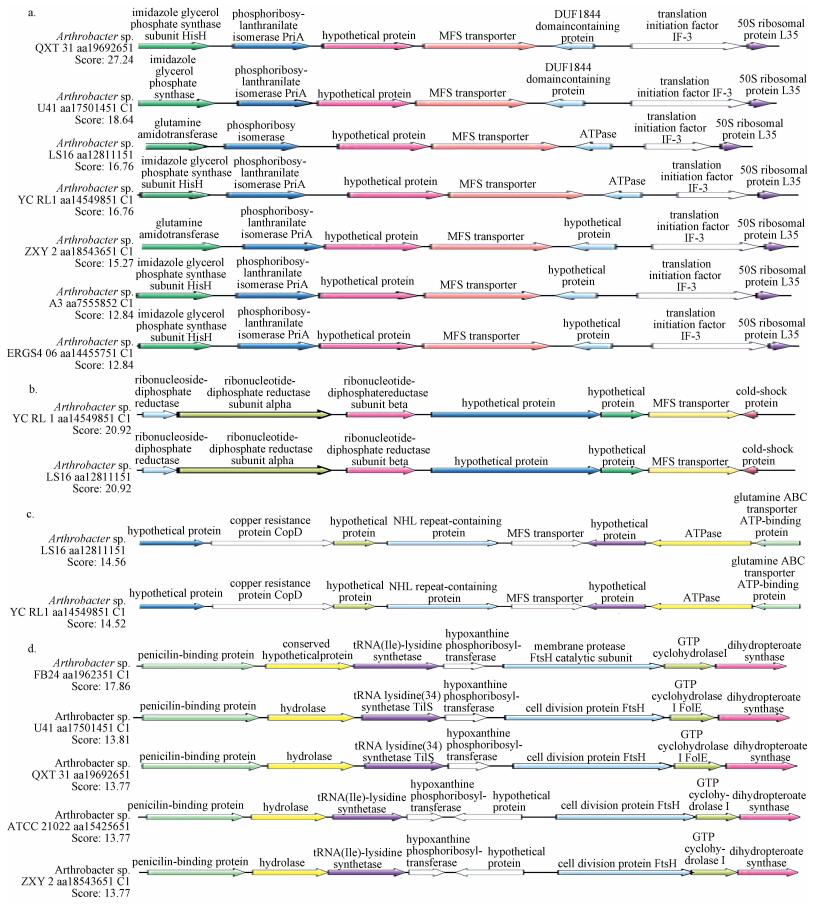 |
| 图 4 菌株TC-1抗性蛋白编码基因在不同节杆菌属间的对比分析(a.四环素转运蛋白, b.氯霉素转运蛋白, c.大环内酯类外排蛋白, d.青霉素结合蛋白) Fig. 4 Genes cluster analysis of encoding genes of antibiotic resistance protein in strain TC-1 between Arthrobacter (a. tetracycline efflux protein, b.chloramphenicol efflux protein, c. macrolide efflux protein, d. penicillin binding protein) |
环境中细菌的重金属抗性通常与抗生素抗性相关(Dey et al., 2012).近年来关于节杆菌属重金属耐受性(王旭梅等, 2010; 张汉波等, 2010)及抗生素抗性(Kim et al., 2016; Tam et al., 2015)的报道也日趋增多, 一些研究表明节杆菌属的微生物同样具有重金属抗性和抗生素抗性(Tam et al., 2015; 杨芬等, 2016).同样, 除上述抗生素抗性外, 本文所筛选的四环素抗性菌株TC-1也具有重金属Hg抗性蛋白, 但对节杆菌属菌株重金属和抗生素的联合抗性的研究尚不透彻(杨芬等, 2016; 张佳奇等, 2016), 仍需进一步的深入研究.
4 结论(Conclusions)1) 本研究从上海市某污水处理厂活性污泥中分离筛选到一株四环素抗性菌TC-1.经过形态观察、生理生化指标测定及16S rDNA序列分析后, 判定菌株TC-1为节杆菌属Arthrobacter sp., 属于革兰氏阳性菌.
2) 根据节杆菌属菌株的全基因组蛋白功能注释可知, 除四环素抗性外, 菌株TC-1同时具有多种抗生素抗性, 包括氯霉素、大环内酯类和青霉素抗性, 且该菌株的抗生素抗性蛋白与节杆菌属的对应功能酶具有很高的同源性, 功能上具有高度相似性.
3) 通过SyntTax数据库对菌株TC-1中的抗性蛋白编码基因进行基因簇同线性分析, 发现其抗性基因与节杆菌属菌株的基因簇同线性较较好, 由此表明四环素外排蛋白、氯霉素转运蛋白、大环内酯类外排蛋白和青霉素结合蛋白编码基因在节杆菌属菌株中具有普遍存在性, 即来源于遗传而非单个菌株的偶然获得.
Burch T R, Sadowsky M J, Lapara T M. 2014. Fate of antibiotic resistance genes and class I integrons in soil microcosms following the application of treated residual municipal wastewater solids[J]. Environmental Science and Technology, 48(10): 5620–5627.
DOI:10.1021/es501098g
|
Chen Q, An X, Li H, et al. 2016. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 92-93: 1–10.
DOI:10.1016/j.envint.2016.03.026
|
Christgen B, Yang Y, Ahammad S Z, et al. 2015. Metagenomics shows that low-energy anaerobic-aerobic treatment reactors reduce antibiotic resistance gene levels from domestic wastewater[J]. Environmental Science and Technology, 49(4): 2577–2584.
DOI:10.1021/es505521w
|
Dey S, Paul A K. 2012. Optimization of chromate reduction by whole cells of sp. SUK 1205 isolated from metalliferous chromite mine environment[J]. Geomaterials, 2: 73–81.
DOI:10.4236/gm.2012.24012
|
Du J, Ren H, Geng J, et al. 2014. Occurrence and abundance of tetracycline, sulfonamide resistance genes, and class I integron in five wastewater treatment plants[J]. Environmental Science and Pollution Research, 21(12): 7276–7284.
DOI:10.1007/s11356-014-2613-5
|
Feichtinger M, Mayrhofer S, Kneifel W, et al. 2016. Tetracycline resistance patterns of Lactobacillus buchneri group strains[J]. Journal of Food Protection, 79(10): 1741–1747.
DOI:10.4315/0362-028X.JFP-15-577
|
Fondi M, Orlandini V, Maida I, et al. 2012. Draft genome sequence of the volatile organic compound-producing Antarctic bacterium Arthrobacter sp. strain TB23, able to inhibit cystic fibrosis pathogens belonging to the Burkholderia cepacia complex[J]. Journal of Bacteriology, 194(22): 6334–6335.
DOI:10.1128/JB.01432-12
|
Gaur R, Jeena G, Shan N, et al. 2015. High density linkage mapping of genomic and transcriptomic SNPs for synteny analysis and anchoring the genome sequence of chickpea[J]. Scientific Reports, 5(13387): 1–11.
|
高俊红, 王兆炜, 张翰瑜, 等. 2016. 兰州市污水处理厂中典型抗生素的污染特征研究[J]. 环境科学学报, 2016, 36(10): 3765–3773.
|
Hegyi H, Gerstein M. 1999. The relationship between protein structure and function: A comprehensive survey with application to the yeast genome[J]. Journal of Molecular Biology, 288(1): 147–164.
DOI:10.1006/jmbi.1999.2661
|
Huang J J, Xi J, Hu H Y, et al. 2016. UV light tolerance and reactivation potential of tetracycline-resistant bacteria from secondary effluents of a wastewater treatment plant[J]. Journal of Environmental Sciences, 41(3): 146–153.
|
Huang K L, Tang J, Zhang X X, et al. 2014. A comprehensive insight into tetracycline resistant bacteria and antibiotic resistance genes in activated sludge using next-generation sequencing[J]. International Journal of Molecular Sciences, 15(6): 10083–10041.
DOI:10.3390/ijms150610083
|
Jiang L, Hu X, Xu T, et al. 2013. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 458-460(3): 267–272.
|
Kim H, Young M H, Park J E, et al. 2013. Sulfonamides and tetracyclines in livestock wastewater[J]. Chemosphere, 91(7): 888–894.
DOI:10.1016/j.chemosphere.2013.02.027
|
Kim J H, Kuppusamy S, Song Y K, et al. 2016. Occurrence of sulfonamide class of antibiotics resistance in Korean paddy soils under long-term fertilization practices[J]. Journal of Soils and Sediments, 17(6): 1–8.
|
Li B, Zhang X, Guo F, et al. 2013. Characterization of tetracycline resistant bacterial community in saline activated sludge using batch stress incubation with high-throughput sequencing analysis[J]. Water Research, 47(13): 4207–4216.
DOI:10.1016/j.watres.2013.04.021
|
Li L, Ye L, Zhang S, et al. 2016. Isolation and identification of aerobic bacteria carrying tetracycline and sulfonamide resistance genes obtained from a meat processing plant[J]. Journal of Food Science, 81(6): 1480–1484.
DOI:10.1111/1750-3841.13318
|
Liu C, Pan F, Guo J, et al. 2016. Hospital acquired pneumonia due to Achromobacter spp. in a geriatric ward in China: clinical characteristic, genome variability, biofilm production, antibiotic resistance and integron in isolated strains[J]. Frontiers in Microbiology, 7(621): 1–11.
|
Topal M, Şenel G U, Öbek E, et al. 2016. Investigation of relationships between removals of tetracycline and degradation products and physicochemical parameters in municipal wastewater treatment plant[J]. Journal of Environmental Management, 173: 1–9.
|
Poole K. 2005. Efflux-mediated antimicrobial resistance[J]. Journal of Antimicrobial Chemotherapy, 56(1): 20–51.
DOI:10.1093/jac/dki171
|
Pruden A. 2014. Balancing water sustainability and public health goals in the face of growing concerns about antibiotic resistance[J]. Environmental Science and Technology, 48(1): 5–14.
DOI:10.1021/es403883p
|
Rizzo L, Manaia C, Merlin C, et al. 2013. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment: A review[J]. Science of the Total Environment, 447(9): 345–360.
|
Sauvage E, Kerff F, Terrak M, et al. 2008. The penicillin-binding proteins: structure and role in peptidoglycan biosynthesis[J]. FEMS Microbiology Reviews, 32(2): 234–258.
DOI:10.1111/j.1574-6976.2008.00105.x
|
Tam H K, Wong C, Yong S T, et al. 2015. Multiple-antibiotic-resistant bacteria from the maritime Antarctic[J]. Polar Biology, 38(8): 1129–1141.
DOI:10.1007/s00300-015-1671-6
|
Wang F H, Qiao M, Chen Z, et al. 2015. Antibiotic resistance genes in manure-amended soil and vegetables at harvest[J]. Journal of Hazardous Materials, 299: 215–221.
DOI:10.1016/j.jhazmat.2015.05.028
|
王旭梅, 盛楠, 王红旗. 2010. 铅抗性细菌的筛选及其对铅活化的研究[J]. 东北农业大学学报, 2010, 41(6): 64–67.
|
杨芬, 巢波, 杨统一, 等. 2016. 土壤中一株抗生素和重金属交叉抗性菌的筛选及抗性研究[J]. 江苏科技大学学报(自然科学版), 2016, 30(3): 303–308.
|
张汉波, 程立忠. 2010. 云南会泽铅锌矿渣中节杆菌的生理特征和系统发育[J]. 微生物学通报, 2010, 37(6): 817–822.
|
张佳奇, 徐艳, 罗义, 等. 2016. 重金属协同选择环境细菌抗生素抗性及其机制研究进展[J]. 农业环境科学学报, 2016, 35(3): 409–418.
DOI:10.11654/jaes.2016.03.001 |
Zeng X, Lin J. 2013. Beta-lactamase induction and cell wall metabolism in Gram-negative bacteria[J]. Frontiers in Microbiology, 4(128): 128.
|
Zhang T, Zhang M, Zhang X. 2009. Tetracycline resistance genes and tetracycline resistant lactose-fermenting Enterobacteriaceae in activated sludge of sewage treatment plants[J]. Environmental Science and Technology, 43(10): 3455–3460.
DOI:10.1021/es803309m
|
Zhang X H, Xu Y B, He X L, et al. 2016. Occurrence of antibiotic resistance genes in landfill leachate treatment plant and its effluent-receiving soil and surface water[J]. Environmental Pollution, 218: 1255–1261.
DOI:10.1016/j.envpol.2016.08.081
|
Zhang X X, Zhang T. 2011. Occurrence, abundance, and diversity of tetracycline resistance genes in 15 sewage treatment plants across China and other global locations[J]. Environmental Science and Technology, 45(7): 2598–2604.
DOI:10.1021/es103672x
|
 2017, Vol. 37
2017, Vol. 37


