靛蓝是一种古老的色素, 广泛应用于医药、印染和食品等行业(Ensley et al., 1983; 韩晓红等, 2008).目前, 靛蓝的合成主要采用化学法, 但化学合成中的原料、催化剂以及中间产物等均具有一定的毒性, 给人类和环境造成严重的危害(Murdock et al., 1993).微生物法合成靛蓝具有高效、高选择性和绿色安全等优点, 近年来逐渐受到研究者的广泛关注.
研究表明, 很多芳烃降解菌能够以吲哚为底物绿色合成靛蓝(O′Connor et al., 1998).这些菌株在不同芳香化合物诱导下产生单加氧酶或双加氧酶, 如苯乙烯单加氧酶(O′Connor et al., 1997)、苯酚羟化酶(Kim et al., 2005)以及萘双加氧酶(Ensley et al., 1983)等, 随后吲哚被不同加氧酶氧化为3-羟基吲哚(吲哚酚), 最后吲哚酚在空气中氧化二聚生成靛蓝(Meyer et al., 2002; Mcclay et al., 2005).在靛蓝合成过程中通常还会有靛红、靛玉红、羟基吲哚等中间产物及其他副产物生成.目前报道的合成菌株大多为假单胞菌属(Pseudomonas), 对于其他菌属合成靛蓝的研究相对较少.丛毛单胞菌(Comamonas)是一类具有多种芳香化合物代谢能力的菌属, 能够用于菲、萘、苯酚及硝基苯等芳烃污染物的降解.关于丛毛单胞菌合成靛蓝的研究相对有限, 目前仅报道了丛毛单胞菌MQ能够在萘的诱导下利用萘双加氧酶转化吲哚合成靛蓝(Zhang et al., 2013; Qu et al., 2012b; 李诗阳等, 2012).
本实验室前期从活性污泥中分离得到一株具有吲哚降解能力的丛毛单胞菌IDO2(Ma et al., 2015), 本文对菌株IDO2转化吲哚合成靛蓝的特性进行研究.考察了菌株在不同芳烃化合物诱导剂合成靛蓝的能力, 并利用表面响应法对菌株IDO2合成靛蓝的条件进行优化.本研究旨在为靛蓝的绿色合成探索高效的微生物菌剂.
2 材料与方法(Materials and methods) 2.1 实验材料 2.1.1 菌株丛毛单胞菌属IDO2为本实验室前期从活性污泥中分离筛选得到的吲哚降解菌, 其16S rRNA已登录GenBank, 收录号为KP895479.
2.1.2 培养基本实验所用培养基为含0.1%酵母浸粉的无机盐培养基, 其中无机盐培养基组成为(g·L-1):(NH4)2SO4 2.0、KH2PO4 2.0、Na2HPO4 1.3、FeCl3 0.00025;pH 6.7.
2.1.3 试剂与仪器靛蓝、吲哚等均购自百灵威公司, 其余试剂均为国产分析纯.使用的主要仪器:Beckman AvantiTM J-30型高速低温冷冻离心机(美国), Shimadzu LC-20A型高效液相色谱(HPLC) (日本), Agilent 6224型液相色谱/飞行时间/质谱联用仪(LC/TOF/MS) (美国).
2.2 实验方法 2.2.1 靛蓝的微生物合成菌株IDO2以10%的接菌量接种于灭菌后的无机盐加酵母浸粉(0.1%)的培养基中, 分别加入不同诱导剂苯酚、萘、苯、菲, 浓度为200 mg·L-1, 吲哚浓度均为100 mg·L-1, 以只加吲哚的实验组作为对照.在摇床中(150 r·min-1, 30 ℃)反应48 h后, 利用HPLC对产物进行定量分析.菌株的生长通过测定菌体胞内蛋白量进行表征(Qu et al., 2012).
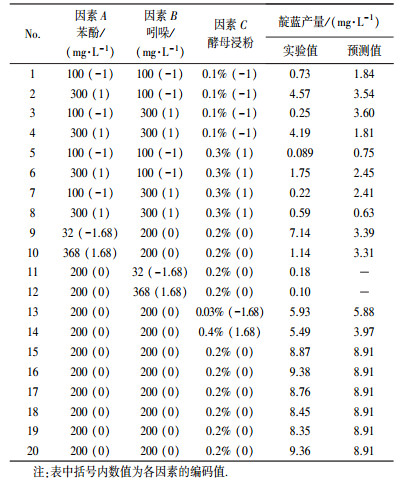
2.2.2 菌株IDO2合成靛蓝条件的优化为考察多因素对菌株转化吲哚合成靛蓝的影响, 采用表面响应法对菌株IDO2合成靛蓝的条件进行优化, 以反应体系靛蓝的产量为响应目标(Y), 考察的影响因素包括苯酚浓度(因素A)、吲哚浓度(因素B)以及培养基中酵母浸粉浓度(因素C).中心组合设计(Central composite design, CCD)是表面响应法中常用的一种5水平实验设计方法, 该方法能够通过有限次实验对影响生物过程的因子及其相互作用进行分析与评价, 利用响应面等高线图能够直观的描绘实验结果, 并通过实验模型和响应面等高线图可以对各个因子进行优化, 从而获得反应过程的最佳条件(刘建忠等, 2002).该方法已成功应用于多种生物过程的优化(李诗阳等, 2012; 曹湘禹等, 2012; Wang et al., 2015).在该设计中, 各因素中心点值组合通常会重复4~6次, 从而得到较准确的实验误差估量.从统计学计算角度, 按以下方程来计算实际值与编码值的关系:Ui= (Xi-X0)/ΔX.其中, Ui为变量的编码值, Xi为变量的实际试验水平值, X0为Xi的中心点值, ΔX为因素Xi水平变化值(Shi et al., 2013; Zhang et al., 2014).使用软件Design Expert 8.0.6对实验进行设计和分析, 苯酚和吲哚的浓度梯度为100~300 mg·L-1, 酵母浸粉的浓度梯度为0.1%~0.3%, 得到设计条件如表 1所示.利用方差分析(ANOVA)对模型及各影响因素进行检验, 当p值<0.05时, 表明该模型或相关影响因素具有显著性.
2.2.3 金属离子对菌株IDO2合成靛蓝的影响考察不同金属离子对菌株IDO2合成靛蓝量的影响.选定的金属离子为:Fe3+、Ca2+、Mg2+、Cu2+、Zn2+、Ni2+、Ba2+、Mn2+(阴离子均为Cl-), 在反应体系中的浓度为0.5 mmol·L-1.以不加金属离子的实验组为对照, 反应结束后用HPLC分析各体系靛蓝的产量.
2.2.4 产物的定量及定性分析将合成的产物在10000 r·min-1条件下离心10 min, 沉淀用二甲基亚砜(DMSO)重悬, 超声10 min, 再次离心(10000 r·min-1, 10 min)得到的蓝色上清液过0.45 μm有机膜, 采用HPLC对靛蓝进行定量分析.HPLC条件如下:Hypersil ODS2色谱柱(5 μm, 250 mm × 4.6 mm);进样量为10 μL;流速为1 mL·min-1;检测波长为280 nm;流动相为甲醇/水, 梯度洗脱:0~20 min, 60%~70%甲醇.
为分析转化过程的中间产物, 用乙酸乙酯萃取对合成物质萃取后, 利用LC/TOF/MS进行定性分析, 其中质谱采用电喷雾电离源(ESI), 负模式进行扫描.
3 结果与讨论(Results and discussion) 3.1 菌株IDO2在不同芳烃化合物诱导下转化吲哚合成靛蓝丛毛单胞菌IDO2能够以吲哚为唯一碳源进行生长, 并可对其降解, 菌株内可能含有多种芳烃加氧酶基因(Ma et al., 2015).前期研究表明, 芳烃化合物可以诱导菌株内特异性加氧酶的表达, 如萘双加氧酶、苯乙烯单加氧酶、苯酚羟化酶等, 吲哚能够被这些加氧酶羟化或氧化, 进而合成靛蓝(马桥等, 2012).因此, 本实验考察了不同芳烃化合物诱导下菌株IDO2转化吲哚合成靛蓝的特性, 分别采用苯酚、萘、苯和菲作为诱导剂.如图 1所示, 菌株IDO2对苯酚、萘、苯、菲及吲哚具有较好的降解性能, 体系中这几种物质的去除率均在80%以上, 说明菌株IDO2可以利用和降解多种芳香化合物, 具有较广的底物适应性, 其中对苯酚的去除效率最高.在这几种芳香化合物诱导下, 菌株IDO2均可转化吲哚合成靛蓝, 吲哚去除率均在90%以上, 但是合成的靛蓝产量具有一定差异.与萘、苯和菲相比, 苯酚诱导下菌株IDO2合成的靛蓝产量最高(4.45 mg·L-1), 说明苯酚诱导表达的羟化酶可能更有利于吲哚的氧化及靛蓝合成.
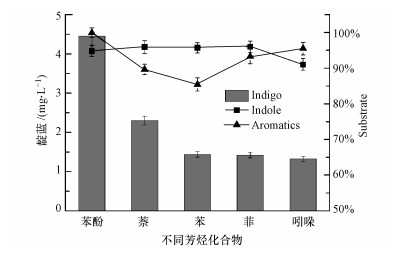 |
| 图 1 菌株IDO2在不同芳烃化合物诱导下的靛蓝产量及底物的去除率 Fig. 1 Indigo synthesis by strain IDO2 induced by different aromatic hydrocarbons and the removal rates of substrate |
菌株IDO2可以在苯酚诱导下以吲哚为底物合成靛蓝, 苯酚、吲哚以及培养基中的酵母浸粉可能对菌株生长及靛蓝合成过程有重要影响.因此, 采用表面响应法, 以靛蓝的产量为响应目标, 以吲哚、苯酚及酵母浸粉浓度为考察因素, 对菌株IDO2合成靛蓝的条件进行优化, 实验设计及结果如表 1所示.
| 表 1 菌株IDO2合成靛蓝的优化实验设计 Table 1 RSM design and results for the biosynthesis of indigo by strain IDO2 |
利用软件Design Expert 8.0.6对实验数据进行回归分析, 得到的响应目标对各编码值的回归方程为:
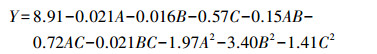
|
式中, Y为靛蓝产量, A为苯酚浓度, B为吲哚浓度, C为酵母浸粉浓度.根据此回归方程计算得到的靛蓝产量预测值列于表 1中.
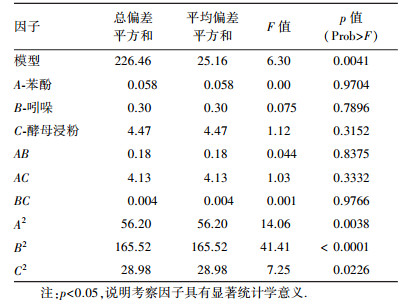
对上述回归方程进行ANOVA方差分析, 分析结果示于表 2中.模型的F值为6.30, 对应的p值为0.0041, 小于0.05, 说明该模型具有显著性.模型的R2为0.85, 说明该模型具有85%的可信度.此外, A2、B2、C2的p值均小于0.05, 说明在菌株IDO2转化吲哚合成靛蓝的过程中, 苯酚浓度、吲哚浓度及酵母浸粉浓度都是显著影响因素.
| 表 2 模型的ANOVA方差分析 Table 2 ANOVA results for response surface model |
利用软件分析还可得到各个单独变量之间相互作用对靛蓝产量影响的3D效果图(图 2).由图 2a、b可以观察到, 当吲哚浓度由100 mg·L-1增加到197.76 mg·L-1时, 靛蓝产量逐渐增加;当吲哚浓度为197.76 mg·L-1时, 靛蓝产量达到最大.由于吲哚是菌株合成靛蓝的直接底物, 其浓度在一定范围内逐渐增加时, 有利于靛蓝的产生;但是吲哚对菌株也具有一定的毒性作用, 过量的吲哚对菌株生长及表达的酶活性有一定影响, 从而造成靛蓝产量下降(Zhang et al., 2013; Qu et al., 2012b).由图 2a、c可观察到, 当苯酚的浓度由100 mg·L-1增加到203.39 mg·L-1时, 靛蓝产量呈上升趋势;在苯酚浓度达到203.39 mg·L-1时靛蓝产量达到最大.苯酚可诱导菌株内芳烃加氧酶的表达, 如苯酚羟化酶, 适宜的苯酚浓度可以提高相关加氧酶的表达量及活性, 从而有利于靛蓝的合成, 但是过量的苯酚也对菌株具有毒害作用(Wang et al., 2015).由图 2b、c可观察到, 随着酵母浸粉浓度的增加, 靛蓝产量先增加后降低, 在酵母浸粉浓度为0.18%时靛蓝产量达到最大.酵母浸粉富含多种营养元素, 如维生素、微量元素等, 这些元素可能有利于菌株的生长以及菌株内芳烃加氧酶的表达, 而过量的酵母浸粉可能不利于菌株对吲哚的羟化, 造成靛蓝产量下降.
 |
| 图 2 菌株IDO2合成靛蓝过程中各因素相互作用的3D响应图 (a.苯酚浓度(A)和吲哚浓度(B), b.吲哚浓度(B)和酵母浸粉浓度(C), c.苯酚浓度(A)和酵母浸粉浓度(C)) Fig. 2 Response surface plot showing the effects of the three factors on indigo production by strain IDO2 (a. Phenol (A) and indole concentrations (B), b. Indole concentrations (A) and yeast extracts (C), c. Phenol (B) and yeast extracts (C)) |
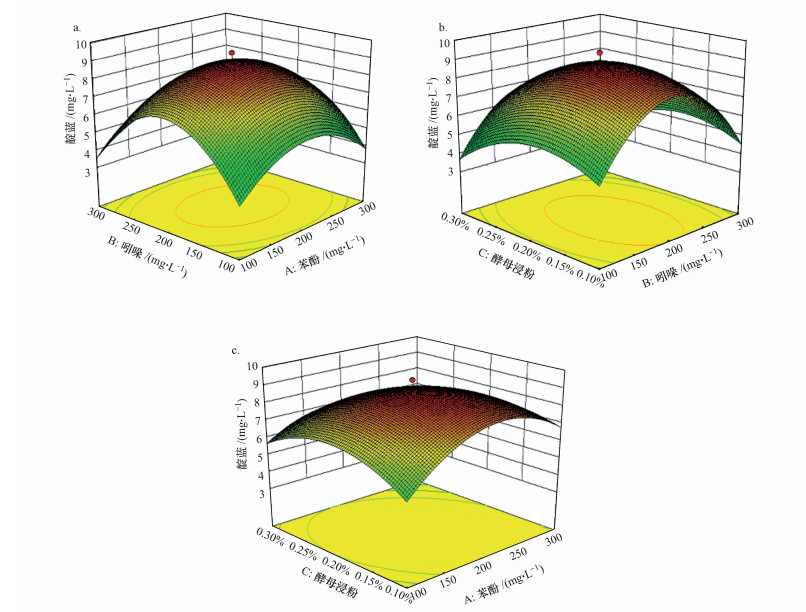
根据模型分析, 得到菌株IDO2合成靛蓝的最优条件为:吲哚197.76 mg·L-1, 苯酚203.39 mg·L-1, 酵母浸粉0.18%.利用该最优条件对菌株IDO2进行培养, 结果如图 3所示.菌株IDO2生长迟缓期较短, 4 h后开始进入对数生长期, 24 h后菌株进入稳定生长期.菌株IDO2在对数生长期时对吲哚具有较好的转化能力, 8 h后吲哚被菌株IDO2迅速利用, 靛蓝产量也逐渐增加, 24 h后吲哚转化率与靛蓝产量基本趋于稳定.靛蓝的产量可达到9.0 mg·L-1, 与初始条件下(吲哚100 mg·L-1, 苯酚200 mg·L-1, 酵母浸粉0.1%)相比, 靛蓝产量提高了1.04倍.
 |
| 图 3 菌株IDO2生长及靛蓝合成曲线 (a.菌株生长曲线, b.吲哚降解及靛蓝合成曲线) Fig. 3 Time course of growth and indigo production (a. Growth of strain IDO2, b. Indole consumption and indigo production) |
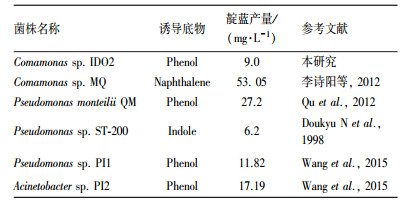
在前期研究中, 李诗阳等(2012)报道丛毛单胞菌MQ在萘诱导下转化吲哚合成靛蓝的产量可达53.05 mg·L-1;Qu等(2012)发现蒙氏假单胞菌QM可在苯酚诱导下合成靛蓝, 其产量为27.2 mg·L-1;此外, 假单胞菌ST-200可直接利用吲哚合成靛蓝, 产量为6.2 mg·L-1;Wang等(2015)报道假单胞菌PI1及不动杆菌PI2可在苯酚诱导下转化吲哚合成靛蓝, 产量分别为11.82 mg·L-1和17.19 mg·L-1(表 3).本研究的丛毛单胞菌IDO2与上述菌株相比, 靛蓝合成产量较低.由于菌株IDO2能够以吲哚为唯一碳源进行生长, 其对吲哚具有较好的降解效率, 可能大部分吲哚在反应过程中发生了降解, 而只有少部分被转化为靛蓝.因此, 后续研究有必要对菌株IDO2合成靛蓝的条件进行进一步优化, 可以从基因组学、蛋白组学、代谢组学等角度考虑, 增强菌株对吲哚的转化能力, 减少副产物的生成, 从而提高靛蓝的产量.
| 表 3 不同菌株合成靛蓝产量的比较 Table 3 Comparison of indigo yields produced by different bacterial strains |
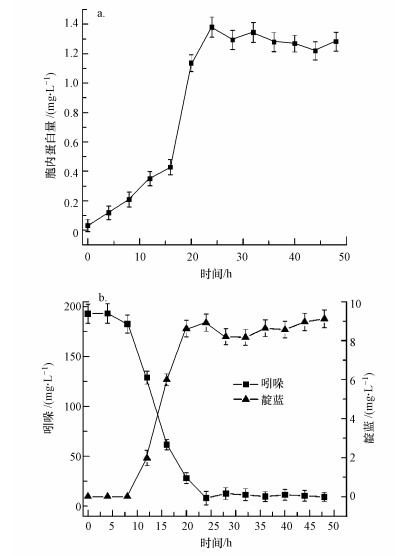
金属离子对靛蓝的合成有着重要影响, 本文考察了金属离子Ba2+、Ca2+、Mg2+、Fe3+、Cu2+、Mn2+、Ni2+、Zn2+对菌株IDO2转化吲哚合成靛蓝的影响, 结果如图 4所示.Ba2+和Ca2+对靛蓝的合成有明显的促进作用, Mg2+几乎无影响, 而Cu2+、Mn2+、Ni2+、Zn2+的抑制效果最为明显, 几乎无靛蓝产生.前期报道发现, Acinetobacter sp. PP-2在苯酚诱导下转化吲哚合成靛蓝时, Ba2+的投加能够明显提高靛蓝产量(Qu et al., 2010).
 |
| 图 4 不同金属离子对IDO2合成靛蓝量的影响 Fig. 4 Effects of different metal ions on indigo synthesis by strain IDO2 |
对菌株IDO2合成的蓝色物质进行HPLC分析, 结果显示(图 5a)在11.0 min时有较大峰出现, 其与标准靛蓝样品具有相同的保留时间, 其紫外可见-吸收光谱的最大吸收波长为241、286、335和616 nm, 与文献中报道的结果基本一致, 初步判断此蓝色产物为靛蓝.此外, 在2.4 min处有个小峰, 其紫外可见-吸收光谱的最大吸收波长为241和299 nm, 与标准靛红样品有相同的特征.利用LC/TOF/MS对菌株IDO2转化吲哚合成的产物进行分析, 结果显示(图 5b、c)在LC中11.0 min峰对应的分子离子峰(M—H)-为261.0668(图 5b), 进一步证实该物质为靛蓝;而2.4 min峰对应的分子离子峰(M—H)-为146.0246(图 5c), 推测该物质可能为靛红.然而, 产物中未检测到靛玉红、羟基吲哚等副产物或其他中间产物的存在.
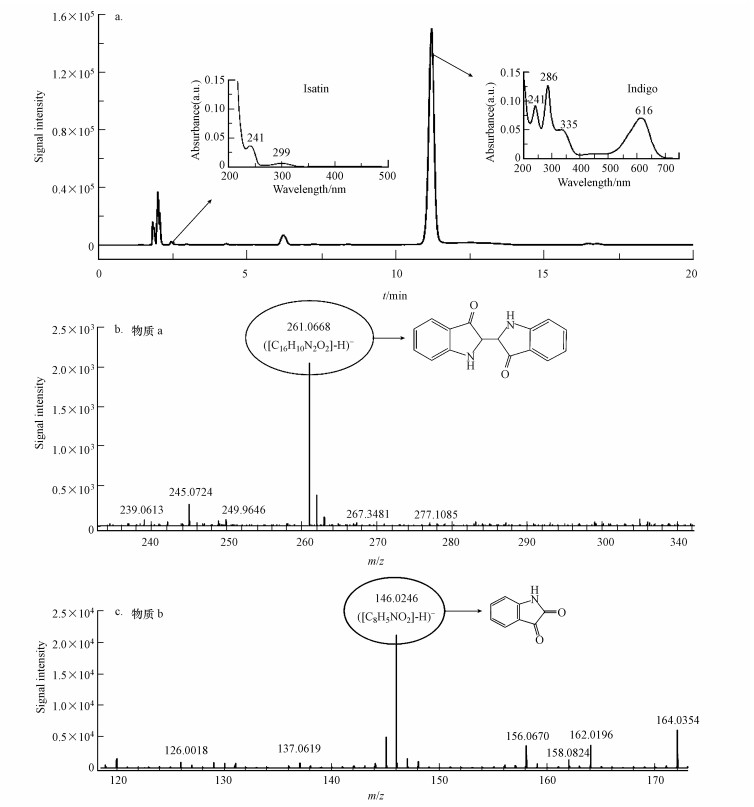 |
| 图 5 菌株IDO2合成产物的HPLC图谱 (a.内置图为对应吸收峰的紫外-可见吸收图谱)及LC/TOF/MS图谱(b、c) Fig. 5 HPLC (a, the inserts show the UV-visible spectrum of the chemicals) and LC/TOF/MS spectra (b, c) of the products produced by strain IDO2 |
根据LC/TOF/MS的产物分析结果及前期研究报道, 提出菌株IDO2转化吲哚合成靛蓝的可能途径(图 6).菌株IDO2可能携带苯酚羟化酶基因, 在苯酚的刺激下能够诱导苯酚羟化酶的表达.苯酚羟化酶在吲哚的C-3位对其氧化合成吲哚酚, 吲哚酚在空气中极不稳定, 能自发二聚合成靛蓝(Qu et al., 2012a; Wang et al., 2015).而吲哚酚进一步氧化能够生成靛红, 合成的靛蓝在光解或生物分解作用下也会产生靛红.通过本论文的研究, 进一步探索了丛毛单胞菌合成靛蓝的基本特性, 为靛蓝类色素的绿色合成提供了新颖的菌种资源.
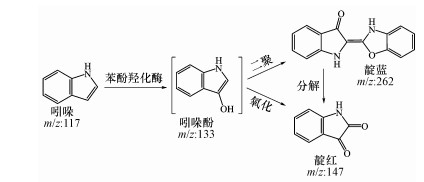 |
| 图 6 菌株IDO2合成靛蓝机理推测 Fig. 6 Proposed synthesis mechanism of indigo by strain IDO2 |
1) 菌株IDO2能在苯酚、萘、苯及菲诱导下转化吲哚合成靛蓝, 其中苯酚诱导合成的靛蓝产量最高.
2) 利用表面响应法对菌株IDO2合成靛蓝进行条件优化, 其最优条件为:吲哚197.76 mg·L-1, 苯酚203.39 mg·L-1, 酵母浸粉0.18%;得到的靛蓝产量为9.0 mg·L-1.金属离子Ba2+、Ca2+对菌株合成靛蓝有促进作用.
3) 利用LC/TOF/MS对菌株IDO2转化吲哚合成的产物进行了分析, 表明合成产物为靛蓝和靛红, 并推测可能的合成途径.
| [${referVo.labelOrder}] | 曹湘禹, 黄益养, 时胜男, 等. 2012. 苯酚羟化酶基因工程菌表达优化及特性研究[J]. 环境科学学报, 2012, 32(7): 1549–1556. |
| [${referVo.labelOrder}] | Doukyu N, Aono R. 1998. Purification of extracellular cholesterol oxidase with high activity in the presence of organic solvents from Pseudomonas sp. strain ST-200[J]. Applied and environmental microbiology, 64(5): 1929–1932. |
| [${referVo.labelOrder}] | Ensley B D, Ratzkin B J, Ossulund T D, et al. 1983. Expression of naphthalene oxidation genes in Escherichia coli results in the biosynthesis of indigo[J]. Science, 222(4260): 167–169. |
| [${referVo.labelOrder}] | 韩晓红, 王伟, 肖兴国. 2008. 靛蓝及其同类色素的微生物生产与转化[J]. 生物工程学报, 2008, 24(6): 921–926. |
| [${referVo.labelOrder}] | Kim J Y, Kim J K, Lee S O, et al. 2005. Multicomponent phenol hydroxylase-catalysed formation of hydroxyindoles and dyestuffs from indole and its derivatives[J]. Letters in Applied Microbiology, 41: 163–168. DOI:10.1111/lam.2005.41.issue-2 |
| [${referVo.labelOrder}] | 李诗阳, 袁琪, 黄益养, 等. 2012. 萘降解菌MQ合成靛蓝的研究[J]. 环境科学学报, 2012, 32(8): 1807–1813. |
| [${referVo.labelOrder}] | 刘建忠, 熊亚红, 翁丽萍, 等. 2002. 生物过程的优化[J]. 中山大学学报(自然科学版), 2002, 41: 132–137. DOI:10.3321/j.issn:0529-6579.2002.z1.038 |
| [${referVo.labelOrder}] | Ma Q, Qu Y, Zhang X, et al. 2015. Systematic investigation and microbial community profile of indole degradation processes in two aerobic activated sludge systems[J]. Scientific Reports, 5: 17674. |
| [${referVo.labelOrder}] | 马桥, 曲媛媛, 张旭旺, 等. 2012. 靛蓝的微生物合成研究新进展[J]. 应用与环境生物学报, 2012, 18(2): 344–350. |
| [${referVo.labelOrder}] | Mcclay K, Boss C, Keresztes I, et al. 2005. Mutations of toluene-4-monooxygenase that alter regiospecificity of indole oxidation and lead to production of novel indigoid pigments[J]. Applied and Environmental Microbiology, 71(9): 5476–5483. DOI:10.1128/AEM.71.9.5476-5483.2005 |
| [${referVo.labelOrder}] | Meyer A, Wursten M, Schmid A, et al. 2002. Hydroxylation of indole by laboratory-evolved 2-hydroxybiphenyl 3-monooxygenase[J]. Journal of Biological Chemistry, 277: 34161–34167. DOI:10.1074/jbc.M205621200 |
| [${referVo.labelOrder}] | Murdock D, Ensley B D, Serdar C, et al. 1993. Construction of metabolic operons catalyzing the de novo biosynthesis of indigo in Escherichia coli[J]. Nature Biotechnology, 11(3): 381–386. DOI:10.1038/nbt0393-381 |
| [${referVo.labelOrder}] | O'Connor K E, Dobson A D W, Hartmans S. 1997. Indigo formation by microorganisms expressing styrene monooxygenase activity[J]. Applied and Environmental Microbiology, 63(11): 4287–4291. |
| [${referVo.labelOrder}] | O'connor K E, Hartmans S. 1998. Indigo formation by aromatic hydrocarbon-degrading bacteria[J]. Biotechnology Letters, 20(3): 219–223. DOI:10.1023/A:1005361415496 |
| [${referVo.labelOrder}] | Qu Y, Pi W, Ma F, et al. 2010. Influence and optimization of growth substrates on indigo formation by a novel isolate Acinetobacter sp. PP-2[J]. Bioresource Technology, 101(12): 4527–4532. DOI:10.1016/j.biortech.2010.01.033 |
| [${referVo.labelOrder}] | Qu Y, Shi S, Zhou H, et al. 2012a. Characterization of a novel phenol hydroxylase in indoles biotranformation from a strain Arthrobacter sp. W1[J]. PLoS One, 7(9): e44313. DOI:10.1371/journal.pone.0044313 |
| [${referVo.labelOrder}] | Qu Y, Zhang X, Ma Q, et al. 2012b. Indigo biosynthesis by Comamonas sp. MQ[J]. Biotechnology Letters, 34(2): 353–357. DOI:10.1007/s10529-011-0778-2 |
| [${referVo.labelOrder}] | Shi S, Ma F, Sun T, et al. 2013. Biotransformation of indole to indigo by the whole cells of phenol hydroxylase engineered strain in biphasic systems[J]. Applied Biochemistry and Biotechnology, 169(4): 1088–1097. DOI:10.1007/s12010-012-0069-y |
| [${referVo.labelOrder}] | Wang J, Zhang X, Fan J, et al. 2015. Indigoids biosynthesis from indole by two phenol-degrading strains, Pseudomonas sp. PI1 and Acinetobacter sp. PI2[J]. Applied Biochemistry and Biotechnology, 176(5): 1263–1276. DOI:10.1007/s12010-015-1644-9 |
| [${referVo.labelOrder}] | Zhang X, Qu Y, Ma Q, et al. 2013. Cloning and expression of naphthalene dioxygenase genes from Comamonas sp. MQ for indigoids production[J]. Process Biochemistry, 48(4): 581–587. DOI:10.1016/j.procbio.2013.02.008 |
| [${referVo.labelOrder}] | Zhang X, Qu Y, Ma Q, et al. 2014. Production of indirubin from tryptophan by recombinant Escherichia coli containing naphthalene dioxygenase genes from Comamonas sp. MQ[J]. Applied Biochemistry and Biotechnology, 172(6): 3194–3206. DOI:10.1007/s12010-014-0743-3 |
 2017, Vol. 37
2017, Vol. 37