2. 台州学院生命科学学院, 台州 317000;
3. 哈尔滨工业大学城市水资源与水环境国家重点实验室, 哈尔滨 150090
2. School of Life Science, Taizhou University, Taizhou 317000;
3. State Key Laboratory of Urban Water Resource and Environment, Harbin Institute of Technology, Harbin 150090
近年来, 随着纺织印染工业的发展, 我国染料产量日益增长.其中, 蒽醌染料具有结构稳定、易于合成和颜色亮丽等良好性能, 作为高端染料呈现出良好的发展势头.1-氨基蒽醌-2-磺酸(ASA-2)不仅是一种合成蒽醌染料的中间体, 而且可经过溴化合成重要的蒽醌染料中间体溴氨酸, 其水溶液色彩鲜艳.高产能下, 蒽醌染料及其中间体在生产、合成和使用过程中有相当比重直接被排放到环境中, 成为工业废水的重要污染源.在我国淡水资源短缺、水体污染加剧和环境问题凸显的大背景下, 蒽醌染料及其中间体的无害化处理受到了广泛关注.生物方法因其能耗小、二次污染少和环境友好等优点而被广泛采用(Slokar and Majcen, 1998;Taha et al., 2014).已报道许多微生物如Bacillus benzeovorans(Walker and Weatherley, 2000)、Rhodopseudomonas XL-1(Dong et al., 2002)、Citrobacter sp. C-7(林海et al., 2010)和Pichia pastoris SMD1168H(Zheng et al., 2014)能够降解蒽醌染料及其中间体.然而工业废水体系复杂, 含多种复合成分, 如多种蒽醌类化合物及其中间体共存, 高盐, 有一定的酸碱度等, 这些都给降解菌株的底物广谱性及恶劣环境耐受性提出了新的挑战.因此, 筛选出高效、广谱性且能够适应工业废水复杂环境条件的降解菌株对工业蒽醌染料废水处理尤为重要.
近年来, 嗜吡啶红球菌凭借其对难降解污染物独特的降解能力受到了许多学者的关注, 成为一类具有潜力的新型微生物资源.已有文献报道, 嗜吡啶红球菌可以降解多氯联苯(Yoon et al., 2000)、石油充氧剂甲基叔丁醚(Vošahlíková-Kolářová et al., 2008)、内分泌干扰物玉米烯酮(Kriszt et al., 2012)和4-硝基甲苯(Kundu et al., 2013)等多种环境污染物.可见, 嗜吡啶红球菌在污染物治理方面有着巨大潜力, 但尚未有关于蒽醌染料及中间体降解方面的报道.
本实验室从受溴氨酸污染的泥土中筛选分离出一株新的蒽醌染料中间体1-氨基蒽醌-2-磺酸(缩写为ASA-2)降解菌, 通过形态学观察和16S rDNA序列分析, 鉴定其为嗜吡啶红球菌(Rhodococcus pyridinovorans).以ASA-2为模式污染物, 研究了菌株GF3对其生物脱色的特性, 并鉴定了ASA-2降解的终产物.此外, 研究发现该菌株还能够降解溴氨酸、蒽醌-2-磺酸钠和蒽醌-2-羧基等蒽醌化合物.
2 材料和方法(Material and methods) 2.1 实验材料菌株分离所用的泥土样品取自国内浙江台州某染料工厂排污口溴氨酸污染的泥土.1-氨基蒽醌-2-磺酸(缩写为ASA-2)由大连理工大学实验室提供, 分子量为303, 配置成5.0 g·L-1的ASA-2母液待用.氨基酸购买于Solarbio公司;甲醇、乙酸和三乙胺为高效液相色谱纯;其他试剂均为国产分析纯.
2.2 培养基富集培养基:蛋白胨10.0 g·L-1, 酵母膏5.0 g·L-1, NaCl 10.0 g·L-1(pH 7.2).
无机盐培养基:KH2PO40.5 g·L-1, Na2HPO4·12H2O 0.82 g·L-1, 0.002 5 g·L-1 FeCl3(pH 7.0).
2.3 实验方法 2.3.1 菌株的筛选将10 g污泥样品投加到200 mL富集培养基(含50 mg·L-1的ASA-2)中培养后, 取培养液接入渐减的富集培养基(酵母膏和蛋白胨含量是上一个培养基的一半)中驯化, 当培养基中ASA-2完全褪色后转接到下一个培养基内, 接种量为10%(体积比).经过大约40 d的驯化培养, 反复不断平板涂布, 最后选出1株具有较强降解能力的菌株(命名为GF3), 保存在15%甘油水溶液中置于-80 ℃冰箱.
2.3.2 菌体形态及16S rDNA测序用扫描电镜(SU80IO, Japan Hitachi)观察菌株GF3的外观形态, 放大倍数为10000倍.16S rDNA的测序工作由大连宝生物公司完成, 将测得的序列用BLAST软件与GenBank中已知的16S rDNA序列进行同源性比较.
2.3.3 菌体生长量与ASA-2浓度的测定菌体干重X(g·L-1)与菌液浊度(OD660nm)存在线性关系, 经实验得OD660nm=4.009 7 X + 0.0553(R2=0.9872).测量样品溶液在660 nm下的吸光度, 然后根据上述公式转换成菌体干重浓度.ASA-2浓度C(mg·L-1)测定采用分光光度计(JASCO UV-560, Japan)法, 即从ASA-2脱色体系取菌液在10000 r·min-1下离心5 min, 取上清液在ASA-2特征吸收波长处(475 nm)测量其吸光度, 根据ASA-2浓度标准曲线C=55.189 OD475 nm-0.7637(R2=0.9990)换算为浓度.ASA-2脱色率的计算公式:
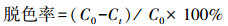
|
(1) |
式中, C0和Ct分别表示初始时刻和t时刻底物的浓度(mg·L-1).
| 表 1 外加营养物质的组成 Table 1 Composition of nutrients in medium |
将保藏在-80 ℃冰箱中的菌种接入20 mL无机盐培养基(含0.6 g·L-1酵母膏, 20 mg·L-1ASA-2)中, 于30 ℃、150 r·min-1培养至ASA-2完全脱色后, 按2%(体积比)的接种量将菌液接入50 mL无机盐培养基(含2.0 g·L-1酵母膏, 50 mg·L-1 ASA-2)中扩大培养.当ASA-2完全脱色时, 离心(10000 r·min-1, 10 min, 4 ℃), 并用磷酸缓冲溶液(20 mmol·L-1, pH=7.0)洗涤3次, 弃上清液, 重悬于10 mL磷酸盐缓冲溶液中.将制备的GF3菌悬液接入到不同的反应体系中, 考察不同外加营养物质、pH(5~10)、温度(20~40 ℃)和转速(0~250 r·min-1)对ASA-2脱色的影响.外加营养物质成分如表 1所示, 其中将氨基酸按照Sørensen′s(Sørensen et al., 2002)方法分为A1、A2、A3、A4, 共4组.
2.5 ASA-2脱色产物分析最适条件下, 将菌株GF3接入含有100 mg·L-1的ASA-2的无机盐培养基中, 定时取样, 测定不同时刻生物量和ASA-2浓度, 并进行紫外-可见波谱扫描.同时, 取100 mg·L-1 ASA-2降解过程中样品, 在10000 r·min-1, 4 ℃下离心(AvantiTM J-30 I, Beckman, USA)分离10 min, 取上清液过0.22 μm滤膜, 进行TOC(Multi N/C 2100S, Germany Jena)和高效液相色谱-质谱分析(HP1100 LC/MSD, Agilent, USA).
高效液相色谱(HPLC)分析条件:流动相超纯水和甲醇(体积比):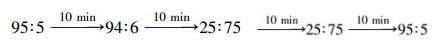
高效液相色谱-质谱联用分析条件:流动相甲醇和超纯水(含0.2%的乙酸和三乙胺), 甲醇体积分数2%, 流速0.2 mL·min-1, 直接质谱进样10 μL.电离方式ES-API(负模式), 检测模式Full scan(50~600 amu), 电离电压20 psig, 离子源温度350 ℃, 去溶剂气流量6.0 L·min-1.
2.6 菌株GF3降解底物广谱性最适条件下, 在外加20 mg·L-1的L-亮氨酸的无机盐培养基中, 加入不同的蒽醌化合物, 定时从反应体系取样, 测定蒽醌化合物的浓度变化.考察菌株GF3对其他蒽醌化合物的降解情况, 初步探索其降解底物广谱性.
3 结果与讨论(Results and discussion) 3.1 ASA-2脱色菌的分离与鉴定经过多次传代驯化、反复平板涂布, 分离纯化得到一株能够有效脱色ASA-2的菌株, 命名为GF3.固体培养基上, 菌株GF3形成的菌落较小, 边缘整齐, 呈圆形、淡红色.扫描电镜观察结果如图 1所示, GF3为球状或短杆状、无鞭毛, 大小约6~12 μm.该菌株对ASA-2有很强的脱色能力, 随着培养时间的延长, ASA-2原有的红色逐渐褪去, 最后完全消失.
 |
| 图 1 好氧培养菌株GF3的形态(×10000) Fig. 1 Morphology of strain GF3 under aerobic conditions (×10000) |
测定菌株GF3的16S rDNA序列, 并与GenBank数据库中的序列比对, 结果显示菌株GF3和Rhodococcus pyridinivorans(NR025033、KF381498)的16S rDNA序列相似性达到100%.因此, 该菌株鉴定为嗜吡啶红球菌(Rhodococcus pyridinivorans).将菌株序列提交至GenBank库中, 获得基因登录序列号为KF953541.
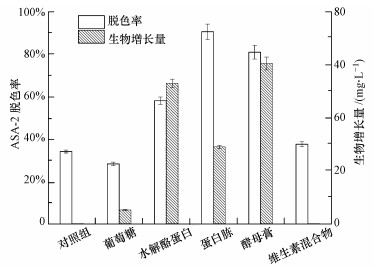
3.2 脱色条件优化 3.2.1 外加营养物质对ASA-2生物脱色的影响菌株GF3能够使ASA-2脱色, 但不能以它为唯一碳源生长.Fan等(Fan et al., 2009)曾报道外加碳源如葡萄糖可提高鞘氨醇单胞菌FL对溴氨酸的脱色率.基于此, 本文研究了外加营养物质酵母膏、蛋白胨、水解酪蛋白、葡萄糖和维生素混合物对ASA-2脱色的影响.实验结果如图 2所示, 在22 h内, 外加蛋白胨、酵母膏和水解酪蛋白的反应体系中ASA-2脱色率依次达到90%、81%和58%, 而外加葡萄糖和维生素混合物体系的脱色率与对照组相似, 仅为30%左右.可见, 蛋白胨、酵母膏和水解酪蛋白对ASA-2脱色有明显促进作用.
 |
| 图 2 外加营养物质对ASA-2脱色的影响 Fig. 2 Effect of additional nutrients on ASA-2 decolorization |
同时, 含有这3种营养物质的反应体系中生物量也分别增长了29、61和53 mg·L-1.虽然酵母膏和水解酪蛋白体系中菌株GF3的生物量都明显高于蛋白胨体系, 但是ASA-2脱色率达到最高的却是外加蛋白胨体系.Dong等(Dong et al., 2003)曾报道蛋白胨可刺激红环菌Rhodocyclus gelatinosus XL-1对蒽醌类化合物活性艳蓝KN-R的生物降解, 进而缩短降解所需的时间.推测蛋白胨中的营养成分可能提高了菌株的脱色活性.
3.2.2 不同氨基酸对ASA-2脱色的影响蛋白胨的主要成分是氨基酸, 水解酪蛋白是酪蛋白经酸水解后形成的氨基酸混合物, 酵母膏中也含有一些氨基酸, 因此推测氨基酸可能在加速ASA-2生物脱色过程中起到了重要作用.已有相关文献报道一些氨基酸能够加速特定污染物的生物降解.Ronen等(Ronen et al., 2005)在研究Achromobacter piechaudii TBPZ降解2, 4, 6-三溴苯酚过程中, 发现色氨酸和苯丙氨酸的加入大大提高了菌株TBPZ生物降解活性, 进而加速了2, 4, 6-三溴苯酚的生物降解.Wu等(Wu et al., 2011)曾报道, 氨基酸是加速伯克氏菌Burkholderia sp. GS3C对十六烷生物降解的主要影响因素.鉴于此, 考察了4组氨基酸混合物(A1、A2、A3、A4, 见表 1)对ASA-2脱色的影响, 结果如图 3a所示.
 |
| 图 3 外加氨基酸对ASA-2脱色的影响(A.氨基酸组合;B. A1中单个氨基酸) Fig. 3 Effect of additional amino acids on ASA-2 decolorization (A. Amino acid mixtures; B. Single amino acids in A1) |
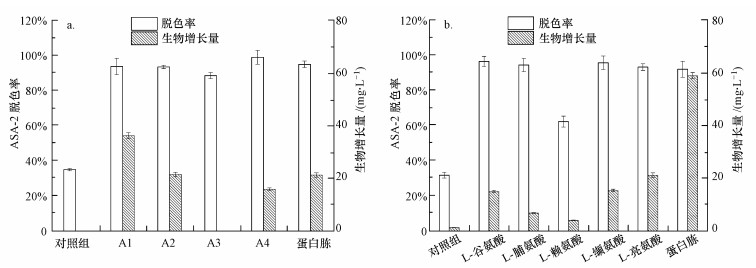
4组氨基酸混合物均对菌株GF3脱色ASA-2有明显促进作用, 其促进作用与500 mg·L-1蛋白栋相当或者更优.A1、A2和A4的添加使ASA-2脱色率分别提高至94%、93%和99% (空白对照组22 h后ASA-2脱色率为35%).同时, 菌株GF3的细胞浓度依次增长了36、21和16 mg·L-1.可见, A1比其它实验组能够更好地促进菌株GF3的细胞增殖.因此, 对A1中单个氨基酸的影响分别进行了研究, 结果如图 3b所示.
A1中5种氨基酸均能在促进ASA-2脱色的同时促进菌株GF3自身细胞增殖.除了L-赖氨酸, 其它4种氨基酸体系ASA-2脱色率均达93%以上, 且与蛋白胨的促进效果相当.并且这4种氨基酸也能较好地促进细胞增殖.这种现象表明, 这4种氨基酸一方面可能是合成相关脱色酶的原料, 另一方面这些氨基酸可能通过促进细胞的增殖来促进ASA-2脱色.据报道, 鞘氨醇单胞菌降解溴氨酸和ASA-2的过程中, 只有脯氨酸的促进效果与蛋白胨相似, 并且发现脯氨酸与脱色酶活性密切相关(Lu et al., 2015).Ronen和Wu也曾报道少数氨基酸可加速难降解物质的生物降解(Ronen et al., 2005;Wu et al., 2011), 但单独氨基酸的促进作用都小于混合氨基酸的促进作用(Wu et al., 2011).然而本实验A1组的L-谷氨酸、L-脯氨酸、L-缬氨酸和L-亮氨酸的添加对ASA-2脱色的促进效果均与这4种氨基酸混合物的促进效果相当, 不过细胞生长量是明显低于这4种氨基酸混合物体系.可见, 过多氨基酸的添加也仅仅是促进了细胞更好的生长.
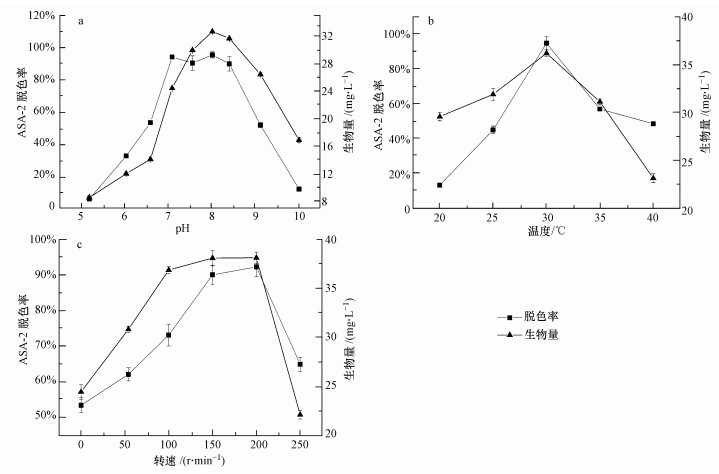
3.2.3 环境因素对ASA-2脱色的影响pH、温度和溶解氧是影响微生物生存与生长以及对污染物降解的重要因素.在外加20 mg·L-1亮氨酸作为共代谢碳源的条件下, 考察了不同pH、温度和摇床转速对菌株GF3脱色ASA-2的影响.实验结果如图 4所示.
 |
| 图 4 不同环境因子对ASA-2脱色的影响 Fig. 4 Effect of environmental factors on ASA-2 decolorization |
当培养基初始pH在7.0~8.5之间时, ASA-2的脱色率最高, 达90%以上.其中pH为8.0时, 反应体系中细胞浓度最高.当初始pH值在8.5~9.0范围内菌株GF3仍然生长良好, 一方面表明该菌株对偏碱环境具有较强的适应能力, 另一方面也可能是因为ASA-2脱色过程中产生了偏酸性产物造成.在测定的20~40 ℃范围内, ASA-2脱色的最适宜温度为30 ℃, 脱色率达到95%, 菌株GF3细胞浓度也达到最高.过高或过低的温度, 都使ASA-2脱色率和细胞生长量明显下降.ASA-2脱色的适宜转速范围在150~200 r·min-1之间, 此时ASA-2脱色率达90%以上, 菌株GF3生长也最好.综合考虑环境因素影响的实验结果, ASA-2脱色的适宜环境条件为pH 8.0、30 ℃和150 r·min-1.
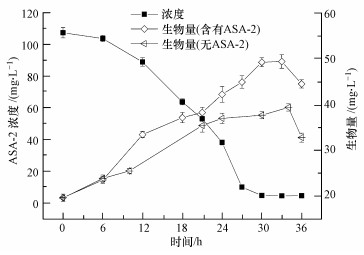
3.3 菌株GF3脱色ASA-2的产物分析 3.3.1 ASA-2的生物脱色过程最适条件下, 研究了ASA-2的生物脱色过程, 实验结果如图 5所示.反应开始6 h内, ASA-2的脱色率只有3%, 含有ASA-2和亮氨酸体系的细胞生长速率和只含有亮氨酸体系的细胞生长速率基本一致.可见, 这期间主要是亮氨酸促进了细胞的生长繁殖.6 h后, ASA-2开始快速脱色, 菌株GF3快速增长, 到30 h ASA-2脱色率已经达95%以上, 此时细胞浓度增加到49 mg·L-1(0 h为20 mg·L-1), 并且明显高于只有亮氨酸作为碳源的对照体系.由此推测菌株GF3可能利用了ASA-2的脱色产物来生长.在随后30~33 h内, ASA-2脱色率不变, 细胞略有增长, 推测这一阶段细胞也是利用中间代谢产物进行生长.
 |
| 图 5 ASA-2脱色过程曲线 Fig. 5 Time course of ASA-2 decolorization |
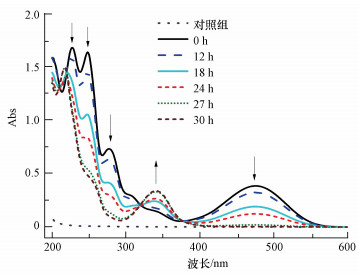
取脱色过程中的样品, 稀释5倍进行紫外-可见波谱分析, 结果如图 6所示.随着反应的进行, ASA-2在紫外光区227、248 nm处和可见光区475 nm处的特征吸收峰逐渐减弱, 最终完全消失, 这与反应液的颜色由红色渐变为无色相一致.由此推断ASA-2蒽醌环结构被破坏, 发色基团发生明显变化.同时在340 nm处逐渐产生了新的特征吸收峰, 峰值逐渐增大.并且, 在475 nm处的特征吸收峰完全消失时, 340 nm处的峰值达到最大.这一现象说明, 在ASA-2的脱色过程中有产物生成, 且不能够被菌株GF3利用.
 |
| 图 6 ASA-2脱色过程的紫外-可见波谱 Fig. 6 UV-VIS during ASA-2 decolorization |
与鞘氨醇单胞菌QYY脱色ASA-2不同, 本实验在340 nm处产生了新的特征吸收峰, 且吸收峰强度达最大后不再减弱.而菌株QYY脱色ASA-2(Lu et al., 2008)过程中是在375 nm处出现了新峰, 并且随着脱色时间的推移, 375 nm处的吸收峰逐渐减弱.因此推测菌株GF3和QYY在对ASA-2的脱色过程中, 蒽醌环断裂的方式可能不同, 导致最终降解产物也不相同, ASA-2被菌株GF3脱色后生成新的化合物.
实验进一步测定了108 mg·L-1 ASA-2脱色过程中总有机碳TOC(扣除了L-亮氨酸的TOC值)的变化.在培养36 h的过程中, 总有机碳由最初的53 mg·L-1, 逐渐减少为20 mg·L-1, 总有机碳降低了约62%.并且随着反应时间的推移, 总有机碳不再变化.可见, 菌株GF3只利用了部分有机碳, 并未将ASA-2完全矿化.
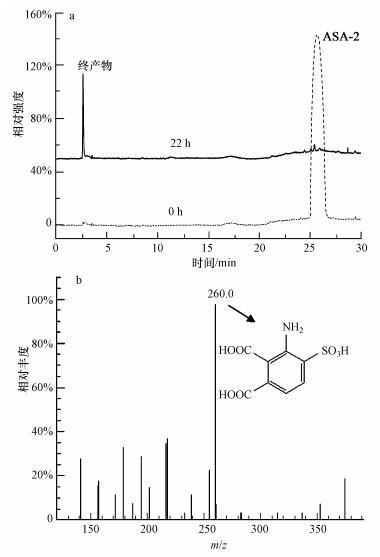
3.3.2 ASA-2脱色产物分析对ASA-2完全脱色后的样品进行液相色谱-质谱分析, 实验结果如图 7所示, ASA-2脱色终产物质荷比为260, 初步推测产物为3-氨基-4-磺酸基邻苯二甲酸.这与菌株QYY降解ASA-2的终产物不同.并由此推测菌株GF3裂解蒽醌环的方式与以往文献报道的蒽醌环裂解方式不同(Fan et al., 2008).可见, 菌株GF3通过一种新的降解途径将ASA-2降解.
 |
| 图 7 ASA-2降解终产物的液相-质谱图 Fig. 7 LC-MS spectra of ASA-2 decolorization product |
在外加20 mg·L-1的L-亮氨酸的无机盐培养基中, 加入不同的蒽醌化合物, 定时从反应体系取样, 测定蒽醌化合物的浓度变化.实验结果显示, 除了1-氨基蒽醌-2-磺酸外, 菌株GF3还能够降解100 mg·L-1的溴氨酸、蒽醌-2-磺酸钠和蒽醌-2-羧酸, 使它们的降解率分别达到90%、99%和94%.但是, 菌株GF3不能够降解蒽醌-2, 6-二磺酸钠、活性艳蓝KN-R和茜素红.
4 结论(Conclusions)1)新分离出一株1-氨基蒽醌-2-磺酸降解菌株, 鉴定为嗜吡啶红球菌(Rhodococcus pyridinivorans).
2)蛋白胨、酵母膏、水解酪蛋白可加速菌株GF3脱色ASA-2, 其中蛋白胨促进效果最好.单个氨基酸中L-亮氨酸对ASA-2生物脱色的促进效果最好, 同时能显著促进菌株GF3的细胞增殖.ASA-2生物脱色的最适环境条件为:pH 8.0、30 ℃和150 r·min-1.30 h内菌株可使108 mg·L-1 ASA-2降解率达95%, 总有机碳去除率62%.
3)菌株GF3能够使ASA-2的蒽醌环裂解, 终产物为3-氨基-4-磺酸基邻苯二甲酸.菌株GF3不仅能降解1-氨基蒽醌-2-磺酸, 还能够降解溴氨酸、蒽醌-2-磺酸钠和蒽醌-2-羧酸.但是菌株GF3不能够降解蒽醌-2, 6-二磺酸钠、活性艳蓝KN-R和茜素红.
| [${referVo.labelOrder}] | Balch W E, Fox G E, Magrum L J, et al. 1979. Methanogens:reevaluation of a unique biological group[J]. Microbiological Reviews , 43 (2) : 260. |
| [${referVo.labelOrder}] | Dong X L, Zhou J T, Wang J. 2002. Decolorization of Anthraquinone dye by Rhodopseudomonas XL-1[J]. HighTechnology Letters , 8 (2) : 11–14. |
| [${referVo.labelOrder}] | Dong X L, Zhou J T, Liu Y. 2003. Peptone-induced biodecolorization of Reactive Brilliant Blue (KN-R) by Rhodocyclus gelatinosus XL-1[J]. Process Biochemistry , 39 (1) : 89–94. DOI:10.1016/S0032-9592(02)00319-9 |
| [${referVo.labelOrder}] | Fan L, Zhu S, Liu D, et al. 2009. Decolorization of 1-amino-4-bromoanthraquinone-2-sulfonic acid by a newly isolated strain of Sphingomonas herbicidovorans[J]. International Biodeterioration & Biodegradation , 63 (1) : 88–92. |
| [${referVo.labelOrder}] | Fan L, Zhu S, Liu D, et al. 2008. Decolorization mechanism of 1-amino-4-bromoanthraquinone-2-sulfonic acid using Sphingomonas herbicidovorans FL[J]. Dyes and Pigments , 78 (1) : 34–38. DOI:10.1016/j.dyepig.2007.10.004 |
| [${referVo.labelOrder}] | Kriszt B, Táncsics A, Cserháti M, et al. 2012. De novo genome project for the aromatic degrader Rhodococcus pyridinivorans strain AK37[J]. Journal of Bacteriology , 194 (5) : 1247–1248. DOI:10.1128/JB.06603-11 |
| [${referVo.labelOrder}] | Kundu D, Hazra C, Dandi N, et al. 2013. Biodegradation of 4-nitrotoluene with biosurfactant production by Rhodococcus pyridinivorans NT2: metabolic pathway, cell surface properties and toxicological characterization[J]. Biodegradation , 24 (6) : 775–793. DOI:10.1007/s10532-013-9627-4 |
| [${referVo.labelOrder}] | 林海, 程琳, 胡琼.2010. 降解高含盐溴氨酸菌种的选育与鉴定[J]. 北京科技大学学报 , 2010, 32 (11) : 1400–1405. |
| [${referVo.labelOrder}] | Lu H, Guan X F, Wang J, et al. 2015. Enhanced bio-decolorization of 1-amino-4-bromoanthraquinone-2-sulfonic acid by Sphingomonas xenophaga with nutrient amendment[J]. Journal of Environmental Sciences , 27 : 124–130. DOI:10.1016/j.jes.2014.05.041 |
| [${referVo.labelOrder}] | Lu H, Zhou J T, Wang J, et al. 2008. Decolorization of 1-aminoanthraquinone-2-sulfonic acid by Sphingomonas xenophaga[J]. World Journal of Microbiology and Biotechnolog , 24 (7) : 1147–1152. DOI:10.1007/s11274-007-9586-1 |
| [${referVo.labelOrder}] | Ronen Z, Visnovsky S, Nejidat A. 2005. Soil extracts and co-culture assist biodegradation of 2, 4, 6-tribromophenol in culture and soil by an auxotrophic Achromobacter piechaudii strain TBPZ[J]. Soil Biology and Biochemistry , 37 (9) : 1640–1647. DOI:10.1016/j.soilbio.2005.02.001 |
| [${referVo.labelOrder}] | Slokar Y M, Le Marechal A M. 1998. Methods of decoloration of textile wastewaters[J]. Dyes and pigments , 37 (4) : 335–356. DOI:10.1016/S0143-7208(97)00075-2 |
| [${referVo.labelOrder}] | Sørensen S R, Ronen Z, Aamand J. 2002. Growth in coculture stimulates metabolism of the phenylurea herbicide isoproturon by Sphingomonas sp. strain SRS2[J]. Applied and Environmental Microbiology , 68 (7) : 3478–3485. DOI:10.1128/AEM.68.7.3478-3485.2002 |
| [${referVo.labelOrder}] | Taha M, Adetutu E M, Shahsavari E, et al. 2014. Azo and anthraquinone dye mixture decolorization at elevated temperature and concentration by a newly isolated thermophilic fungus, Thermomucor indicae-seudaticae[J]. Journal of Environmental Chemical Engineering , 2 (1) : 415–423. DOI:10.1016/j.jece.2014.01.015 |
| [${referVo.labelOrder}] | Vošahlíková-Kolářová M, Krejčík Z, Cajthaml T, et al. 2008. Biodegradation of methyl tert-butyl ether using bacterial strains[J]. Folia Microbiologica , 53 (5) : 411–416. DOI:10.1007/s12223-008-0062-6 |
| [${referVo.labelOrder}] | WuR R, Dang Z, Yi X Y, et al. 2011. The effects of nutrient amendment on biodegradation and cytochrome P450 activity of an n-alkane degrading strain of Burkholderia sp. GS3C[J]. Journal of Hazardous Materials , 186 (2) : 978–983. |
| [${referVo.labelOrder}] | Walker G M, Weatherley L R. 2000. Biodegradation and biosorption of acid anthraquinone dye[J]. Environmental Pollution , 108 (2) : 219–223. DOI:10.1016/S0269-7491(99)00187-6 |
| [${referVo.labelOrder}] | Yoon J H, Kang S S, Cho Y G, et al. 2000. Rhodococcus pyridinivorans sp. nov., a pyridine-degrading bacterium[J]. International Journal of Systematic And Evolutionary Microbiology , 50 (6) : 2173–2180. DOI:10.1099/00207713-50-6-2173 |
| [${referVo.labelOrder}] | Zheng M, Chi Y, Yi H, et al. 2014. Decolorization of Alizarin Red and other synthetic dyes by a recombinant laccase from Pichia pastoris[J]. Biotechnology Letters , 36 (1) : 39–45. DOI:10.1007/s10529-013-1323-2 |
 2016, Vol. 36
2016, Vol. 36


