海洋是地球上最大的硫库,海水中硫酸盐平均含量高达29 mmol ·L-1;而沉积物是海洋硫酸盐最重要的“汇”,是典型的富硫生境(吴自军等,2013).在海洋渔业生产过程中,大量饵料的投放导致养殖水体中有机营养过剩.在沉积物-水界面附近,好氧微生物在降解有机饵料过程中,消耗大量氧气,进而导致海水-沉积物界面层以下形成缺氧环境(Purdy et al.,2003).在缺氧条件下,硫酸盐还原菌(SRB)将继续降解有机物,并通过厌氧硫酸盐呼吸作用,将电子传递给硫酸盐产生硫化物.在沉积物-水界面及其以下生境中,虽然硫化物能够同重金属结合形成不溶性化合物,从而降低重金属的毒性(Garcia et al.,2011);但是,残留的高浓度溶解性硫化物,则极易毒害底栖生物和水生生物(Vaquer-Sunyer and Duarte,2010).因此,在沉积物-水界面探讨硫化物的生物产生过程及微生物特征类群,对于硫化物的控制和消除、形成健康的养殖环境具有重要的理论意义.
沉积物-水界面系统中积累的硫化物,全部由硫酸盐的异化还原作用或有机物的矿化作用产生,其中SRB发挥着主体作用;这些硫化物约有99%将被水体中的溶解氧或微生物进一步氧化,而硫氧化相关的微生物类群担当重要角色.可见,养殖沉积环境中,硫化物以微生物为基础,通过产生-消除的动态平衡和微循环过程长期维持着稳定分布(Lavik et al.,2009).而在很多研究中,对这些环境中硫循环微生物的研究,均以培养为基础进行,限制了对硫循环微生物种类的全面认识,进而阻碍了对硫化物产生过程的控制.另外,在沉积环境硫化物控制过程中,虽然有研究采用颗粒活性碳吸附或通过固定氧化铁颗粒氧化的方式去除,并取得较好的效果(Asaoka et al.,2009;Sun et al.,2013);但相比而言,利用微生物对“老化”沉积物的修复更具有持续性,可见,应用硫氧化型微生物对沉积物中硫化物的控制,具有较好的应用前景,将是养殖区沉积物-水界面硫化物污染控制的重要方向.而深入揭示该生境中参与硫循环的微生物群落特征,将为硫化物的生物控制提供重要的理论依据.
本研究通过人为模拟“老化”的养殖区沉积环境,应用变性梯度凝胶电泳技术(DGGE)和高通量测序技术(High-throughput sequencing)解析微生物群落,尤其是硫循环相关微生物类群的垂直分布规律,并与硫化物、溶解氧等环境因子相比较,为筛选快速有效的硫化物控制微生物以及修复养殖环境提供理论基础.
2 材料与方法(Materials and methods) 2.1 养殖环境模型模型为长方体,采用有机玻璃制作,高40 cm,容积25 L,侧壁及顶端不同间隔设取样口以及设备监测口.自胶州湾采集海水养殖区沉积物并与实验室原有硫酸盐还原反应器污泥混合,置于模型中,泥高约20 cm;上覆约15 cm的高浓度硫酸盐养殖水体.将模型置于室温下,静置2年以上,直到形成清晰稳定的水-沉积物界面(图 1).
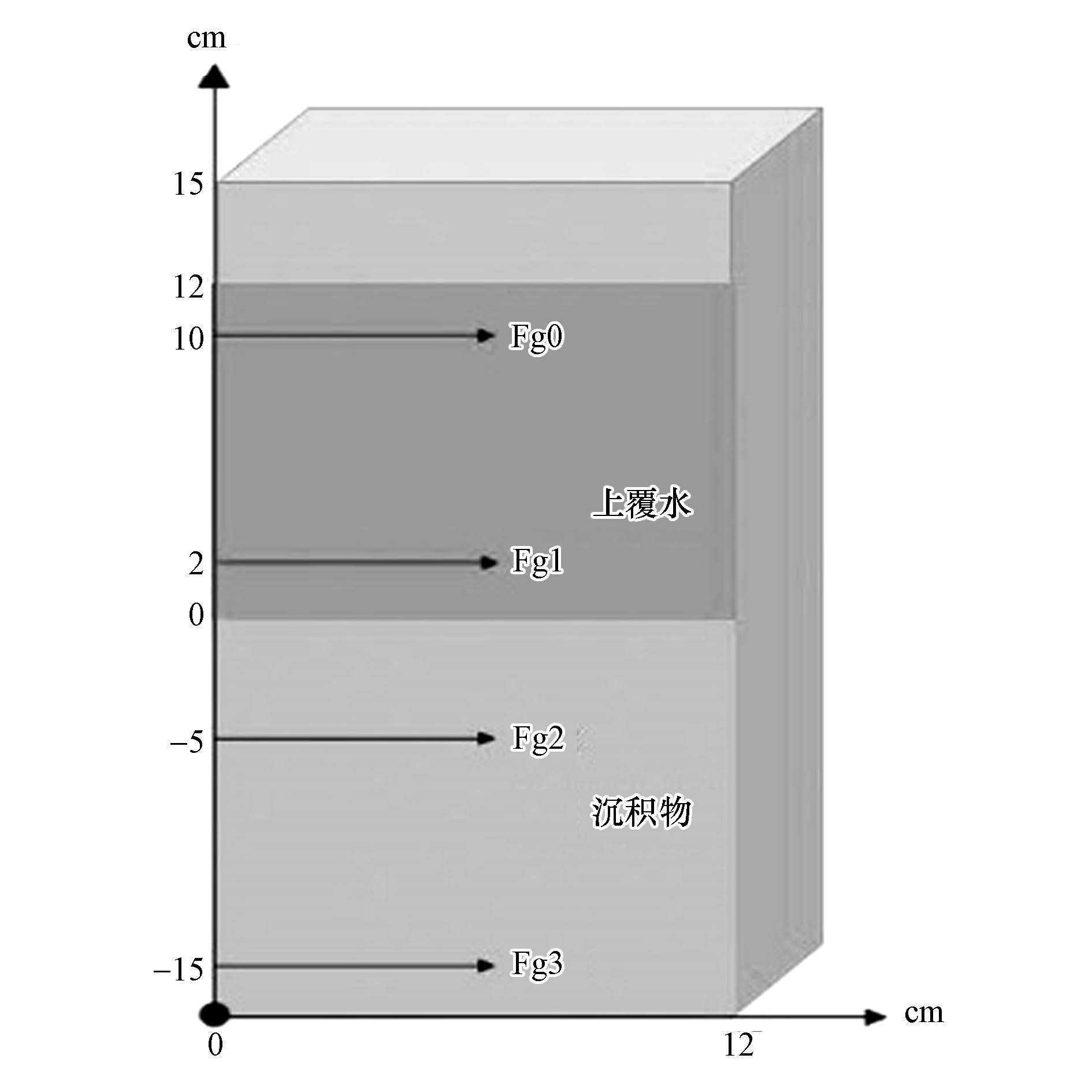 |
| 图 1 本研究的实验模型(Fg0~Fg1:上覆水取样位置;Fg2~Fg3:沉积物取样位置) Fig. 1 The experimental model of this study(Fg0~Fg1:Water sampling locations;Fg2~Fg3:Sediment sampling location) |
以水-沉积物界面为0点,分别于上覆水+10 cm位置(Fg0)、上覆水+2 cm位置(Fg1)、沉积物-5 cm位置(Fg2)、沉积物-15 cm位置(Fg3)取样,用于化学分析;另取10 mL样品,-20 ℃保存,用于微生物分子生物学分析.
2.2 化学分析方法分别检测硫化物、溶解氧、pH、硫酸盐、有机质等环境参数.pH和溶解氧直接采用雷磁DZS-706型多参数水质仪测定;硫化物采用酸挥发分离-碘量法(水和废水检测分析方法);硫酸根采用离子色谱仪测定(ICS-2100,DIONEX),分离柱型号AS20(4.0 mm×250 mm),电导检测器35 ℃,淋洗液:KOH(浓度梯度10~40 mmol·L-1),流速1.00 mL·min-1,进样量25 μL;有机质采用重铬酸钾氧化-比色法(GB9834—1988).
2.3 微生物群落分析水样Fg0、Fg1和沉积物Fg2、Fg3采用高效土壤DNA提取试剂盒(Mobio,美国)提取总DNA,取5 μL跑琼脂糖凝胶电泳,检测其纯度和质量,其中水样Fg0生物量极少,未能获得足够DNA,因此后续微生物分析中仅针对Fg1~Fg3样品.
针对微生物群落的DGGE分析,按文献(Zhao et al.,2014)方法,以总DNA为模板,采用细菌16S rDNA通用引物BA101F:5′-TGGCGGACGGGT GAGTAA-3′; BA534R:5′-ATTACCGCGGCTGCTGG-3′,进行PCR扩增以及对PCR产物进行DGGE分析.引物BA534R的5′端有一个40 bp的GC夹,引物由Invitrogen(上海)合成.应用Gel-Pro 4.5对DGGE图谱进行数字化,提取各条带的灰度值.以同一迁移率下,有条带计为1,无条带计为0,形成DGGE图谱二维矩阵.采用SPSS软件(SPSS Inc.,Chicago IL)对二维矩阵进行聚类分析,比较群落的差异.
参考文献(Gao et al.,2014)方法,以微生物总DNA为模板,采用16S rDNA通用引物515F(5′-GTGCCAGCAGCCGCGGTAA-3′)和806R(5′- GGACTACCAGGGTATCTAAT-3′),对V4区进行高通量测序,委托北京诺禾公司采用基于Illumina公司Miseq平台进行测序.测得的序列通过筛选后,以相似性97%为标准获得操作分类单元(OTU),OTU数据用RDP数据库(http://rdp.cme.msu.edu/)中的Classifer程序进行分类;按文献(Gao et al.,2014)方法,对微生物群落的多样性(包括Shannon指数、稀释曲线)、种类组成和相对丰度、群落在属水平差异进行分析.将获得的16S rDNA 高通量测序序列提交至MG-RAST公共数据库,登录号分别为:4633199.3,4633200.3,4633201.3.
3 结果(Results) 3.1 水-沉积物环境状况以沉积物-水界面为0点,分别向上、向下进行垂向监测,获得的相关环境参数如表 1.在自上而下的垂向延伸过程中,环境的pH有较小升高,但均在7左右,近中性;而溶解氧和硫化物的浓度分布恰好相反,在上层水相中,溶解氧的浓度可达4.7 mg·L-1,而此时硫化物则几乎监测不到;在沉积物相中,在5 cm以下的环境中的硫化物浓度高达190 mg·L-1以上.由于该生境为人工创制的高硫酸盐环境,其硫酸盐含量高于一般海水及沉积物中浓度,检测发现整个环境中硫酸盐含量在6200 mg·L-1左右.
| 表 1 沉积物-水界面附近环境参数的垂向变化 Table 1 Vertical changes of environmental parameters near the sediment-water interface |
分别获取+2、-5和-15 cm位置的水或沉积物样品,提取DNA后进行以16S rDNA为基础的DGGE分析,并对图谱进行聚类分析,结果如图 2.比较发现3个水平的细菌群落随着深度而发生明显变化,尤其是界面层上下的变化更加明显.
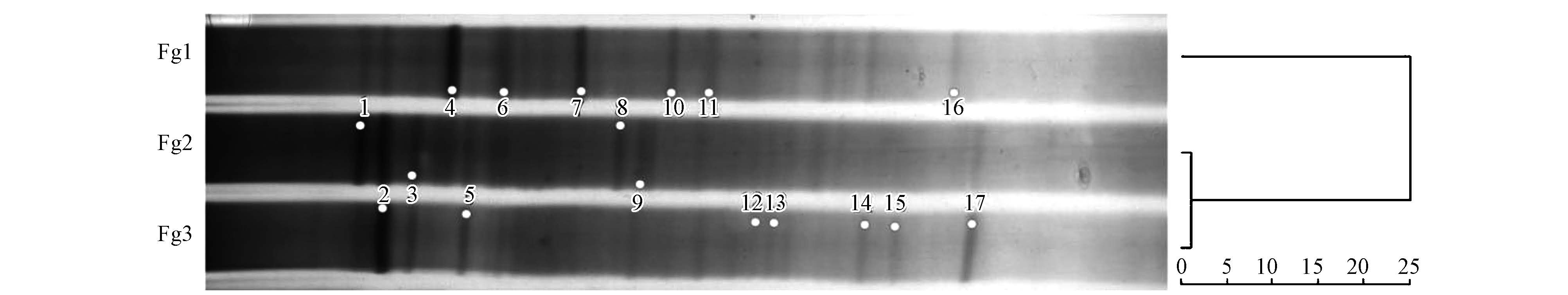 |
| 图 2 不同微生物样品的DGGE图谱及聚类分析 Fig. 2 DGGE profile of samples at different location and cluster analysis for DGGE profiles |
聚类分析可以看出沉积物中的Fg2和Fg3群落结构相似性较高,其群落分离距离小于2%;然而,水相中微生物群落结构与沉积物中微生物群落结构存在较大的分离,达到25%.这种微生物群落结构的不同,应该是由于环境条件的差异导致的,如在水相中溶解氧含量为4.70 mg·L-1,而界面层以下则几乎检测不到氧气;硫化物浓度在水相中非常低,沉积物中则可达到190 mg·L-1以上.可见,界面层之上的水相是比较典型的好氧环境,硫化物和有机质含量低;相反,界面层以下则是典型的厌氧区域,无氧气分布,硫化物和有机质含量较高,这些环境条件的差异富集了不同的微生物类群,导致了群落结构的较大差异.从DGGE谱图中可以看出,条带4、6、7、10、16等对应的微生物种群是好氧的水相中独有类群;而条带1、2、3、5、9、17等是厌氧的沉积物中独特的种群;沉积物随着深度变化,也出现了不同的条带,如8、14、15等.
3.3 微生物群落组成及丰度变化对3个环境微生物样品进行高通量测序分析,分别获得10万、9万和11万条有效序列,覆盖度达到90%以上.对序列进行稀释曲线和多样性指数分析,结果如图 3;对细菌序列在门/纲水平上进行群落组成和聚类分析,结果如图 4所示.
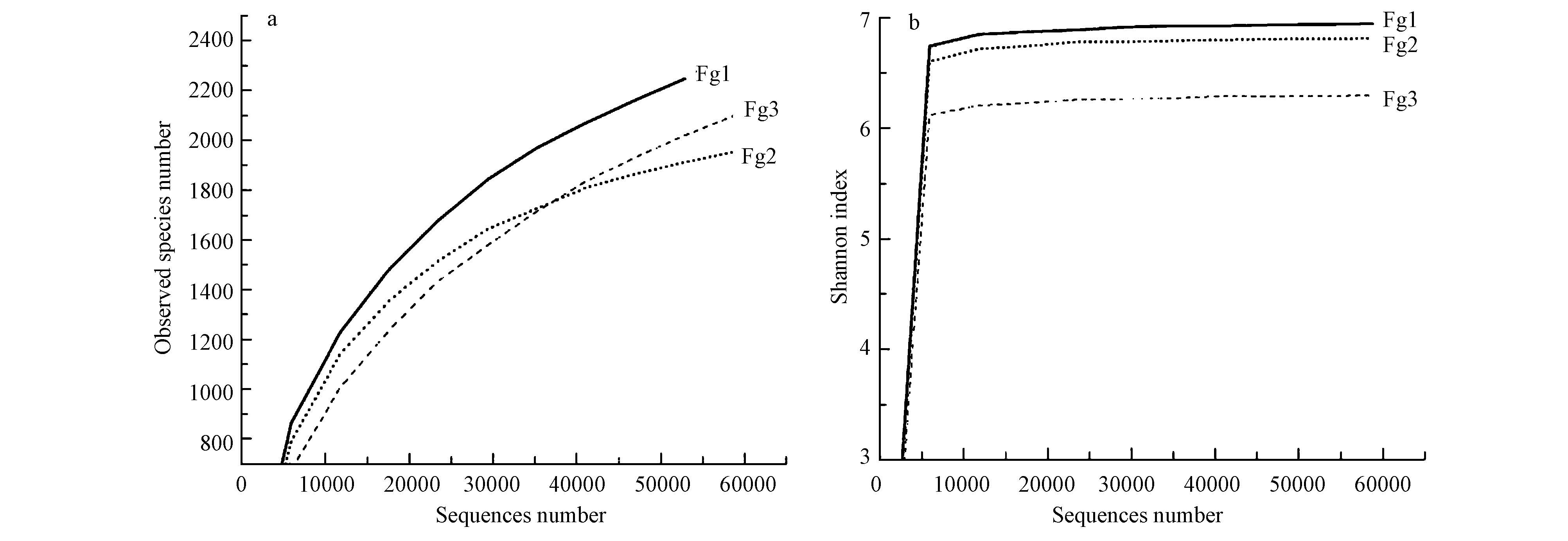 |
| 图 3 不同微生物群落高通量序列的稀释曲线(a)及多样性指数(b) Fig. 3 Rarefaction-plots(a)and Shannon index(b)for high-throughput sequences of different microbial samples |
 |
| 图 4 细菌群落在门/纲水平上的组成和相对丰度(a)及聚类分析(b) Fig. 4 Bacterial composition and relative abundance(a)and clustering analysis(b)in phylum or class level |
根据图 3,水相样品Fg1中的微生物群落多样性指数最高,为6.8,其微生物种类数量可达2200种以上,高于Fg2、Fg3样品中微生物数量.有研究表明,好氧环境中往往含有更多种类的微生物数量,更高的微生物群落多样性指数;而厌氧环境中微生物种类则要低的多(Gao et al.,2014),这主要是由环境条件和微生物的特性来决定的.厌氧微生物的营养生态位相对较窄,能够代谢的底物比较有限,而且往往需要更苛刻的环境条件,这就造成在许多条件下厌氧微生物无法生存,进而导致其种类在该环境中的下降;相反,好氧微生物能够利用的底物相当宽泛,而且部分好氧微生物可以进行兼性的厌氧呼吸,这就形成了在某些生境中好氧微生物种类更为丰富的特征.
根据图 4,水相样品Fg1和沉积物样品Fg2、Fg3中的微生物在包括古菌域和细菌域的18个门中均有较高丰度的分布,其中,细菌域中的变形菌门(Protebacteria)丰度为7.6%~32.8%、绿弯菌门(Chloroflexi)为丰度13.6%~22.3%、古菌域中的广古菌门(Euryarchaeota)丰度为19.3%~29.2%,这3个门为微生物群落中的绝对优势类群.对微生物群落进行聚类分析得到了与DGGE分析一致的结果,均表明水相中微生物群落与沉积物相存在明显差异;而通过对高通量测序序列的聚类分析,也发现在沉积物的不同水平层次中,微生物群落结构也存在比较大的差异.
4 讨论(Discussion)应用高通量测序技术对环境中的微生物群落进行分析,不但可以揭示某些微生物类群的存在以及相对丰度,更可以根据相似的分离菌种的特性推断其可能的生物学功能,以及其在特殊环境中的分类地位.越高的分类阶元,如门、纲等,微生物类群具有共同特征则越少,则越难于推断其功能特性;相反,获得微生物类群在更小的分类阶元之中的分布和丰度,如种、属水平,则可能获得更清晰的生物学背景和更确切的生态功能.目前,单以部分16S rDNA序列的比较分类,较难确认到种水平,而多可以鉴定至属水平,因此本研究对已经鉴定至属的微生物分布、丰度及其特性进行了归纳,结果见表 2.
| 表 2 微生物群落在属的水平上的相对丰度 Table 2 Changes in relative abundance of the microbial communities in genus level |
根据表 2中微生物种属的特性和在沉积物-水环境中的垂向分布特征,我们发现微生物功能特性与其分布具有非常好的相关性.厌氧微生物类群,如产甲烷菌和SRB,主要分布于水-沉积物界面层以下的厌氧区,在该区域氧气几乎检测不到,而硫化物浓度较高.然而,虽然产甲烷菌和硫酸盐还原菌均为厌氧微生物,但其在厌氧区的分布层次存在明显不同.产甲烷菌主要分布于更深的-15 cm以下,在该区域中,溶解氧含量会更低,更不易受到外界溶解氧条件的扰动,比较适于对生长条件要求苛刻的产甲烷类群的生存;相比而言,δ变形菌纲中的硫酸盐还原菌则大多分布于-5 cm以上,甚至+2 cm的好氧层,这与许多研究的结论是一致的(Ramsing et al.,1993),许多SRB不但能够耐受一定浓度的溶解氧,而且有些SRB甚至能够以氧为电子受体进行有氧呼吸,因此它们并非严格的厌氧微生物(Marschall et al.,1993).在营养生态位上,二者均能够利用小分子的挥发酸,而两者之间将会存在较大竞争(Yoda et al.,1987);然而从空间生态位上来看,本研究中,两微生物在空间是分离的,充分占据较大宽度的生态位,重叠部分尽量缩小,这样降低了两类微生物的竞争,对于生态环境的稳定至关重要.
相对于厌氧微生物来说,好氧微生物则多分布于界面以上好氧的水相,在该区域氧气含量可高达4.70 mg·L-1,如硝化细菌、α和β变形菌纲中有机物降解、硫氧化微生物类群均主要分布于该层中,可见溶解氧是决定微生物群落分布的最重要的环境因子.当然,也有一些微生物类群并不完全遵循氧气决定的规律,如Methanosaeta属(Patel and Sprott,1990)产甲烷菌虽然在厌氧层中的丰度最高,但在好氧水相中的丰度也达到16.60%;严格厌氧的绿弯菌门(Chloroflexi)中的Anaerolinea属(Sekiguchi et al.,2003)微生物在好氧水相中的丰度远高于其厌氧沉积物中,达到5.60%.虽然文献(Patel and Sprott,1990)和(Marschall and Sprott,1993)对这两属微生物的需氧特征描述均为严格厌氧型,然而由于微生物类群的多样性以及环境特征的不确定性,某些特殊种类微生物在特定环境条件下可能会改变原有的适应性特征,提高对氧的耐受能力,而具体原因还有待进一步深究.
本研究的模型模拟的是“老化”的海水养殖沉积物环境,具有典型的高盐、高硫特征.由于该环境存在明显的厌氧区,因此硫酸盐的还原将在所难免(Reese et al.,2008),参与硫酸盐还原的微生物主要是δ变形菌纲中的SRB,它们能够在厌氧条件下,通过氧化小分子有机物,将电子传递给硫酸盐,实现硫酸盐呼吸过程,进而导致生境中硫化物的积累(Patel and Sprott,1990).根据表 2发现,δ变形菌纲中的SRB主要分布于界面层以下的-5 cm,好氧水相区域也有较多分布,这表明该类微生物虽然主要在厌氧条件下发挥还原硫酸盐的作用,同时具有较强的氧的耐受性.根据表 1,在该区域,还原产生的硫化物浓度高达192 mg·L-1,高浓度的硫化物将通过梯度渗透和扩散作用向界面层移动,移动过程中被上层中的溶解氧、硫氧化微生物进一步氧化,产生硫酸盐或单质硫.在本研究过程中,在沉积物和水界面处发现一层薄薄的淡黄色沉淀物,则极有可能是单质硫,它们主要是硫化物被β和γ变形菌纲中的硫氧化微生物,如Thiobacillus Acidithiobacillus和Halothiobacillus属微生物氧化的产物.可见,通过SRB和硫氧化微生物的作用,实现了硫元素的循环过程,即“SO2-4-H2S-S0”的循环,完成了硫单质的积累,而硫单质将会被硫氧化微生物进一步氧化为硫酸盐,完成整个循环过程.参与循环的微生物以及本模型中硫的循环过程如图 5所示.
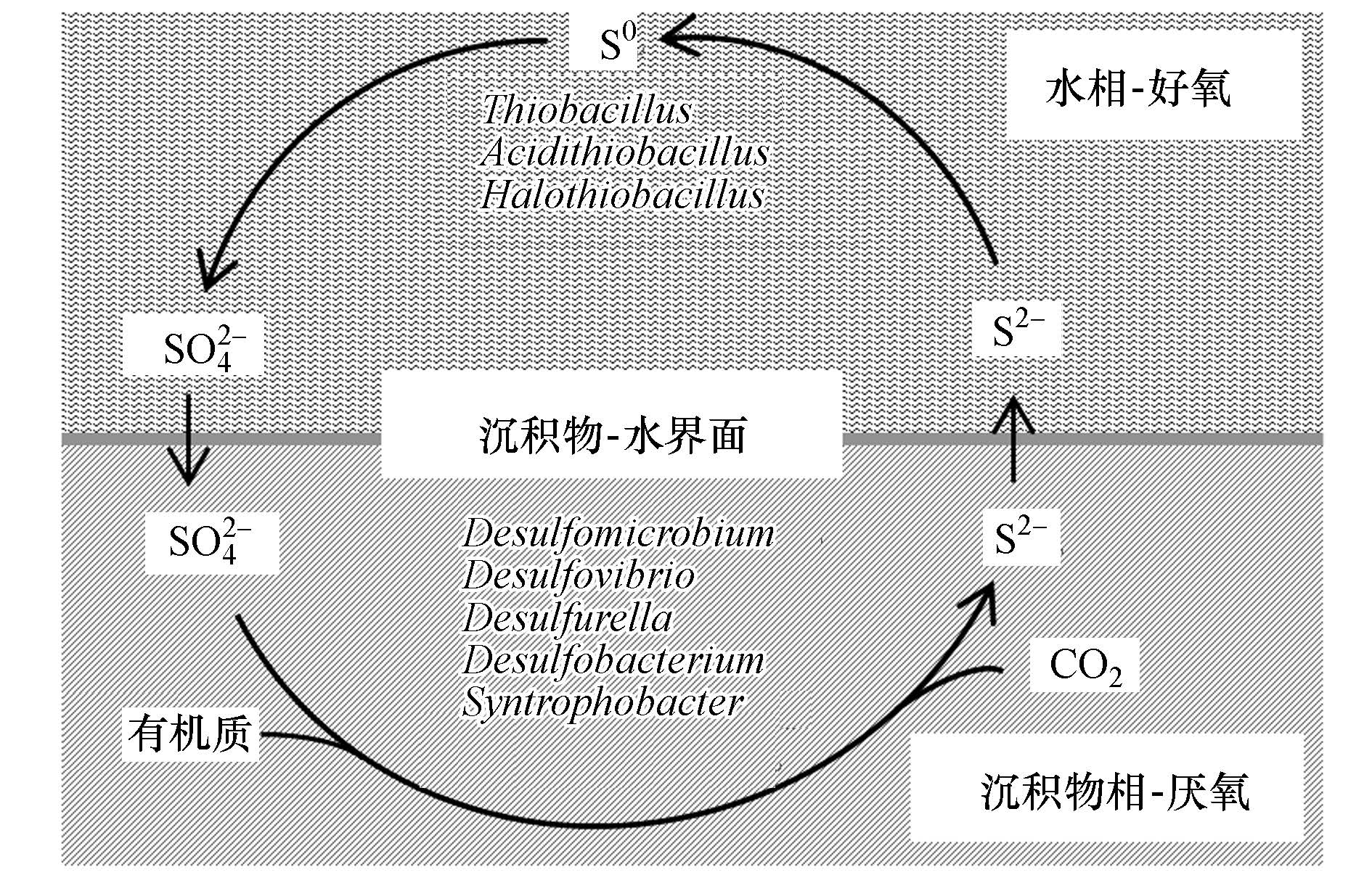 |
| 图 5 本研究中微生物参与的硫循环过程 Fig. 5 Microbal-driven sulfur recycle in the present artificial model |
在该生境中,在硫循环的过程中,需要有持续的小分子挥发酸物质的不断输入作为碳源和能源,才能推动该循环的进行;而该小分子的挥发酸,则是由该生境中的有机物矿化、发酵而来,这些微生物主要包括拟杆菌门(Bacteroides)、绿变菌门(Chloroflexi)和厚壁菌门(Firmicutes)中的 Bacteroides,Anaerolinea,Clostridium,Veillonella等属.这些微生物与硫还原、氧化微生物一起推动着硫元素在该生境中的循环过程,并实现有机物的持续降解.
5 结论(Conclusions)应用不依赖于培养的DGGE和高通量测序技术,探讨了富含硫化物的沉积物中微生物群落的垂向分布特征.微生物群落分布与环境特征具有很好的吻合性,在水-沉积物垂向剖面中,细菌域中的变形菌门、绿弯菌门以及古菌域中的广古菌门是微生物群落中的绝对优势类群.在该模型中存在微生物主导的硫循环过程,产酸发酵微生物与硫还原、氧化微生物两者共同作用,一起推动着硫元素在该生境中的循环过程,并实现有机物的持续降解.
| [1] | Asaoka S, Yamamoto T, Yoshioka I, et al. 2009. Remediation of coastal marine sediments using granulated coal ash[J]. Journal of Hazardous Materials , 172 (1) : 92–98. DOI:10.1016/j.jhazmat.2009.06.140 |
| [2] | Bonch-Osmolovskaya E A, Sokolova T G, Kostrikina N A, et al. 1990. Desulfurella acetivorans gen[J]. nov. and sp. nov.-a new thermophilic sulfur-reducing eubacterium[J]. Archives of Microbiology , 153 (2) : 151–155. |
| [3] | Bowman J P, Nichols D S. 2002. Aequorivita gen[J]. nov., a member of the family Flavobacteriaceae isolated from terrestrial and marine Antarctic habitats[J]. International Journal of Systematic and Evolutionary Microbiology , 52 (5) : 1533–1541. |
| [4] | Dahle H, Birkeland N K. 2006. Thermovirga lienii gen[J]. nov., sp. nov., a novel moderately thermophilic, anaerobic, amino-acid-degrading bacterium isolated from a North Sea oil well[J]. International Journal of Systematic and Evolutionary Microbiology , 56 (7) : 1539–1545. |
| [5] | DiPippo J L, Nesbø C L, Dahle H, et al. 2009. Kosmotoga olearia gen[J]. nov., sp. nov., a thermophilic, anaerobic heterotroph isolated from an oil production fluid[J]. International Journal of Systematic and Evolutionary Microbiology , 59 (12) : 2991–3000. |
| [6] | Galagan J E, Nusbaum C, Roy A, et al. 2002. The genome of Macetivorans reveals extensive metabolic and physiological diversity. Genome Research[M]. : 532 -542. |
| [7] | Garcia C A B, de Andrade Passos E, do Patrocínio Hora Alves J. 2011. Assessment of trace metals pollution in estuarine sediments using SEM-AVS and ERM-ERL predictions[J]. Environmental Monitoring and Assessment , 181 (1/4) : 385–397. |
| [8] | Gao C Y, Wang A J, Wu W M, et al. 2014. Enrichment of anodic biofilm inoculated with anaerobic or aerobic sludge in single chambered air-cathode microbial fuel cells[J]. Bioresource Technology , 167 : 124–132. DOI:10.1016/j.biortech.2014.05.120 |
| [9] | Kelly D P, Wood A P. 2000. Reclassification of some species of Thiobacillus to the newly designated genera Acidithiobacillus gen[J]. nov., Halothiobacillus gen. nov. and Thermithiobacillus gen. nov[J]. International Journal of Systematic and Evolutionary Microbiology , 50 (2) : 511–516. |
| [10] | Lavik G, Stuhrmann T, Brüchert V, et al. 2009. Detoxification of sulphidic African shelf waters by blooming chemolithotrophs[J]. Nature , 457 (7229) : 581–586. DOI:10.1038/nature07588 |
| [11] | Lomans B P, Maas R, Luderer R, et al. 1999. Isolation and characterization of Methanomethylovorans hollandica gen[J]. nov., sp. nov., isolated from freshwater sediment, a methylotrophic methanogen able to grow on dimethyl sulfide and methanethiol[J]. Applied and Environmental Microbiology , 65 (8) : 3641–3650. |
| [12] | Marschall C, Frenzel P, Cypionka H. 1993. Influence of oxygen on sulfate reduction and growth of sulfate-reducing bacteria[J]. Archives of Microbiology , 159 (2) : 168–173. DOI:10.1007/BF00250278 |
| [13] | Patel G B, Sprott G D. 1990. Methanosaeta concilii gen[J]. nov., sp. nov. ("Methanothrix concilii") and Methanosaeta thermoacetophila nom. rev., comb. nov[J]. International Journal of Systematic Bacteriology , 40 (1) : 79–82. |
| [14] | Postgate J R, Campbell L L. 1966. Classification of Desulfovibrio species, the nonsporulating sulfate-reducing bacteria[J]. Bacteriological Reviews , 30 (4) : 732–738. |
| [15] | Purdy K J, Munson M A, Cresswell-Maynard T, et al. 2003. Use of 16S rRNA-targeted oligonucleotide probes to investigate function and phylogeny of sulphate-reducing bacteria and methanogenic archaea in a UK estuary[J]. FEMS Microbiology Ecology , 44 (3) : 361–371. DOI:10.1016/S0168-6496(03)00078-3 |
| [16] | Ramsing N B, Kühl M, Jørgensen B B. 1993. Distribution of sulfate-reducing bacteria, O2, and H2S in photosynthetic biofilms determined by oligonucleotide probes and microelectrodes[J]. Applied and Environmental Microbiology , 59 (11) : 3840–3849. |
| [17] | Reese B K, Anderson M A, Amrhein C. 2008. Hydrogen sulfide production and volatilization in a polymictic eutrophic saline lake, Salton Sea, California[J]. Science of the Total Environment , 406 (1/2) : 205–218. |
| [18] | Sekiguchi Y, Yamada T, Hanada S, et al. 2003. Anaerolinea thermophila gen[J]. nov., sp. nov. and Caldilinea aerophila gen. nov., sp. nov., novel filamentous thermophiles that represent a previously uncultured lineage of the domain Bacteria at the subphylum level[J]. International Journal of Systematic and Evolutionary Microbiology , 53 (Pt 6) : 1842–1851. |
| [19] | Sun J L, Shang C, Kikkert G A. 2013. Hydrogen sulfide removal from sediment and water in box culverts/storm drains by iron-based granules[J]. Water Science and Technology , 68 (12) : 2626–2631. DOI:10.2166/wst.2013.543 |
| [20] | Vaquer-Sunyer R, Duarte C M. 2010. Sulfide exposure accelerates hypoxia‐driven mortalit[J]. Limnology and Oceanography , 55 (3) : 1075–1082. DOI:10.4319/lo.2010.55.3.1075 |
| [21] | 吴自军, 任德章, 周怀阳.2013. 海洋沉积物甲烷厌氧氧化作用(AOM)及其对无机硫循环的影响[J]. 地球科学进展 , 2013, 28 (7) : 765–773. |
| [22] | Yoda M, Kitagawa M, Miyaji Y. 1987. Long term competition between sulfate-reducing and methane-producing bacteria for acetate in anaerobic biofilm[J]. Water Research , 21 (12) : 1547–1556. DOI:10.1016/0043-1354(87)90140-0 |
| [23] | Zhao Y G, Feng G, Bai J, et al. 2014. Effect of copper exposure on bacterial community structure and function in the sediments of Jiaozhou Bay, China[J]. World Journal of Microbiology and Biotechnology , 30 (7) : 2033–2043. DOI:10.1007/s11274-014-1628-x |
 2016, Vol. 36
2016, Vol. 36


