蛋白质与核酸都是重要的生物大分子,它们可以与很多外源性的小分子、离子发生相互作用。从分子层面探讨生物大分子之间以及生物大分子与小分子之间的相互作用,对于认识生物分子的基本构造与功用、指导药物结构设计等都具有十分重要的意义。
传统的分子相互作用研究方法有很多,比如酶联免疫吸附、荧光共振能量转移、质谱等检测蛋白质相互作用的研究技术,染色体免疫共沉淀等检测核酸相互作用的技术。然而,多数技术都属于终点标记的方法,并且只能定性检测有无相互作用,不能反映结合力的强弱。随着研究的不断深入,基于光学检测的分析方法逐渐进入公众的视野,其具有超高的灵敏度、较少的样品量、可定量检测结合力及原位的检测方法等优势,这对于生命科学的研究大有裨益。主流的基于光学检测的相互作用分析技术有表面等离子体共振(surface plasmon resonance,SPR)技术、生物膜干涉(biolayer interferometry,BLI)技术、背向散射干涉(back-scattering interferometry,BSI)技术、微量热泳动(microscale thermophoresis,MST)技术,本文对这4种分子相互作用表征技术的原理、优缺点及应用进行综述与比较,为分子间相互作用表征技术的选择提供参考。
1 SPR技术 1.1 SPR的技术原理与方法特点SPR技术是利用光在不同介质中产生消逝波后与等离子波产生共振,进而构建分子间相互作用的生物传感分析技术。1902年,Wood[1]首先发现了衍射光栅上的一种反常衍射现象,这是由于表面等离子体波激发导致。1957年,Ritchie[2]从理论上证实了金属表面等离子体激发的存在,SPR假说取得重大进展。1982年,Nylander等[3]证明了SPR在气体检测和生物传感器中的应用,为进一步测量生物分子间相互作用奠定了基础[4-6]。
SPR技术的检测原理如图 1A所示,平面偏振光在全内反射条件下撞击金属芯片,导致金属中的自由电子产生表面等离子体激元。如图 1B所示,SPR检测过程主要分为6个步骤,首先配体即生物分子通过偶联反应被固定在生物传感芯片表面,获得初始基线;而后分析物被注入并流过传感器芯片的表面,分子间相互作用的发生引起芯片表面质量浓度的变化进而导致光折射率的变化,这种变化可以通过共振单位(response unit,RU)进行量化和表征,RU增大对应着结合事件的发生;当结合和解离达到平衡时,RU也维持在一个稳定值;接着停止注入分析物,解离阶段开始,分析物在缓冲液的冲洗下从配体的结合位点逐步解离,此步骤对应着RU的逐渐降低;再生步骤就是将分析物完全从配体结合位点去除,随后再次校正,使RU回到基线水平,为新的检测周期做好准备。

|
图 1 表面等离子体共振(SPR)技术的检测原理(A)及表征方法(B) |
SPR在检测时响应速度很快,且样品量比较少,待分析物也无需标记,通过实时、动态监测RU的变化,从结合、解离反应中得到分子间的结合常数(association constant,Ka)、解离常数(dissociation constant,Kd)以及平衡解离常数(dissociation equilibrium constant,KD),反映分子间作用力的大小及特异性等信息。SPR具有高通量检测的能力,检测浓度可低至pmol/L。
1.2 SPR技术在分子间相互作用检测中的应用近年来SPR已发展成为行业内的金标准,在分子间相互作用领域发挥了举足轻重的作用。
1.2.1 检测同源分子间的相互作用如Nan等[7]利用SPR技术揭示1型跨膜蛋白CD147直接与Ran结合蛋白1(Ran-binding protein 1,RanBP1)发生相互作用,证据显示CD147能够调节癌细胞的迁移[8]。而CD147与RanBP1的结合导致了紫杉醇治疗癌症时的耐药性,表明CD147是紫杉醇敏感性的关键调节因子,对癌症化疗具有重要意义。又如Jarczewska等[9]利用SPR技术测定了单链脱氧核糖核酸(single-stranded deoxyribonucleic acid,ssDNA)和肽核酸(peptide-nucleic acid,PNA)探针与miRNA的相互作用。感染部位的巨噬细胞被认为是预防脓毒症发展的潜在治疗靶点,而Kelch样环氧氯丙烷相关蛋白1/核因子E2相关因子2(Kelch-like epoxy chloropropane-associated protein 1/nuclear factor erythroid 2-related factor 2,Keap1/Nrf2)系统是巨噬细胞抗菌活性的关键调节剂,Wang等[10]利用SPR技术验证了一种独特的七次甲基染料IR-61能够直接抑制Keap1/Nrf2蛋白质间相互作用并激活Nrf2,从而增强巨噬细胞的抗菌功能。
1.2.2 检测异源分子间的相互作用贺毅等[11]首先利用分子对接对3 000多种化合物与白细胞介素15受体α(interleukin 15 receptor α,IL-15Rα)进行模拟对接,并根据其特异性结合情况予以打分。在此基础上,再利用SPR技术测量化合物与IL-15Rα重组蛋白的亲和力,最终筛选出分子量为556.6的甘西鼠尾新酮A(neoprzewaquinone A,Neo)与IL-15Rα的特异性结合亲和力最高,结果表明Neo可能是一种新的以IL-15Rα为靶点的潜在活性药。Zhou等[12]利用SPR技术测定了加热过程中的肌原纤维蛋白(myofibrillar protein,MP)和β-胡萝卜素(beta-carotene,β-C)之间的相互作用,结果表明,在37 ℃左右MP和β-C之间的相互作用从范德华力变为了疏水相互作用,为β-C输运系统合适温度的选择提供了重要依据。
作为SPR的核心部件,其传感器针对不同检测目的采取了不同的设计,如半导体、金属薄膜、纳米材料等组成的无机界面传感器,硫醇、聚乙二醇等涂覆的有机界面传感器,以及核酸、抗体等构成的生物界面传感器等。这些设计有助于提高检测的灵敏度和选择性,在用于复杂体系检测时可以最大限度减少样品在等离子体表面的污染现象[13]。
2 BLI技术 2.1 BLI的技术原理与方法特点BLI技术是一种实时的光学生物传感技术,同SPR技术一样,用于检测无标记生物分子间的相互作用[14]。该方法基于光干涉的原理,使用光纤生物传感器来收集信号。这些信号随着分子间相互作用的发生,实时反映传感器层光学厚度的变化。在此系统中,分析物与固定在传感器表面的配体相互作用形成单分子层,这会导致反射光的干涉谱按一定比例移动。
BLI技术的检测过程主要分为5个步骤,如图 2所示,首先是将生物传感器浸入到缓冲液中进行平衡;接着将传感器浸入到已知浓度的固化溶液中,溶液中经修饰的生物分子结合到生物传感器的表面,使其表面膜层厚度增加;接着将固化完已知浓度生物分子的生物传感器浸入缓冲液中做基线;然后再将该生物传感器浸入到含有待测分析物的样品溶液中,由于待测分析物与生物分子的特异性结合而导致膜层厚度增加;最后将结合分析物的生物传感器浸入到缓冲液中进行解离,分析物从生物传感器表面脱落导致膜层厚度减少[15]。
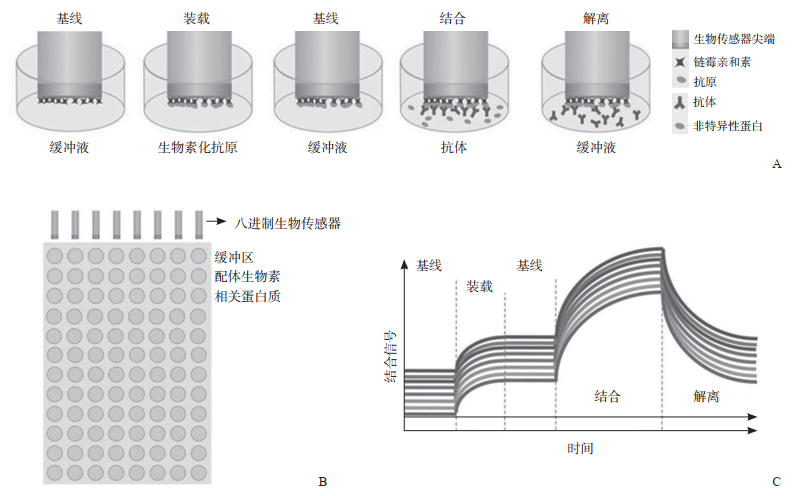
|
图 2 生物膜干涉(BLI)技术的检测原理及表征方法[15] A: 检测步骤;B:分析物承载台;C:检测结果表征 |
BLI通过对光干涉信号的实时监测即可得到待测样品的Ka、Kd、KD等信息,具有操作简单、检测时间短、样品消耗量少、无需标记等优点,广泛应用于生物分子间相互作用的分析和快速检测,检测浓度可低至pmol/L。
2.2 BLI技术在分子间相互作用检测中的应用自2006年产业化以来,BLI技术已广泛应用于各类生物体系的测定,可以实现无标记分子间相互作用分析的实时检测。
2.2.1 检测同源分子间的相互作用Dubrow等[16]以甲型流感病毒的NS1和磷酸肌醇3激酶为模型蛋白,利用在BLI中加入蔗糖等糖类作为阻滞剂,测量蛋白质之间的弱相互作用。BLI在定量分析蛋白质之间的相互作用时,有时会存在分析物和生物传感器之间的非特异性结合(nonspecific binding,NSB),特别是在测量弱相互作用时,NSB对BLI的准确性影响较大,糖类尤其是蔗糖是优秀的NSB阻滞剂。Neira等[17]利用BLI测量了组氨酸磷酸载体蛋白(histidine phosphocarrier protein,HPr)前38个N端残基(HPr38)、前58个N端残基(HPr58)及前70个N端残基(HPr70)这3种蛋白质与天蓝色链球菌的N端结构域等蛋白质的相互作用,其结果与完整的HPr和同种蛋白质的结合常数处于一个量级。
2.2.2 检测异源分子间的相互作用Guo等[18]利用BLI技术证实了用于治疗炎症性疾病的传统中药配方二妙散中的活性化合物槲皮素、汉黄芩素和芸香碱与影响风湿性关节炎的核心靶蛋白TNF-α、IL-6及IL-1β存在相互作用,进而证明二妙散通过抑制炎症细胞因子在抗炎中发挥重要作用。金黄色葡萄球菌被认为是一种常见的食源性病原微生物,通常会引起食物中毒和各种传染病[19]。C54A突变体LysGH15能够特异性地识别和结合金黄色葡萄球菌,Liu等[20]利用LysGH15作为受体结合BLI建立了一种检测金黄色葡萄球菌的新方法,可以直接、快速、准确、高特异性地检测金黄色葡萄球菌,为食品安全及相关领域提供了高效、可靠的诊断工具。
BLI技术可应用于蛋白质与蛋白质的相互作用分析、抗体筛选、核酸与蛋白相互作用分析、抗体与小分子药物相互作用分析等多个方面[21-24]。相较于传统的检测技术,BLI技术无需荧光标记或发光基团即可直接检测,能最大程度保证实验的真实性[25];相较于SPR技术,BLI技术具有高通量、成本低、易维护等优点。
3 BSI技术 3.1 BSI的技术原理与方法特点BSI技术是通过监测通道内光折射率的变化测定分子间相互作用的技术。He-Ne激光照射到含有待测物质的微流体通道上,在通道的内外界面会产生多角度的反射和折射,在环绕管轴360°的范围内产生干涉条纹,称之为BSI。当通道内液体的折射率改变时,干涉条纹会相对于初始位置产生移动,移位的大小可用于确定分子间相互作用。
BSI技术的检测过程如图 3所示,激光器发出的激光经镜面反射后射入到通有待测溶液的石英毛细管中,激光在毛细管内外界面发生多次反射、折射,其中一部分的反射和折射光再经过镜面发射到电荷耦合器件(charged-coupled device,CCD)线阵处,这些反射和折射的光在CCD处发生干涉并产生干涉条纹。当毛细管内的液体发生相互作用时,其折射率会发生改变,CCD处的干涉条纹也会随之发生改变,获取条纹移动对应的相位值的变化,进而获得一系列的曲线,从而确定分子相互作用的相关信息[26]。
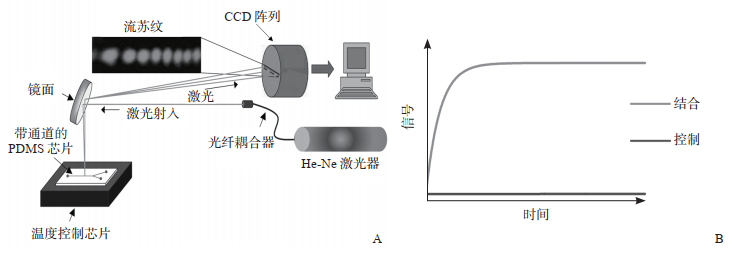
|
图 3 BSI技术的检测原理(A)及表征方法(B)[26] BSI:背向散射干涉;CCD:电荷耦合器件;PDMS:聚二甲基硅氧烷. |
BSI技术是目前唯一可以以无标记方式确定结合亲和力的技术,它不依赖于热变化或表面内固定,可以用于包括有机溶剂在内的许多不同介质中,此外,BSI技术的检测下限明显低于其他自由溶液技术(可低至pmol/L),可以在生理浓度范围内进行检测。然而,BSI对实验条件要求苛刻,易受温、湿度等的影响,目前为止还没有实现仪器的商业化。
3.2 BSI技术在分子间相互作用检测中的应用BSI技术可在生理相关条件下进行无标记和无固定的相互作用分析,灵敏度高且体积小。
3.2.1 检测同源分子间的相互作用Abbas等[27]利用BSI技术测量神经元钙传感器恢复素与其目标G蛋白偶联受体激酶1(G protein-coupled receptor kinase 1,GRK1)的相互作用,得到的结果与等温量热滴定法及圆二色谱法获得的结果一致。Jepsen等[28]建立了BSI系统来测量蛋白A和IgG之间的相互作用,证明BSI具有一定的温度和时间稳定性。
3.2.2 检测异源分子间的相互作用Haddad等[29]利用BSI技术测量乙酰胆碱酯酶(acetylcholinesterase,AChE)与几种已知的新型抑制剂之间的相互作用,结果发现,在没有底物的情况下即可量化AChE与抑制剂之间的相互作用;通过BSI的结合数据与IC50进行比较,可以区分竞争性、非竞争性等不同类型的抑制剂。传统检测AChE相互作用的方法侧重于底物水解的间接测量,且需要探针和繁琐的程序,而BSI技术可实现简单、无标记的直接测量。除此以外,Baksh等[30]利用BSI技术测量了多种物质的结合相互作用,包括胰蛋白酶- 抗胰蛋白酶、胰蛋白酶- 对氨基苯甲脒、抗凝血酶- 肝素以及蛋白A-IgG,所得的结合曲线与Kd值及其他经典方法得到的结果一致。
BSI技术虽然尚未商用,但可以相对容易地用标准光学部件自行搭建,且可用于包括有机溶剂在内的许多不同介质中。BSI技术采用了特殊设计的微流体通道,而当前药物发现和优化的重点也已从传统方法转向快速微流体筛选[31],BSI作为量化分子间相互作用的生物传感平台,将在药物发现中发挥重要作用。
4 MST技术 4.1 MST的技术原理与方法特点MST技术是一种利用荧光实时监测在温度梯度的环境中分子在毛细管内定向泳动,进而检测分子间相互作用的技术。1856年,Ludwig首次发现了热泳动现象,又称为Soret效应[32],它描述了分子在温度梯度内诱导产生的热量和定向运动。2010年,德国NanoTemper Technologies公司开发出第1台商用微量热泳动仪[33]。
MST技术的检测过程如图 4所示,在初始状态下红外激光是关闭的,荧光分子在整个毛细管中均匀分布,在这个阶段采集到的稳定荧光信号称为初始荧光。当红外激光器被打开时毛细管的局部区域立即被加热,在热电导入之前荧光强度会剧烈下降,这种现象被称为温度跃迁(temperature jump,T-Jump)。随后,荧光分子发生热泳运动,热区的荧光信号会减弱,直到在质量扩散效应的反作用下达到稳定状态。这一过程通常持续30 s,被称为热浸。最后,红外激光器关闭,荧光分子就会扩散回来,荧光强度又会增加,形成典型的MST曲线。不同浓度的配体对荧光分子的热电泳性有不同的影响,从而产生一系列MST特性曲线,T-Jump和热泳信号可以分别拟合以获得结合亲和力值[34]。
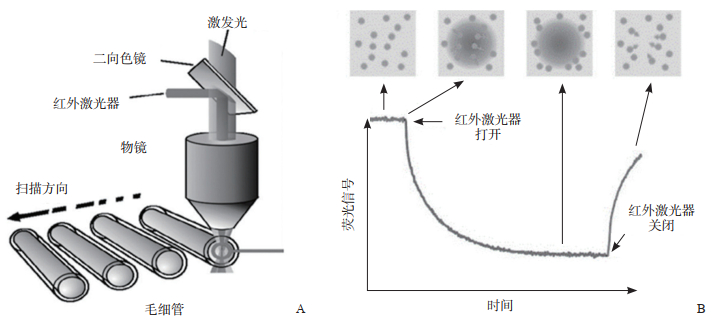
|
图 4 微量热泳动(MST)的检测原理(A)及表征方法(B)[34] |
MST主要通过监测在微观温度梯度下配体结合过程中由于大小、表面电荷和水化层的微小变化而引起的荧光分子的热电性变化,定量地分析生物分子间的相互作用。通过将荧光检测的精确性和热泳动的灵敏性结合起来,MST技术提供了一种灵活、稳定、快速的检测方法,不受分子大小、种类的影响,可检测低至pmol/L浓度的分子间相互作用。MST技术是目前应用于分子间相互作用分析的一项最新的超灵敏技术。
4.2 MST技术在分子间相互作用检测中的应用MST技术是一种能够灵敏、高通量、快速地定量分析分子间相互作用的新兴技术,是近10年来研究分子间相互作用强有力的工具。
4.2.1 检测同源分子间的相互作用Zhang等[35]将5(6)-羧基荧光素标记到活化T细胞核因子c1(nuclear factor of activated T cells c1,NFATc1)、Na+/H+交换器1(Na+/H+ exchanger 1,NHE1)、非洲猪瘟病毒(African swine fever virus,ASFV)编码的蛋白抑制剂A238L和钙调磷酸酶调节蛋白1(regulator of calcineurin 1,RCAN1)短肽的N末端,而后利用MST技术详细测量被标记的多肽与钙调磷酸酶之间的相互作用,并得到很好的结果,对于了解生命活动的调节具有重要意义。又如Jordan等[36]以小RNA41(small RNA41,sRNA41)及其天然靶标mRNA-MM2089为例,用MST表征其结合亲和力,结果显示MST技术可以快速、准确地表征RNA之间的相互作用。
4.2.2 检测异源分子间的相互作用Huang等[37]利用MST技术研究了牛乳铁蛋白与二氢杨梅素、杨梅素2种黄酮化合物的结合作用,发现杨梅素对于牛乳铁蛋白有更强的亲和力,可以诱导牛乳铁蛋白更多的构象变化。Yu等[38]以镉离子和黄曲霉素B1适配体为研究对象,比较在适配体上修饰不同的荧光基团以及将荧光基团修饰在适配体的不同位置,导致2个相同的异源分子在同一条件下的MST出现不同的测量结果,为合理设计MST方法提供了基础。
近年来,MST技术被成功地用于分析各种生物分子相互作用,从蛋白质与蛋白质之间的相互作用、蛋白质与多肽、核酸、小分子之间的相互作用,到小分子之间的相互作用等[39-43]。MST根据是否需要荧光基团标记分为2种类型,一种类型的MST需要用荧光基团标记,另一种系统依赖于紫外范围内芳香族氨基酸的固有荧光。这2个系统都通过红外激光的热效应来检测分子的运动,各有其优点和缺点,无标记的MST可以使用未标记的天然蛋白质,然而许多分析物在紫外线范围内发出的荧光可能会干扰KD值的准确测定;相比之下,标记的MST允许使用荧光标记的探针连接到配体上,在可见光范围内具有可测量的光密度而不是紫外线,从而避免了来自分析物的信号干扰。
5 小结本文介绍了SPR、BLI、BSI、MST这4种常用的基于光学检测的分子间相互作用表征技术。其中SPR、BLI及BSI技术都是直接检测分子间相互作用后引起的光学效应的变化,MST则是应用荧光作为检测手段来监测局部分子间发生相互作用时的热运动。相较于前3种技术,MST技术可以测量天然状态环境中的分子相互作用,结合毛细管的使用大大降低了样品的消耗量,实现了高通量、短时间的测定。这4种技术在检测原理上大相径庭,各具特点,可以提供多维度的数据信息,也都存在着优点与不足。
SPR技术可以实时、动态、免标记地完成对分析物的检测,检测浓度可低至pmol/L,由于其生物传感器具有高特异性,可以检测其他技术难以研究的高度动态的复合物,但获得一个样品的亲和力值需要1~2 h,且对于低浓度和存在非特异性结合的样品检测准确度不高。BLI技术对分析物的纯度要求不高,可以检测多个浓度样品的结合信息,它获得一个样品的亲和力值同样需要1~2 h,适合大量样品的筛选,对SPR技术起到了非常好的补充作用,但其检测范围有限,试剂消耗量较大,对于弱相互作用检测准确度不高。BSI技术所需的样品量少,有极高的灵敏度,只是其极易受温度变化的影响,有的待测物质产生的光热效应会引起折射率的变化,进而影响测量结果的准确性。MST技术对待测分子的大小和分子量无任何限制,且可以在血浆、细胞裂解液等几乎任何缓冲液中进行检测,同时其分析物的用量少(仅需要几微升),约20 min即可获得亲和力值,检测浓度可低至pmol/L,比较适合微量样品的表征分析,但其检测成本较高,仪器造价昂贵。
综上所述,在研究不同的目标分子时选择一种或几种适宜的分子间相互作用检测技术进行交叉验证,多维度地去表征分子间相互作用,有助于了解药物转运和代谢过程,为药物设计和筛选提供有价值的信息,进而指导药物设计,推动医药研究发展。
| [1] |
WOOD R W. On a remarkable case of uneven distribution of light in a diffraction grating spectrum[J]. Proc Phys Soc London, 1902, 18(1): 269-275. DOI:10.1088/1478-7814/18/1/325 |
| [2] |
RITCHIE R H. Plasma losses by fast electrons in thin films[J]. Phys Rev, 1957, 106(5): 874-881. DOI:10.1103/physrev.106.874 |
| [3] |
NYLANDER C, LIEDBERG B, LIND T. Gas detection by means of surface plasmon resonance[J]. Sens Actuat, 1982, 3: 79-88. DOI:10.1016/0250-6874(82)80008-5 |
| [4] |
LIEDBERG B, NYLANDER C, LUNSTRÖM I. Surface plasmon resonance for gas detection and biosensing[J]. Sens Actuat, 1983, 4: 299-304. DOI:10.1016/0250-6874(83)85036-7 |
| [5] |
LIEDBERG B, NYLANDER C, LUNDSTRÖM I. Biosensing with surface plasmon resonance: how it all started[J]. Biosens Bioelectron, 1995, 10(8): ⅰ-ⅸ. DOI:10.1016/0956-5663(95)96965-2 |
| [6] |
HOMOLA J, YEE S S, GAUGLITZ G. Surface plasmon resonance sensors: review[J]. Sens Actuat B Chem, 1999, 54(1/2): 3-15. DOI:10.1016/s0925-4005(98)00321-9 |
| [7] |
NAN G, ZHAO S H, WANG T, et al. CD147 supports paclitaxel resistance via interacting with RanBP1[J]. Oncogene, 2022, 41(7): 983-996. DOI:10.1038/s41388-021-02143-3 |
| [8] |
CUI H Y, WANG S J, MIAO J Y, et al. CD147 regulates cancer migration via direct interaction with Annexin A2 and DOCK3-β-catenin-WAVE2 signaling[J]. Oncotarget, 2016, 7(5): 5613-5629. DOI:10.18632/oncotarget.6723 |
| [9] |
JARCZEWSKA M, BOJARSKI W, MAJEWSKA A, et al. Studies on the application of single-stranded DNA and PNA probes for electrochemical detection of miRNA 141[J]. Bioelectrochemistry, 2023, 150: 108363. DOI:10.1016/j.bioelechem.2022.108363 |
| [10] |
WANG Y, TANG B, LI H, et al. A small-molecule inhibitor of Keap1-Nrf2 interaction attenuates sepsis by selectively augmenting the antibacterial defence of macrophages at infection sites[J]. Ebio Medicine, 2023, 90: 104480. DOI:10.1016/J.EBIOM.2023.104480 |
| [11] |
贺毅, 王海霞, 刘敏, 等. 基于分子对接和表面等离子体共振技术筛选IL-15Rα小分子抑制剂[J]. 生理学报, 2023, 75(5): 623-628. DOI:10.13294/j.aps.2023.0064 |
| [12] |
ZHOU T, LIU H, DIAO X, et al. Molecular interaction between myofibrillar protein and beta-carotene during heating[J]. Food Chem, 2024, 435: 137588. DOI:10.1016/j.foodchem.2023.137588 |
| [13] |
D'AGATA R, BELLASSAI N, SPOTO G. Exploiting the design of surface plasmon resonance interfaces for better diagnostics: a perspective review[J]. Talanta, 2024, 266: 125033. DOI:10.1016/j.talanta.2023.125033 |
| [14] |
CONCEPCION J, WITTE K, WARTCHOW C, et al. Label-free detection of biomolecular interactions using BioLayer interferometry for kinetic characterization[J]. Comb Chem High Throughput Screen, 2009, 12(8): 791-800. DOI:10.2174/138620709789104915 |
| [15] |
张艳, 姜丽艳, 张晓光, 等. 生物膜干涉技术在生物分子间相互作用检测中的应用[J]. 生命科学仪器, 2019, 17(2): 49-52, 42. DOI:10.11967/2019170408 |
| [16] |
DUBROW A, ZUNIGA B, TOPO E, et al. Suppressing nonspecific binding in biolayer interferometry experiments for weak ligand-analyte interactions[J]. ACS Omega, 2022, 7(11): 9206-9211. DOI:10.1021/acsomega.1c05659 |
| [17] |
NEIRA J L, PALOMINO-SCHÄTZLEIN M. Folding of the nascent polypeptide chain of a histidine phosphocarrier protein in vitro[J]. Arch Biochem Biophys, 2023, 736: 109538. DOI:10.1016/j.abb.2023.109538 |
| [18] |
GUO B, ZHAO C, ZHANG C, et al. Elucidation of the anti-inflammatory mechanism of Er Miao San by integrative approach of network pharmacology and experimental verification[J]. Pharmacol Res, 2022, 175: 106000. DOI:10.1016/j.phrs.2021.106000 |
| [19] |
HUANG Z, YU X, YANG Q, et al. Aptasensors for Staphylococcus aureus risk assessment in food[J]. Front Microbiol, 2021, 12: 714265. DOI:10.3389/fmicb.2021.714265 |
| [20] |
LIU X, HUANG C, QIU C, et al. Rapid and sensitive detection of Staphylococcus aureus using biolayer interferometry technology combined with phage lysin LysGH15[J]. Biosens Bioelectron, 2022, 198: 113799. DOI:10.1016/j.bios.2021.113799 |
| [21] |
JUNG V, ROGER K, CHHUON C, et al. BLI-MS: combining biolayer interferometry and mass spectrometry[J]. Proteomics, 2022, 22(9): E2100031. DOI:10.1002/PMIC.202100031 |
| [22] |
NIKKHOI S K, LI G, ELEYA S, et al. Bispecific killer cell engager with high affinity and specificity toward CD16a on NK cells for cancer immunotherapy[J]. Front Immunol, 2023, 13: 1039969. DOI:10.3389/fimmu.2022.1039969 |
| [23] |
EL-KAMAND S, DU PLESSIS M D, BREEN N, et al. A distinct ssDNA/RNA binding interface in the Nsp9 protein from SARS-CoV-2[J]. Proteins, 2022, 90(1): 176-185. DOI:10.1002/prot.26205 |
| [24] |
ZHANG D, HAMDOUN S, CHEN R, et al. Identification of natural compounds as SARS-CoV-2 entry inhibitors by molecular docking-based virtual screening with bio-layer interferometry[J]. Pharmacol Res, 2021, 172: 105820. DOI:10.1016/j.phrs.2021.105820 |
| [25] |
SANDERS M, MCPARTLIN D, MORAN K, et al. Comparison of enzyme-linked immunosorbent assay, surface plasmon resonance and biolayer interferometry for screening of deoxynivalenol in wheat and wheat dust[J]. Toxins, 2016, 8(4): 103. DOI:10.3390/toxins8040103 |
| [26] |
BORNHOP D J, LATHAM J C, KUSSROW A, et al. Free-solution, label-free molecular interactions studied by back-scattering interferometry[J]. Science, 2007, 317(5845): 1732-1736. DOI:10.1126/science.1146559 |
| [27] |
ABBAS S, KOCH K W. Label-free quantification of direct protein-protein interactions with backscattering interferometry[J]. Bio Protoc, 2021, 11(24): e4256. DOI:10.21769/BioProtoc.4256 |
| [28] |
JEPSEN S T, JØRGENSEN T M, ZONG W, et al. Evaluation of back scatter interferometry, a method for detecting protein binding in solution[J]. Analyst, 2015, 140(3): 895-901. DOI:10.1039/c4an01129e |
| [29] |
HADDAD G L, YOUNG S C, HEINDEL N D, et al. Back-scattering interferometry: an ultrasensitive method for the unperturbed detection of acetylcholinesterase-inhibitor interactions[J]. Angew Chem Int Ed Engl, 2012, 51(44): 11126-11130. DOI:10.1002/anie.201203640 |
| [30] |
BAKSH M M, FINN M G. An experimental check of backscattering interferometry[J]. Sens Actuators B Chem, 2017, 243: 977-981. DOI:10.1016/j.snb.2016.12.055 |
| [31] |
梁怡萧, 潘建章, 方群. 基于微流控技术的细胞水平高通量药物筛选系统的研究进展[J]. 色谱, 2021, 39(6): 567-577. DOI:10.3724/SP.J.1123.2020.07014 |
| [32] |
DUHR S, BRAUN D. Why molecules move along a temperature gradient[J]. Proc Natl Acad Sci USA, 2006, 103(52): 19678-19682. DOI:10.1073/pnas.0603873103 |
| [33] |
WIENKEN C J, BAASKE P, ROTHBAUER U, et al. Protein-binding assays in biological liquids using microscale thermophoresis[J]. Nat Commun, 2010, 1: 100. DOI:10.1038/ncomms1093 |
| [34] |
BREITSPRECHER D, SCHLINCK N, WITTE D, et al. Aptamer binding studies using MicroScale thermophoresis[J]. Methods Mol Biol, 2016, 1380: 99-111. DOI:10.1007/978-1-4939-3197-2_8 |
| [35] |
ZHANG N, LIU Y, SHI X, et al. Microscale thermophoresis and fluorescence polarization assays of calcineurin-peptide interactions[J]. Anal Biochem, 2022, 646: 114626. DOI:10.1016/j.ab.2022.114626 |
| [36] |
JORDAN B, NICKEL L, SCHMITZ R A. Microscale thermophoresis to study RNA-RNA binding affinity[J]. Methods Mol Biol, 2022, 2516: 291-303. DOI:10.1007/978-1-0716-2413-5_15 |
| [37] |
HUANG J, HE Z, CHENG R, et al. Assessment of binding interaction dihydromyricetin and myricetin with bovine lactoferrin and effects on antioxidant activity[J]. Spectrochim Acta Part A Mol Biomol Spectrosc, 2020, 243: 118731. DOI:10.1016/j.saa.2020.118731 |
| [38] |
YU H, ZHAO Q. Profiling additional effects of aptamer fluorophore modification on microscale thermophoresis characterization of aptamer-target binding[J]. Anal Chem, 2023, 95(46): 17011-17019. DOI:10.1021/acs.analchem.3c03603 |
| [39] |
AL-JUBAIR T, STEFFEN J H, MISSEL J W, et al. Characterization of human aquaporin protein-protein interactions using microscale thermophoresis (MST)[J]. STAR Protoc, 2022, 3(2): 101316. DOI:10.1016/j.xpro.2022.101316 |
| [40] |
JIANG H, COLE P A. N-terminal protein labeling with N-hydroxysuccinimide esters and microscale thermophoresis measurements of protein-protein interactions using labeled protein[J]. Curr Protoc, 2021, 1(1): e14. DOI:10.1002/cpz1.14 |
| [41] |
EL ATAB O, SCHNEITER R. Measuring the interaction of sterols and steroids with proteins by microscale thermophoresis[J]. Methods Mol Biol, 2023, 2704: 173-181. DOI:10.1007/978-1-0716-3385-4_10 |
| [42] |
JI C H, LEE M J, KIM S B, et al. Analyzing the interaction of arginylated proteins and Nt-Arg-mimicking chemical compounds to N-recognins[J]. Methods Mol Biol, 2023, 2620: 253-262. DOI:10.1007/978-1-0716-2942-0_27 |
| [43] |
BERWANGER A, STEIN S C, KANY A M, et al. Disrupting Kaposi's sarcoma-associated herpesvirus (KSHV) latent replication with a small molecule inhibitor[J]. J Med Chem, 2023, 66(15): 10782-10790. DOI:10.1021/acs.jmedchem.3c00990 |
 2024, Vol. 45
2024, Vol. 45


