2. 海军军医大学(第二军医大学)第一附属医院消化内科, 上海 200433
2. Department of Gastroenterology, The First Affiliated Hospital of Naval Medical University (Second Military Medical University), Shanghai 200433, China
人类机体是社会和环境的重要组成部分,不仅促进了社会和环境的复杂多元化发展,其自身也是变化多端的复杂系统。病理学作为研究生命机体发展机制的重要桥梁,从微观角度展现了机体发育、分化、成熟、衰老和死亡的复杂变化状态,揭示了组织器官中各类细胞的功能和宿命。近年来,随着单细胞分析技术的发展[1],病理学系统呈现出全新的复杂性。持续演变的细胞处于变化着的复杂生态系统之中,如何揭示这些复杂的单细胞表型及其与空间、时间等信息之间的因果关系成为亟待解决的关键问题。本文以肿瘤为例,阐述基于复杂系统中因果关系的单细胞病理学的意义和研究方法,为同行提供参考。
1 复杂系统中的因果关系复杂系统是由多种因素相互作用形成的复杂集体,具有敏锐的信息传递与处理能力,也是一种自身能够不断进化以适应外部变化的系统[2-3]。目前研究认为复杂系统普遍具有非线性、不确定性、不可预测性等特征[4],因此,对复杂系统的建模与研究十分困难。在复杂系统的众多作用中,因果关系是最重要的一种关系[5-6]。因果关系是一个或多个事件(“因”)对另外一个或多个事件(“果”)的作用关系,其中后一事件被认为是前一事件的结果。为了佐证因果关系而产生的因果推论是实证科学研究至关重要的一环。因果关系的推断有一部分是基于统计学上归纳推理的相关关系,因此复杂系统中因果关系的鉴定常与相关关系混淆[7-8]。
由于复杂系统中需要考虑的因素众多,特定条件下可以通过非复杂系统中的方法来判定不同事件之间的因果关系。非复杂系统根据因果关系发生的时间和形式可分为过程性因果和事件性因果[9]。过程性因果是一个动态的发生过程,其因果关系的判定远比事件性因果更加复杂,研究中采用因果环路图表示不同事件间的因果关系[10]。事件性因果更加注重特定时间点上事件的因果关系,常用随机对照试验[11-12]、双重差分法[13]、贝叶斯累加回归树模型[14]、元学习(meta learning)[15]等方法来判定不同事件间的因果关系。复杂系统中因素众多,并且存在大量的非线性关系,因果关系的识别非常困难。人们对复杂系统中因果关系的判定方法做了大量研究,包括收敛交叉映射算法[16]、彼得-克拉克瞬时条件独立性算法[17]和压缩感知[18]等。
随着人工智能技术的迅速发展,各种观测数据的因果效应估计方法应运而生。贝叶斯网络可以捕获复杂数据中的因果关系,借助数值技术可对该网络进行连续优化,提高其计算效率[19]。Prosperi等[20]通过机器学习实现因果推理和反事实预测,判定数据驱动的预测模型为精准医疗带来的干预(决策)是否具有可信度。Cui等[21]阐明了机器学习模型的风险来源,将因果推断思想引入机器学习,建立了面向线性模型和深度非线性模型的稳定学习方法体系。这些方法的建立为揭示复杂系统的因果关系提供了有力手段。
2 病理学是复杂系统病理学作为一门基础医学科学,融合了疾病的发生机制和发展规律、细胞的生理和病理形态、机体的功能代谢异常和遗传学改变等因素,是显微镜下的复杂系统。肿瘤病理学是病理学研究中的重要分支,由肿瘤细胞与其周围的间质细胞、血管、淋巴细胞等构成的肿瘤微环境是隶属于病理学下的子复杂系统。现代医学认为肿瘤的发生是基因突变不断积累的结果。细胞中包含原癌基因和抑癌基因,原癌基因促使细胞分裂增殖,抑癌基因抑制细胞增殖分化,两类基因各司其职,共同维持生物体的发展与平衡[22]。当正常细胞受到致癌因素(精神因素、物理因素和化学因素等)的诱导时,原癌基因与致癌基因发生突变,使细胞发生无限增殖不受控制的癌变,破坏生命机体的正常工作状态,形成癌症,这仅仅是隐藏在肿瘤这一子复杂系统中癌细胞起源的单向因果关系。此外,肿瘤细胞及肿瘤微环境中蛋白的异常空间表达促使肿瘤细胞向邻近组织(间质、血管、神经)的侵犯与远处转移,也是众多因素包含因果关系在内的多种相互关系共同作用的结果。对于肿瘤病理学这一复杂系统(图 1)的研究,需要在收集、分析大量病理切片的基础上,统计、归纳、推理出肿瘤的发生、发展机制,可视化演绎各种因素下的相互作用关系,从而为开发新的肿瘤治疗策略和改善患者预后提供科学依据。
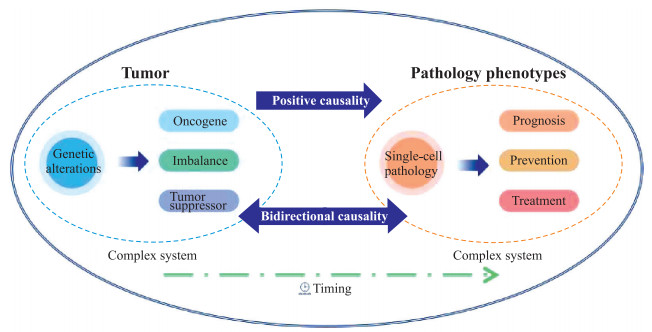
|
图 1 肿瘤病理学复杂系统 Fig 1 Complex systems of tumor pathology |
3 单细胞病理学
肿瘤的组织学和细胞学表型能够判定预后和指导治疗。目前,肿瘤主要根据组织结构及细胞形态进行病理学诊断和分级。细胞是生物学的基本单位,随着肿瘤学的研究越来越深入,传统的病理学研究方法已不能完全反映不同肿瘤细胞之间的差异性、不同患者之间肿瘤细胞的异质性及同一患者同一肿瘤内的异质性。由于分子和细胞生物学、计算生物学、软件工程、数据信息科学和人工智能的飞速发展[23],以及显微技术、染色技术和成像技术的日趋成熟,单细胞分析的精度、速度和样本通量都得到了提升和保障,单细胞研究成为现实,因此我们提出了单细胞病理学的概念。我们把单细胞病理学定义为病理学的一个分支,它主要研究单个细胞的形态、结构和功能,以及这些特性在疾病发生、发展和转归中的作用,通过分析单个细胞的形态结构、遗传学、分子生物学和生物化学特性,深入了解疾病的本质和发生机制,为疾病的诊断、治疗和预防提供有力支持。
3.1 单细胞病理学的科学性在单细胞水平上阐明组织器官的形态结构、空间分布和分子表型,可以了解机体发育进化规律和疾病演变轨迹。Greenbaum等[24]通过对66例胎盘样本的50万个细胞和588条子宫螺旋动脉进行细胞蛋白质标记和追踪分析,详细描述了孕早期的动脉重塑变化,确定了每个阶段存在哪些类型的母体免疫细胞,以及胎盘滋养细胞的确切位置。Lake等[25]基于45名健康供体和48例肾脏疾病患者的肾脏样本构建了人类肾脏细胞图谱,通过空间转录组学和3D成像分析技术绘制了肾脏51种主要细胞类型的高分辨率分布图谱,其中有28种细胞类型在肾损伤时发生状态改变。通过对肿瘤细胞的单细胞分析,人们可以观察到肿瘤细胞的异质性,理解肿瘤的复杂性和其对治疗的反应[26]。这种深入到单个细胞的观察和分析,使人们对疾病的本质有了更深入、更精确的理解。
3.2 单细胞病理学的实用性单细胞分析不仅可以帮助人们深入理解疾病的本质,还可以为临床诊断、预测疾病进展和指导治疗提供有力的支持。通过人体胎盘图谱发现,母体免疫细胞在动脉及其周围区域发挥着监控作用,免疫细胞的组成像一个时钟,基于某一样本中免疫细胞的类型即能判断出胎龄[24]。通过肾脏细胞图谱,可以识别急性肾损伤和慢性肾病的疾病亚群,为后续的个体化疗法提供依据[25]。通过对单个肿瘤细胞进行分析,医师可以更准确地判断患者的病情,制定更有效的治疗方案[26]。此外,单细胞分析技术还可以用于药物的研发和筛选,大大缩短新药的研发周期[1]。
3.3 单细胞病理学的可行性在过去20年间,科学技术飞速发展,早期的免疫组织化学和原位杂交技术,以及陆续涌现的质谱流式细胞术、单分子荧光原位杂交、多重容错性荧光原位杂交和基于空间条形码的技术等,能够弥补单细胞测序技术无法获取细胞形态结构和空间分布信息的缺陷。新近广泛开展的基于石蜡样本的空间组学技术可通过检测蛋白质及其修饰或mRNA等,研究细胞在组织样品中的相对位置关系,揭示细胞空间分布关系对疾病的影响[27]。
4 基于因果关系复杂系统的单细胞病理学研究方法 4.1 单细胞病理学研究的技术基础将形态学特征、人工智能分割技术与单细胞测序和空间转录组结合起来是研究单细胞病理学的技术基础。现有的单细胞研究主要集中在单细胞转录组测序上,利用10x Genomics、BD Rhapsody等平台分析每一个单个细胞的序列信息,揭示细胞、基因、组学上的异质性。例如,Wu等[26]利用10x Genomics的单细胞转录组测序和空间转录组学联合分析方法解析了结肠癌肝转移的免疫图谱;Fawkner-Corbett等[28]利用10x Genomics的单细胞转录组测序结合Visium空间基因表达解决方案,创建了人类肠道发育的大规模单细胞时空图谱;Boyd等[29]借助10x Genomics的空间转录组学方法发现了损伤反应性肺成纤维细胞通过血小板反应蛋白解整合素金属肽酶4损害肺功能;Moncada等[30]整合微阵列的空间转录组学和单细胞转录组测序揭示了胰腺导管腺癌的组织结构。单细胞转录组测序观察的是单个细胞的基因表达谱,忽略了整个微环境中细胞间的相互作用及空间特征,并且分析成本昂贵,样本要求高(单细胞数量达105~106个,且活细胞数目需超过80%)。
单细胞分析已经揭示了人类肿瘤之间和肿瘤内部的广泛异质性,但复杂的单细胞表型及其空间背景目前还没有反映在组织学分层中,而组织学分层是许多临床决策的基础。2020年,Jackson等[31]利用成像质谱仪同时量化乳腺癌的35个生物标志物,揭示了肿瘤微环境的多细胞特征和与不同临床结局相关的乳腺癌细胞新亚群。该研究利用肿瘤标志物量化了乳腺癌组织中的肿瘤和肿瘤微环境的多细胞特征,根据细胞组成和组织结构生成了单细胞表型和细胞群落及其与疾病关系的详细空间图,为单细胞病理学的研究奠定了基础。单细胞中蛋白质定位具有多样性和复杂性,从基本类别如细胞核和细胞质到单个蛋白质复合物的精细定位,基于蛋白质组学绘制高度解析的亚细胞定位图谱有助于了解细胞的生物学行为和病理过程,也为疾病诊治、药物筛选、药物分析等提供了新的研究手段[32]。人类生物分子图谱计划在2023年发表了人体肠道、肾脏和母胎界面(胎盘及其周围母体组织)的参考细胞图谱[24-25, 33]。该计划旨在运用前沿的单细胞组学技术和成像方法,以前所未有的规模和单细胞分辨率描述人体的主要结构和器官,构建健康组织的细胞3D图谱,揭示各类型细胞排列及其与人体不同组织和器官相互作用的新信息,为疾病的早期发现和早期治疗提供重要依据。
4.2 单细胞病理学研究的研究步骤病理切片是临床医学中疾病诊断的基础,一张病理切片中包含非常多的细胞,不管是肿瘤细胞还是组织细胞,其细胞形态特征和空间分布情况与患者的病情发展、治疗方法密切相关。利用数字病理切片结合人工智能和组学技术,有望揭示每一个细胞的形态学结构及其蕴含的生物学信息[34]。基于因果学习的人工智能技术能够像人类一样进行推理和做出决策,相对于传统的机器学习,其通过因果推理代替关联推理,最终达到反事实思考,为医师和患者提供治疗方案优化、治疗效果预测等智能服务。基于因果关系复杂系统的单细胞病理学研究大致可分为6个步骤(图 2)。
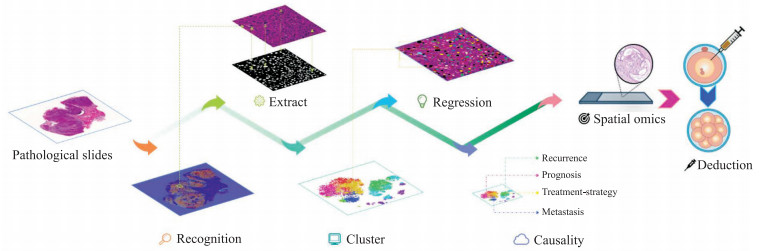
|
图 2 基于因果关系复杂系统的单细胞病理学研究步骤 Fig 2 Research steps of single-cell pathology based on complex causal systems |
(1)数据收集与整理。首先,需要准备质量合格的样本,包括新鲜组织和石蜡组织样本,确保RNA的完整性,并尽可能减少污染。组织样本制成H-E染色的高质量病理切片,并予以数字化。其次,收集患者临床信息、治疗情况和预后情况(如3年或5年生存率等)。在这一阶段,纳入的样本数量和类型越多,通过人工智能技术获得的有效特征就越多。
(2)细胞核分割与特征提取。利用图像处理算法(包括传统算法或深度学习)自动分割病理切片中的细胞核和/或全细胞分割。由于H-E染色的细胞质和细胞膜对比性不强,全细胞分割需要借助特殊染色,而细胞核染色对比性强,且特征明显,本文目前聚焦于细胞核分割。对分割后的细胞核提取特征,如纹理信息、面积大小、最大直径、最小直径等。
(3)单细胞聚类和回归。基于细胞核提取到的特征,通过聚类的方法将细胞聚成不同的类别,随后将不同类别回归到原始病理切片上。
(4)复杂系统的因果判定。对收集到的病理图像进行深度分析与处理,建立不同细胞群落与患者疗效、预后之间的复杂系统模型,最后通过现有的因果判定方法进一步揭示肿瘤的生长机制与现有细胞种类之间的关系。
(5)空间组学分析。上述研究过程会挖掘出与疗效和预后关联的一系列细胞的形态学特征。破译这些细胞和组织结构的分子谱和空间信息的技术就是空间组学技术。空间组学技术有多种,其中空间条形码技术是通过使用空间条形码寡核苷酸阵列捕获组织RNA,将RNA序列及其空间位置关联起来[27]。该技术能够将细胞位置和RNA表达结合起来,揭示细胞空间分布关系对疾病的影响。因果人工智能发现的细胞形态学特征可以在病理切片上可视化,而具有此类特征的细胞可以基于常规病理切片进行空间转录组测序,能够以最小的成本获得与疾病转归最直接的遗传学信息。
(6)演绎。空间组学初步揭示了形态学特征蕴含的信息和演变规律,利用这些信息能够识别更多样本中此类细胞的状态及其与临床特征的关系。针对发现的关键基因,在体外进行细胞学和功能学研究,了解其与疗效和预后相关性的内在逻辑,有助于更好地认识肿瘤的发生和发展机制,并辅助临床决策。
4.3 单细胞病理学研究的挑战单细胞病理学的发展主要依赖于人工智能与空间转录组技术的有机结合。目前空间转录组技术还存在一定局限性:(1)分辨率有限。目前基于测序的空间转录组技术无法实现真正意义上的单个细胞分辨率,而是检测含有5~10个细胞大小的组织区域。(2)基因通量有限。基于成像的空间转录组技术能检测到的基因通量有限,有待技术突破。(3)实验操作复杂。空间转录组实验需要精细的操作和烦琐的步骤,包括细胞固定、染色、成像和分析等,这增加了实验的难度和成本。(4)数据分析和解读的挑战。空间转录组数据具有高维度和高噪声的特点,需要先进的算法和计算资源来进行处理和分析,同时对结果的解读需要丰富的生物学知识和经验。尽管空间转录组技术存在上述缺陷,但随着技术的不断发展和优化,相信这些问题会逐渐得到解决。
5 结语经典的细胞分类并不能有效区分细胞个体功能差异,没有两个细胞是完全相同的。人工智能和单细胞病理学分析技术给细胞分类带来了重要变革,其可以定义和精准分类整个组织内每一个细胞及每个细胞组成的空间结构,并发现新的细胞类型、新的细胞功能状态和新的信号通路。基于因果关系的单细胞病理学能够从病理和临床数据中发现因果模型和因果关系,并根据因果模型来研究相应的现象或推断变量的因果变化,其最终的目的就是指导治疗方案的选择、判断转归和预后,提高患者的治疗效果和生活质量。
| [1] |
胡静涵, 胡懿凡, 蒋俊锋, 等. 单细胞转录组测序技术及其在肿瘤研究中的应用进展[J]. 海军军医大学学报, 2023, 44(7): 800-807. HU J, HU Y, JIANG J, et al. Single-cell transcriptome sequencing and its application in tumor research: recent progress[J]. Acad J Naval Med Univ, 2023, 44(7): 800-807. DOI:10.16781/j.CN31-2187/R.20230182 |
| [2] |
MCGILL E, ER V, PENNEY T, et al. Evaluation of public health interventions from a complex systems perspective: a research methods review[J]. Soc Sci Med, 2021, 272: 113697. DOI:10.1016/j.socscimed.2021.113697 |
| [3] |
SIEGENFELD A F, BAR-YAM Y. An introduction to complex systems science and its applications[J]. Complexity, 2020, 2020: 6105872. DOI:10.1155/2020/6105872 |
| [4] |
LADYMAN J, LAMBERT J, WIESNER K. What is a complex system?[J]. Eur J Philos Sci, 2013, 3: 33-67. DOI:10.1007/s13194-012-0056-8 |
| [5] |
WAGNER A. Causality in complex systems[J]. Biol Philos, 1999, 14(1): 83-101. DOI:10.1023/A:1006580900476 |
| [6] |
STAVROGLOU S K, PANTELOUS A A, STANLEY H E, et al. Unveiling causal interactions in complex systems[J]. Proc Natl Acad Sci USA, 2020, 117(14): 7599-7605. DOI:10.1073/pnas.1918269117 |
| [7] |
HUME D, LINDSAY A D. A treatise of human nature[J]. Philos Q, 1958, 8(33): 379. DOI:10.2307/2216614 |
| [8] |
BRADY H E. Models of causal inference: going beyond the Neyman-Rubin-Holland theory[C/OL]//Annual Meetings of the Political Methodology Group. Chicago, Illinois, 2003-03-30[2023-12-04]. https://public.websites.umich.edu/~wmebane/midx2003.pdf.
|
| [9] |
PEARL J. Causal diagrams and the identification of causal effects[M]. Cambridge: Cambridge University Press, 2009: 65-106.
|
| [10] |
COLETTA V R, PAGANO A, PLUCHINOTTA I, et al. Causal loop diagrams for supporting nature based solutions participatory design and performance assessment[J]. J Environ Manage, 2021, 280: 111668. DOI:10.1016/j.jenvman.2020.111668 |
| [11] |
MORGAN K L, RUBIN D B. Rerandomization to improve covariate balance in experiments[J]. Ann Statist, 2012, 40(2): 1263-1282. |
| [12] |
WU J Q, HOREWEG N, DE BRUYN M, et al. Automated causal inference in application to randomized controlled clinical trials[J]. Nat Mach Intell, 2022, 4: 436-444. DOI:10.1038/s42256-022-00470-y |
| [13] |
LECHNER M. The estimation of causal effects by difference-in-difference methods[J]. Econometrics, 2011, 4(3): 165-224. DOI:10.1561/0800000014 |
| [14] |
CHIPMAN H A, GEORGE E I, MCCULLOCH R E. BART: Bayesian additive regression trees[J]. Ann Appl Stat, 2010, 4: 266-298. DOI:10.1214/09-aoas285 |
| [15] |
TON J F, SEJDINOVIC D, FUKUMIZU K. Meta learning for causal direction[J]. arXiv E Prints, 2020: arXiv: 2007.02809. DOI: 10.48550/arXiv.2007.02809.
|
| [16] |
SUGIHARA G, MAY R, YE H, et al. Detecting causality in complex ecosystems[J]. Science, 2012, 338(6106): 496-500. DOI:10.1126/science.1227079 |
| [17] |
RUNGE J, NOWACK P, KRETSCHMER M, et al. Detecting and quantifying causal associations in large nonlinear time series datasets[J]. Sci Adv, 2019, 5(11): eaau4996. DOI:10.1126/sciadv.aau4996 |
| [18] |
ITEN R, METGER T, WILMING H, et al. Discovering physical concepts with neural networks[J]. Phys Rev Lett, 2020, 124(1): 010508. DOI:10.1103/PhysRevLett.124.010508 |
| [19] |
LUO Y, PENG J, MA J. When causal inference meets deep learning[J]. Nat Mach Intell, 2020, 2: 426-427. DOI:10.1038/s42256-020-0218-x |
| [20] |
PROSPERI M, GUO Y, SPERRIN M, et al. Causal inference and counterfactual prediction in machine learning for actionable healthcare[J]. Nat Mach Intell, 2020, 2: 369-375. DOI:10.1038/s42256-020-0197-y |
| [21] |
CUI P, ATHEY S. Stable learning establishes some common ground between causal inference and machine learning[J]. Nat Mach Intell, 2022, 4: 110-115. DOI:10.1038/s42256-022-00445-z |
| [22] |
KONTOMANOLIS E N, KOUTRAS A, SYLLAIOS A, et al. Role of oncogenes and tumor-suppressor genes in carcinogenesis: a review[J]. Anticancer Res, 2020, 40(11): 6009-6015. DOI:10.21873/anticanres.14622 |
| [23] |
于观贞, 刘西洋, 张彦春, 等. 人工智能在临床医学中的应用与思考[J]. 第二军医大学学报, 2018, 39(4): 358-365. YU G Z, LIU X Y, ZHANG Y C, et al. Artificial intelligence in clinical medicine: application and thinking[J]. Acad J Sec Mil Med Univ, 2018, 39(4): 358-365. DOI:10.16781/j.0258-879x.2018.04.0358 |
| [24] |
GREENBAUM S, AVERBUKH I, SOON E, et al. A spatially resolved timeline of the human maternal-fetal interface[J]. Nature, 2023, 619(7970): 595-605. DOI:10.1038/s41586-023-06298-9 |
| [25] |
LAKE B B, MENON R, WINFREE S, et al. An atlas of healthy and injured cell states and niches in the human kidney[J]. Nature, 2023, 619(7970): 585-594. DOI:10.1038/s41586-023-05769-3 |
| [26] |
WU Y, YANG S, MA J, et al. Spatiotemporal immune landscape of colorectal cancer liver metastasis at single-cell level[J]. Cancer Discov, 2022, 12(1): 134-153. DOI:10.1158/2159-8290.CD-21-0316 |
| [27] |
BRESSAN D, BATTISTONI G, HANNON G J. The dawn of spatial omics[J]. Science, 2023, 381(6657): eabq4964. DOI:10.1126/science.abq4964 |
| [28] |
FAWKNER-CORBETT D, ANTANAVICIUTE A, PARIKH K, et al. Spatiotemporal analysis of human intestinal development at single-cell resolution[J]. Cell, 2021, 184(3): 810-826.e23. DOI:10.1016/j.cell.2020.12.016 |
| [29] |
BOYD D F, ALLEN E K, RANDOLPH A G, et al. Exuberant fibroblast activity compromises lung function via ADAMTS4[J]. Nature, 2020, 587(7834): 466-471. DOI:10.1038/s41586-020-2877-5 |
| [30] |
MONCADA R, BARKLEY D, WAGNER F, et al. Integrating microarray-based spatial transcriptomics and single-cell RNA-seq reveals tissue architecture in pancreatic ductal adenocarcinomas[J]. Nat Biotechnol, 2020, 38(3): 333-342. DOI:10.1038/s41587-019-0392-8 |
| [31] |
JACKSON H W, FISCHER J R, ZANOTELLI V R T, et al. The single-cell pathology landscape of breast cancer[J]. Nature, 2020, 578(7796): 615-620. DOI:10.1038/s41586-019-1876-x |
| [32] |
KOBAYASHI H, CHEVERALLS K C, LEONETTI M D, et al. Self-supervised deep learning encodes high-resolution features of protein subcellular localization[J]. Nat Methods, 2022, 19(8): 995-1003. DOI:10.1038/s41592-022-01541-z |
| [33] |
HICKEY J W, BECKER W R, NEVINS S A, et al. Organization of the human intestine at single-cell resolution[J]. Nature, 2023, 619(7970): 572-584. DOI:10.1038/s41586-023-05915-x |
| [34] |
王斐, 魏培莲, 潘军, 等. 人工智能技术在组织和细胞形态学评估中的应用[J]. 第二军医大学学报, 2018, 39(8): 923-927. WANG F, WEI P L, PAN J, et al. Application of artificial intelligence technology in tissue and cell morphology assessment[J]. Acad J Sec Mil Med Univ, 2018, 39(8): 923-927. DOI:10.16781/j.0258-879x.2018.08.0923 |
 2024, Vol. 45
2024, Vol. 45


