2. 海军军医大学(第二军医大学)转化医学研究中心干细胞与再生医学研究室, 上海 200433;
3. 上海市细胞工程重点实验室, 上海 200433
2. Laboratory of Stem Cell and Regenerative Medicine, Center of Translational Medicine, Naval Medical University (Second Military Medical University), Shanghai 200433, China;
3. Shanghai Key Laboratory of Cell Engineering, Shanghai 200433, China
免疫系统包括免疫器官、免疫细胞和免疫分子,通过复杂的免疫网络在机体中对“自己”或“异己”的物质产生生物学效应并维持内环境的稳态。免疫细胞是免疫系统和免疫功能的基本单位。固有免疫细胞通过模式识别受体识别病原体相关分子模式或损伤相关分子模式,诱发炎症反应,活化抗原提呈细胞启动抗原的加工,将抗原提呈给适应性免疫细胞,实现免疫学功能——免疫防御、免疫监视和免疫自稳。因此,识别和表征免疫细胞的异质性是免疫学研究的重要内容。随着分子生物学技术的兴起,细胞的分类标准从形态、发育部位和功能发展为分化群、细胞因子、DNA和RNA等分子标记。传统的基于大批量群体的技术只能描述样本的均值或优势值,发现和应用的标志物十分有限,掩盖了可能发挥关键作用的稀有信号或细胞亚群[1],研究者们迫切需要一种具备同时、高通量和无偏分析的技术,以揭示免疫系统的组成和变化[2]。
单细胞技术是以单个细胞分辨率为基础建立分子图谱,独立地收集单细胞基因组、转录组、蛋白质组、代谢组和空间位置的信息,描述单细胞特征的新技术[3-4]。近年来,单细胞技术的发展和应用革新了以往免疫学研究中对于细胞类型、细胞功能状态、细胞暴露于外部刺激时的可塑性及细胞分化或重编程轨迹的认识,其中最成熟和最广泛应用的技术是单细胞RNA测序(single-cell RNA sequencing,scRNA-seq)技术,scRNA-seq可以全面测量单个细胞中基因的表达水平。本文对目前单细胞转录组测序技术的发展、流程、功能和其在免疫学领域的应用进展进行阐述。
1 单细胞转录组测序技术及其发展单细胞转录组测序技术通常采用scRNA-seq,一般流程(图 1)为:(1)细胞分离(部分技术同时用分子条形码标记细胞以保留原始信息);(2)细胞裂解并提取RNA分子;(3)RNA反转录为cDNA并扩增,同时用唯一分子标识符(unique molecular identifier,UMI)对每个分子进行标记;(4)UMI的汇集和下游文库的构建及测序;(5)数据分析[1]。流程中最受关注的是细胞分离和数据分析。
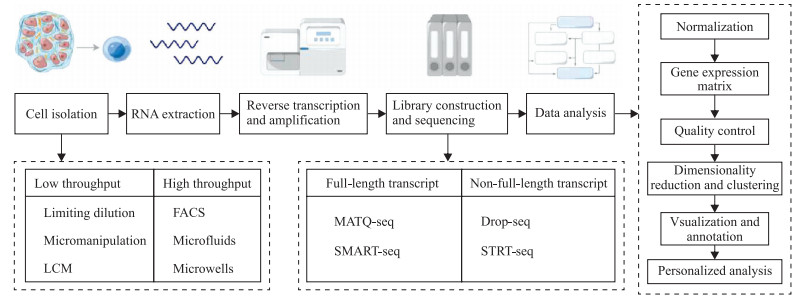
|
图 1 scRNA-seq技术流程(使用FigDraw平台) Fig 1 Scheme of scRNA-seq (by FigDraw) scRNA-seq: Single-cell RNA sequencing; LCM: Laser capture microdissection; FACS: Fluorescence activated cell sorting; MATQ-seq: Multiple annealing and dC-tailing-based quantitative single-cell RNA sequencing; SMART-seq: Switching mechanism at 5' end of the RNA transcript; Drop-seq: Droplet-based single-cell RNA sequencing; STRT-seq: Single-cell tagged reverse transcription sequencing. |
细胞分离是scRNA-seq的第1步,分离效果是影响下游结果的重要因素。将整个组织变成细胞悬液通常是通过化学消化或机械解离来实现。主要的细胞分离方法[2]包括限制性稀释、显微操作、荧光激活细胞分选(fluorescence-activated cell sorting,FACS)、激光捕获显微切割(laser capture microdissection,LCM)、微流控和微孔法。限制性稀释法和显微操作法无法进行大规模分析;LCM法能够保留空间信息,但受限于吞吐量和样本形式;FACS法能在测序前分离出具有特异性标记的细胞,但是对细胞进行的主观过滤可能丢失样本中有价值的隐藏信息;微流控法和微孔法能够高通量地将单个细胞分离到单个液滴或平板中的单个微孔中,但不能保证分离的准确性。
文库构建是通过反转录和cDNA扩增来放大单个细胞RNA的微弱信号,根据对转录本的覆盖范围分为全长和非全长转录本测序方法:前者包括RNA转录物5' 端转换机制测序(switching mechanism at 5' end of the RNA transcript,SMART-seq)[5]、单细胞通用poly(A)-独立的RNA测序[6]及多重退火和基于dC尾的定量单细胞RNA测序[7],能够检测到稀有转录本信号;后者包括液滴单细胞RNA测序(droplet-based single-cell RNA sequencing,Drop-seq)[8]和单细胞标记的逆转录测序(single-cell tagged reverse transcription sequencing,STRT-seq)[9]。为了避免在这个过程中丢失细胞来源的信息,在同一个微孔或液滴(可能来自不同细胞)中的mRNA被标记上UMI,以定量分析来自同一基因的不同转录本。
数据分析一般分为7步[10]:(1)对原始数据进行标准化处理,主要是去除背景噪音和提取条形码、UMI、基因的序列;(2)校正和整合数据;(3)构建基因的表达矩阵,了解各个基因的表达水平;(4)对细胞表达数据进行质控,3个常用的过滤指标为基因数、mRNA分子总数和线粒体基因占比;(5)降维、聚类及可视化;(6)找到每一个细胞簇的标记基因并注释;(7)个性化分析。
scRNA-seq技术的发展过程见图 2[11]。2009年scRNA-seq第1次用于描述小鼠的四细胞期卵裂球[12],这开辟了研究单个细胞的先河。2011年的一个重要进展是STRT-seq,该技术运用分子条形码标记单个细胞,实现数十个细胞的并行检测,为高通量单细胞测序技术的发展奠定了基础。2013年开始报道scRNA-seq在免疫细胞中的应用,描述了体外骨髓来源的树突状细胞(dendritic cell,DC)[13],并生成了源于组织的免疫细胞的第1个无偏scRNA-seq图谱[14]。2016年,Stubbington等[15]借助大规模平行RNA单细胞测序(massively parallel RNA single-cell sequencing,MARS-seq)技术,利用单个细胞的转录标志物,生成了一份包含超过4 000个小鼠脾脏免疫细胞的转录和蛋白质表面标记图谱,并通过将转录标志物与经典表面蛋白质标记进行整合对不同的细胞群体进行了基准测试。研究者利用这种方法鉴定出了不同的小鼠DC和单核细胞亚型,进一步提出了免疫细胞分类的问题,推动了对细胞异质性的深入研究。之后,免疫系统的scRNA-seq研究数量急剧增加。近年来,SMART-seq3[16]、Drop-seq、单细胞组合索引RNA测序(single-molecule combinatorial indexing RNA sequencing,sci-RNA-seq)[17]和10x Genomics[18]等高通量的检测方法陆续被开发和应用。
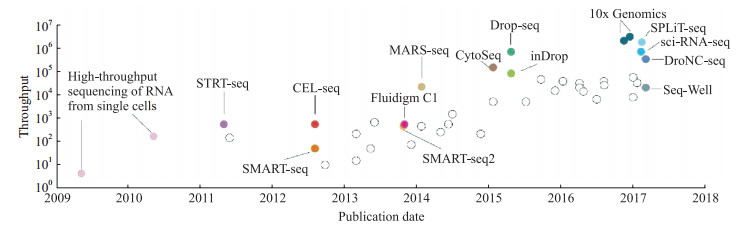
|
图 2 scRNA-seq技术的发展过程[11] Fig 2 Development of scRNA-seq[11] scRNA-seq: Single-cell RNA sequencing; STRT-seq: Single-cell tagged reverse transcription sequencing; CEL-seq: Sensitive highly-multiplexed single-cell RNA sequencing; SMART-seq: Switching mechanism at 5' end of the RNA transcript; MARS-seq: Massively parallel RNA single-cell sequencing; CytoSeq: Gene expression cytometry; Drop-seq: Droplet-based single-cell RNA sequencing; inDrop: Indexing droplet RNA sequencing; SPLiT-seq: Split pool ligation-based transcriptome sequencing; sci-RNA-seq: Single-molecule combinatorial indexing RNA sequencing; DroNC-seq: Massively parallel single-nucleus RNA sequencing. |
目前常用的scRNA-seq技术有4种类型(表 1)[1]。第1种技术是利用FACS将单细胞分选进含有特异性分子条形码的96孔或384孔板,然后对单个细胞进行标记和测序,包括MARS-seq2.0[19]和SMART-seq3。第2种被称为微流控,利用不同的乳液和微流控设备将单个细胞划分为微小液滴,并与带有分子条形码和oligo-dT的磁珠随机结合,然后对处理后的液滴进行测序,能够在一次实验中对成千上万的细胞进行分析,包括Drop-seq、索引液滴RNA测序(indexing droplet RNA sequencing,inDrop)和10x Genomics。第3种被称为微孔,借助内置携带分子条形码磁珠的微孔阵列分离样本中的单个细胞进行高通量测序,开源的平台有Seq-Well和Microwell-seq等[20]。第4种是使用组合索引,进行多轮的标记和混合来获取单个细胞的数据,如sci-RNA-seq能够以相对较低的成本生成大量的组织图谱。此外,Lovatt等[21]提出了体内转录组分析,可以保留单个细胞的空间和微环境的信息,但是无法进行高通量的分析。
2 单细胞转录组测序技术的功能
相比传统的免疫学研究技术,scRNA-seq技术具有高分辨率和高灵敏度,能够分析单个细胞的转录组、检测细胞中低表达的基因,允许在没有先验知识的情况下对细胞进行聚类,在识别免疫细胞异质性和推断免疫细胞分化轨迹方面功能优越。
2.1 在识别免疫细胞异质性方面的功能免疫细胞的异质性是指细胞间的多样性和差异性,主要体现在细胞的类型、亚群、状态和数量等方面。传统的细胞分类方法如流式细胞术和免疫荧光组织化学方法,局限于少数选定的标记分子,掩盖了稀有亚群的特殊标志,使研究者难以构画免疫细胞的全面图谱。scRNA-seq能够避免群体平均值和已有标记的干扰,完善对细胞异质性的解释[22]。Björklund等[23]对来自人类扁桃体和小肠的固有淋巴样细胞(innate lymphoid cell,ILC)进行scRNA-seq和无偏聚类分析,将细胞分为预期的4个不同群体——ILC1、ILC2、ILC3和自然杀伤(natural killer,NK)细胞,这4种已知类型细胞的转录组有助于识别未知的标记基因(图 3A);根据细胞因子反应及其分泌谱,研究揭示了3个从未报道的ILC3细胞亚群,同时预测了此亚群不同的功能。Crinier等[24]对小鼠及人的外周血和脾脏NK细胞进行了scRNA-seq分析,在小鼠、人的脾脏中分别鉴定出3、4个亚群,在小鼠和人血液中各鉴定出2个亚群。他们还比较了物种内和物种间NK细胞的转录谱,发现2个主要亚群NK1和NK2在不同器官和物种间的相似性,定义NK1为人CD56dim和小鼠CD27-CD11b+ NK细胞,NK2为人CD56bright和小鼠CD27+CD11b- NK细胞(图 3B)。
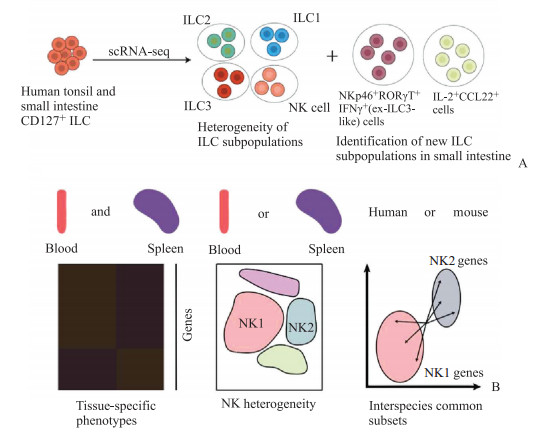
|
图 3 scRNA-seq技术在识别免疫细胞异质性方面的应用[24-25] Fig 3 scRNA-seq in identifying immune cell heterogeneity[24-25] A: Human tonsil and small intestine CD127+ ILC include all of the previously characterized ILC subpopulations (ILC1, ILC2, and ILC3) and NK and scRNA-seq allowed for the discovery of further heterogeneity of ILC subpopulations; B: scRNA-seq not only revealed the heterogeneity of NK in the blood and spleen, but also identified 2 major NK subsets transcriptionally similar across organs and species. scRNA-seq: Single-cell RNA sequencing; ILC: Innate lymphoid cell; NK: Natural killer cell; ROR: Retinoic acid receptor-related orphan receptor; IFN: Interferon; IL: Interleukin; CCL: C-C motif chemokine ligand. |
B细胞和T细胞利用V(D)J重组产生不同的B细胞受体(B cell receptor,BCR)和T细胞受体(T cell receptor,TCR),这是影响免疫细胞异质性的重要因素。在同一个个体内几乎不会出现2个完全相同的V(D)J体细胞重排,所以TCR和BCR序列可作为T细胞和B细胞克隆的独特标志。Stubbington等[15]开发了第1个从scRNA-seq重建TCR的工具TRaCer,从全长mRNA的scRNA-seq数据中检测TCR异源二聚体的多样性。TRaCer可以用来提取单个细胞TCR来源的测序结果,并将它们映射到包含所有V(D)J片段可能组合的TCR参考池中。结合转录组测序和TCR重建允许多个实验组在淋巴细胞克隆性和对感染的异质性反应之间建立联系。Rizzetto等[26]开发了VDJPuzzle。BASIC是目前唯一专门为B细胞设计的算法,也是唯一自行组装V(D)J受体序列的方法[27-28]。以上3种方法都遵循过滤和组装两大步骤。过滤步骤获取与V(D)J重组中使用的所有可能基因片段一致的scRNA-seq读数或综合数据库数据。TRaCer和VDJPuzz都利用从头转录组组装软件包Trinity来构建TCR;BASIC在受体的5'(V基因)和3'(恒定区)末端使用已识别的保守序列来执行半从头的BCR组装。
在单细胞水平上建立免疫组库有助于研究T细胞和B细胞的克隆性、受体特异性、受体亲和力和互补决定区变异性之间的联系,从而提供抗原驱动免疫反应的信息,对免疫系统疾病和新的治疗靶点的认知具有重要价值。
胸腺髓质上皮细胞(medullary thymic epithelial cell,MTEC)随机表达自体抗原是免疫系统用来实现自身耐受的一种策略。Brennecke等[29]使用scRNA-seq对MTEC转录组进行了表征,发现MTEC具有不同的异位组织限制性抗原(tissue-restricted antigen,TRA)表达模式,而被称为自身免疫调节因子的转录因子在MTEC亚群体中诱导了不同的TRA基因簇的表达。MTEC异质性的研究有助于进一步阐释T细胞的发育过程。
2.2 在推断免疫细胞分化轨迹方面的功能scRNA-seq可以帮助阐明免疫细胞被捕获时的时间状态,允许从静态快照推断时间序列,并揭示跨时间点的细胞状态连续体。这种未被观察到的维度被称为“伪时间(pseudotime)”,它可以描述细胞在发育和分化过程中的进展(图 4)[30]。
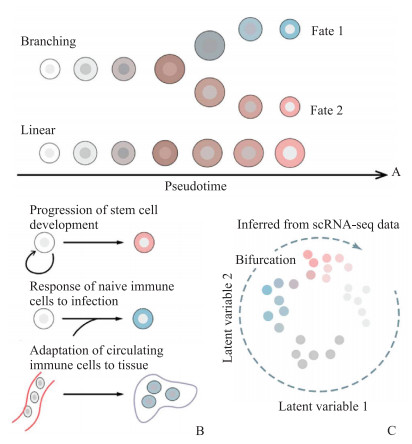
|
图 4 scRNA-seq在推断免疫细胞分化轨迹方面的应用[30] Fig 4 scRNA-seq in inferring immune cellular trajectories[30] A: Single cell shows progression of differentiation align with a "pseudotime"; B: Cell trajectory analysis involves biological processes such as the progression of stem cells towards terminal differentiation fate, the initial immune cell response to infection, and the adaptation of circulating immune cells to their eventual resident tissue; C: Pseudotime branching trajectory inferred from scRNA-seq data generated from a mouse malaria infection model. scRNA-seq: Single-cell RNA sequencing. |
SCUBA、Monocle和Wishbone等伪时序分析可以根据scRNA-seq数据利用降维算法执行轨迹推断[27],连接在空间上相近和转录本相似的细胞。类似的观点已被应用于造血干细胞的单细胞qPCR数据,使早期造血干细胞分化事件的识别、造血干细胞的分化重建和MLL-AF9型急性髓系白血病中不同细胞层次的检测成为可能[25]。
Schlitzer等[31]分析了250个经FACS分选的单细胞,这些细胞来自3个不同的前DC组。他们鉴定出一组DC祖细胞在转入cDC1或cDC2谱系时表达水平随命运选择而改变的标记基因。See等[32]使用单细胞mRNA测序结合生物信息学方法,推导出一组可分化为CD1和CD2的前DC细胞。然后他们使用质谱流式细胞技术(cytometry by time of flight,CyTOF)发现3类细胞的表面分别有CD123+、CD33+和CD45RA+标记。scRNA-seq、CyTOF和功能测定技术的综合运用证实该细胞群具有CX3CR1+CD33+标记以及独特的极化、诱导初始CD4+ T细胞增殖和产生IL-2的能力,从而将前DC与常规pDC区别开来。
Li等[33]将功能测定、质谱流式细胞术、scRNA-seq和高通量TCR测序相结合表征人胎儿肠道22个CD4+ T细胞群,接着对T细胞分化轨迹进行分析。该研究发现细胞间的转变涉及1 000多个表达基因的调控,同时这些基因可分为3个模块,结合测序前的CD4+标记发现第3模块的表达谱主要涉及细胞活化和免疫调节功能,提示了调节性T细胞的一个发育途径。
Paul等[34]将scRNA-seq应用于2 730个骨髓细胞(KIT+SCA1-Lineage-),发现转录因子CCAAT增强子结合蛋白α(CCAAT enhancer binding proteinalpha,CEBPA)和CCAAT增强子结合蛋白ε(CCAATenhancer binding protein epsilon,CEBPE)分别参与决定单核细胞和中性粒细胞的特异性分化,并且CEBPA和CEBPE的敲除导致造血阻滞。这些时间轨迹可以是单一的分叉,如CD4+T细胞命运的选择,也可以是像造血过程的复杂多分支。这些研究提供了对早期骨髓细胞、淋巴细胞和巨噬细胞分化调节的更深入的理解,也为研究疾病的发展提供了一个新思路。
3 单细胞转录组测序技术在免疫学研究中的应用 3.1 肿瘤免疫到目前为止,许多免疫分子如α干扰素、β干扰素、粒细胞-巨噬细胞集落刺激因子、IL-2、程序性死亡受体配体1等已被批准用于癌症治疗,嵌合抗原受体T细胞在治疗血液系统恶性肿瘤领域也取得了积极的成果[35]。由于免疫机制具有复杂性和多样性,上述免疫疗法临床实践并没有达到理想效果。越来越多的证据表明免疫治疗的效果与肿瘤免疫微环境(tumor immune microenvironment,TIME)的复杂性密切相关。对TIME中的单细胞进行高维分析不仅能够评估肿瘤、免疫和基质细胞的异质性,而且可以绘制与临床阶段、疾病亚型和肿瘤位置相关的TIME图谱[36]。scRNA-seq技术可以对实体瘤和血液肿瘤中的免疫细胞进行高通量基因表达谱测定,揭示免疫细胞的异质性。同时,通过对单个免疫细胞进行时间序列分析,可以推断免疫细胞在TIME中的分化和功能变化,理解TIME的组成和调控机制。
3.1.1 实体瘤实体瘤是一类起源于固体组织的肿瘤,其TIME中的免疫细胞具有高度异质性。Peng等[37]利用scRNA-seq技术对胰腺导管腺癌组织进行了分析,发现胰腺导管腺癌中T细胞、B细胞、巨噬细胞和成纤维细胞分别有5、6、5和8个亚群。他们对T细胞和巨噬细胞进行深入研究发现,着丝粒蛋白-A和着丝粒蛋白-E等细胞周期相关基因在某些亚型中优先表达,表明这些细胞具有增殖能力。此外,他们还进行了伪时间分析,显示了初始T细胞和记忆T细胞向调节性T细胞的转变过程。这些分析丰富了胰腺癌免疫微环境的基本信息,为深入理解胰腺癌的发生、发展提供新的视角。
Scheper等[38]从肿瘤常驻功能障碍T细胞中分离出TCRα/β基因进行scRNA-seq分析,并将其转染到功能供体T细胞中,测试后发现这些TCR不能识别抗原,表明TIME中的功能障碍型TCR可能作为免疫治疗的新靶点。Zheng等[39]结合scRNA-seq和TCR测序技术,在6例肝细胞癌患者的肿瘤组织、癌旁正常组织和外周血中鉴定出11个T细胞亚群,还发现CD8+ T细胞中的1个标记基因与γ干扰素下调和不良预后有关。肿瘤浸润性髓系细胞(tumor-infiltrating myeloid cell,TIM)也是诊断和治疗的重要靶点。Zilionis等[40]利用inDrop技术对人和小鼠肺癌样本中的TIM进行了测序,发现外周血髓系细胞和浸润肿瘤部位的髓系细胞显示出明显的差异,而且TIM中的一些特定基因与患者生存有密切关系。
3.1.2 血液肿瘤血液肿瘤是指起源于造血系统的恶性肿瘤,包括白血病、淋巴瘤、多发性骨髓瘤等,这些疾病通常会导致造血系统失控,使异常的白细胞、红细胞或血小板大量增生,进而占据正常造血细胞的位置和功能,导致免疫系统受损,出现贫血和出血症状。scRNA-seq可用于描述和比较血液肿瘤的转录组,寻找可能的耐药原因或病理特征。Giustacchini等[41]提出了一种将FACS、高灵敏度突变检测和全转录组分析相结合应用于同一单细胞的灵敏方法,通过这种方法研究人员分析了患者的慢性髓细胞性白血病(chronic myelogenous leukemia,CML)干细胞,区分出一个具有耐药性的亚群,对CML干细胞和正常干细胞的比较也表明CML相关的细胞存在外源性破坏的可能。Yan等[42]从急性髓系白血病患者骨髓中分离出的20个细胞,根据基因表达谱鉴定了潜在的原始细胞,并进行主成分分析以识别异常细胞群。scRNA-seq技术有助于提高诊断准确性,并改善急性髓系白血病的靶向治疗。Saadatpour等[43]研究MLL-AF9小鼠模型中急性髓系白血病肿瘤异质性,识别白血病细胞的2种不同亚型——一种类似于粒细胞/单核细胞祖细胞,另一种类似于巨噬细胞和DC,功能实验表明这些亚型在增殖率和克隆表型上存在差异。这些发现为血液肿瘤的诊断和免疫治疗提供了新的方法。
3.2 自身免疫性疾病自身免疫性疾病是机体对自身抗原的免疫反应造成的组织损伤,发病机制涉及先天和后天因素。自身免疫性疾病的临床病程在不同发病类型和个体之间有很大差异,许多发病机制也尚不清楚。类风湿性关节炎(rheumatoid arthritis,RA)是一类典型的自身免疫性疾病,它的一个重要特征是由淋巴细胞、巨噬细胞和DC组成的滑膜内层病理性增厚[2]。Stephenson等[44]把5例RA患者的滑膜组织3D打印后进行scRNA-seq分析,主要分析了成纤维细胞对纤维组织形成的关键影响,还鉴定了纤维组织中典型的免疫细胞,包括CD4+ T细胞、B细胞和NK细胞。研究发现一种NK细胞亚群表达与成纤维细胞分泌相关的趋化因子XCL1和XCL2。研究者认为,该结果提供了造血细胞和成纤维细胞在自身免疫性疾病中的第1个图谱。Kernfeld等[45]利用单细胞测序和机器学习相结合的方法来分析整个胸腺,首次发现了一些罕见的非常规淋巴细胞。从自身免疫性疾病患者获得的髓系淋巴细胞和非常规淋巴细胞显示出自身免疫基因的显著上调。这些单细胞分析有望揭示自身免疫性疾病的细胞病理特征。
3.3 移植免疫器官移植已经成为一种相对成熟的抢救终末期患者的治疗方法,但它仍然面临着新的问题——免疫系统猛烈攻击异体器官。虽然免疫抑制剂已经投入使用,但免疫排斥反应对移植物的有害影响仍然是致命的。此外,移植后的免疫抑制治疗会导致多种并发症,可能缩短患者的生存期。Wu等[46]首次尝试在人类肾脏样本上进行scRNA-seq分析,发现与健康人的肾脏相比,移植后发生免疫排斥患者的肾脏具有广泛的炎症反应。研究者还发现了一类特殊的非经典单核细胞群——同时表达典型的单核细胞标记和一组成熟DC标记,他们将这组单核细胞与外周血单核细胞数据集进行比较,发现组与组之间有明显的界线,表明一类新的细胞群似乎正在从单核细胞向DC分化。他们对假定的疾病相关基因也进行了重新评估,发现与T细胞介导排斥反应相关的30个基因主要在T细胞中表达,为基于这些基因的预测提供了依据。他们还发现一些以前认为与内皮细胞相关的异体移植病理基因在内皮细胞中没有特异性表达,有些甚至在非内皮细胞中特异性表达。这些发现为理解移植免疫中的免疫反应及预测排斥反应的程度提供了重要的思路。Yang[47]等对正常移植肝和脂肪变性移植肝组织细胞进行scRNA-seq,发现脂肪变性移植肝中富含高表达集落刺激因子3的库普弗细胞,这一促炎表型可能参与了移植过程中的肝损伤。scRNA-seq技术为供体肝脏的修复提供了新的靶点,有助于减轻脂肪肝移植损伤和扩大移植肝供体库。
4 展望单细胞转录组测序技术不断发展和完善,已经成为免疫学研究的强大工具,能够帮助我们更深入地了解复杂的免疫网络——发现新型免疫细胞类型和亚型、揭示免疫应答的分子机制、分析免疫细胞的功能状态和研究免疫细胞的发育分化等[48-49]。然而,随着单细胞技术的成熟和公布的数据集指数级增长,统一注释和命名的缺乏限制了免疫学家利用单细胞数据进行临床研究的范围。为此,研究者们正在构建来自各种技术、物种和数据类型(如RNA、蛋白质、表观遗传学、空间数据等)的数据集成工具,以便注释和表征指定的细胞亚群,实现学术界、工业界和临床机构之间的数据共享和合作。目前,一些数据集已经具有上述实用性,如人类细胞图谱[50]和CZIcellxgene[51](https://github.com/chanzuckerberg/cellxgenedocumentation/blob/main/README.md)。
以scRNA-seq为代表的单细胞测序技术本身依然面临着很大挑战。在数据处理方面,存在的问题主要包括批次效应、缺失效应和扩增偏差[27]。技术批次效应会导致数据人为地逐批聚类,掩盖生物学变异;缺失效应会导致非生物学零点值的富集,导致双峰表达谱,不利于分组分析;某些基因可能会优先扩增,引入难以调整的偏差。后续技术的改进将逐步克服这些问题,如UMI的使用有助于检测扩增偏差,更准确地定量起始的RNA水平。
细胞的状态是由其基因组、表观基因组、转录组、蛋白质组、代谢组和微环境共同决定的[52],单组学技术如scRNA-seq只能提供单一层面的静态分子信息,无法反映免疫系统从基因组到表型组的分子规律。在单细胞和空间分辨率上的多组学技术能够研究基因调控的分子动力学,从而识别内源性和外源性因子对细胞异质性和功能状态的影响[53]。单细胞多组学分析可以根据分析物的性质以及用细胞条形码的方法进行分类。单细胞转录组和表观遗传组联合分析的方法有物理分离、预扩增和分割、序列分割或组合索引[54]。常见表观遗传的信息,如DNA甲基化、染色质可及性、组蛋白修饰、转录因子和染色质重塑复合物等,都可以在单个细胞中与转录组平行检测。Ma等[55]开发了同时高通量ATAC和RNA表达测序(simultaneous high-throughput ATAC and RNA expression with sequencing,SHARE-seq)技术,实现单个细胞高通量同步转座酶检测染色质可及性和RNA测序。单细胞转录组与蛋白质组联合分析中借助通过测序扩展成簇规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)兼容的转录组和表位的细胞索引(expanded CRISPR-compatible cellular indexing of transcriptomes and epitopes by sequencing,ECCITE-seq)并行检测免疫细胞转录组与表面蛋白、克隆型和CRIPSR扰动[3]。
空间多组学分析涉及分辨率、样本类型、分析顺序和分析物的数量。2022年,空间多组学技术被Nature杂志列为年度最值得关注的7项技术之一[56]。目前大多数正在进行的空间多组学的基本原则主要有6类[54]:基于阵列的空间转录组学、微流控确定性条形码、DNA抗体标签、多重单分子荧光原位杂交、原位测序和质谱成像。其中,基于阵列的空间转录组方法允许通过以下方式进行全转录组分析:从固定和透化的组织切片中捕获聚腺苷酸化的RNA,使用数千个带有空间条形码的寡dT点阵列。随后进行逆转录和文库制备,通过读取带有条形码的cDNA高通量测序结果,将转录本定位到成像组织切片中的特定位置。
这些单细胞数据将通过加速开发可靠的诊断和预后标志物、建立模型以在单个患者的数据基础上实现精准治疗、发现新的治疗靶点和开发新的免疫治疗分子等方面彻底改变我们诊断和治疗疾病的能力[1]。
| [1] |
GINHOUX F, YALIN A, DUTERTRE C A, et al. Single-cell immunology: past, present, and future[J]. Immunity, 2022, 55(3): 393-404. DOI:10.1016/j.immuni.2022.02.006 |
| [2] |
CAO Y, QIU Y, TU G, et al. Single-cell RNA sequencing in immunology[J]. Curr Genomics, 2020, 21(8): 564-575. DOI:10.2174/1389202921999201020203249 |
| [3] |
ZHU C, PREISSL S, REN B. Single-cell multimodal omics: the power of many[J]. Nat Meth, 2020, 17: 11-14. DOI:10.1038/s41592-019-0691-5 |
| [4] |
LOUIE R H, LUCIANI F. Recent advances in single-cell multimodal analysis to study immune cells[J]. Immunol Cell Biol, 2021, 99(2): 157-167. DOI:10.1111/imcb.12432 |
| [5] |
RAMSKÖLD D, LUO S, WANG Y C, et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells[J]. Nat Biotechnol, 2012, 30(8): 777-782. DOI:10.1038/nbt.2282 |
| [6] |
FAN X, ZHANG X, WU X, et al. Single-cell RNA-seq transcriptome analysis of linear and circular RNAs in mouse preimplantation embryos[J]. Genome Biol, 2015, 16(1): 148. DOI:10.1186/s13059-015-0706-1 |
| [7] |
SHENG K, CAO W, NIU Y, et al. Effective detection of variation in single-cell transcriptomes using MATQ-seq[J]. Nat Methods, 2017, 14(3): 267-270. DOI:10.1038/nmeth.4145 |
| [8] |
KLEIN A M, MAZUTIS L, AKARTUNA I, et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells[J]. Cell, 2015, 161(5): 1187-1201. DOI:10.1016/j.cell.2015.04.044 |
| [9] |
NATARAJAN K N. Single-cell tagged reverse transcription (STRT-seq)[J]. Methods Mol Biol, 2019, 1979: 133-153. DOI:10.1007/978-1-4939-9240-9_9 |
| [10] |
LUECKEN M D, THEIS F J. Current best practices in single-cell RNA-seq analysis: a tutorial[J]. Mol Syst Biol, 2019, 15(6): e8746. DOI:10.15252/msb.20188746 |
| [11] |
SVENSSON V, VENTO-TORMO R, TEICHMANN S A. Exponential scaling of single-cell RNA-seq in the past decade[J]. Nat Protoc, 2018, 13(4): 599-604. DOI:10.1038/nprot.2017.149 |
| [12] |
TANG F, BARBACIORU C, WANG Y, et al. mRNA-seq whole-transcriptome analysis of a single cell[J]. Nat Methods, 2009, 6(5): 377-382. DOI:10.1038/nmeth.1315 |
| [13] |
SHAPIRO E, BIEZUNER T, LINNARSSON S. Single-cell sequencing-based technologies will revolutionize whole-organism science[J]. Nat Rev Genet, 2013, 14(9): 618-630. DOI:10.1038/nrg3542 |
| [14] |
JAITIN D A, KENIGSBERG E, KEREN-SHAUL H, et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types[J]. Science, 2014, 343(6172): 776-779. DOI:10.1126/science.1247651 |
| [15] |
STUBBINGTON M J T, LÖNNBERG T, PROSERPIO V, et al. T cell fate and clonality inference from single-cell transcriptomes[J]. Nat Methods, 2016, 13(4): 329-332. DOI:10.1038/nmeth.3800 |
| [16] |
HAGEMANN-JENSEN M, ZIEGENHAIN C, CHEN P, et al. Single-cell RNA counting at allele and isoform resolution using Smart-seq3[J]. Nat Biotechnol, 2020, 38(6): 708-714. DOI:10.1038/s41587-020-0497-0 |
| [17] |
CAO J, PACKER J S, RAMANI V, et al. Comprehensive single-cell transcriptional profiling of a multicellular organism[J]. Science, 2017, 357(6352): 661-667. DOI:10.1126/science.aam8940 |
| [18] |
GAO C, ZHANG M, CHEN L. The comparison of two single-cell sequencing platforms: BD Rhapsody and 10x Genomics chromium[J]. Curr Genomics, 2020, 21(8): 602-609. DOI:10.2174/1389202921999200625220812 |
| [19] |
KEREN-SHAUL H, KENIGSBERG E, JAITIN D A, et al. MARS-seq2.0: an experimental and analytical pipeline for indexed sorting combined with single-cell RNA sequencing[J]. Nat Protoc, 2019, 14(6): 1841-1862. DOI:10.1038/s41596-019-0164-4 |
| [20] |
HAN X, WANG R, ZHOU Y, et al. Mapping the mouse cell atlas by Microwell-seq[J]. Cell, 2018, 172(5): 1091-1107.e17. DOI:10.1016/j.cell.2018.02.001 |
| [21] |
LOVATT D, RUBLE B K, LEE J, et al. Transcriptome in vivo analysis (TIVA) of spatially defined single cells in live tissue[J]. Nat Meth, 2014, 11: 190-196. DOI:10.1038/nmeth.2804 |
| [22] |
EFREMOVA M, VENTO-TORMO R, PARK J E, et al. Immunology in the era of single-cell technologies[J]. Annu Rev Immunol, 2020, 38: 727-757. DOI:10.1146/annurev-immunol-090419-020340 |
| [23] |
BJÖRKLUND Å K, FORKEL M, PICELLI S, et al. The heterogeneity of human CD127+ innate lymphoid cells revealed by single-cell RNA sequencing[J]. Nat Immunol, 2016, 17(4): 451-460. DOI:10.1038/ni.3368 |
| [24] |
CRINIER A, MILPIED P, ESCALIÈRE B, et al. High-dimensional single-cell analysis identifies organ-specific signatures and conserved NK cell subsets in humans and mice[J]. Immunity, 2018, 49(5): 971-986.e5. DOI:10.1016/j.immuni.2018.09.009 |
| [25] |
PAPALEXI E, SATIJA R. Single-cell RNA sequencing to explore immune cell heterogeneity[J]. Nat Rev Immunol, 2018, 18(1): 35-45. DOI:10.1038/nri.2017.76 |
| [26] |
RIZZETTO S, KOPPSTEIN D N P, SAMIR J, et al. B-cell receptor reconstruction from single-cell RNA-seq with VDJPuzzle[J]. Bioinformatics, 2018, 34(16): 2846-2847. DOI:10.1093/bioinformatics/bty203 |
| [27] |
NEU K E, TANG Q, WILSON P C, et al. Single-cell genomics: approaches and utility in immunology[J]. Trends Immunol, 2017, 38(2): 140-149. DOI:10.1016/j.it.2016.12.001 |
| [28] |
CANZAR S, NEU K E, TANG Q, et al. BASIC: BCR assembly from single cells[J]. Bioinformatics, 2017, 33(3): 425-427. DOI:10.1093/bioinformatics/btw631 |
| [29] |
BRENNECKE P, REYES A, PINTO S, et al. Single-cell transcriptome analysis reveals coordinated ectopic gene-expression patterns in medullary thymic epithelial cells[J]. Nat Immunol, 2015, 16(9): 933-941. DOI:10.1038/ni.3246 |
| [30] |
STUBBINGTON M J T, ROZENBLATT-ROSEN O, REGEV A, et al. Single-cell transcriptomics to explore the immune system in health and disease[J]. Science, 2017, 358(6359): 58-63. DOI:10.1126/science.aan6828 |
| [31] |
SCHLITZER A, SIVAKAMASUNDARI V, CHEN J, et al. Identification of cDC1- and cDC2-committed DC progenitors reveals early lineage priming at the common DC progenitor stage in the bone marrow[J]. Nat Immunol, 2015, 16(7): 718-728. DOI:10.1038/ni.3200 |
| [32] |
SEE P, DUTERTRE C A, CHEN J, et al. Mapping the human DC lineage through the integration of high-dimensional techniques[J]. Science, 2017, 356(6342): eaag3009. DOI:10.1126/science.aag3009 |
| [33] |
LI N, VAN UNEN V, ABDELAAL T, et al. Memory CD4+ T cells are generated in the human fetal intestine[J]. Nat Immunol, 2019, 20(3): 301-312. DOI:10.1038/s41590-018-0294-9 |
| [34] |
PAUL F, ARKIN Y, GILADI A, et al. Transcriptional heterogeneity and lineage commitment in myeloid progenitors[J]. Cell, 2015, 163(7): 1663-1677. DOI:10.1016/j.cell.2015.11.013 |
| [35] |
LIU Z, LI H, DANG Q, et al. Integrative insights and clinical applications of single-cell sequencing in cancer immunotherapy[J]. Cell Mol Life Sci, 2022, 79(11): 577. DOI:10.1007/s00018-022-04608-4 |
| [36] |
GOHIL S H, IORGULESCU J B, BRAUN D A, et al. Applying high-dimensional single-cell technologies to the analysis of cancer immunotherapy[J]. Nat Rev Clin Oncol, 2021, 18: 244-256. DOI:10.1038/s41571-020-00449-x |
| [37] |
PENG J, SUN B F, CHEN C Y, et al. Single-cell RNA-seq highlights intra-tumoral heterogeneity and malignant progression in pancreatic ductal adenocarcinoma[J]. Cell Res, 2019, 29(9): 725-738. DOI:10.1038/s41422-019-0195-y |
| [38] |
SCHEPER W, KELDERMAN S, FANCHI L F, et al. Low and variable tumor reactivity of the intratumoral TCR repertoire in human cancers[J]. Nat Med, 2019, 25(1): 89-94. DOI:10.1038/s41591-018-0266-5 |
| [39] |
ZHENG C, ZHENG L, YOO J K, et al. Landscape of infiltrating T cells in liver cancer revealed by single-cell sequencing[J]. Cell, 2017, 169(7): 1342-1356.e16. DOI:10.1016/j.cell.2017.05.035 |
| [40] |
ZILIONIS R, ENGBLOM C, PFIRSCHKE C, et al. Single-cell transcriptomics of human and mouse lung cancers reveals conserved myeloid populations across individuals and species[J]. Immunity, 2019, 50(5): 1317-1334.e10. DOI:10.1016/j.immuni.2019.03.009 |
| [41] |
GIUSTACCHINI A, THONGJUEA S, BARKAS N, et al. Single-cell transcriptomics uncovers distinct molecular signatures of stem cells in chronic myeloid leukemia[J]. Nat Med, 2017, 23(6): 692-702. DOI:10.1038/nm.4336 |
| [42] |
YAN B, HU Y, BAN K H K, et al. Single-cell genomic profiling of acute myeloid leukemia for clinical use: a pilot study[J]. Oncol Lett, 2017, 13(3): 1625-1630. DOI:10.3892/ol.2017.5669 |
| [43] |
SAADATPOUR A, GUO G, ORKIN S H, et al. Characterizing heterogeneity in leukemic cells using single-cell gene expression analysis[J]. Genome Biol, 2014, 15(12): 525. DOI:10.1186/s13059-014-0525-9 |
| [44] |
STEPHENSON W, DONLIN L T, BUTLER A, et al. Single-cell RNA-seq of rheumatoid arthritis synovial tissue using low-cost microfluidic instrumentation[J]. Nat Commun, 2018, 9(1): 791. DOI:10.1038/s41467-017-02659-x |
| [45] |
KERNFELD E M, GENGA R M J, NEHERIN K, et al. A single-cell transcriptomic atlas of Thymus organogenesis resolves cell types and developmental maturation[J]. Immunity, 2018, 48(6): 1258-1270.e6. DOI:10.1016/j.immuni.2018.04.015 |
| [46] |
WU H, MALONE A F, DONNELLY E L, et al. Single-cell transcriptomics of a human kidney allograft biopsy specimen defines a diverse inflammatory response[J]. J Am Soc Nephrol, 2018, 29(8): 2069-2080. DOI:10.1681/ASN.2018020125 |
| [47] |
YANG X, LU D, WANG R, et al. Single-cell profiling reveals distinct immune phenotypes that contribute to ischaemia-reperfusion injury after steatotic liver transplantation[J]. Cell Prolif, 2021, 54(10): e13116. DOI:10.1111/cpr.13116 |
| [48] |
LI Y, ZHAI T, CHEN J, et al. Water-dispersible gold nanoclusters: synthesis strategies, optical properties, and biological applications[J]. Chemistry, 2022, 28(10): e202103736. DOI:10.1002/chem.202103736 |
| [49] |
LI Y, LU H, QU Z, et al. Phase transferring luminescent gold nanoclusters via single-stranded DNA[J]. Sci China Chem, 2022, 65(6): 1212-1220. DOI:10.1007/s11426-022-1238-2 |
| [50] |
REGEV A, TEICHMANN S A, LANDER E S, et al. The human cell atlas[J]. Elife, 2017, 6: e27041. DOI:10.7554/elife.27041 |
| [51] |
NER-GAON H, MELCHIOR A, GOLAN N, et al. JingleBells: a repository of immune-related single-cell RNA-sequencing datasets[J]. J Immunol, 2017, 198(9): 3375-3379. DOI:10.4049/jimmunol.1700272 |
| [52] |
MACAULAY I C, PONTING C P, VOET T. Single-cell multiomics: multiple measurements from single cells[J]. Trends Genet, 2017, 33(2): 155-168. DOI:10.1016/j.tig.2016.12.003 |
| [53] |
STUART T, SATIJA R. Integrative single-cell analysis[J]. Nat Rev Genet, 2019, 20: 257-272. DOI:10.1038/s41576-019-0093-7 |
| [54] |
VANDEREYKEN K, SIFRIM A, THIENPONT B, et al. Methods and applications for single-cell and spatial multi-omics[J]. Nat Rev Genet, 2023, 24(8): 494-515. DOI:10.1038/s41576-023-00580-2 |
| [55] |
MA S, ZHANG B, LAFAVE L M, et al. Chromatin potential identified by shared single-cell profiling of RNA and chromatin[J]. Cell, 2020, 183(4): 1103-1116.e20. DOI:10.1016/j.cell.2020.09.056 |
| [56] |
EISENSTEIN M. Seven technologies to watch in 2022[J]. Nature, 2022, 601: 658-661. DOI:10.1038/d41586-022-00163-x |
 2024, Vol. 45
2024, Vol. 45


