2. 海军军医大学(第二军医大学)基础医学院组织胚胎学教研室, 上海 200433
2. Department of Histology and Embryology, College of Basic Medical Sciences, Naval Medical University(Second Military Medical University), Shanghai 200433, China
自1977年Sanger发明双脱氧链终止法开启了第一代测序技术后,测序技术逐渐成为人类探索基因最有用的工具。随着技术的不断进步,测序的样本逐渐由组织或大量细胞发展至如今的单细胞层面[1],即单细胞测序,该技术包括单细胞基因组测序、单细胞转录组测序和单细胞表观遗传组测序等,其中以单细胞转录组测序最为通用。单细胞转录组测序技术始于2009年,Tang等[2]基于高通量测序平台对单个小鼠卵裂球和卵母细胞的转录组进行的测序,此后该技术方法不断改进革新,商业化的单细胞高通量测序也已发展成熟并得到广泛应用,对生命科学的研究起到了巨大的推动作用。
单细胞转录组测序通过对单个细胞中转录本进行非靶向定量,实现了在单细胞分辨率下解析基因表达情况的目标,能够更加深入地研究细胞类型、细胞功能、细胞亚群及其异质性、细胞谱系等,还常用于识别新的细胞类型、确认罕见的细胞群、构建细胞状态和系统发育的图谱。该技术具有独特的优势,已成为生命科学研究的焦点技术,改变了生物界和医学界许多领域的研究方式[3]。在肿瘤领域,单细胞转录组测序技术已在肿瘤异质性、肿瘤转移、肿瘤微环境、抗肿瘤药物研发等方面的研究中广泛应用,拓展并加深了人们对肿瘤的认识,为肿瘤的防治提供了新的思路和策略。
1 单细胞转录组测序技术流程及技术发展单细胞转录组测序常使用单细胞RNA测序(single-cell RNA sequencing,scRNA-seq)获取每一个细胞的RNA表达数据,前提是使用新鲜组织或在组织保存液中暂时保存的组织;对于冷冻保存的组织样本,则可采用单细胞核RNA测序(single-nucleus RNA sequencing,snRNA-seq)[4]。2017年,“带有DNA标签的抗体”被引入该技术体系,通过联合基于抗体检测的蛋白标记技术,进一步发展出细胞索引转录组和表位测序(cellular indexing of transcriptomes and epitopes by sequencing,CITE-seq)[5]及RNA表达和蛋白测序(RNA expression and protein sequencing,REAP-seq)[6],实现了单细胞维度下的蛋白质信息与转录组信息的整合。单细胞多组学测序可以同时对单个细胞中的基因组、转录组、表观遗传组和染色质可及性中的2种及以上同时测序,并开展联合分析[7]。
单细胞转录组测序主要流程包括单细胞的分离获取、核酸的扩增、测序文库的构建、测序和分析(图 1)。
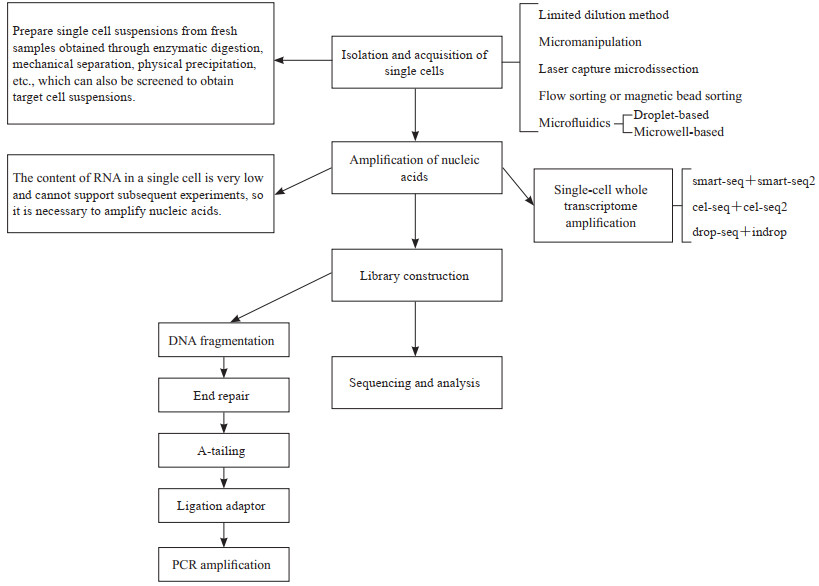
|
图 1 单细胞转录组测序技术的流程和方法 Fig 1 Process and methods of single-cell transcriptome sequencing smart-seq: Switching mechanism at 5' end of the RNA transcript; smart-seq2: Switching mechanism at 5' end of the RNA transcript 2; cel-seq: Cell expression by linear amplification and sequencing; cel-seq2: Cell expression by linear amplification and sequencing 2; drop-seq: Droplet-based scRNA-seq; indrop: Indexing droplet RNA sequencing; PCR: Polymerase chain reaction. |
获取高活性比例的单细胞悬液是单细胞测序成功的前提。样本通过酶解、机械分离、物理沉淀等方法制成单细胞悬液,亦可进行筛选得到靶细胞悬液。单细胞测序发展之初由于单细胞分离获取技术受限,通常使用有限稀释法、显微操作法、激光捕获显微切割、流式分选或磁珠分选等方法,但只能检测数量有限的细胞(约几十至几百个)。随着条形码单细胞标识技术的出现,单细胞测序进入了高通量时代。微珠携带有数百万条寡核糖核苷酸,包含细胞条形码和唯一分子标识符(unique molecular identifier,UMI),分别用于标识细胞和核酸。使用微珠时,主要通过微滴技术和微孔技术来获取单细胞及其中的核酸:前者通过微流控技术使细胞悬液中的单个细胞仅与一个微珠结合,随后输入油相孔道,形成“油包水”的微滴;后者通过微流控技术将细胞悬液中的单个细胞分散至各个微孔中,再分别与一个微珠结合[8]。
单细胞中所含全部RNA(即全转录组)含量极低,通常为pg级,无法达到建库测序的需要,因此先要进行核酸扩增,再进行cDNA文库构建、测序和分析。近十年来,单细胞全转录组扩增测序方法主要包括RNA转录物5'端转换机制测序(switching mechanism at 5' end of the RNA transcript,smart-seq)[9]及RNA转录物5'端转换机制测序2(switching mechanism at 5' end of the RNA transcript 2,smart-seq2)[10]、细胞表达线性扩增测序(cell expression by linear amplification and sequencing,cel-seq)[11]及细胞表达线性扩增测序2(cell expression by linear amplification and sequencing 2,cel-seq2)[12]、液滴单细胞RNA测序测序(droplet-based single-cell RNA sequencing,drop-seq)[13]和索引液滴RNA测序(indexing droplet RNA sequencing,indrop)[14]等技术(表 1),不同方法各具优势和不足。近年来还不断有新的方法涌现,如使用RNA转录物5'端转换机制测序3(switching mechanism at 5' end of the RNA transcript 3,smart-seq3)在等位基因和同源异构体分辨率下检测单细胞RNA[15]和利用dA尾对单细胞进行大量转录组分析测序(vast transcriptome analysis of single cells by dA-tailing,VASA-seq)来检测单细胞中的总转录组[16]。与此同时,研究者们也意识到组织原位空间生态位内单细胞研究的必要性并开发了相应的技术[17],将单细胞转录组测序与空间转录组测序联合应用,更好地解释了细胞间的相互联系和相互作用,加深了对细胞微环境的认识[18]。
|
|
表 1 单细胞全转录组扩增方法 Tab 1 Methods of single-cell whole transcriptome amplification |
2 单细胞转录组测序技术在肿瘤研究中的应用 2.1 肿瘤异质性及细胞亚群划分研究
肿瘤异质性是指恶性肿瘤在生长过程中,经过多次分裂增殖后,其子代细胞呈现出基因或分子生物学方面的改变,使肿瘤的生长速度、侵袭能力、对药物的敏感性和预后等方面产生差异。单细胞转录组测序可以清晰地显示肿瘤内部细胞构成,将肿瘤细胞划分成不同亚群并追踪谱系,有助于理解肿瘤异质性的起源和形成机制。Neftel等[19]通过包括scRNA-seq在内的整合分析,发现胶质母细胞瘤中的恶性细胞以4种主要细胞状态存在,并揭示了其转录特征。Couturier等[20]使用scRNA-seq研究胶质母细胞瘤,发现肿瘤中容易分裂的癌细胞往往与神经胶质祖细胞有着共同的基因表达特征,提示这类细胞可能是肿瘤难以彻底消灭的原因。Castellan等[21]从scRNA-seq数据中分析鉴定了胶质母细胞瘤中胶质母细胞瘤干细胞(glioblastoma stem cell,GSC)的基因调控网络,进而发现Yes相关蛋白(Yes-associated protein,YAP)/PDZ结合基序转录共激活因子(transcriptional coactivator with PDZ-binding motif,TAZ)是调节GSC干性和可塑性这一肿瘤生物学核心事件的关键分子引擎。
Zhang等[22]利用scRNA-seq分析原发性胃腺癌的转录异质性,确定了5个具有不同表达谱的细胞亚群,并用一组分化相关基因揭示了肿瘤内部和肿瘤之间分化程度的高度多样性。Chen等[23]对膀胱癌进行scRNA-seq分析,发现其包含8种类型的细胞,其中上皮细胞又分为17个亚群,表明膀胱癌具有高度异质性。He等[24]揭示了携带表皮生长因子受体(epidermal growth factor receptor,EGFR)突变的早期肺腺癌的腺癌细胞异质性,并提示肿瘤细胞、基质细胞和免疫浸润细胞之间的复杂相互作用。Leader等[25]通过scRNA-seq和CITE-seq对人非小细胞肺癌进行分析,研究了瘤突变负荷和肿瘤蛋白53突变情况,构建了非小细胞肺癌的细化分类及患者分层的数据库。Van Galen等[26]借助scRNA-seq技术对40份骨髓样本(来自16例急性髓系白血病患者和5名健康供体)进行数据分析,将造血干细胞的分化类型分为15个主要的细胞亚群,并通过机器学习区分良恶性细胞。这些研究极大地加深了人们对肿瘤异质性的认知,划分出的细胞亚群有利于肿瘤发展轨迹的识别,为寻找肿瘤治疗的靶细胞和靶点提供了基础数据。
2.2 肿瘤细胞转移研究肿瘤转移是患者死亡的重要原因之一,从单细胞水平分析影响肿瘤转移的危险因素,可以为肿瘤转移防治提供新的策略。Xu等[27]通过scRNA-seq鉴定了9个乳腺癌细胞亚群,其中乳腺癌干细胞存在于原发性肿瘤中,并且可以浸润转移到淋巴结,提示应用scRNA-seq可以提前发现具有潜在转移风险的癌细胞亚群,尽早做出干预以取得更好的预后。Kim等[28]通过scRNA-seq发现在肺腺癌中包含3种肿瘤细胞状态(tumor cell state,tS),包括不同比例的tS1和tS2细胞及数量较少的tS3细胞,其中tS2信号基因的高表达与肺腺癌患者总生存期较低、肿瘤进展和肿瘤转移密切相关,因此通过靶向药物阻断tS2相关基因的高表达有望延长患者总生存期。同时该研究还证实,基质和免疫细胞变化创造了促肿瘤和免疫抑制的微环境,并且在肿瘤组织和转移淋巴结中发现大量髓系细胞,提示髓系细胞在肿瘤转移中可能发挥重要作用。
循环肿瘤细胞(circulating tumor cell,CTC)是存在于外周血中的各类肿瘤细胞的统称,被认为是肿瘤转移性前体细胞,少部分CTC能逃逸机体的免疫监视进而产生转移灶。CTC-Tracer是在域适应基础上开发出的一种深度迁移学习方法,将从血液样本中分离的CTC的scRNA-seq表达谱作为输入,通过有效地映射原发肿瘤的scRNA-seq图谱,CTC-Tracer可以识别CTC并追踪其病变来源,经验证CTC-Tracer具有极高的稳定性和准确性,将极大地推动液体活检技术在基础研究和临床研究中的应用[29]。
2.3 肿瘤微环境研究肿瘤微环境是指肿瘤细胞所处的周围微环境,包括肿瘤细胞、成纤维细胞、免疫细胞、骨髓源性细胞和细胞外基质、微血管及各种信号分子。肿瘤微环境中各种成分相互制约、相互促进,构成复杂的调控网络。Katzenelenbogen等[30]提出了INs-seq(intracellular staining and sequencing,一种细胞内蛋白免疫检测联合scRNA-seq的整合技术),通过该技术发现对小鼠髓系细胞触发受体2(triggering receptor expressed on myeloid cells-2,TREM2)进行基因消融可抑制肿瘤内调节性骨髓细胞的积聚,使功能失调的CD8+ T细胞显著减少,而自然杀伤细胞和细胞毒性T细胞增加,肿瘤生长也随之减缓。Molgora等[31]通过scRNA-seq发现TREM2的缺失和抗性均与肿瘤浸润中缺乏甘露糖受体C型1(mannose receptor C-type 1,MRC1+)和C-X3-C基序趋化因子受体1(C-X3-C motif chemokine receptor 1,CX3CR1+)巨噬细胞相关,髓系亚群表达免疫刺激分子促进T细胞反应的改善。通过测序分析肿瘤进程中T细胞、B细胞、巨噬细胞等免疫细胞的变化,可以发现自身免疫系统功能紊乱与肿瘤演变息息相关。由此推断,可通过调整自身免疫细胞的功能状态及数量减少自身免疫系统功能紊乱,增加髓系亚群表达免疫刺激分子,达到杀灭肿瘤的目的。
免疫检查点抑制剂能够利用肿瘤免疫逃逸的机制加强机体对肿瘤的监视,通过自身的免疫细胞来杀灭肿瘤细胞。在三阴性乳腺癌和人EGFR2过表达的乳腺癌中,肿瘤浸润淋巴细胞的数量是患者的关键预后因素。Savas等[32]通过对乳腺癌中分选T细胞的scRNA-seq,发现具有肿瘤浸润淋巴细胞的乳腺癌中含有CD8+组织驻留记忆T细胞(tissue-resident memory T cell,TRM),并且CD8+ TRM在乳腺癌中具有较高的细胞毒性活性,与三阴性乳腺癌患者更好的预后相关。CD8+ TRM有助于乳腺癌免疫监视,并且是免疫检查点抑制调节的关键靶标。Xu等[27]还发现,由nectin细胞黏附分子2(nectin cell adhesion molecule 2,NECTIN2)-具有免疫球蛋白和免疫受体酪氨酸抑制基序结构域的T细胞免疫受体(T-cell immunoreceptor with immunoglobulin and immunoreceptor tyrosine-based inhibitory motif domains,TIGIT)介导的转移性乳腺癌细胞和肿瘤微环境细胞之间的相互作用促进了免疫逃逸和淋巴结转移的发生,提示干预肿瘤细胞和肿瘤微环境细胞之间的相互联系可以破坏肿瘤微环境,减少肿瘤转移。
成纤维细胞是肿瘤微环境的另一核心成分。Zhou等[33]借助单细胞多组学测序发现,结直肠癌肿瘤组织中带有体细胞拷贝数改变的成纤维细胞比例远高于相邻的正常组织,其中7号染色体的富集最为强烈,同时发现5个基因是结直肠癌预后较差的成纤维细胞特异性生物标志物。Chen等[23]通过scRNA-seq在膀胱癌肿瘤微环境中鉴定出19种细胞类型,其中包含2种成纤维细胞亚型,即炎症性肿瘤相关成纤维细胞(inflammatory cancer-associated fibroblast,iCAF)和肌肿瘤相关成纤维细胞(myo cancer-associated fibroblast,mCAF),其中iCAF与不良预后密切相关。借助scRNA-seq对比正常组织和不同时期肿瘤组织中成纤维细胞的亚型,探究其在肿瘤进展中的变化,有助于判断肿瘤的预后,并有望通过针对成纤维细胞的靶向治疗调控甚至杀灭肿瘤。
胶质母细胞瘤细胞至少存在3种亚型,即前神经元型、经典型和间充质型。Jain等[34]将scRNA-seq与空间转录组技术结合,证明在胶质母细胞瘤中肿瘤相关成纤维细胞(cancer-associated fibroblast,CAF)与间充质GSC、内皮细胞和M2巨噬细胞邻近。CAF被GSC趋化吸引,并能够富集GSC,具有促肿瘤的作用。Hoogstrate等[35]通过对比原发性和复发性胶质母细胞瘤,发现复发性肿瘤细胞表现为优先向间充质型进展,并通过scRNA-seq和免疫组织化学染色等证实复发性胶质母细胞瘤主要通过肿瘤微环境重新组织进化而非肿瘤细胞的分子进化,这些特征与复发生存率显著降低相关。上述研究结果为胶质母细胞瘤的治疗提供了全新的见解和思路。
肿瘤相关微生物群也是肿瘤微环境的固有组成部分。Galeano等[36]通过将scRNA-seq与空间转录组技术应用于口腔鳞状细胞癌和结直肠癌研究,揭示了空间、细胞和分子宿主-微生物相互作用,并通过引入靶向细菌16S rRNA保守区域的引物开发了一种scRNA-seq方法,即入侵-黏附-定向表达测序,用于识别肿瘤细胞相关细菌和与之相互作用的宿主细胞,通过功能研究发现被细菌感染的癌细胞以单细胞的形式侵入周围环境,并将髓系细胞募集到细菌区域。同时数据显示肿瘤内微生物群的分布不是随机的,主要集中在具有支持肿瘤进展功能的免疫和上皮细胞微生态位中。对肿瘤内微生物群的功能效应进行研究和评估,将有助于确定预防和治疗此类肿瘤的分子和细胞靶点。
2.4 抗肿瘤药物研发肿瘤耐药性是制约肿瘤化疗药物和靶向药物疗效的一大难题,又分为内在性耐药(未接触药物时已存在)和获得性耐药(接触药物后产生)两大类,肿瘤异质性决定了使用单一药物的局限性,同时肿瘤细胞的高度可塑性也是耐药性产生的根源。Marjanovic等[37]分析了基因工程小鼠肺肿瘤从肿瘤前增生到腺癌7个阶段的单细胞转录组,结果提示转录状态的多样性随时间的推移而增加,并通过计算预测异质性的肿瘤细胞通过共同的、过渡性的高可塑性细胞状态(high-plasticity cell state,HPCS)介导产生。进一步研究发现,从小鼠肿瘤和人类患者来源的异种移植中分离的HPCS细胞显示出较高的分化和增殖能力,HPCS与人类肿瘤生存率低有关,并在小鼠中显示出耐药性。该研究认为HPCS是大部分异质性的起点,提出了靶向HPCS的治疗策略。Cohen等[38]通过scRNA-seq发现了多发性骨髓瘤的有效治疗靶点,寻找新的治疗靶点可以有效解决因长期使用抗肿瘤药物所致的肿瘤细胞耐药性问题。
Chang等[39]开发了跟踪不同克隆反应的单细胞RNA测序(tracking differential clonal response by scRNA-seq,TraCe-seq)系统,使用慢病毒载体将3' scRNA-seq兼容的条形码库(长度30 nt)稳定整合到肿瘤细胞基因组中,在亚群和单细胞分辨率下同时跟踪肿瘤起源并比较肿瘤细胞对不同疗法的即时反应,解析决定药物反应和耐药性的转录特征。该系统使直接比较不同疗法的疗效和作用机制成为可能。Chang等[39]使用TraCe-seq揭示了应用传统EGFR激酶抑制剂与双重EGFR抑制剂-降解剂后不同的转录反应和耐药机制,两者的区别在于EGFR蛋白是否持续存在,使用降解剂降低EGFR水平可能会阻断抑制剂的疗效,而由抑制剂结合EGFR引起的内质网应激的诱导对于实现完全疗效至关重要。TraCe-seq技术将大大加速药物反应机制及耐药机制的研究,加快抗肿瘤药物的研发进度。
3 小结单细胞转录组测序技术在肿瘤学方面的应用取得了突飞猛进的发展,极大地推动着肿瘤学研究的进步,在单细胞水平进行监测可以更好地研究肿瘤发生、发展及演变过程。使用该技术可以从肿瘤异质性、肿瘤转移、肿瘤微环境、免疫学治疗、肿瘤耐药性等方面多角度、多层次更深入地了解肿瘤,最终实现防治肿瘤的目标。单细胞转录组测序技术的发展日新月异,方法日趋成熟和多样,应用也越来越广泛,但该技术仍然存在一定的局限性,这些局限性目前正在被不断发现、认识和解决。例如,单一的scRNA-seq虽然可以识别异质性的细胞类型,甚至发现罕见细胞群,但对于细胞在组织原位的空间分布却无法识别,联合使用空间转录组测序可以解决这一局限,更好地解释细胞间联系和微环境相互作用;单细胞的核酸含量极低,容易发生数据丢失,同时核酸含量低、易降解等性质使扩增难度增大,还可能出现非特异性扩增等,需要不断优化实验条件、改进创新技术方法;在分离单细胞时会打破组织和器官的整体性,其中稀有细胞应该如何获取以及分离细胞时活性细胞受到刺激、细胞内部有无发生应激变化无法证实,通过对比解离样本的scRNA-seq数据和冷冻样本的snRNA-seq数据可能有助于发掘和解读相关信息。此外,虽然目前获得的与肿瘤相关的海量单细胞转录组数据已极大地推动了肿瘤学研究的发展,但如何加速开发相应的生物信息学分析手段、更好地进行发掘利用也是丞待解决的关键问题。单细胞转录组测序技术正朝着低成本、高通量和高质量的方向发展,随着高通量单细胞多组学测序技术的不断开发应用,以及机器学习和人工智能识别的革命性发展,未来一定会在临床肿瘤诊断、治疗、预后等方面得到更多的应用。
| [1] |
STARK R, GRZELAK M, HADFIELD J. RNA sequencing: the teenage years[J]. Nat Rev Genet, 2019, 20(11): 631-656. DOI:10.1038/s41576-019-0150-2 |
| [2] |
TANG F, BARBACIORU C, WANG Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell[J]. Nat Methods, 2009, 6(5): 377-382. DOI:10.1038/nmeth.1315 |
| [3] |
JOVIC D, LIANG X, ZENG H, et al. Single-cell RNA sequencing technologies and applications: a brief overview[J]. Clin Transl Med, 2022, 12(3): e694. DOI:10.1002/ctm2.694 |
| [4] |
SLYPER M, PORTER C B M, ASHENBERG O, et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors[J]. Nat Med, 2020, 26(5): 792-802. DOI:10.1038/s41591-020-0844-1 |
| [5] |
STOECKIUS M, HAFEMEISTER C, STEPHENSON W, et al. Simultaneous epitope and transcriptome measurement in single cells[J]. Nat Methods, 2017, 14(9): 865-868. DOI:10.1038/nmeth.4380 |
| [6] |
PETERSON V M, ZHANG K X, KUMAR N, et al. Multiplexed quantification of proteins and transcripts in single cells[J]. Nat Biotechnol, 2017, 35(10): 936-939. DOI:10.1038/nbt.3973 |
| [7] |
LEE J, HYEON D Y, HWANG D. Single-cell multiomics: technologies and data analysis methods[J]. Exp Mol Med, 2020, 52(9): 1428-1442. DOI:10.1038/s12276-020-0420-2 |
| [8] |
PAN Y, CAO W, MU Y, et al. Microfluidics facilitates the development of single-cell RNA sequencing[J]. Biosensors, 2022, 12(7): 450. DOI:10.3390/bios12070450 |
| [9] |
RAMSKÖLD D, LUO S, WANG Y C, et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells[J]. Nat Biotechnol, 2012, 30(8): 777-782. DOI:10.1038/nbt.2282 |
| [10] |
PICELLI S. Full-length single-cell RNA sequencing with Smart-seq2[J]. Methods Mol Biol, 2019, 1979: 25-44. DOI:10.1007/978-1-4939-9240-9_3 |
| [11] |
HASHIMSHONY T, WAGNER F, SHER N, et al. CEL-Seq: single-cell RNA-Seq by multiplexed linear amplification[J]. Cell Rep, 2012, 2(3): 666-673. DOI:10.1016/j.celrep.2012.08.003 |
| [12] |
HASHIMSHONY T, SENDEROVICH N, AVITAL G, et al. CEL-Seq2:sensitive highly-multiplexed single-cell RNA-Seq[J]. Genome Biol, 2016, 17: 77. DOI:10.1186/s13059-016-0938-8 |
| [13] |
MACOSKO E Z, BASU A, SATIJA R, et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets[J]. Cell, 2015, 161(5): 1202-1214. DOI:10.1016/j.cell.2015.05.002 |
| [14] |
KLEIN A M, MAZUTIS L, AKARTUNA I, et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells[J]. Cell, 2015, 161(5): 1187-1201. DOI:10.1016/j.cell.2015.04.044 |
| [15] |
HAGEMANN-JENSEN M, ZIEGENHAIN C, CHEN P, et al. Single-cell RNA counting at allele and isoform resolution using Smart-seq3[J]. Nat Biotechnol, 2020, 38(6): 708-714. DOI:10.1038/s41587-020-0497-0 |
| [16] |
SALMEN F, DE JONGHE J, KAMINSKI T S, et al. High-throughput total RNA sequencing in single cells using VASA-seq[J]. Nat Biotechnol, 2022, 40(12): 1780-1793. DOI:10.1038/s41587-022-01361-8 |
| [17] |
MOSES L, PACHTER L. Museum of spatial transcriptomics[J]. Nat Methods, 2022, 19(5): 534-546. DOI:10.1038/s41592-022-01409-2 |
| [18] |
ZHANG J, SONG C, TIAN Y, et al. Single-cell RNA sequencing in lung cancer: revealing phenotype shaping of stromal cells in the microenvironment[J]. Front Immunol, 2022, 12: 802080. DOI:10.3389/fimmu.2021.802080 |
| [19] |
NEFTEL C, LAFFY J, FILBIN M G, et al. An integrative model of cellular states, plasticity, and genetics for glioblastoma[J]. Cell, 2019, 178(4): 835-849. DOI:10.1016/j.cell.2019.06.024 |
| [20] |
COUTURIER C P, AYYADHURY S, LE P U, et al. Single-cell RNA-seq reveals that glioblastoma recapitulates a normal neurodevelopmental hierarchy[J]. Nat Commun, 2020, 11: 3406. DOI:10.1038/s41467-020-17186-5 |
| [21] |
CASTELLAN M, GUARNIERI A, FUJIMURA A, et al. Single-cell analyses reveal YAP/TAZ as regulators of stemness and cell plasticity in glioblastoma[J]. Nat Cancer, 2021, 2(2): 174-188. DOI:10.1038/s43018-020-00150-z |
| [22] |
ZHANG M, HU S, MIN M, et al. Dissecting transcriptional heterogeneity in primary gastric adenocarcinoma by single cell RNA sequencing[J]. Gut, 2021, 70(3): 464-475. DOI:10.1136/gutjnl-2019-320368 |
| [23] |
CHEN Z, ZHOU L, LIU L, et al. Single-cell RNA sequencing highlights the role of inflammatory cancer-associated fibroblasts in bladder urothelial carcinoma[J]. Nat Commun, 2020, 11(1): 5077. DOI:10.1038/s41467-020-18916-5 |
| [24] |
HE D, WANG D, LU P, et al. Single-cell RNA sequencing reveals heterogeneous tumor and immune cell populations in early-stage lung adenocarcinomas harboring EGFR mutations[J]. Oncogene, 2021, 40(2): 355-368. DOI:10.1038/s41388-020-01528-0 |
| [25] |
LEADER A M, GROUT J A, MAIER B B, et al. Single-cell analysis of human non-small cell lung cancer lesions refines tumor classification and patient stratification[J]. Cancer Cell, 2021, 39(12): 1594-1609.e12. DOI:10.1016/j.ccell.2021.10.009 |
| [26] |
VAN GALEN P, HOVESTADT V, WADSWORTH Ⅱ M H, et al. Single-cell RNA-seq reveals AML hierarchies relevant to disease progression and immunity[J]. Cell, 2019, 176(6): 1265-1281.e24. DOI:10.1016/j.cell.2019.01.031 |
| [27] |
XU K, WANG R, XIE H, et al. Single-cell RNA sequencing reveals cell heterogeneity and transcriptome profile of breast cancer lymph node metastasis[J]. Oncogenesis, 2021, 10(10): 66. DOI:10.1038/s41389-021-00355-6 |
| [28] |
KIM N, KIM H K, LEE K, et al. Single-cell RNA sequencing demonstrates the molecular and cellular reprogramming of metastatic lung adenocarcinoma[J]. Nat Commun, 2020, 11(1): 2285. DOI:10.1038/s41467-020-16164-1 |
| [29] |
GUO X, LIN F, YI C, et al. Deep transfer learning enables lesion tracing of circulating tumor cells[J]. Nat Commun, 2022, 13(1): 7687. DOI:10.1038/s41467-022-35296-0 |
| [30] |
KATZENELENBOGEN Y, SHEBAN F, YALIN A, et al. Coupled scRNA-seq and intracellular protein activity reveal an immunosuppressive role of TREM2 in cancer[J]. Cell, 2020, 182(4): 872-885.e19. DOI:10.1016/j.cell.2020.06.032 |
| [31] |
MOLGORA M, ESAULOVA E, VERMI W, et al. TREM2 modulation remodels the tumor myeloid landscape enhancing anti-PD-1 immunotherapy[J]. Cell, 2020, 182(4): 886-900.e17. DOI:10.1016/j.cell.2020.07.013 |
| [32] |
SAVAS P, VIRASSAMY B, YE C, et al. Single-cell profiling of breast cancer T cells reveals a tissue-resident memory subset associated with improved prognosis[J]. Nat Med, 2018, 24(7): 986-993. DOI:10.1038/s41591-018-0078-7 |
| [33] |
ZHOU Y, BIAN S, ZHOU X, et al. Single-cell multiomics sequencing reveals prevalent genomic alterations in tumor stromal cells of human colorectal cancer[J]. Cancer Cell, 2020, 38(6): 818-828. DOI:10.1016/j.ccell.2020.09.015 |
| [34] |
JAIN S, RICK J W, JOSHI R S, et al. Single-cell RNA sequencing and spatial transcriptomics reveal cancer-associated fibroblasts in glioblastoma with protumoral effects[J]. J Clin Investig, 2023, 133(5): e147087. DOI:10.1172/JCI147087 |
| [35] |
HOOGSTRATE Y, DRAAISMA K, GHISAI S A, et al. Transcriptome analysis reveals tumor microenvironment changes in glioblastoma[J]. Cancer Cell, 2023, 41(4): 678-692.e7. DOI:10.1016/j.ccell.2023.02.019 |
| [36] |
GALEANO NIÑO J L, WU H, LACOURSE K D, et al. Effect of the intratumoral microbiota on spatial and cellular heterogeneity in cancer[J]. Nature, 2022, 611(7937): 810-817. DOI:10.1038/s41586-022-05435-0 |
| [37] |
MARJANOVIC N D, HOFREE M, CHAN J E, et al. Emergence of a high-plasticity cell state during lung cancer evolution[J]. Cancer Cell, 2020, 38(2): 229-246.e13. DOI:10.1016/j.ccell.2020.06.012. |
| [38] |
COHEN Y C, ZADA M, WANG S Y, et al. Identification of resistance pathways and therapeutic targets in relapsed multiple myeloma patients through single-cell sequencing[J]. Nat Med, 2021, 27(3): 491-503. DOI:10.1038/s41591-021-01232-w |
| [39] |
CHANG M T, SHANAHAN F, NGUYEN T T T, et al. Identifying transcriptional programs underlying cancer drug response with TraCe-seq[J]. Nat Biotechnol, 2022, 40(1): 86-93. DOI:10.1038/s41587-021-01005-3 |
 2023, Vol. 44
2023, Vol. 44


