2. 上海交通大学医学院附属上海儿童医学中心心内科,上海 200127;
3. 上海智慧儿科临床诊治技术工程技术研究中心,上海 200127;
4. 上海理工大学健康科学与工程学院智能医学影像与计算医疗实验室,上海 200093
2. Department of Pediatric Cardiology, Shanghai Children's Medical Center, Shanghai Jiao Tong University School of Medicine, Shanghai 200127, China;
3. Shanghai Engineering Research Center of Intelligence Pediatrics (SERCIP), Shanghai 200127, China;
4. Intelligent Medical Imaging and Computational Medicine Laboratory, School of Health Sciences and Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China
先天性心脏病(congenital heart disease,CHD)是最常见的出生缺陷之一,活产婴儿中CHD的发病率约为0.9%,是0~5岁儿童的主要死亡原因[1]。中国每年约有10万~15万新生儿被诊断为CHD,而且自2016年我国全面实施二胎政策以来,CHD的发病率呈逐渐升高的趋势[2],因此,早期准确诊断CHD具有重要的临床意义。经胸超声心动图(transthoracic echocardiography,TTE)可以实时动态观察心脏,快速检测心脏的各种异常,具有无创、无辐射和低成本等优点,在CHD的诊断和治疗中有非常重要的价值[3]。TTE主要包括标准切面获取、动态图像扫描和测量等步骤,其中,准确获取标准切面是后续生物学特征测量和最终诊断CHD的先决条件。
然而,心脏的解剖结构和空间构型复杂多变,通过TTE进行准确诊断复杂且费时,还严重依赖有经验的心脏病学专家对每个TTE切面的准确判断。根据美国超声心动图学会的建议,采用标准成像技术进行心脏的二维、M型和彩色多普勒超声检查[4],这意味着图像是按照相同的协议以可重复的方式获得的。事实上,图像需要在一个特定的平面上获得,才能对特定结构进行测量,并减少观察者之间和观察者内部的差异[5]。因此,应用自动智能识别儿童心脏超声标准切面的深度学习技术非常必要,其可为智能诊断CHD奠定基础,还可作为基层心脏超声医师进行切面扫查的标准化培训,具有很好的临床应用价值。
在过去的几年里,基于人工智能的图像识别技术,特别是深度学习,已越来越多地被用于超声心动图[6-12]。据报道,深度学习在CHD的评估、诊断和预后方面有很大潜力[13-16]。传统的机器学习通常需要在输入图像中推导出预定义的特征[17]。相比之下,深度学习从图像中自动估计结果,而不需要预先定义特定的成像特征[18]。此外,卷积神经网络的深层结构能够从原始图像中提取详细的低层次信息,并将这些信息组合成高阶结构信息,从而能够从图像中识别复杂的实体,这种技术在分析各种心脏疾病的超声心动图结果时可能很有用[19-20]。因此,本研究通过改进密集连接网络(dense convolutional network,DenseNet)中的瓶颈层,降低了参数量和计算量,提出了一个轻量、高效的深度学习模型用于儿童心脏超声标准切面的自动智能识别。
1 资料和方法 1.1 数据采集及图像预处理回顾性分析上海交通大学医学院附属上海儿童医学中心心脏超声诊断中心影像归档和通信系统数据库中,2022年9月至10月行心脏超声检查儿童的4 035张心脏超声图像(DICOM格式)。将所有图像转换为JPG格式、大小512像素×384像素的图像,并且对每张图像进行严格脱敏处理。
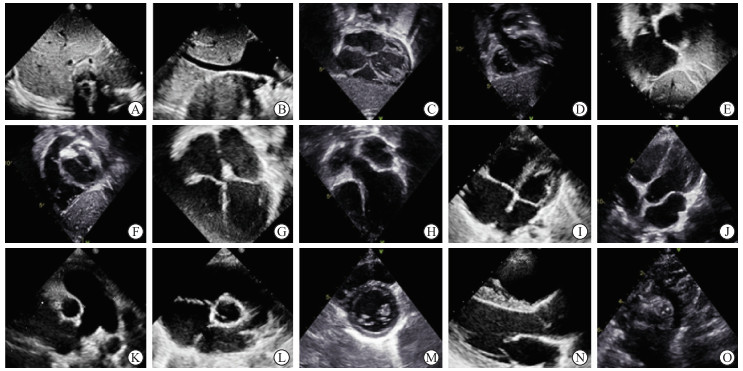
1.2 仪器与方法所有图像均由GE Vivid E90型或Philips EPIQ 7C型号超声诊断仪采集,探头频率为2.0~5.0 MHz。行超声心动图检查时,患儿取平卧位或左侧卧位及仰卧头后伸位,按顺序依次扫查剑突下、心尖、胸骨旁、胸骨上窝等切面,15个标准切面依次为剑突下下腔静脉和降主动脉横断切面、剑突下下腔静脉长轴切面、剑突下四腔心切面、剑突下五腔心切面、剑突下双心房切面、剑突下右心室流出道切面、心尖四腔心切面、心尖五腔心切面、低位胸骨旁四腔心切面、低位胸骨旁五腔心切面、胸骨旁肺动脉及分支切面、胸骨旁大动脉短轴切面、胸骨旁左心室短轴切面、胸骨旁左心室长轴切面、胸骨上窝主动脉弓切面(图 1)。不是上述15个标准切面的图像都归属于非标准切面。共选取4 035张超声图像,包括标准切面3 423张和非标准切面612张,按照6∶2∶2的比例将4 035张图像随机划分为训练集(2 421张)、验证集(807张)、测试集(807张),分别用于模型行训练、调参和评估。将所提出的深度学习模型与DenseNet121、InceptionV3、MobileNetV3 3种模型进行对照,评价模型对儿童心脏超声标准切面和非标准切面的识别性能。
|
图 1 15个儿童心脏超声标准切面 Fig 1 Fifteen pediatric heart ultrasound standard views A: Transverse section of subxiphoid inferior vena cava and descending aorta; B: Long-axis view of subxiphoid inferior vena cava; C: Subxiphoid 4-chamber view; D: Subxiphoid 5-chamber view; E: Subxiphoid sagittal view of atrium septum; F: Subxiphoid short-axis view through right ventricular outflow tract; G: Apical 4-chamber view; H: Apical 5-chamber view; I: Low parasternal 4-chamber view; J: Low parasternal 5-chamber view; K: Parasternal view of pulmonary artery; L: Parasternal short-axis view of large artery; M: Parasternal short-axis view of left ventricle (at the level of the mitral valve); N: Parasternal long-axis view of left ventricle; O: Suprasternal long-axis view of entire aortic arch. |
1.3 深度学习模型的设计
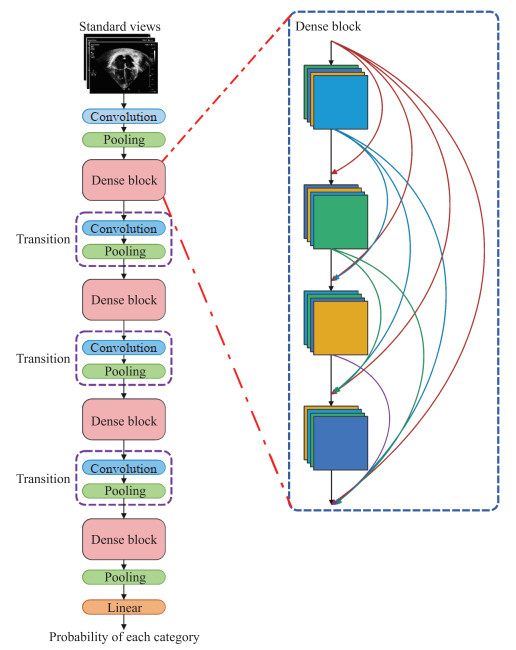
针对临床需求及超声设备硬件限制,受DenseNet[21]和Inception[22] 2个模型诸多优点的启发,基于它们的设计思想,开发了一个轻量、高效的深度学习模型,对心脏超声切面进行自动识别。在模型的密集层中设计了2个分支,其中1个是大小为7×7卷积核的深度可分离卷积,另1个是大小为3×3卷积核的深度可分离卷积。然后将它们的输出特征图相加,最后再和瓶颈层的输入特征图拼接作为瓶颈层的输出特征图。通过设计这种多尺度卷积核可获得不同大小的感受野,提升模型的性能。同时深度可分离卷积因采用分组卷积的技术,降低了参数量及计算量。此外,将4个阶段密集块堆叠的比例设置为1∶1∶3∶1,其每个阶段具体的层数分别为8、8、24和8。这种在高层堆叠较多层的方式能够有效提取出高级语义信息。提出的儿童心脏超声切面深度学习模型的整体网络框架如图 2所示。
|
图 2 本研究提出的儿童心脏超声切面深度学习模型的整体网络框架 Fig 2 Overall network framework of the proposed deep learning model in this study for pediatric echocardiography views |
提出的深度学习模型对儿童心脏超声切面的分类方法步骤如下:(1)对心脏超声标准切面图像进行预处理,全部转换为固定大小并归一化;(2)将一批图像送入模型前向传播;(3)通过损失函数量化模型输出值与真实值的偏差;(4)通过反向传播算法,计算梯度,更新模型权重。提出的模型具有深度学习神经网络算法的典型模块,包括卷积层、池化层、激活函数、全连接层等。在模型的训练过程中使用的优化器为AdamW,损失函数为交叉熵,初始学习率为0.01。训练软件环境为Python 3.6编程语言、PyTorch 1.12.1深度学习框架。硬件环境为英特尔酷睿i9-10900X处理器、32 GB内存、2块英伟达RTX3080图形处理器、22 GB显存。
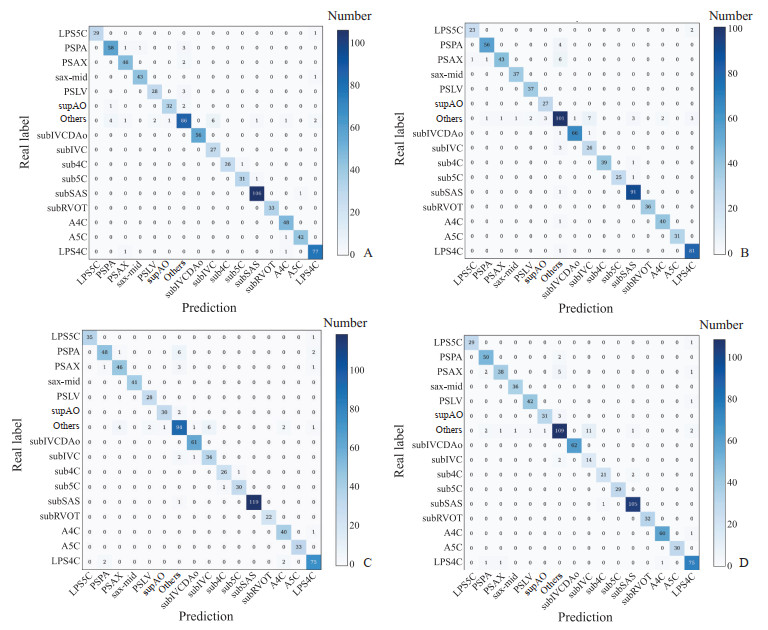
1.4 深度学习模型识别效果的评价标准由2位有5年以上临床超声工作经验的医师对所有图像进行手动标注,若2位医师的手工标注不一致则该图像不纳入。以人工手动标注结果为金标准,采用准确度、精确率、特异度、召回率和F1指数评估DenseNet121、InceptionV3、MobileNetV3和所提出模型的识别性能。准确度(%)=(TP+TN)/(TP+TN+FP+FN)×100%,精确率(%)=TP/(TP+FP)×100%,特异度(%)=TN/(TN+FP)×100%,召回率(%)=TP/(TP+FN)×100%,F1指数(%)=2×(精确率×召回率)/(精确率+召回率)×100%,其中TP表示真阳性数量,TN表示真阴性数量,FP表示假阳性数量,FN表示假阴性数量。使用参数量、模型大小和浮点运算数3个指标评估模型的识别效率。运用混淆矩阵和热力图衡量模型对儿童心脏超声标准切面识别效果。混淆矩阵作为一种可视化的工具,可以直观反映模型的分类结果,其中混淆矩阵的纵坐标代表了该类的真实标签,横坐标代表了该类的预测标签,对角线上的颜色越深说明模型预测正确的样本数越多。热力图可以直观地显示出模型对图像某些特征区域的关注度,颜色越偏暖色说明模型对该区域关注度越高,反之模型对该区域关注度越低。
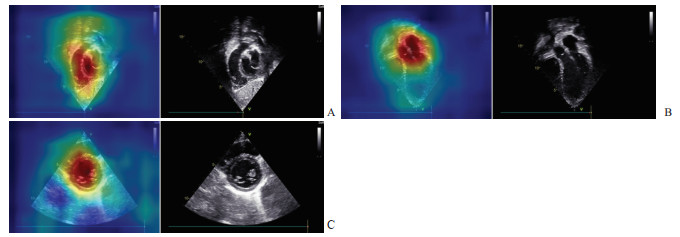
2 结果DenseNet121、InceptionV3、MobileNetV3模型和所提出的深度学习模型识别15个儿童心脏超声标准切面和非标准切面的平均F1指数分别为94.59%、95.13%、92.41%、94.73%,4种模型对儿童心脏超声切面的识别结果表现良好,所提出的深度学习模型F1指数稍低于InceptionV3模型(表 1、表 2),但所提出的深度学习模型的参数量仅为1.8×106(表 3),说明所提出的深度学习模型不仅表现出非常好的识别性能,且轻量高效。图 3是4种模型的图像分类混淆矩阵,从混淆矩阵中可以得出所提出的深度学习模型在多个儿童心脏超声切面中的识别准确率均高于DenseNet121、InceptionV3、MobileNetV3模型。图 4是剑突下右心室流出道切面、心尖五腔心切面和胸骨旁左心室短轴切面的热力图,从热力图中可以看出所提出的模型能够关注到儿童心脏超声切面中的关键特征区域。
|
|
表 1 DenseNet121、InceptionV3、MobileNetV3模型对儿童心脏超声切面的识别性能 Tab 1 Performance of DenseNet121, InceptionV3 and MobileNetV3 for identification of pediatric echocardiographic views |
|
|
表 2 本研究提出的深度学习模型对儿童心脏超声切面的识别性能 Tab 2 Performance of the proposed deep learning model in this study for identification of pediatric echocardiographic views |
|
|
表 3 DenseNet121、InceptionV3、MobileNetV3和本研究提出的深度学习模型对儿童心脏超声切面识别的运行效率 Tab 3 Operational efficiency of DenseNet121, InceptionV3, MobileNetV3 and the proposed deep learning model in this study for identification of pediatric echocardiographic views |
|
图 3 4种模型识别儿童心脏超声切面效果的混淆矩阵图 Fig 3 Confusion matrix diagram of performance of 4 models for identifying views of pediatric echocardiography A: DenseNet121; B: InceptionV3; C: MobileNetV3; D: The proposed deep learning model in this study. LPS5C: Low parasternal 5-chamber view; PSPA: Parasternal view of pulmonary artery; PSAX: Parasternal short-axis view of large artery; sax-mid: Parasternal short-axis view of left ventricle (at the level of the mitral valve); PSLV: Parasternal long-axis view of left ventricle; supAO: Suprasternal long-axis view of entire aortic arch; subIVCDAo: Transverse section of subxiphoid inferior vena cava and descending aorta; subIVC: Long axis view of subxiphoid inferior vena cava; sub4C: Subxiphoid 4-chamber view; sub5C: Subxiphoid 5-chamber view; subSAS: Subxiphoid sagittal view of atrium septum; subRVOT: Subxiphoid short-axis view through right ventricular outflow tract; A4C: Apical 4-chamber view; A5C: Apical 5-chamber view; LPS4C: Low parasternal 4-chamber view. |
|
图 4 本研究提出的深度学习模型对3个儿童心脏超声标准切面中区域关注度的热力图 Fig 4 Heatmap of the proposed deep learning model in this study for regional concerns in 3 pediatric echocardiography standard views A: Subxiphoid short-axis view through right ventricular outflow tract; B: Apical 5-chamber view; C: Parasternal short-axis view of left ventricle (at the level of the mitral valve). |
3 讨论
近年来,由于人工智能技术的发展,深度学习模型在图像识别领域内获得了广泛应用。然而,深度学习模型普遍要求足够多的训练数据集,否则网络训练就会出现过拟合问题[23]。在不同的图像领域,数据采集的难度不尽相同,一般来说自然图像数据集的规模往往远大于医学图像数据集。因此,在医学图像领域,深度学习模型应用的最大困难在于数据集规模的限制。
本研究提出了一种轻量、高效的模型,不仅没有在小样本数据集上出现过拟合现象,还表现出了非常好的综合性能,对剑突下右心室流出道切面、心尖五腔心切面和胸骨旁左心室短轴切面这3个切面的识别率最高,达到近100%的准确率。这是因为剑突下右心室流出道切面中流出道至肺动脉的特征比较明显,心尖五腔心切面中主动脉的特征比较明显,胸骨旁左心室短轴切面中左心室呈圆形的特征比较明显。此外,由于深度学习模型饱受黑盒诟病,使用类激活图(class activation mapping,CAM)算法[24]对模型进行可解释分析,此算法可通过热力图来分析模型对剑突下右心室流出道切面、心尖五腔心切面和胸骨旁左心室短轴切面这3个切面中图像特征区域的关注度,从热力图中可以看出提出的深度学习模型对这3个切面中的图像特征区域关注度较高,而对图像的其他区域关注度较低。对于15个标准切面和非标准切面,识别率相对低的是非标准切面,原因在于非标准切面数据中含有大量与其他15个切面差异较小的图像,这对于识别有较大影响。总体而言,本研究提出的模型在识别儿童心脏超声标准切面上表现良好。
本研究提出的深度学习模型不仅性能较高,运行效率也比较高,提出的模型参数量仅为1.8×106,模型大小仅为3.588 MB,均低于DenseNet121、InceptionV3、MobileNetV3模型。此外,本研究提出模型的浮点运算数低于DenseNet121和InceptionV3模型,虽计算量略高于MobileV3模型,但性能较其高。总体而言,本研究提出的模型适合应用于硬件条件受限的临床场景中,如偏远落后地区、基层社区医疗机构的低性能超声设备和一些移动便携超声设备都可以搭载本研究提出的模型,辅助基层心脏超声医师在进行切面扫查时打出标准切面,从而提高心脏超声诊断CHD的准确率。
本研究存在以下局限性:(1)为单中心数据,超声检查机器和扫查方式不同可能会导致图像质量存在差异,未来还需要在不同中心和不同质量的图像数据集上验证所提出深度学习模型的性能。(2)心脏超声图像数据集相对较少,未来还将继续扩大数据集进行深入研究。(3)使用的是静态心脏超声图像,而心脏超声视频序列可能会反映更多的心脏结构信息。(4)仅对儿童心脏超声标准切面进行了初步分类,并未将患儿的临床资料纳入研究,未来会将患者的临床资料和图像所对应的CHD种类进行统计以辅助解决临床问题。(5)所提出的模型运用了较多的深度可分离卷积,会造成较高的内存访问率,这在一定程度上限制了运行速率,未来将设计更合理的模型结构以进一步释放模型的运行速率。
综上所述,本研究探讨了深度学习在儿童心脏超声标准切面自动智能识别中应用的可行性和准确性,所提出的轻量、高效深度学习模型在15个儿童心脏超声标准切面和非标准切面分类任务中取得了令人满意的结果,且模型的参数量较少,运行效率较高,为智能诊断CHD提供了良好的技术基础,也为基层心脏超声医师练习心脏切面扫查提供了标准化培训的新方法。
| [1] |
ZHAO Q M, LIU F, WU L, et al. Prevalence of congenital heart disease at live birth in China[J]. J Pediatr, 2019, 204: 53-58. DOI:10.1016/j.jpeds.2018.08.040 |
| [2] |
王维. 新生儿先天性心脏病防控措施研究进展[J]. 武警后勤学院学报(医学版), 2021, 30(1): 80-84. DOI:10.16548/j.2095-3720.2021.01.025 |
| [3] |
American College of Cardiology Foundation Appropriate Use Criteria Task Force, American Society of Echocardiography, American Heart Association, et al. ACCF/ASE/AHA/ASNC/HFSA/HRS/SCAI/SCCM/SCCT/SCMR 2011 appropriate use criteria for echocardiography. A report of the American College of Cardiology Foundation Appropriate Use Criteria Task Force, American Society of Echocardiography, American Heart Association, American Society of Nuclear Cardiology, Heart Failure Society of America, Heart Rhythm Society, Society for Cardiovascular Angiography and Interventions, Society of Critical Care Medicine, Society of Cardiovascular Computed Tomography, Society for Cardiovascular Magnetic Resonance American College of Chest Physicians[J]. J Am Soc Echocardiogr, 2011, 24(3): 229-267. DOI:10.1016/j.echo.2010.12.008 |
| [4] |
LOPEZ L, COLAN S D, FROMMELT P C, et al. Recommendations for quantification methods during the performance of a pediatric echocardiogram: a report from the Pediatric Measurements Writing Group of the American Society of Echocardiography Pediatric and Congenital Heart Disease Council[J]. J Am Soc Echocardiogr, 2010, 23(5): 465-495. DOI:10.1016/j.echo.2010.03.019 |
| [5] |
BURGOS-ARTIZZU X P, CORONADO-GUTIÉRREZ D, VALENZUELA-ALCARAZ B, et al. Evaluation of deep convolutional neural networks for automatic classification of common maternal fetal ultrasound planes[J]. Sci Rep, 2020, 10(1): 10200. DOI:10.1038/s41598-020-67076-5 |
| [6] |
SUDARSHAN V, ACHARYA U R, NG E Y, et al. Automated identification of infarcted myocardium tissue characterization using ultrasound images: a review[J]. IEEE Rev Biomed Eng, 2015, 8: 86-97. DOI:10.1109/RBME.2014.2319854 |
| [7] |
JAHREN T S, STEEN E N, AASE S A, et al. Estimation of end-Diastole in cardiac spectral Doppler using deep learning[J]. IEEE Trans Ultrason Ferroelectr Freq Control, 2020, 67(12): 2605-2614. DOI:10.1109/TUFFC.2020.2995118 |
| [8] |
KRITTANAWONG C, JOHNSON K W, ROSENSON R S, et al. Deep learning for cardiovascular medicine: a practical primer[J]. Eur Heart J, 2019, 40(25): 2058-2073. DOI:10.1093/eurheartj/ehz056 |
| [9] |
SCHUURING M J, IŠGUM I, COSYNS B, et al. Routine echocardiography and artificial intelligence solutions[J]. Front Cardiovasc Med, 2021, 8: 648877. DOI:10.3389/fcvm.2021.648877 |
| [10] |
LONG Q, YE X, ZHAO Q. Artificial intelligence and automation in valvular heart diseases[J]. Cardiol J, 2020, 27(4): 404-420. DOI:10.5603/cj.a2020.0087 |
| [11] |
CHEN C, QIN C, QIU H, et al. Deep learning for cardiac image segmentation: a review[J]. Front Cardiovasc Med, 2020, 7: 25. DOI:10.3389/fcvm.2020.00025 |
| [12] |
NARANG A, BAE R, HONG H, et al. Utility of a deep-learning algorithm to guide novices to acquire echocardiograms for limited diagnostic use[J]. JAMA Cardiol, 2021, 6(6): 624. DOI:10.1001/jamacardio.2021.0185 |
| [13] |
BLAIVAS M, ADHIKARI S, SAVITSKY E A, et al. Artificial intelligence versus expert: a comparison of rapid visual inferior vena cava collapsibility assessment between POCUS experts and a deep learning algorithm[J]. J Am Coll Emerg Physicians Open, 2020, 1(5): 857-864. DOI:10.1002/emp2.12206 |
| [14] |
JIAN Z N, WANG X P, ZHANG J Z, et al. Diagnosis of left ventricular hypertrophy using convolutional neural network[J]. BMC Med Inform Decis Mak, 2020, 20: 243. DOI:10.1186/s12911-020-01255-2 |
| [15] |
HONG W, SHENG Q, DONG B, et al. Automatic detection of secundum atrial septal defect in children based on color Doppler echocardiographic images using convolutional neural networks[J]. Front Cardiovasc Med, 2022, 9: 834285. DOI:10.3389/fcvm.2022.834285 |
| [16] |
ULLOA CERNA A E, JING L, GOOD C W, et al. Deep-learning-assisted analysis of echocardiographic videos improves predictions of all-cause mortality[J]. Nat Biomed Eng, 2021, 5(6): 546-554. DOI:10.1038/s41551-020-00667-9 |
| [17] |
JORDAN M I, MITCHELL T M. Machine learning: trends, perspectives, and prospects[J]. Science, 2015, 349(6245): 255-260. DOI:10.1126/science.aaa8415 |
| [18] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539 |
| [19] |
KUSUNOSE K, ABE T, HAGA A, et al. A deep learning approach for assessment of regional wall motion abnormality from echocardiographic images[J]. JACC Cardiovasc Imaging, 2020, 13(2 Pt 1): 374-381. DOI:10.1016/j.jcmg.2019.02.024 |
| [20] |
ZHANG J, GAJJALA S, AGRAWAL P, et al. Fully automated echocardiogram interpretation in clinical practice[J]. Circulation, 2018, 138(16): 1623-1635. DOI:10.1161/CIRCULATIONAHA.118.034338 |
| [21] |
HUANG G, LIU Z, VAN DER MAATEN L, et al. Densely connected convolutional networks[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), July 21-26, 2017, Honolulu, HI, USA: IEEE, 2017: 2261-2269. DOI: 10.1109/CVPR.2017.243.
|
| [22] |
SZEGEDY C, LIU W, JIA Y, et al. Going deeper with convolutions[C]//2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). June 7-12, 2015, Boston, MA, USA. IEEE, 2015: 1-9. DOI: 10.1109/CVPR.2015.7298594.
|
| [23] |
余镇, 吴凌云, 倪东, 等. 基于深度学习的胎儿颜面部超声标准切面自动识别[J]. 中国生物医学工程学报, 2017, 36(3): 267-275. DOI:10.3969/j.issn.0258-8021.2017.03.002 |
| [24] |
ZHOU B, KHOSLA A, LAPEDRIZA A, et al. Learning deep features for discriminative localization[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). June 27-30, 2016, Las Vegas, NV, USA. IEEE, 2016: 2921-2929. DOI: 10.1109/CVPR.2016.319.
|
 2023, Vol. 44
2023, Vol. 44


